小鼠不同细胞间组蛋白修饰变化对MafA基因转录表达的影响
李明岳,鲍世韵,刘嘉林,郑锦锋,林宝行,余小舫
(暨南大学第二临床医学深圳市人民院,深圳 518020)
组蛋白在翻译后的修饰中会发生改变,从而提供一种识别的标志,为其它蛋白与DNA的结合产生协同或拮抗效应,是一种动态转录调控成分[1]。染色质免疫共沉淀-实时定量PCR法(chromatin immunoprecipitation-real time quantitative PCR methods,CHIP-PCR)技术可以检测特定DNA序列染色质的结构,能够充分反映生理条件下的DNA与蛋白质相互作用的真实情况[2]。MafA基因又称亮氨酸拉链蛋白Maf家族A(v-maf musculoaponeurotic fibrosarcoma oncogene family protein A,MafA),是一个关键的基因特异性表达的调节器,对于胰腺生长和β细胞的分化及β细胞正常功能的维持起重要作用[3]。本研究采用CHIP-PCR技术比较小鼠胚胎干细胞(mouse embryonic stem cell,mES)、NIH小鼠成纤维细胞(NIH 3T3)和小鼠胰岛素瘤β细胞(NIT-1)之间MafA基因转录起始区的组蛋白修饰变化,以管家基因MLH1为对照,探讨组蛋白修饰对MafA基因转录表达的作用。
1 材料和方法
1.1 主要材料和仪器
(1)129/J小鼠胚胎干细胞(40 XY)和γ射线照射处理的第3代昆明小鼠胚胎成纤维细胞由赛业(广州)生物科技有限公司提供。小鼠成纤维细胞株NIH3T3细胞和小鼠β细胞株NIT-1细胞由中山医科大学动物实验中心细胞库提供,上述两细胞均来自美国ATCC细胞库;(2)组蛋白H3K4三甲基化(histone 3 lysine 4 trimethylation,H3K4m3)抗体(Abcam)、组蛋白H3K9三甲基化(histone 3 lysine 9 trimethylation,H3K9m3)抗体(Abcam)、组蛋白H3乙酰化抗体(Upstate)、抗小鼠Ig-G磁珠(BioLabs);(3)超声仪器Bioruptor(Diagenode)、Nanodrop(Agilent)、TaKaRa PCR Thermal Cycler(宝生生物工程有限公司)、ABI 7700 Realtime PCR仪(ABI);(4)引物设计软件:Primer 5.0。
1.2 细胞的准备
1.2.1 mES细胞的培养:预先准备由明胶包被的六孔板培养器皿,饲养层细胞是经γ射线照射处理的第3代昆明小鼠胚胎成纤维细胞。加入解冻后的mES细胞悬液和129/J小鼠胚胎干细胞完全培养基,在37℃、5%CO2、80%相对湿度的培养箱中培养。复苏后的第2天换用DMEM完全培养液,定期更换培养液,当mES细胞克隆较大或克隆间出现融合时,予传代。培养时间为2周。
1.2.2 NIT-1细胞的培养:将解冻后细胞悬液移入培养器皿中,加入足量的DMEM完全培养基,置于37℃、5%CO2、80%相对湿度的培养箱中培养。复苏后第2天更换培养液,待细胞长满培养器皿80%后进行传代。培养时间为2周。
1.2.3 NIH 3T3细胞的培养:方法同NIT-1细胞。
1.3 实验方法
1.3.1 CHIP-PCR技术检测组蛋白修饰
CHIP-PCR技术是研究蛋白与DNA之间在染色质环境下相互作用的一种常用方法,用于检测每个候选靶基因的启动子区域是否存在能与组蛋白及特定转录因子结合的特定DNA序列。一般有两种方法,一种是使用标准微球菌核酸酶来消化细胞核;另一种是在细胞中加入甲醛或将细胞暴露在紫外射线使得染色质发生交联。接着用超声波将染色质切成300bp到1000bp之间的小片段,然后加入组蛋白修饰抗体以及偶联Ⅱ抗磁珠进行孵育反应,利用磁铁的吸引力将组蛋白修饰的DNA片段从待测样品分离出来,采用酚-氯仿法纯化DNA片段。最后用定量PCR法扩增DNA片段,其扩增产物的量即可反映该启动子区中组蛋白修饰水平。
1.3.1.1 组蛋白修饰检测样本的制备:将3种细胞(1×107个细胞/瓶)分别用胰酶消化,用Bioruptor进行超声处理后于2%琼脂糖凝胶电泳,结果见图1。将断裂后的DNA片段分成两份,一份进行免疫共沉淀反应,一份作为input对照(不做任何处理直接进行PCR反应)。在第一份DNA样品中其余依次加入组蛋白H3K4m3抗体(或组蛋白H3K9m3抗体,或组蛋白H3乙酰化抗体)、G蛋白磁珠、抗小鼠IgG磁珠(磁珠偶联Ⅱ抗),孵育后,缓冲液洗涤,去除未结合的DNA片段,蛋白酶K消化抗体,用酚-氯仿溶液进行DNA纯化后,Nanodrop测DNA浓度。

图1 DNA超声处理后电泳图Fig.1 DNA after ultrasonic treatment Electrophoresis
1.3.1.2 实时荧光定量聚合酶联反应扩增目的基因启动子区:将3种细胞纯化的DNA进行实时定量PCR。采用ABI 7700 Realtime PCR仪对免疫沉淀中MafA基因和MLH1基因的DNA、input control和阴性对照进行扩增。测定标准曲线和熔解曲线,确保扩增的准确性和专一性,用Ct值表示样品中模板的相对含量,数据采用2-△△CT法进行分析。引物设计如下:MafA(mm9_cpgIslandExt_CpG:187.chr15:75576482-75578594.长度185bp):5′-CCTTGTACAGGTCCCGCTC-3′,5′-GGAGGTCATCC GACTGAAAC-3′;MLH1(mm9_cpgIslandExt_CpG:117.chr9:11117496 4-111175156.长度193bp):5′-TTAACCGAGCCAGGGACTTT-3′,5′-GCTTCCAG AATTGCCGACAT-3′。所有基因PCR程序一致,均如下:95℃、10min,40个PCR循环(95℃、15s,60℃、60s,收集荧光)。为了建立PCR产物的熔解曲线,扩增反应结束后,95℃、2min,60℃、20s,99℃、10s,并从60℃缓慢加热到99℃,退火温度59℃。实验重复3次。
1.3.2 实时定量逆转录PCR:检测3种细胞MafA和MLH1基因的表达水平。Trizol抽提RNA,鉴定RNA纯度及定量后,RNA逆转录成cDNA。以GAPDH基因作为内参。设计引物序列如下:MafA:5′-AAAGCGGTGCTGGAGGAT-3′,5′-GGTTCAGGTG GTGCTGGTA-3′;MLH1:5′-GC GGTTAGTCGAGAAC TGATA-3′,5′-AGTGAACTTCGTGCTTGGTG-3′;GAP DH:5′-GTCCAT GCCATCACTGCCAC-3′,5′-ATGA CCTTGCCCACAGCCTT-3′。荧光定量PCR反应体系及扩增条件与前相同。测定标准曲线和熔解曲线,确保扩增的准确性和专一性,用Ct值表示样品中模板的相对含量,数据采用2-△△CT法进行分析。实验重复3次。
1.3.3 统计学分析:数据经SPSS软件13.0统计,采用单因素方差分析,两两比较则用t检验,进行相关分析。
2 结果
2.1 组蛋白修饰的检测结果
用染色质免疫共沉淀法检测3种细胞中基因的组蛋白修饰的状况。以mES细胞为参照,结果提示:NIT-1细胞中MafA基因转录起始区的H3乙酰化和H3K4m3修饰水平明显增高(P<0.05),H3K9m3修饰水平明显降低(P<0.05);基因MLH1转录起始区的H3乙酰化修饰水平降低而H3K4m3修饰水平增高,但无统计学意义(P<0.05)。在NIH 3T3细胞中,MafA基因转录起始区的H3乙酰化和H3K9m3修饰水平明显增高(P<0.05),H3K4m3修饰水平明显降低(F=0.277,P=0.013);管家基因MLH1转录起始区的H3K9m3修饰水平增高(P<0.05)(表1)。
表1 基因在不同细胞中组蛋白修饰的比较(±s,n=3)Tab.1 Gene histone modifications in different cells comparisons(±s,n=3)
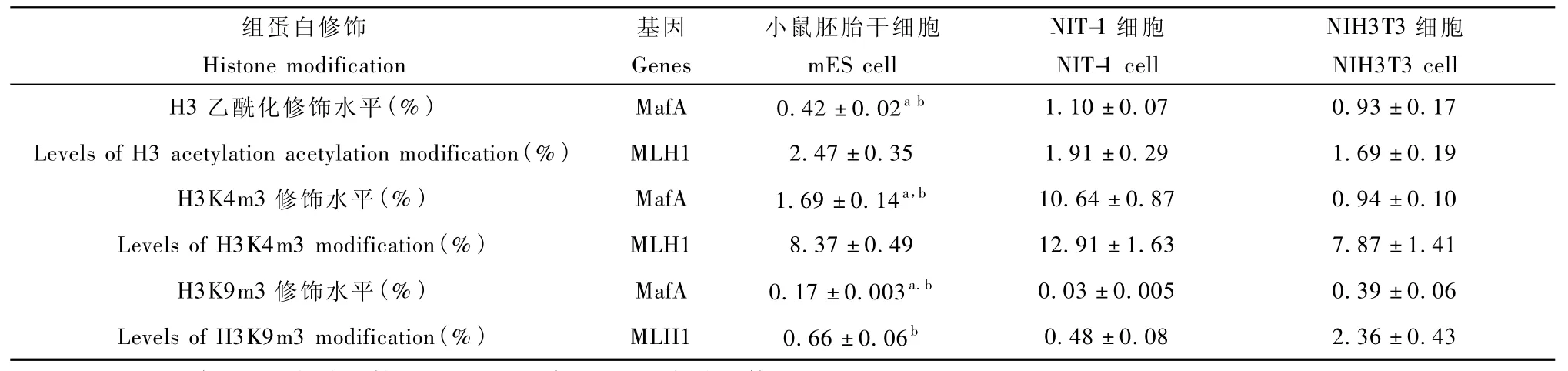
表1 基因在不同细胞中组蛋白修饰的比较(±s,n=3)Tab.1 Gene histone modifications in different cells comparisons(±s,n=3)
注:a.P<0.05与NIT-1细胞比较;b.P<0.05与NIH3T3细胞比较Note:a.P<0.05 vs NIT-1 cell.b.P<0.05 vs NIH3T3 cell
组蛋白修饰 基因 小鼠胚胎干细胞 NIT-1细胞 NIH3T3细胞Histone modification Genes mES cell NIT-1 cell NIH3T3 cell H3乙酰化修饰水平(%) MafA 0.42±0.02a b 1.10±0.07 0.93±0.17 Levels of H3 acetylation acetylation modification(%) MLH1 2.47±0.35 1.91±0.29 1.69±0.19 H3K4m3修饰水平(%) MafA 1.69±0.14a,b 10.64±0.87 0.94±0.10 Levels of H3K4m3 modification(%) MLH1 8.37±0.49 12.91±1.63 7.87±1.41 H3K9m3修饰水平(%) MafA 0.17±0.003a.b 0.03±0.005 0.39±0.06 Levels of H3K9m3 modification(%) MLH1 0.66±0.06b 0.48±0.08 2.36±0.43
2.2 基因mRNA的表达水平
实时定量逆转录PCR检测基因表达,MafA在NIT-1细胞表达,而在NIH 3T3和mES细胞基本不表达,前者与后两者存在统计学差异(P<0.05)。MLH1在3种细胞均表达,3者之间的表达水平无统计学差异(表2)。
表2 基因在不同细胞中mRNA表达水平比较(±s,n=3)Tab.2 mRNA expression level of gene in different cells comparisons(±s,n=3)

表2 基因在不同细胞中mRNA表达水平比较(±s,n=3)Tab.2 mRNA expression level of gene in different cells comparisons(±s,n=3)
注:★P<0.05 3者之间存在统计学差异Note:★P<0.05 statistical differences among three type cells
基因 小鼠胚胎干细胞 NIT-1细胞 NIH3T3细胞Genes mES cell NIT-1 cell NIH3T3 cell MafA 4.89E-05±0.24E-05 4.72E-03±0.17E-03★1.15E-04±0.19E-04 MLH1 1.10E-03±0.22E-03 1.21E-03±0.27E-03 0.97E-03±0.25E-03
2.3 相关分析
对基因的组蛋白修饰和基因表达进行相关分析。结果表明,MafA基因的表达与H3K4m3修饰存在直线相关(相关系数0.995,P<0.01);与H3K9m3修饰存在直线负相关(相关系数-0.751,P<0.05),与H3乙酰化修饰无相关性。管家基因MLH1的表达与所检测的组蛋白修饰无相关性。
3 讨论
NIT-1细胞来源于NOD小鼠,是由SV40和大鼠胰岛素基因的启动子经转基因方式产生永生化的β细胞系,具有与正常β细胞相似的基因表型,在葡萄糖的刺激下能够分泌胰岛素[4]。由于大量高纯度的β细胞在获取上存在困难,本实验采用NIT-1细胞代替正常β细胞作为研究对象。
组蛋白修饰是表观遗传学的重要组成部分,可在基因的DNA序列不发生改变时,使特异性基因的表达发生改变,并且这种改变还能通过有丝分裂和减数分裂进行遗传[5]。在机体中,细胞基本生命活动所必需的管家基因可在各个细胞中表达;特异性基因MafA虽存在于所有的细胞,随着胰腺的发育,其表达最终仅局限在β细胞[6],提示组蛋白修饰可能参与其中的调控。
组蛋白有多种共价修饰方式,其中H3乙酰化、H3K4m3和H3K9m3是修饰基因转录调控的重要机制[7]。H3乙酰化修饰使DNA与组蛋白间的静电引力和空间位阻增大,两者间的相互作用减弱,DNA易于解聚,染色质呈转录活性结构,进而激活基因转录。H3K4m3使组蛋白和DNA的结构由紧变松,靶基因才能和转录复合物相互作用,基因得以转录。H3K9m3能使组蛋白形成一个异染色质蛋白HP1(heterochromatin protein 1)或其他抑制性染色质因子的结合位点,HP1的结合进而会导致DNA分子上特定CpG岛的甲基化和稳定的基因沉默。不同组蛋白修饰可依次发挥作用或组合在一起,形成一个修饰的级联,它们通过协同或拮抗来共同发挥作用[8]。本研究发现,MafA基因在mES、NIT-1和NIH 3T3三种细胞中的差异表达与H3K9m3修饰存在直线负相关;与H3K4m3修饰存在直线相关。MafA基因的表达与H3乙酰化修饰无相关性,这可能跟H3K9m3和H3乙酰化修饰两者间存在竞争性有关[9]。而MLH1基因的表达与组蛋白修饰不存在相关性。结果表明,H3K9m3与H3K4m3修饰能协同作用于MafA基因转录起始区,调控其表达。
MafA基因的表达可在β细胞分化的最后阶段检测到,其除了与Pdx-1和BE-TA2一起协同刺激胰岛素基因表达[10],还参与Pdx-1基因的表达调控,,是一个关键的基因特异性表达的调节器[11]。Zhao等[12]发现MafA基因敲除小鼠出生时的胰岛在形态学上是正常的,但随着年龄的增长,小鼠表现为葡萄糖刺激胰岛素分泌受损和胰岛组织结构的异常。可见,MafA基因与β细胞的分化密切相关。在胚胎干细胞分化过程中,随着转录起始区H3K9m3与H3K4m3修饰水平的改变,MafA基因选择性转录,胚胎干细胞能向β细胞定向分化;当MafA基因选择性失活时,胚胎干细胞则失去向β细胞定向分化的能力。因而,在胚胎干细胞向β细胞分化过程中,MafA基因的H3K4m3与H3K9m3修饰调控机制具有重要的作用。
综上所述,H3K4m3与H3K9m3修饰能相互协调,共同调控MafA基因的表达,对胚胎干细胞向β细胞分化具有重要的意义。
[1]SternerDE,Berger SL.Acetylation of histones and transcription related factors[J].Microbiol Mol Biol Rev,2000,64(2):435-45
[2]Kuras L.Charaeterization of protein-DNA associationin vivoby chromatinimmunoprecipitation.[J].Methods Mol Bio,2004, 284:147-162.
[3]Coolen M,Sii-Felice K,Bronchain O,et al.Phylogenomic analysis and expression pattern of large Maf genes inXenopus tropicalisprovide new insights into the functional evolution of the gene family in osteichthyans.[J].Dev Genes Evol,2005,215(7):327-329.
[4]Hamaguchi K,GaskinsHR,LeiterEH,etal.NIT-1,a pancreatic beta-cell line established from a transgenic NOD/L tmouse[J].Diabetes,1991,40(7):842-849.
[5]Peterson CL,Laniel MA.Histones and histone modifications[J].Curr Biol,2004,14(14):546-551.
[6]Matsuoka TA,ArtnerI,HendersonE,etal.TheMafA transcription factor appears to be responsible for tissuespecific expression of insulin.[J].Proc Natl Acad Sci USA,2004,101(9):2930-2933.
[7]Jenuwein T,Allis CD.Translating the histone code[J].Science,2001,293(5532):1074-1080.
[8]Strahl BD,Allis CD.The language of covalent histone modifications[J].Nature,2000,403(6765):41-45.
[9]Litt MD,Simpson M,Gaszner M,et al.Correlation between histone lysine methylation and developmental changes at the chicken beta-globin locus[J].Science,2001,293(5539):2453-2455.
[10]Aramata S,Han SI,Yasuda K,et al.Synergistic activation of the insulin gene promoter by the β cell enriched transcription factors MafA,Beta2,and Pdx1[J].Biochim Biophys Acta,2005,1730:41-46.
[11]Samaras SE,Zhao L,Means A,et al.The islet beta cellenriched RIPE3b1/Maftranscription factorregulates pdx-1 expression[J].Biol Chem,2003,278:12263-12270.
[12]Zhao L,Gao M,Matsuoka TA,et al.The islet beta cellenriched MafA cativator is a key regulator of insulin gene transcription[J].JBiolChem,2005,280(12):11887-11894.

