肠道病毒71型VP1蛋白二级结构及B细胞表位的预测*
王 锦,许国章,倪红霞
肠道病毒71型VP1蛋白二级结构及B细胞表位的预测*
王 锦1,许国章2,倪红霞2
目的预测C4基因亚型肠道病毒71型的VP1蛋白二级结构及B细胞表位,为EV71疫苗研制提供理论基础。方法本研究将宁波地区EV71分离株与国内外EV71代表株进行同源性分析和并构建亲缘进化树。以VP1基因序列为材料,应用EX-PASY服务器上的GOR4、SOPMA两种方法分析预测蛋白质二级结构,并结合蛋白质的亲水性、柔韧性、表面可能性等指标综合评价VP1蛋白的B细胞抗原表位。结果同源性分析和亲缘进化分析得出,2010年宁波EV71分离株属于C4a基因亚型,与基因库检索到的C4a基因亚型代表株核苷酸和氨基酸同源性较高,分别为93.6%~99.3%和98.3%~100%。VP1蛋白二级结构以无规则卷曲和β-片层为主,有多处抗原指数较高的区段,结合亲水性、柔韧性、表面可能性等指标,综合预测VP1蛋白的B细胞抗原表位在第39~40、159~167、212~220、287~290位氨基酸残基的可能性较大。结论EV71 VP1蛋白多个区段具有抗原潜力通过生物信息学方法预测VP1蛋白二级结构及B细胞表位为EV71疫苗研制提供理论基础。
肠道病毒71型;VP1;蛋白质二级结构;B细胞表位
肠道病毒71型(Enterovirus 71,EV71)是小RNA病毒科(Picornaviridae)肠道病毒(Enterovirus,EV)A组成员,1969年由Schmidt等从美国加利福尼亚州患有中枢神经系统疾病的婴儿粪便标本中分离到。分子流行病学研究显示,EV71的VP1蛋白不但具有与血清型相对应的遗传多样性,并且是特异性中和表位所在区域,直接影响病毒的抗原性。目前,我国流行的EV71以C4亚型为主,采用生物信息学方法对EV71分离株进行种系分析以及VP1蛋白二级结构和B细胞表位预测。旨在为EV71疫苗的研究提供理论依据,同时为各类诊断试剂的研发提供依据。
1 材料与方法
1.1 样本来源 2010年宁波市疾病预防控制中心病毒研究所共收集全市177份HFMD患者标本,患者的年龄范围在0~24岁,男女比例是2.04∶1。其中粪便、咽拭子、肛拭子、脑脊液标本各89、26、59、3份。临床标本接种非洲绿猴肾细胞(VERO)进行病毒分离,置于36℃培养,每天观察细胞培养管的细胞病变效应(CPE)并记录,每份标本至少传3代,如果出现EV特异性CPE则保存于-70℃条件下待用。本研究的31株EV71均来自此次分离的EV71流行株,其它EV71和柯萨奇病毒A组16型(Coxsackievirus,Cox A16)代表株来自 NCBI的GenBank Database。
1.2 病毒分离物VP1编码区RT-PCR基因扩增和鉴定 取分离培养液200μL采用QIAGEN Reansy Mini Kit试剂盒提取RNA,并进行全长VP1编码区的核苷酸序列测定和分析。特异性引物序列:VP1-F:5’-GCAGCCCAGAAGAACTTCAC-3’;VP1-R:5’-ACCACTCTAAAGTTGCCCAC-3’,RT-PCR反应参数:42℃反转录30min;95℃预变性4min;以95℃45s、58℃90s、72℃1min扩增35个循环;72℃后延伸10min。使用1.25%琼脂糖凝胶电泳鉴定PCR产物。
1.3 序列测定 扩增产物委托英潍捷基(上海)贸易有限公司进行基因测序。
1.4 EV71 VP1区同源性和亲缘进化分析 VP1序列分析整理和同源性分析使用DNAStar V7.10软件;多序列比对和邻接法(neighbor-joining,NJ)构建系统发生树使用 Mega4.0.2软件[1]。
1.5 VP1蛋白二级结构预测 对31份分离株的VP1序列分别应用EX-PASY服务器上的GOR4[2]和SOPMA两种方法分析预测EV71 VP1蛋白的二级结构[3]。
1.6 EV71 VP1蛋白亲水性、柔韧性、表面可能性和B细胞抗原表位的预测 应用DNAStar软件的Protean程序,采用 Hopp-Woods和 Kyte-Doolittle方法预测氨基酸的亲水性,采用Karplus-Schultz和Emini方法预测柔韧性[4]及表面可能性,采用Jameson-Wolf方法预测潜在的B细胞抗原表位,综合评价VP1蛋白的B细胞抗原表位。
2 结 果
2.1 宁波EV71流行株VP1蛋白同源性和遗传进化分析 将31株EV71病毒分离物,进行全长VP1区的核苷酸序列测定分析,结果显示31株EV71 VP1区核苷酸序列长度均为891bp,编码297个氨基酸,没有核苷酸的插入和缺失。本研究的31株EV71 VP1序列虽有一定差异,但核苷酸和氨基酸同源性很高,分别在97.1%~100%和99.3%~100%之间,其中4株的同源性为100%;与基因库(GenBank Database)检索到各基因型和基因亚型EV71代表株VP1序列进行同源性分析发现,31株EV71的VP1蛋白与C4a基因亚型的核苷酸和氨基酸同源性最高在93.6%~99.3%和98.3%~100%之间;与Cox A16核苷酸和氨基酸同源性最低在62.8%~63.3%和71.8%~72.5%之间。宁波分离株与2008-2010年中国大陆流行株的核苷酸序列具有高度同源性。根据EV71的VP1编码区核苷酸序列,运用Mega4.0.2软件将宁波EV71分离株与GenBank Database检索到EV71代表株构建亲缘进化树(见图1),宁波分离株属于C4基因亚型的C4a进化分支,并存在多个传播链,与2008-2010年国内其他地方的分离流行株亲缘关系较近,进化途径相似,说明2010年EV71宁波株对近几年中国大陆EV71流行株具有一定代表性。
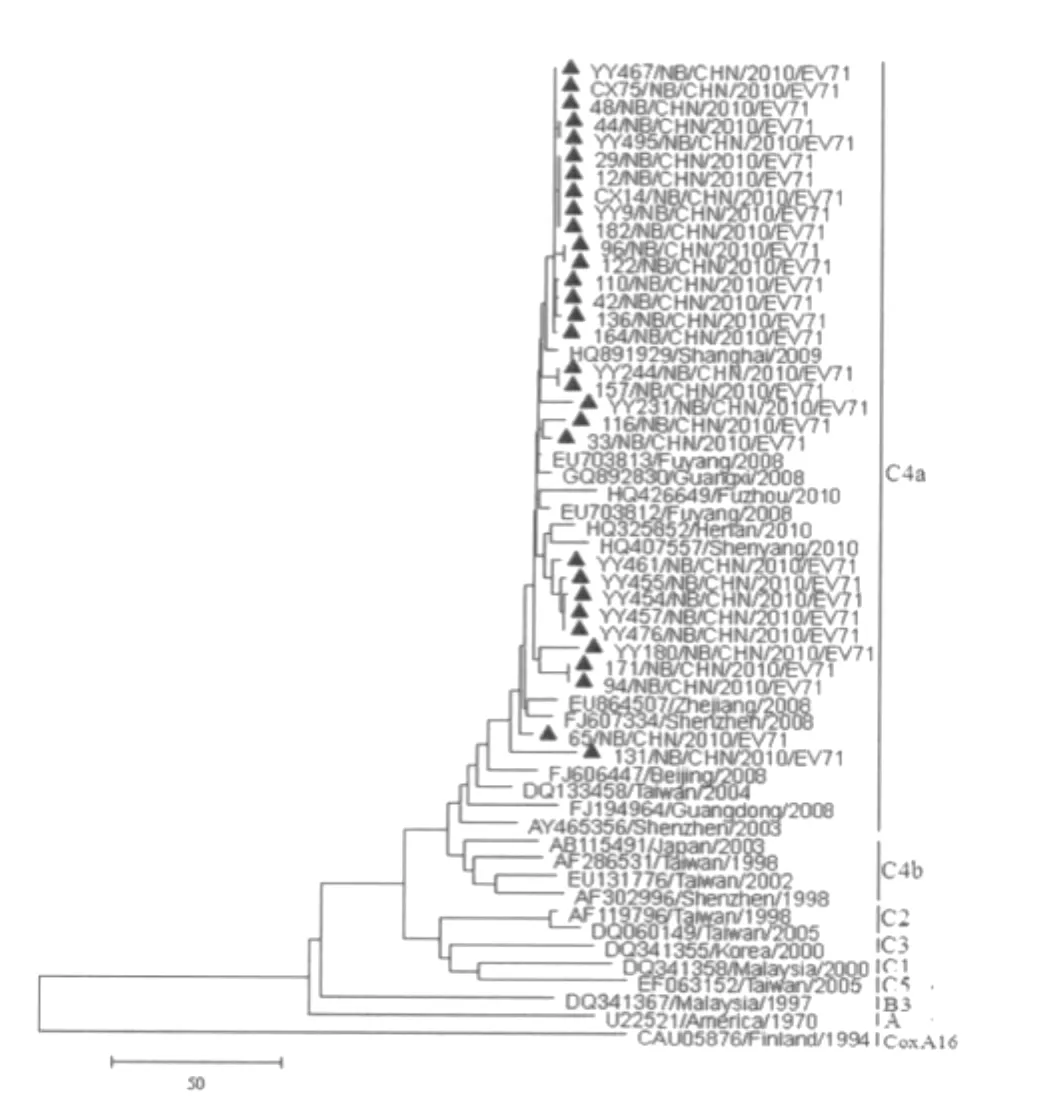
图1 EV71分离株和其他各个基因型EV71代表株全长VP1区亲缘进化树注:▲代表本研究中的EV71宁波株Fig.1 The phylogenetic tree of VP1 gene among 31 strains and the representative strains of EV71▲On behalf of the study of strains EV71 in Ningbo
2.2 EV71 VP1二级结构 运用EX-PASY服务器上的GOR4和SOPMA两种方法预测EV71 VP1蛋白的二级结构,两种方法的预测结果无显著差异,均提示EV71 VP1的二级结构以无规则卷曲和β-片层为主,并有多个α-螺旋的形成。31份标本VP1蛋白的无规则卷曲重合区域主要位于26~31、37~40、54~60、71~76、96~106、132~133、156~157、160~166、172~178、184~189、208~212、225~231、239~246、262~270、274~282位氨基酸;β-片层重合区域主要位于68~70、93~94、109~113、135~139、203~206、221~223、232~238、247~253、294~296位氨基酸;α-螺旋重合区域主要位于16~21、46~51、63~67、80~86、116~126位氨基酸。
2.3 EV71 VP1蛋白亲水性、柔韧性、表面可能性和B细胞抗原表位的预测 对本研究的31份氨基酸序列分别进行亲水性,柔韧性,表面可能性分析,并根据各项指标综合预测B细胞抗原表位。
2.3.1 亲 水 性 分 析 运 用 Kyte-Doolittle 和Karplus-Schulz方法分析VP1蛋白的亲水性,结果显示该蛋白存在较多亲水性区域,56~64、118~124、159~169、212~222位的氨基酸亲水性指数普遍最高,提示该区域暴露于表面并成为抗原表位的可能性较大。
2.3.2 柔韧性分析 运用 Karplus-Schulz方法分析蛋白的柔韧性,该蛋白骨架的柔韧性区域较多且呈均匀分布,主要分布在26~36、39~44、98~105、157~168、206~220、237~245、280~294位氨基酸。
2.3.3 表面可能性分析 运用Emini方法分析蛋白的表面可能性,结果显示该蛋白氨基酸的表面可能性较大,主要分布在37~40、159~167、212~221、241~245、264~271位氨基酸,易被溶剂分子接触,成为抗原表位。
通过 Antigenic index-Jameson-Wolf法预测 B细胞抗原决定簇:结果显示该蛋白存在多个抗原可能性较大的区域,并均匀分布,其中第35~43、53~65、70~80、118~124、158~169、206~224、238~245、262~270、275~293的抗原指数较高,提示该区段含有潜在的抗原决定簇。结合VP1蛋白的亲水性,柔韧性,表面可能性[5]的重合区域,并分析VP1的蛋白质二级结构,预测VP1蛋白以第39~40、159~167、212~220、287~290位氨基酸成为抗原表位的可能性较大。(由于版面所限,图2为其中4株EV71的亲水性、柔韧性、表面可能性软件截图示例)。图2可见,不同株C4基因亚型EV71 VP1的B细胞抗原表位软件分析结果基本一致。

图2 VP1蛋白的亲水性、柔韧性、表面可能性分析Fig.2 Analysis of hydrophilicity、flexility and surface probability in VP1 gene
3 讨 论
EV71分子流行病学显示,EV71的VP1蛋白是最重要的衣壳蛋白,不但具有与血清型相对应的遗传多样性,并且是特异性中和表位所在区域,直接影响病毒的抗原性,研究EV71VP1蛋白的B细胞抗原表位对疫苗研究具有重要意义[6]。VP1蛋白通常用于病毒的基因型别鉴定和遗传进化分析,目前发现EV71至少包括11个基因型(A、B1~B5、C1~C5),中国大陆EV71流行株以C4亚型为主。本研究的31株EV71与国内正在流行的C4a基因亚型的EV71同源性较高,亲缘关系近。构建亲缘进化树也显示宁波分离株属于C4基因亚型的C4a进化分支,并存在多个传播链,与2008-2010年国内其他地方的分离株距离较近[7-8],有共同进化趋势。宁波株与C4a亚型氨基酸的高度同源性,说明使用宁波流行株来预测中国大陆流行株的B细胞抗原表位具有可行性。
应用生物信息学方法预测蛋白质的抗原优势表位,并通过体外合成其肽段进行试验验证,是目前获得抗原优势表位肽段的一种重要方法,即省时省力、又经济高效[9-10]。本研究应用蛋白质二级结构、亲水性、柔韧性、表面可能性来预测EV71 C4a基因亚型中国大陆代表株的B细胞抗原表位。EV71 VP1蛋白的二级结构以无规则卷曲和β-片层为主,α-螺旋较少,而影响蛋白质抗原表位形成的主要因素为具有较多的无规则卷曲,与理论相符。结合EV71 VP1蛋白的亲水性、柔韧性、表面可能性分析的重合区域,第39~40、159~167、212~220、287~290位氨基酸残基成为抗原表位的可能性较大。除了这四个区段,还存在其他抗原指数较高的区段,在蛋白成熟的过程中许多抗原指数高的区段会发生构象变化,被外层的氨基酸残基或糖基所遮蔽,所以预测结果仍需要进一步实验证明。图2可见,不同株C4a基因亚型EV71 VP1的B细胞抗原表位软件分析结果基本一致,可以推测同一基因亚型内抗原表位非常相近。
EV71感染可引起多种临床症状,其感染引发的神经症状常引起儿童死亡,长期以来人们研究EV71的毒力位点,但并未得到确定答案。Mc Minn等对1999年澳大利亚流行的C2基因亚型EV71 VP1区进行分析,发现表现为HFMD的分离株与具有神经毒性的分离株主要区别在于第170位氨基酸的不同,前者为丙氨酸,后者为缬氨酸,可能是此氨基酸的变化,改变的VP1蛋白的空间结构,从而使毒力发生根本性改变,但这点并未得到实验证实。本研究的EV71为C4a基因亚型,其170位氨基酸为丙氨酸,生物学软件综合分析发现其抗原潜力并不大,将170位的丙氨酸人为更改为缬氨酸后,抗原潜力无明显改变,第170位氨基酸的是否为毒力位点仍需动物实验来验证,并且不同型别EV71的毒力位点可能存在差异。Foo DGG根据EV71 VP1蛋白的氨基酸序列合成了95段有部分重叠的肽段,与载体连接后免疫小鼠,取血清进行中和抗体试验,其中SP55(163~177)和SP70(208~222)片段能够诱发中和抗体,其中SP70具有高度保守性,成为EV71多肽疫苗的候选肽段。SP55和SP70分别与本次预测的158~169和212~221区段有一致区域。朵建英等[11]研究EV71亚单位疫苗,其中Vac6用于小鼠攻毒试验后5d,肌肉组织、小肠组织病理观察和免疫组织化学检测不到病理改变和病毒抗原的存在,对幼鼠的保护效果明显,起到与灭活全病毒疫苗相同的抗病毒保护作用。Vac6包含的23~53、47~83、108~149、248~292为氨基酸片段,与本次预测结果有核心重合区域。同时说明生物信息学方法对抗原位点的预测具有一定可行性。本次研究为EV71的抗原性分析提供理论材料,有助EV71新型疫苗的研究和生物学试剂的研发。
[1]Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular Evolutionary Genetics Anzlysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24 :1596-1599.
[2]Garnier J,Gibrat JF,Robson B.GOR method for predicting protein secondary structure from am ino acid sequence[J].Methods Enzymol,1996,266(5):540-553.
[3]Andersen CA,Rost B.Secondary structure assignment[J].Methods Biochem Anal,2003;44:341-363.
[4]Karplus PA,Schultz GE.Prediction of chain flexibility in proteins[J].Naturwissenchaften,1985,72(4):212-213.
[5]冯起甲,徐智,吴国明,等.脂多糖结合蛋白的B细胞抗原表位预测及其效应初步分析[J].第三军医大学学报,2008,30(4):292-294.
[6]檀晓娟,许文波.肠道病毒71型的分子流行病学现况[J].中国疫苗和免疫,2008,14:361-367.
[7]赵生仓,姜双应,刘桂香,等.青海省分离到肠道病毒71型及其VP1区基因特征分析[J].中华实验和临床病毒学杂志,2010(24):436-438.
[8]Yang F,Ren L,Xiong Z,et al.Enterovirus71 outbreak in the People's Republic of China in2008[J].J Clin Microbiol,2009,47,47(7):2351-2352 .
[9]Zhou BP,Chen XC,Liu WL,et al.Expression of recombinant VP1 protein of enterovirus 71 anddevelopment of serological assay for detection of EV71 infection[J].Chinese journal experimental and clinical virology ,2008,22(6):492-494.
[10]Ong KC,Devi S,Cardosa MJ,et al.Formaldehyde-inactivated whole virus vaccine protects a murine model of Enterovirus 71 encephalomyelitis against disease[J].J Virol,2009,84:661-665.
[11]朵建英.EV71小鼠模型的建立及其在疫苗评价总的中应用[D].北京:中国协和医学院,2010:64-74.
Prediction on secondary structure and B cell epitope of enterovirus type 71 for VP1 gene
WANG Jin,XU Guo-zhang,NI Hong-xia
(Medical School of Ningbo University,Ningbo 315211,China)
The purpose of this study was to predict the secondary structure and B cell epitopes of enterovirus type 71 of VP1 Gene of C4 subgenotype,so as to provide theory evidence for the study of vaccine preparation for EV71.EV71 strains were isolated from hand,foot and mouth disease in Ningbo in 2010.Homology and phylogenetic analysis based on VP1 with others EV71 have published in GenBank were conducted.The secondary structure of EV71 based on VP1 was analyzed using the GOR4 and SOPMA,and the B cell epitope of EV71 was predicted by combining the hydrophilicity、flexility and surface probability.The Ningbo strain shared the highest homology of nucleotide and amino acid with the type C4a were 93.6%-99.3%and 98.3%-100%,phylogenetic analysis revealed that Ningbo strains clusterd within the C4a evolution branch of C4 subgenotype.The secondary structure of EV71 VP1 showed random coils andβ-sheet mainly with a number of antigen index higher section.The B cell epitopes was probably located at the regions of 39-40,159-167,212-220,and 287-290 of VP1 Gene.The secondary structure and B cell epitopes of EV71 VP1 could be predicted by bioinformatics methods,which providing theory evidence for the study of vaccine preparation for EV71.
Enterovirus 71;VP1;protein secondary structure;B cell epitope
1002-2694(2011)10-0001-05
R373.2
A
*浙江省卫生厅医药卫生科学研究基金资助项目(2009A190);宁波
市科技局重大科技专项资助项目(2009C50008)
许国章,Email:xugz@nbcdc.org.cn
1.宁波大学医学院,宁波 315211;2.宁波市疾病预防控制中心,宁波 315010
2011-07-25;
2011-09-06

