(S)-酮基布洛芬拆分用脂肪酶基因的克隆及表达
许丽娟,赵玉红,刘瑞恩,赵运英,张金红
南开大学生命科学学院,天津 300071
(S)-酮基布洛芬拆分用脂肪酶基因的克隆及表达
许丽娟,赵玉红,刘瑞恩,赵运英,张金红
南开大学生命科学学院,天津 300071
本实验室筛选出一株具有不对称拆分消旋酮基布洛芬氯乙酯的菌株 NK13为材料,经鉴定为巨大芽孢杆菌Bacillus megaterium。通过构建其基因文库,从中筛选得到阳性克隆重组子pUC-NK1。测序分析表明,该重组子质粒中包含一长度为 633 bp的脂肪酶基因的完整开放阅读框,核苷酸同源性对比证明该脂肪酶基因属首次发现(GenBank Accession No.EU381317),将此基因克隆到原核表达载体pET21b(+)中构建重组表达质粒pET-NKest1,转化Escherichia coli BL21,经Isopropyl-β-D-Thiogalactoside(IPTG)诱导在宿主菌中得到表达,经SDS-PAGE电泳检测证明该脂肪酶成熟蛋白分子量约为20 kDa。薄层层析与HPLC检测结果显示,表达菌株转化外消旋酮基布洛芬氯乙酯得到(S)-酮基布洛芬过量(e.e.%),由野生菌NK13的5.84%提高到75.28%,提高约15倍,说明该脂肪酶具有优先拆分得到(S)-酮基布洛芬的特性。
不对称拆分,(S)-酮基布洛芬,脂肪酶,克隆,表达
Abstract:We screened a strain NK13 for a certain extent asymmetric hydrolysis the rac-ketoprofen Chloroethyl ester to(S)-Ketoprofen.As identified, NK13 was Bacillus megaterium.Digested NK13 genomic DNA with Sau3AΙ partially and recovered the fragment from 2 kb to 6 kb, cleaved the plasmid of pUC18 with BamH Ι, ligated the 2−6 kb fragment of NK13 genomic DNA into pUC18 plasmid, and then transformed an Escherichia coli strain DH5α.We created the gene library of NK13 and obtained a positive clone, pUC-NK1 in the library from the tributyrin flat.The result of sequencing showed that there was a whole open read frame(ORF)of 633 bp lipase gene in the plasmid of pUC-NK1.To compare with the genes of GenBank, this lipase gene was reported firstly(GenBank Accession No.EU381317).The lipase gene was amplified by PCR, using pUC-NK1 plasmid as template, and subcloned into the high expression vector pET21b(+)under the control of T7 promoter.The recombinant plasmid, pET-NKest1, was then transformed into an Escherichia coli strain BL21(DE3)for the production of recombinant lipase protein.After 3 hours of induction by isopropyl-β-D-thiogalactoside(IPTG), lipase was expressed.SDS-PAGE analysis showed that the relative molecular mass of the lipase protein was about 20 kDa.The result of high performance liquid chromatography(HPLC)showed that the conversion rate of the recombinant strain was fifty times than the wild strain NK13’s.The(S)-Ketoprofen enantiomeric excess of the recombinant strain was 75.28%, which indicated that thelipase could hydrolyze(S)-Ketoprofen Chloroethyl ester firstly.If we research the conditions of the hydrolysis rac-ketoprofen Chloroethyl ester of this lipase further, maybe it could offer a foundation to product(S)-Ketoprofen industrially.
Keywords:enantioselectivity,(S)-Ketoprofen, lipase, cloning, expression
2-芳基丙酸类药物属于非甾体消炎镇痛药,是解热镇痛、消炎抗风湿的基本药物和主要产品,它是目前处方药和非处方药处方量最大的一类药物。常用的有布洛芬、奈普生和酮基布洛芬 3种,其 α位有一个手性碳原子存在(S)-构型和(R)-构型一对光学对映体,临床上一般以外消旋体的形式供应。其中酮基布洛芬的消炎镇痛作用是阿司匹林的 150倍,解热作用是阿司匹林的100倍,临床上用于治疗慢性类风湿性关节炎、变性性关节炎、外伤和术后疼痛等。药理实验表明,其(S)-异构体的消炎镇痛作用是外消旋酮基布洛芬的2倍,而(R)-异构体的活性很低,但有其他方面药用价值,它对牙周病的骨质疏松有治疗作用[1]。
目前,(S)-酮基布洛芬的制备方法主要有不对称合成、酶法拆分、非对映体结晶拆分等。其中利用微生物脂肪酶催化的立体选择性拆分[2-6]获得单一对映体具有立体专一性强、拆分效率高、生产条件温和、无环境污染等诸多优点,已经成为研究的热点。然而酶法拆分在工业化生产中也存在着许多缺点,如需对过量的生物量进行处理,对营养供应进行适当的控制,每批反应的产率并不稳定等,目前我国工业化生产条件尚未成熟。
近年来,研究微生物酯酶/脂肪酶拆分酮基布洛芬的报道也有很多[7-9]。意大利Menarini公司开发的(S)-酮基布洛芬于 1996年首次在西班牙上市,现在我国尚无生产此药的厂家。本实验室经过筛选得到了一株具有不对称拆分外消旋酮基布洛芬氯乙酯的野生菌 NK13,前期研究[10]已表明该野生菌中存在不止一种能够拆分消旋酮基布洛芬氯乙酯的酶,其中已经得到一个新的酯酶基因[11],本研究通过构建其基因文库,克隆表达了另一个脂肪酶基因,并对该酶的性质进行了研究。
1 材料与方法
1.1 菌株和质粒
巨大芽孢杆菌Bacillus megaterium NK13菌株,从生产奈普生药厂附近的土壤中分离得到;pUC18为本实验室保存;E.coli DH5α为本实验室保存;pET21b(+)及E.coli BL21(DE3)由南开大学生命科学学院蔡宝立教授惠赠。
1.2 试剂和培养基
各种限制性内切酶、DNA marker、碱性磷酸酶(Alkaline phosphatase)、T4 DNA连接酶、DNA凝胶回收试剂盒购自大连宝生物TaKaRa公司。溶菌酶、RNase、蛋白酶K、氨苄青霉素购自北京鼎国生物技术公司。其他试剂均为分析纯。NK13菌株液体培养基:葡萄糖1.2%;蛋白胨0.5%;NaCl 1%,pH 7.0。LB培养基用于E.coli DH5α培养,SOC培养基用于转化后菌体复苏。
1.3 基因组DNA文库的构建
提取NK13基因组DNA,用Sau3AI进行部分酶切后,琼脂糖凝胶回收2~6 kb的DNA片段,质粒pUC18用BamHⅠ酶切后用去磷酸化酶(CIAP)处理。回收DNA片段和酶切后的去磷酸化载体用T4 DNA连接酶连接后,用电转化[12]方法转化 E.coli DH5α,涂布在含有三丁酸甘油酯和罗丹明B的Amp抗性 LB平板上培养,构建基因文库,筛选有水解圈的阳性克隆,命名为pUC-NK1。将过夜培养的阳性克隆pUC-NK1菌液送三博远志公司进行测序。
1.4 脂肪酶基因的扩增
根据测序结果,在脂肪酶基因两侧设计一对引物,P1:5'-TTCCATATG GCGGAAGCAAACCATAATCCG-3',P2:5'-TTGGATCC GTTTGTATTTTGTCCCCCGC-3'(在上下游引物 5'端分别引入 NdeⅠ和 BamHⅠ酶切位点,下划线标识),以阳性克隆 pUC-NK1的质粒DNA为模板,PCR扩增脂肪酶基因。PCR反应条件:94℃预变性3 min;94℃变性50 s,51℃退火30 s,72℃延伸1 min,共30个循环。
1.5 重组表达质粒pET-NKest1的构建
提取pUC-NK1菌株质粒并以其为模板,用引物P1和P2扩增目的基因,试剂盒回收PCR产物,与T-vector连接转化E.coli DH5α,PCR鉴定的阳性重组质粒用NdeⅠ和BamHⅠ双酶切,回收脂肪酶基因片段,与经同样双酶切处理的pET21b(+)载体连接,构建酯酶基因重组表达质粒 pET-NKest1,转化E.coli BL21(DE3),用于脂肪酶的表达。
1.6 脂肪酶的表达
接pET-NKest1表达菌株单菌落于20 mL氨苄青霉素终浓度为100 µg/mL的 LB培养基中,37℃过夜培养。按 1%接种量转入 500 mL培养基,至OD600值为0.6~0.8之间时,加入IPTG至终浓度为0.8 mmol/L,37℃培养3 h,收获菌体,SDS-PAGE电泳观察结果。
1.7 薄层层析法检测表达菌株对酮基布洛芬氯乙酯转化率
在诱导3 h后的pET-NKest1表达菌株液体培养基中加入10 µL酮基布洛芬氯乙酯(20 mg/mL),间隔 2 h取出一份加入盐酸和乙酸乙酯萃取,取有机相,进行薄层层析(TLC),美国UVP公司 GDS-8000型凝胶成像分析系统测定转化率。
1.8 高压液相色谱(HPLC)检测产物光学纯度
将层析板上与酮基布洛芬对应的点取下分别放到离心管中,再加1 mL乙酸乙酯充分振荡萃取;12 000 r/min离心10 min后取上清液作为HPLC检测样品。在CHIROBIOTIC V柱上,以四氢呋喃:柠檬酸+柠檬酸钠(pH 6.3,0.05 mol/L)=10:90缓冲液为流动相检测酮基布洛芬的光学纯度(e.e.值)。进样量20 µL/次,流速1 mL/min。
2 结果
2.1 基因文库的构建
将Sau3AI部分酶切的NK13基因组DNA和质粒pUC18用BamHⅠ酶切后,用去磷酸化酶(CIAP)处理,产物经1%琼脂糖凝胶电泳(图1)。
构建NK13基因文库得到了约20000个转化子,从其中筛选得到一株具有水解三丁酸甘油酯能力的阳性克隆pUC-NK1(图2)。
2.2 脂肪酶基因的扩增

图1 NK13基因组DNA和pUC18质粒的酶切结果Fig.1 Enzyme digestion of NK13 genomic DNA and plasmid pUC18.M1: λ-Hind Ⅲ marker; M2: DL2000 marker; 1: NK13 DNA digestied with Sau3AI; 2: genomic DNA of NK13; 3:plasmid pUC18; 4: pUC18 digested with BamHⅠ.
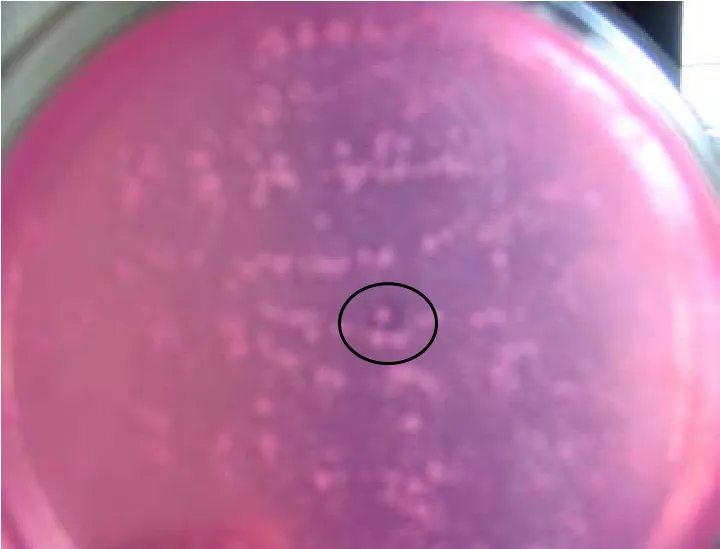
图2 基因文库中的阳性克隆Fig.2 Positive clones of pUC-NK1 in gene libraries.

图3 脂肪酶基因序列Fig.3 Sequence of the lipase gene and its deduced amino acid.
测序结果显示 pUC-NK1阳性质粒中插入的外源片段长度为3038 bp,经DNAStar软件进行分析,发现一长度为633 bp的开放阅读框(图3),编码的蛋白在其N端有一段信号肽序列(斜体部分)。将该核苷酸序列与NCBI的GenBank数据库中的基因核苷酸序列进行比对,结果显示其与 Bacillus subtilis lipase(lipW)基因同源性最高,为77%。其推导的氨基酸序列与Bacillus subtilis subsp.酯酶同源性最高,为85%。该脂肪酶基因已录入NCBI数据库,GenBank登录号为EU381317。
2.3 重组表达质粒的构建
以基因文库中得到的pUC-NK1质粒为模板,用引物 P1和 P2成功扩增出与预期大小相符的条带(图4)。
回收PCR产物,与T-vector连接测序,结果表明重组质粒中的基因片段与预期的脂肪酶基因序列一致。用NdeⅠ和BamHⅠ从质粒T-vector上双酶切脂肪酶酶基因,与经相同酶切的 pET21b(+)载体连接,成功构建重组表达质粒pET-NKest1,转化E.coli BL21(DE3)宿主菌。筛选阳性转化子,提质粒用NdeⅠ和 BamHⅠ进行双酶切检验(图5),证明构建的表达质粒pET-NKest1已成功转入E.coli BL21(DE3)。
2.4 脂肪酶的表达
pET-NKest1表达菌株经IPTG诱导3 h后,目的蛋白得到表达,经10%SDS-PAGE电泳检测,发现重组蛋白的分子量约为20 kDa(图6)。
2.5 表达菌株中酮基布洛芬氯乙酯的转化率
表达菌株中酮基布洛芬氯乙酯转化率随着转化时间的增加而提高,转化20 h时,酮基布洛芬氯乙酯转化率超过50%(图7)。
2.6 HPLC检测产物光学纯度
分别将2~20 h的转化产物进行高效液相色谱分析(图8),其中4 h时的转化产物(S)-酮基布洛芬对映体过量值(e.e.%)最高,达75.28%。

图4 PCR扩增脂肪酶基因产物Fig.4 PCR products of the lipase gene.M: DL2000 marker; 1:PCR products.
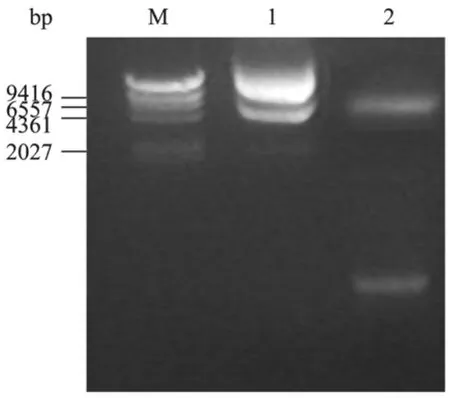
图5 pET-NKest1质粒的酶切鉴定Fig.5 Identification of plasmid pET-NKes1 by enzyme digestion.M: λ-Hind Ⅲ marker; 1: plasmid pET-NKest1; 2:pET-NKest1 digestied with NdeⅠ and BamHⅠ.

图6 SDS-PAGE分析E.coli BL21(DE3)中脂肪酶的表达Fig.6 SDS-PAGE analysis of the recombinant protein expressed in the E.coli BL21(DE3).M: protein marker; 1: the cell of pET-NKest1 induced at 3 h; 2,3: E.coli BL21(DE3)including pET21b(+)plasmid; 4: supernatant of of pET-NKest1 induced at 3 h.

图7 E.coli BL21(DE3)转化酮基布洛芬氯乙酯TLC结果Fig.7 Transformation for ketoprofen chloroethyl ester in E.coli BL21(DE3).M: protein marker; 1−10: the transformated products at 2 h, 4 h, 6 h, 8 h, 10 h, 12 h, 14 h, 16 h, 18 h, 20 h;A: ketoprofen chloroethyl ester; B: ketoprofen.
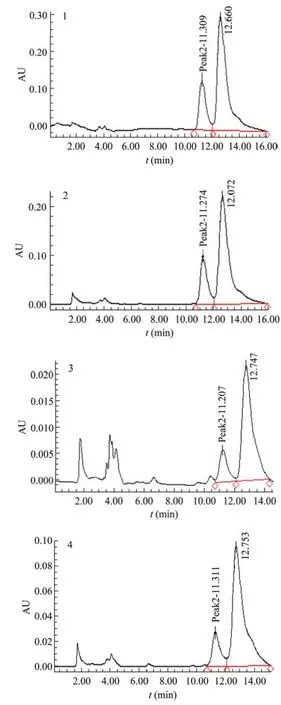
图8 HPLC检测转化产物的光学纯度Fig.8 HPLC analysis of the transformated products.1−4:HPLC analysis of the transformated products at 2 h,4 h, 10 h,20 h.
3 讨论
脂肪酶是一类重要的水解酶,在食品、医药、皮革和洗涤等行业中都有广泛的应用。随着酶学研究的发展,脂肪酶的很多特性被发现,其中脂肪酶可以专一性用于制备许多用化学方法难以合成的手性化合物及其前体,而且由于脂肪酶催化的反应还有选择立体专一性强、拆分效率高、生产条件温和、无环境污染等诸多优点,成为手性分离技术的研究热点之一。国外对脂肪酶的研究报道较多,而国内这方面的研究起步较晚,不同微生物产的脂肪酶在酶活、酶学性质及应用领域等方面都存在很大的差异。
本实验室以自筛选得到的一株具有不对称能力的菌株Bacillus megaterium NK13为材料,前期的研究已发现野生 NK13菌中存在不止一种可以拆分酮基布洛芬氯乙酯的酶,本研究成功地从 NK13中克隆到了其中一个能够拆分酮基布洛芬氯乙酯的脂肪酶基因(GenBank Accession No.EU381317),构建了其重组表达质粒并在大肠杆菌中表达出具有生物活性的脂肪酶,检测了表达菌株对底物的转化效率及产物的光学纯度,4 h时转化产物(S)-酮基布洛芬达到75.28%,是野生菌的15倍。如果进一步优化脂肪酶的表达及拆分条件,或进一步筛选NK13中其他立体选择性更高的酶,将具有更好的工业应用前景。
本研究得到的这个脂肪酶是从 Bacillus megaterium NK13中获得的第2个具有不对称拆分外消旋酮基布洛芬氯乙酯能力的酶。与前期 NK13中发现第一个具有拆分外消旋酮基布洛芬氯乙酯能力的酯酶[11]相比,本试验发现的脂肪酶具有优先选择拆分(S)-酮基布洛芬的能力,拆分得到(S)-酮基布洛芬过量达到了75.28%,但该脂肪酶的转化速率比酯酶要慢,酯酶45 min时对底物的转化率已接近50%,而该脂肪酶需要10 h转化率才能达到一半。这个结果有利于揭示野生菌 Bacillus megaterium NK13对外消旋酮基布洛芬氯乙酯的转化率高、而光学专一性不强是否与微生物中具有转化不同光学对映体化合物的酶的共存直接相关。
REFERENCES
[1]Famaey JP, Paulus HE.Therapeutic Applications ofNSAIDs.New York: Marcel Dekker, 1992.
[2]Kato K, Gong Y, Saito T, et al.Efficient preparation of optically active ketoprofen by Mucojavanicus lipase immobilized on an inorganic support.J Biosci Bioeng,2000, 90(3): 332–334.
[3]Berglund P.Controlling lipase enantioselectivity for organic synthesis.Biomol Eng, 2001, 18(1): 13–22.
[4]Shen D, Xu JH, Gong PF, et al.Isolation of esterase producer trichospron brassicae and its catalytic performance in kinetic resolution of ketoprofen.Microbiology, 2002, 29(1): 46–49.沈端, 许建和, 宫鹏飞, 等.酮基布洛芬拆分用酯酶产生菌的筛选及其催化特性.微生物学通报, 2002, 29(1):46–49.
[5]Wu JC, Chua PS, Chow Y, et al.Extraction of Candida rugosa lipase from aqueous solutions into organic solvents by forming an ionpaired complex with AOT.J Chem Technol Biotechnol, 2006, 81(6): 1003–1008.
[6]Friedrich W, Friedrich AR, Reiner M, et al.Identification of environment strains of Bacillus mycoides by fatty acid analysis and species-specific 16S rDNA oligonucleotide probe.FEMS Microbiol Ecol, 1997, 24(3): 201–209.
[7]Choi GS, Kim JY, Kim JH, et al.Construction and characterization of a recombinant esterase with high activity and enantioselectivity to(S)-ketoprofen ethyl ester.Protein Express Purif, 2003, 29(1): 85–93.
[8]Yoons, Kims, RyuY, et al.Identification and characterization of a novel(S)-ketoprofen-specific esterase.Int J Biol Macromol, 2007, 41(1): 1–7.
[9]Long ZD, Xu JH, Zhao LL, et al.Overexpression of Serratia marcescens lipase in Escherichia coli for efficient bioresolution of racemic ketoprofen.J Mol Catal B:Enzym, 2007, 47(3/4): 105–110.
[10]Zhang JH, Guan R, Tan ZL, et al.Purification and properties of lipases/esterases from a bacillus strain for enantioselective resolution of(S)-Ketoprofen.Art Cells Blood Subst Immob Biotechnol, 2005, 33(4): 435–445.
[11]Xu LJ, Tan ZL, Liu G, et al.Cloning and expression of esterase gene to enantioseletive resolution of(S)-ketoprofen in NK13.Chin J Biotechnol, 2008, 28(2): 32–36.许丽娟, 谭之磊, 刘刚, 等.NK13中(S)-酮基布洛芬拆分用酯酶基因的克隆及表达.中国生物工程杂志, 2008,28(2): 32–36.
[12]Ausubel F, Brent R, Kingston RE, et al.Yan ZY, Wang HL,trans.Short Protocols in Molecular Biology.Beijing:Science Press, 2001.Ausubel F, Brent R, Kingston RE, 等著.颜子颖, 王海林译.精编分子生物学实验指南.北京: 科学出版社, 2001.
Cloning and expression of lipase gene to enantioselective resolution of(S)-ketoprofen
Lijuan Xu, Yuhong Zhao, Ruien Liu, Yunying Zhao, and Jinhong Zhang
College of Life Science of Nankai University, Tianjin 300071, China
Received:July 3, 2009;Accepted:November 5, 2009
Supported by:Natural Science Foundation of Tianjin(No.05YFJMJC01100).
Corresponding author:Jinhong Zhang.Tel: +86-21-23508233; E-mail: jinhzhang@nankai.edu.cn天津市自然科学基金项目(No.05YFJMJC01100)资助。