一种利用基因表达变化检测、鉴别抗生素的新方法
赵冰,孙赵麟,杨亮,梁海华,沈立新,段康民
西北大学生命科学学院 西部资源生物与现代生物技术教育部重点实验室,西安 710069
一种利用基因表达变化检测、鉴别抗生素的新方法
赵冰,孙赵麟,杨亮,梁海华,沈立新,段康民
西北大学生命科学学院 西部资源生物与现代生物技术教育部重点实验室,西安 710069
本研究利用基因表达变化构建一个高通量检测筛选抗生素化合物的新方法。采用含 luxCDABE报道子的质粒pCS26和 pMS402为载体,构建大肠杆菌重组质粒、筛选铜绿假单胞菌以及沙门氏菌的随机启动子库,获得对抗生素有反应的启动子-报道子融合体,构成抗生素筛选体系。通过重复实验筛选出15个启动子-报道子融合体,它们对不同抗生素有不同的反应,通过这一系统可以检测样品中的抗生素,并初步判断与现有抗生素的异同。使用这种方法可以进行高通量筛选潜在的抗菌物质。
抗生素,筛选,启动子,报道子
Abstract:We designed and constructed an antibiotic screening system by using antibiotic responsive genes as reporters.Plasmid pCS26 carrying a promoterless luminescence reporter, luxCDABE, was used as the vector and the promoter regions of antibiotic responsive genes/operons from Escherichia coli were cloned upstream of the lux reporter to form the first part of the screening reporter array.Random promoter library of Salmonella enterica and Pseudomonas aeruginosa were screened for antibiotic responsive clones which consist of the second part of the screening array.The selected final reporter array responded to different antibiotics in distinct patterns and enabled in vivo high-throughput screening for antibiotics.Unknown antibiotics could, in general,be classified by analyzing the response patterns.This screening system is both sensitive and efficient and should prove to be a useful tool for screening new antibiotic compounds.
Keywords:antibiotics, screening, promoter, reporter
由于抗药性的出现和蔓延,临床使用的抗生素在逐渐失去效用,开发新的抗生素已成为人类医学和微生物学的一个紧迫课题[1-3],而新的筛选方法是新型抗生素研究的关键之一。新抗生素筛选方法需要具有高特异性和高灵敏性,而且最好可以对大量样品进行高通量检测[4]。
传统的抗生素筛选模型主要基于病原菌的体外实验检测寻找杀菌或抑菌物,其优点是得到的化合物可以进入病原菌细胞;缺点是作用点不明确,难以进行化学改进,筛选过程有明显的随机性,而且不灵敏[5]。上世纪70、80年代起进行的靶点筛选技术,主要是利用体外筛选体系,寻找生物化学抑制物,优点是比较灵敏,可检测低效物,可用于化学改进,而且筛选方便。但缺点是需要将体外实验转化为体内实验,筛选到的化合物常常面临难以进入细胞的问题[6-7]。基因标记方法是当前筛选方法中最先进的技术之一。通过标记特殊基因,寻找相关生物活性物,优点是灵敏、可检测低效物、可用于化学改进、易筛选、高通量、可进入细胞。但缺点是需要前期知识,需要建立系统[8]。
本实验利用以荧光素酶(Luciferase)基因(lux- CDABE)作为报道子的高通量基因表达检测技术,发现甄别对低于抑制浓度(Sub-inhibitory concentration)已知抗生素有反应的启动子。使用这些启动子-报道子系统进行抗生素的分类筛选。具备高通量、灵敏、高效等特点。
1 材料
1.1 菌株、培养基以及培养条件
铜绿假单胞菌(Pseudomonas aeruginosa,PA)pKD-phzA1[9]、大肠杆菌(Escherichia coli,E.coli)10B,质粒 pMS402、pCS26、铜绿假单胞菌随机启动子文库[10]、沙门氏菌Salmonella enterica随机启动子文库[11]均为本实验室保存。常规使用的培养基为LB,大肠杆菌、沙门氏菌和铜绿假单胞菌常规培养在 LB 培养基中 37℃有氧过夜培养。含有质粒的菌株根据需要分别添加卡那霉素(50 µg/mL)和甲氧苄氨嘧啶(300 µg/mL)。测试用菌使用前接单菌落到5 mL LB液体培养基中,加入相应抗生素,37℃摇床培养14 h后,调整OD600到0.440±0.01。
1.2 主要试剂和仪器
酵母粉、胰蛋白胨为英国Oxoid公司产品;氨苄青霉素(Ampicillin,Amp)、羧苄青霉素(Carbenicillin,Cb)、氯霉素(Chloramphenicol,Cm)、红霉素(Erythromycin,Em)、四环素(Tetracycline,Tc)、庆大霉素(Gentamicin,Gm)、壮观霉素(Spectinomycin,Spe)、甲氧苄氨嘧啶(Trimethoprim,Tmp)、环丙沙星(Ciprofloxacin,Cip)、利福平(Rifampicin,Rif)、磺胺(Sulfanilamide,SAs)、卡那霉素(Kanamycin,Kan)、琼脂粉均来自Wolsen公司;多粘菌素(Polymixin,poly)来自Sigma公司;DMSO、EDTA、Tris-base、培养基均为美国Amresco公司产品;限制性内切酶BamH I、Xho I购自美国 New England BioLabs公司;T4 DNA连接酶由TaKaRa公司提供;Taq酶、dNTPS均购自北京天为时代公司;DNA 产物纯化试剂盒购自北京鼎国公司;其他试剂均为国产分析纯。Wallac 1420 Multilabel Counter购自 PerkinElmer公司;离心机Centrifuge5415D、Centrifuge5810R、PCR仪Mastercycle Gradient均购自 Eppendorf公司;Luminescent Image Analyzer LAS 3000 购自 Fujifilm公司;96孔板、384孔板均为 Costar公司产品;Multi-blot Replicator(48 pins and 96 pins)为V&P公司产品。
2 方法
2.1 大肠杆菌胁迫相关基因重组质粒的构建
PCR扩增6个已知胁迫相关基因的启动子区域并将其克隆到含有luxCDABE 报道子的pCS26报道质粒上。以大肠杆菌染色体基因组为模板,利用特定的引物(表1)PCR扩增各个不同的启动子片段。PCR产物用 0.8%琼脂糖凝胶电泳检测。PCR扩增产物用DNA纯化试剂盒纯化后,用Xho I和BamH I酶切,以T4 DNA连接酶连接到用相同酶切的质粒pCS26上,电转化至10B菌株中,在含有卡那霉素的平板上筛选阳性克隆。获得带有相应基因启动子-lux报道子融合体质粒。相应基因的启动子活性以lux发光量(Counts per second,CPS)来表示。其他分子生物学操作依据报道的方法进行[12]。
2.2 启动子库筛选的方法
通过环丙沙星和羧苄青霉素对铜绿假单胞菌和沙门氏菌随机启动子库进行筛选。对铜绿假单胞菌Cb和 Cip的浓度分别为 5 µg/mL和 0.1 µg/mL;对沙门氏菌 Cb和 Cip的浓度分别为 7.5 µg/mL和0.005 µg/mL。将构建好的随机启动子库[10-11]接到含有上述浓度Cip或是Cb的含有相应抗生素的LB培养基的 384孔板中,37℃过夜培养,然后使用 96针转接器将该库中所有克隆同时转接到384孔板的Cip和Cb培养基中,将384孔板放在Victor Multilabel Counter 中于37℃每隔6 h对菌体的发光情况进行检测,共计检测30 h。以无抗生素培养基中菌体的发光值为对照,将在含有Cip或是Cb培养基中发光值增加3倍或3倍以上变化的克隆挑出,采用同样的方法转接至另一个384孔板中,每隔2 h检测其发光值,共计检测28 h,再将含抗生素条件下发光值有3倍或3倍以上变化的克隆挑出。最后将筛选出的阳性克隆转接到底部透光的黑色96孔板中,在Victor Multilabel Counter中每隔 0.5 h检测其发光值,同时检测其OD600值,共计检测24 h[11]。

表1 大肠杆菌中6个与胁迫相关因子的引物序列Table 1 Sequences of the primers used for PCR amplifying E.coli stress response gene promoters
2.3 DNA测序和序列分析
使 用 pZE.05(5′CCAGCTGGCAATTCCGA-3′)和 pZE.06(5′AATCATCACTTTCGGGAA-3′)分别作为正向和反向引物[11],对筛选出的对抗生素有反应的克隆进行PCR扩增,扩增产物用pZE.06为引物进行测序,并使用Vector NTI软件(Invitrogen公司)进行后续分析,确定有关克隆上的基因。
2.4 双层培养基混菌法抗生素反应的测试
下层为1%固体琼脂LB培养基,上层为0.5%琼脂 LB培养基并混合菌液。将混菌的培养基平板置于无菌操作室,风干20 min。然后在培养基中加入不同抗生素1.5 µL,在不同菌中抗生素的浓度不同(表2)。37℃过夜培养,然后于Las-3000下照相。
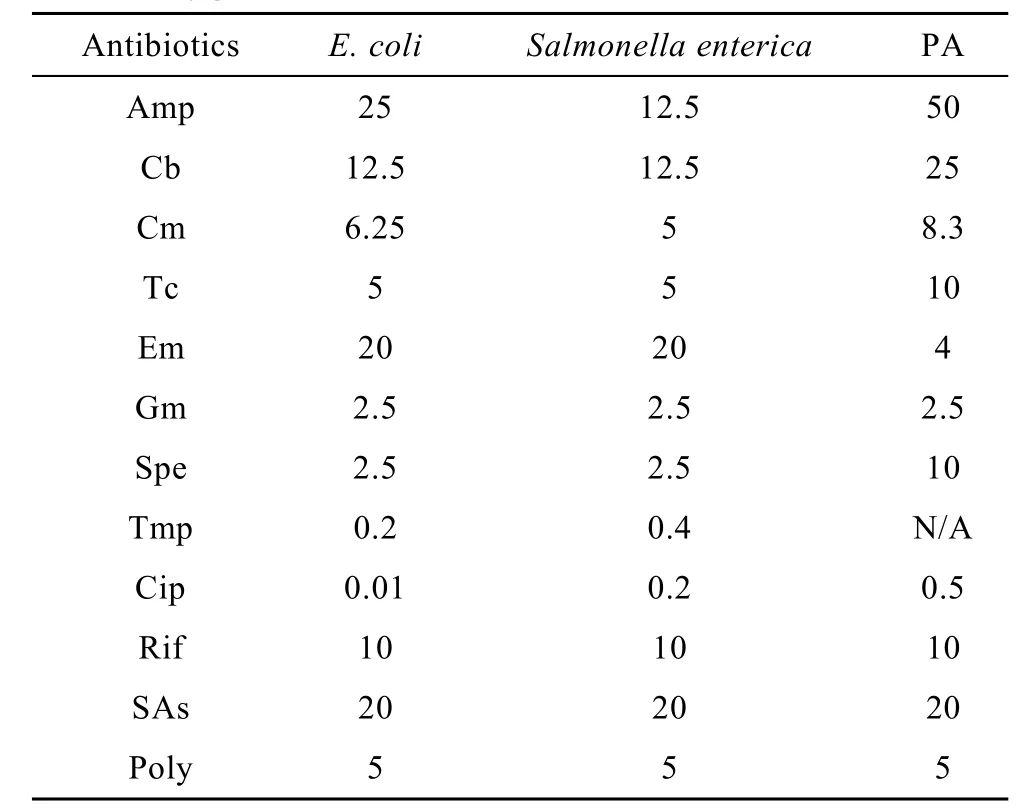
表2 实验中抗生素的使用浓度Table 2 Concentrations of antibiotics used for different bacteria(µg/mL)
2.5 液体培养基中抗生素反应测试
在黑色底部透明 96孔板中加入 80 µL培养基(含相应抗生素)。稀释过夜培养的菌液20倍,即取100 µL菌液到2 mL LB培养基中,然后分别将上述稀释后的菌液加入到96孔板中,每孔加入10 µL。按计算好的抗生素浓度(表 2)将抗生素母液加入每行的第二孔中,然后依次做梯度稀释直到每行的倒数第一个孔,第一个孔加10 µL水为对照,一块96孔板上重复两组进行检测。同时用50 µL矿物油覆盖。在 Wallac 1420 Victor Multilabel Counter上于37℃每隔0.5 h测定 OD600值,共测定24 h。
3 结果
3.1 对抗生素有反应的启动子的筛选
3.1.1 已报道的大肠杆菌胁迫相关基因的重组质粒的建立
细菌普遍存在应对压力的胁迫响应基因(Stress response genes)。一定浓度抗生素的存在对细菌的生长有所影响,是一种生存压力(Stress),胁迫响应基因对其有响应,因而可以作为报道子检测抗生素的存在[13]。实验选取大肠杆菌中6个已报道的胁迫相关基因构建报道重组质粒,用以检验抗生素对胁迫相关基因的影响,并检验以此作为抗生素报道子的可行性。6个与胁迫相关基因的启动子克隆到以荧光素酶基因为报道子的 pCS26质粒上,经PCR扩增后获得重组质粒中启动子序列(图1),并经测序验证。构建好的融合报道质粒分别命名为pBZ-cspA、pBZ-dinD、pBZ-ibpAB、pBZ-recA、pBZ-rpoH、pBZ-uvrA。
3.1.2 对抗生素有反应的铜绿假单胞菌和沙门氏菌随机启动子的筛选
高浓度的抗生素可以杀菌或抑菌,但低于抑制浓度的抗生素可以作为信号分子调节包括致病因子在内的许多基因的表达[14]。这些对抗生素有反应的基因反过来可以用于检测抗生素的存在。从细菌随机启动子库中筛选对抗生素有反应的启动子克隆是构建筛选体系的另一个途径。
本研究以环丙沙星和羧苄青霉素对沙门氏菌和铜绿假单胞菌部分随机启动子库[9-10]进行了筛选。其中铜绿假单胞菌含有质粒pMS402,沙门氏菌含有质粒 pCS26。pMS402质粒和 pCS26质粒带有无启动子的luxCDABE报道基因,插入外源启动子后,通过luxCDABE发光的大小表明启动子的活性。以未添加抑制浓度抗生素条件下的基因表达情况为对照,初步从铜绿假单胞菌随机启动子库的1920个克隆中筛选出在添加抑制浓度抗生素条件下表达情况有3倍或3倍以上变化的克隆,其中对Cb有反应的82个,Cip 218个,随后对这些克隆分别在2种条件下于底部透光的96孔板中进行第2次、第3次筛选。挑选发光值发生3倍或3倍以上变化的克隆,并通过 2次平行实验进行进一步验证,最后确定受 Cb调节的基因有1个,受Cip调节的基因有2个。Cip对其为正向激活作用,即Cip促进其启动子的表达,结果见表2前3个基因。在对沙门氏菌随机启动子库进行筛选最终获得受调节的基因有 19个。12个对Cip敏感,而且Cip对其表现都为正向激活作用。7个基因对Cb敏感,对ycfC、traI 、rrlA为抑制作用,其余都是正调节(表3)。
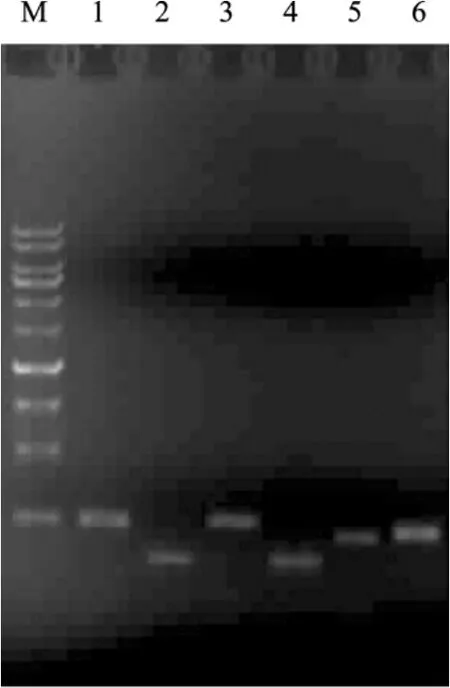
图1 重组质粒的PCR鉴定Fig.1 Identification of recombinant plasmids by PCR.M: DNA marker; 1: cspA; 2: dinD; 3: ibpAB; 4: recA; 5: rpoH; 6: uvrA.

表3 抗生素反应菌株测序比对的相关数据Table 3 List of the genes identified that were regulated by subinhibitory concentrations of antibiotics
3.2 筛选系统的检验
3.2.1 固体平板上抗生素对细菌启动子的影响情况
本试验选择 12种抗菌药物对已筛选出的对抗生素有反应的报道子进行双层培养基法混菌,在含菌的平板上分别点上12种抗生素,在Las-3000下检测启动子的发光强度。
这些对抗生素有反应的启动子使荧光素酶基因表达而发光。发光的强弱显示启动子的活性即通过测定菌体发光的强弱来反映基因的表达水平。图2是混合含有recA基因作为启动子的大肠杆菌和含有glgB作为启动子的沙门氏菌的双层培养以及它们对不同抗生素的反应结果。其中黑色区域代表此处启动子受正向激活作用,荧光素酶发光增强。这些结果表明,同一种抗生素可以引起不同基因表现出不同的反应。而同一基因对不同抗生素的反应也不相同,有的受到激活,有的受到抑制,有的对抗生素没有反应。
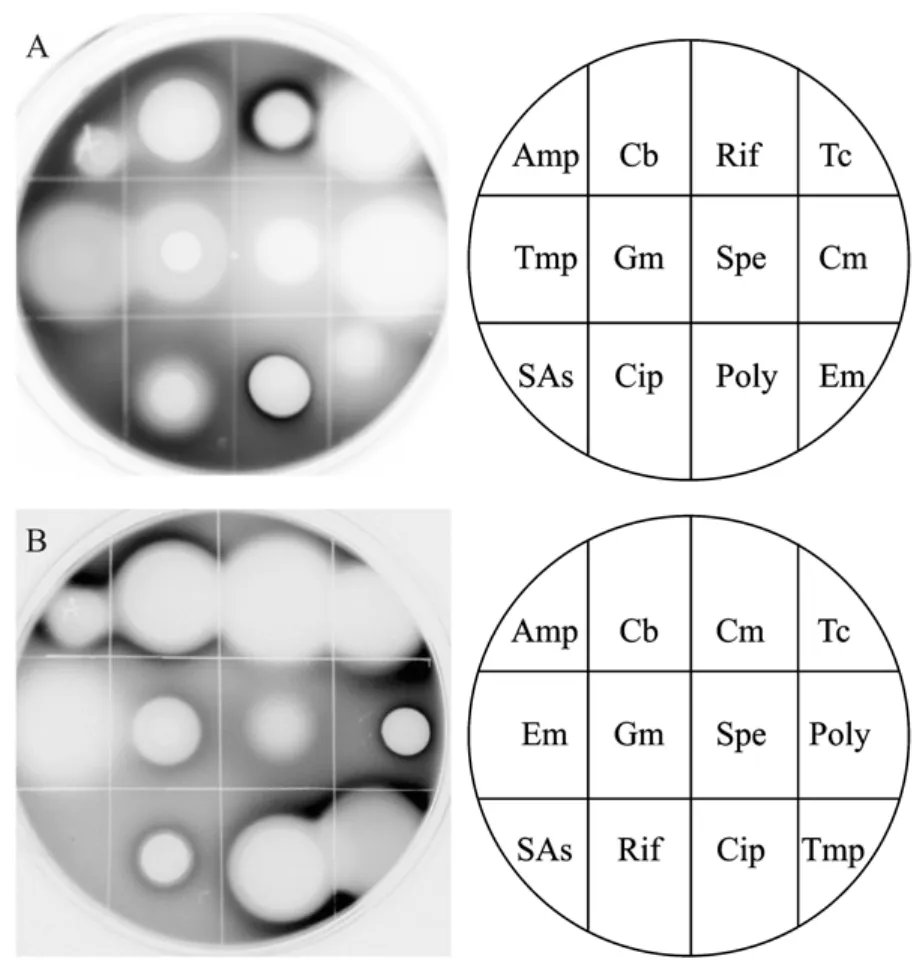
图2 两个代表性启动子-报道子对12种抗生素的反应Fig.2 Representative responses of two promoters to 12 antibiotics.(A)recA.(B)glgB.Black zones on the plates indicate that luminescence is elevated.Imaging obtained using Las-3000.
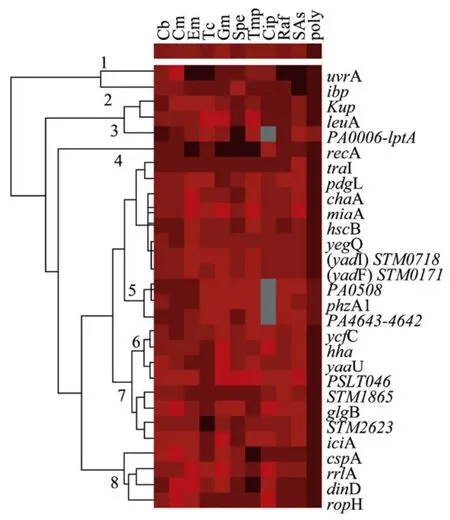
图3 对29种启动子的cluster分析图谱Fig.3 Cluster analysis of antibiotic responsive reporters.
为了比较不同基因的反应,将不同种类抗生素对基因的影响进行cluster分析[15],结果将类似反应的基因归为8个clusters(图3)。从每个cluster中选取代表性报道子,最终从29个基因中筛选出15个基因,分别是uvrA、recA、dinD、glgB、hha、(yadI)STM0718、yaaU、iciA、leuA、yegQ、PSLT046、phzA1、PA4643-4642、PA0508、PA0006-lptA。
3.2.2 液体培养基中抗生素对启动子的影响情况
对这15个基因在液体LB培养基中的表达情况进行验证。发现抗生素对基因的表达影响也存在于液体培养基中。图4是液体培养基中抗生素对2个代表基因的抑制和促进。
3.3 通过报道子检验未知抗生素X
为了进一步检验获得的筛选系统,通过选取的15个报道子对一个抗生素样品进行了检测。采用双层培养基法进行菌液培养,并在平板上点不同抗生素,通过对比抗生素X与其他抗生素对启动子发光的影响,发现X与Tc、Gm、Spe对同种启动子的促进作用的相似性高于其他抗生素。后经验证为硫酸新霉素,属于氨基糖苷类抗生素。说明这种抗生素筛选模型可以进行初步的抗生素类别鉴定。
4 讨论
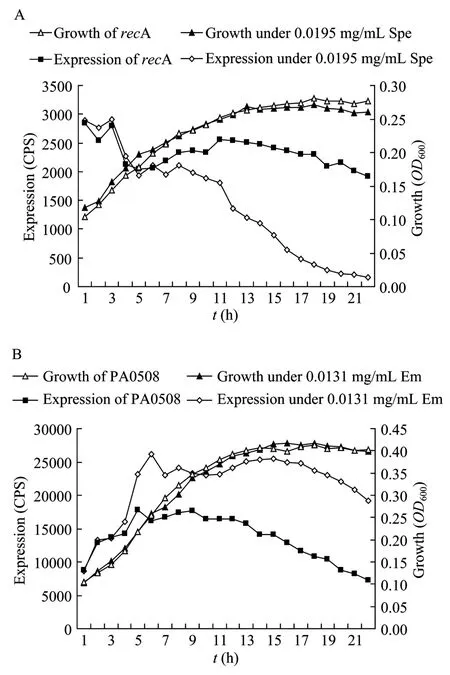
图4 低浓度抗生素对recA和PA0508的表达调节Fig.4 Effect of subinhibitory antibiotic on the expression of recA(A)and PA0508(B).(A)The inhibitory of Spe to recA.(B)The promotery of Em to PA0508.Cps represents the expressin of gene;OD represents the growth of gene.
抗生素是微生物在生长过程中分泌的一类次级代谢物,非微生物基本生命活动所必需。这些化学物质在一定浓度下具有抑制或杀死其他微生物的作用。但是,随着研究的不断深入,抗生素除传统意义上的抑菌和杀菌外,还表现出其他的活性[14]。特别是在低于最小抑制浓度的条件下,抗生素作为一种信号分子对外界基因的表达具有调节作用。低于抑制浓度的抗生素可以调节致病因子的表达[16-18],诱导病原菌的抗药性[19],影响微生物群体中细胞间的相互作用以及微生物与寄主的相互作用[20]。所以高浓度的抗生素对细菌是抑制的,而低浓度时很可能促进细菌的生存[14]。而抗生素对细菌的不同作用也就提供了通过细菌来辨别抗生素的可能性。
本研究采用pMS402和pCS26为载体,通过克隆和随机启动子文库筛选的方法,选取对低于抑制浓度抗生素有反应的报道子构成抗生素筛选体系。这些质粒载体特性是都具有一个多克隆位点,以及缺失启动子的报道基因 luxCDABE,并带有强转录终止子,pMS402具有卡那霉素和甲氧苄氨嘧啶的抗性基因,pCS26具有卡那霉素抗性基因。luxCDABE基因的表达强弱直接体现了启动子活性的大小,因此,通过构建启动子-lux报道子的融合体,便于在固体平板上和液体培养基中检测启动子的活性。可以更直观地检测抗生素对启动子的影响。
实验构建的大肠杆菌重组质粒含有胁迫相关基因:基因recA、uvrA、dinD是已被报道的与大肠杆菌SOS修复相关的基因[21]。SOS修复系统是生物细胞在修复DNA损伤的一套网络系统,在DNA损伤时,LexA调控自身及其下游的其他30多种SOS修复蛋白的表达,其中RecA和UvrA是SOS系统中研究最多的两种修复蛋白,在DNA损伤后表达大大增加[22]。将报告基因置于recA和uvrA启动子调控下,一旦环境样品中存在致畸物,将损伤细胞DNA,从而诱导报告基因的表达,最后通过报告基因表达的强度来判断致畸物的含量。当喹诺酮药物作用于细菌DNA时,引起SOS反应[23],促进recA、uvrA、dinD等一系列SOS相关基因表达,在实验中也可以看到喹诺酮类药物Cip对这3个基因有促进表达的作用。这与文献报道的结果相一致。
筛选体系中大部分报道子是对实验室保存的铜绿假单胞菌和沙门氏菌随机启动子库筛选得到的。测序分析表明这些启动子控制的基因功能多样(表2),并非是由于胁迫相关的基因。这表明低于抑制浓度的抗生素的确对基因组中很多基因具有调节作用,这种情况下的抗生素更可能的是以信号分子起作用[14]。但是,抗生素对这些基因的具体调节还有待进一步研究。
本研究建立的由 15个启动子-报道子融合体构成的抗生素筛选体系对代表各大类不同的抗生素都有不同的反应,可以用来检测样品中是否存在抗生素成分,表明这一体系具有筛选潜在抗生素的功能。可以预计,系统将难以避免获得假阳性结果,这将依赖进一步的检验进行排除。通过比较该筛选体系中启动子对已知抗生素的反应,同时可以初步判断样品抗生素是否与已知的抗生素相同。总之,这一系统将是抗生素物质筛选中有用的新型工具。
REFERENCES
[1]Talbot GH, Bradley J, Edwards JE Jr, et al.Bad bugs need drugs: an update on the development pipeline from the antimicrobial availability task force of the infectious diseases society of america.Clin Infect Dis, 2006, 42(5):657−668.
[2]Norrby SR, Nord CE, Finch R.Lack of development of new antimicrobial drugs: a potential serious threat to public health.Lancet Infect Dis, 2005, 5(2): 115−119.
[3]Projan SJ.Why is big pharma getting out of antibacterial drug discovery? Curr Opin Microbiol, 2003, 6(5):427−430.
[4]Kirsch DR, Lai MH, Gillum AM, et al.The use of beta-galactosidase gene fusions to screen for antibacterial antibiotics.J Antibiot(Tokyo), 1991, 44(2): 210−217.
[5]Silver LL.Does the cell wall of bacteria remain a viable source of targets for novel antibiotics? Biochem Pharmacol, 2006, 71(7): 996−1005.
[6]Payne DJ, Gwynn MN, Rosenberg M, et al.Genomic approaches to antibacterial discovery.Methods Mol Biol,2004, 266: 231−259.
[7]Cappelletti LM, Spagnoli R.Biological transformation of kanamycin A to amikacin(BBK-8).J Antibiot, 1983, 36(3):328−330.
[8]Mills SD.When will the genomics investment pay off for antibacterial discovery? Biochem Pharmacol, 2006, 71(7):1096−1102.
[9]Duan K, Dammel C, Surette MG, et al.Modulation of Pseudomonas aeruginosa gene expression by host microflora through interspecies communication.Mol Microbiol, 2003, 50(5): 1477−1491.
[10]Wang Y, Wei JH, Duan KM, et al.Construction of a Pseudomonas aeruginosa random promoter library and identification of novel iron-regulated genes by high-through- put library screening.Acta Microbiol Sin,2008, 48(7): 863−868.王琰, 魏金花, 段康民, 等.铜绿假单胞菌随机启动子库的构建及受铁调控基因的筛选和鉴定.微生物学报,2008, 48(7): 863−868.
[11]Bjarnason J, Southward CM, Surette MG.Genomic profiling of Iron-responsive genes in Salmonella enterica serovar typhimurium by high-throughput screening of a random promoter library.J Bacteriol, 2003, 185(16): 4973−4982.
[12]Sambrook J, Fritsch EF, Maniatis T.Molecular Cloning: A Laboratory Manual.2nd ed.New York: Cold Spring Harbor Laboratory Press, 1989.
[13]Rocha EP, Matic I, Taddei F.Over-representation of repeats in stress response genes: a strategy to increase versatility under stressful conditions? Nucleic Acids Res,2002, 30(9): 1886−1894.
[14]Grace Y, Helena HW, Julian D.The truth about antibiotics.Int J Med Microbiol, 2006, 296: 163−170.
[15]Eisen MB, Spellman PT, Brown PO, et al.Cluster analysis and display of genome-wide expression patterns.Proc Natl Acad Sci USA, 1998, 95: 14863−14868.
[16]Ohlsen K, Ziebuhr W, Hacker J, et al.Effects of subinhibitory concentrations of antibiotics on alpha-toxin(hla)gene expression of methicillin-sensitive and methicillin-resistant Staphylococcus aureus isolates.Antimicrob Agents Chemother, 1998, 42(11): 2817−2823.
[17]Herbert S, Barry P, Novick RP.Subinhibitory clindamycin differentially inhibits transcription of exoprotein genes in Staphylococcus aureus.Infect Immun, 2001, 69(5):2996−3003.
[18]Sofer D, Gilboa-Garber N, Garber NC, et al.'Subinhibitory' erythromycin represses production of Pseudomonas aeruginosa lectins, autoinducer and virulence factors.Chemotherapy, 1999, 45(5): 335−341.
[19]Walsh C.Where will new antibiotics come from? Nat Rev Microbiol, 2003, 1(1): 65−70.
[20]Piatti G.Bacterial adhesion to respiratory mucosa and its modulation by antibiotics at sub-inhibitory concentrations.Pharmacol Res, 1994, 30(4): 289−299.
[21]Lund PA.Microbial molecular chaperones.Adv Microb Physiol, 2001, 44: 93−140.
[22]Minton KW.DNA repair in the extremely radioresistant bacterium Deinococcus radiodurans.Mol Microbiol, 1994,66: 3856−3867.
[23]Michelle D, Robert EW, Hancock, et al.Ciprofloxacin induction of a susceptibility determinant in Pseudomonas aeruginosa.Antimicrob Agents Chemother, 2005, 49(8):3222−3227.
Construction of a promoter reporter array for antibiotic screening
Bing Zhao, Zhaolin Sun, Liang Yang, Haihua Liang, Lixin Shen, and Kangmin Duan
Key Laboratory of Resources Biology and Biotechnology in Western China, Ministry of Education, Faculty of Life Sciences, Northwest University,Xi’an 710069, China
Received:May 13, 2009;Accepted:November 11, 2009
Supported by:National Natural Science Foundation of China(Nos.30870097, 30611120520).
Corresponding author:Kangmin Duan.Tel: +86-29-88305288; E-mail: kduan@nwu.edu.cn国家自然科学基金(Nos.30870097, 30611120520)资助。