DNA微列阵技术检测戊型肝炎病毒方法的建立及初步应用
李志士,梁海英,于天华
(1.长春中医药大学附属医院,吉林长春 130021;2.吉林大学中日联谊医院输血科)
DNA微列阵技术检测戊型肝炎病毒方法的建立及初步应用
李志士1,梁海英2,于天华2
(1.长春中医药大学附属医院,吉林长春 130021;2.吉林大学中日联谊医院输血科)
目的建立DNA微列阵技术检测戊型肝炎病毒方法,进而探讨该方法用于检测临床标本的可行性。方法通过生物医学数据库戊型肝炎病毒基因进行检索和筛选,应用分子生物学软件,进行序列分析、引物及探针设计,并进行验证,建立了戊型肝炎病毒DNA微列阵技术检测方法。结果试验所设计的探针仅与HEV的PCR产物杂交呈阳性,与乙肝、丙肝等对照病毒的PCR产物杂交呈阴性。敏感性试验显示,该方法比同巢式PCR方法检测戊型肝炎病毒敏感度要高,用该方法检测了长春地区50份疑似HEV临床病料,38份阳性;而用巢式PCR法扩增HEV ORF1基因确诊为阳性的只有35份。结论利用DNA微列阵技术检测戊型肝炎病毒方法,特异性和敏感性强,可作为HEV临床标本检测方法。
戊型肝炎病毒;核酸探针;检测
(Chin J Lab Diagn,2010,14:0996)
目前戊型肝炎病毒的检测方法主要包括两大 类:一是即就用基因工程重组抗原建立的抗体诊断方法,其诊断手段主要是采用间接ELISA方法检测血清中的抗体;另一类是采用RT-PCR为基础建立的基因诊断方法,其诊断手段主要是利用RT-nPCR检测病毒RNA。本文应用DNA微列阵技术检测戊型肝炎病毒[1],现报道如下。
1 材料与方法
1.1 酶及主要试剂
Taq DNA聚合酶购自上海生工;PCR产物纯化试剂盒购自v-gene生物科技有限公司;dNTP购自Promega公司;DNA Marker DL 2000、dNTPs购自大连宝生物工程有限公司;鲑鱼精DNA购自Sigma公司,高纯度病毒核酸提取试剂盒(High Pure Viral Nucleic Acid Kit,Roche Molecular Biochemicals产品)。反转录酶M-MLV、RNA酶均为大连宝生物工程公司及promega等公司产品。
1.2 主要仪器
MicroGrid TAS型芯片点样仪(BioRobotics公司)、384孔板、Personal 4100A型芯片扫描仪(GenePix公司)、GenePix4.1软件(GenePix公司)、PCR 扩增仪(GeneAmp PCR sydt 2400)等。
1.3 引物的合成
查阅相关文献,根据genebank报道的Ag85A序列(X57230),选择戊型肝炎病毒ORF2基因一段高度保守的序列,委托上海生物工程公司合成一对引物,其中引物序列M3:5′-GTC(T)ATGC(T)TGCATACATGGCT-3′(6 010-6 031);M4:5-AGCCGACGAAATC(T)AATTCTGTC-3′(6 336-6 351),引物 5′端进行HEX修饰,用来扩增ORF2基因一段高度保守的序列。
1.4 探针的设计与合成
应用Array Designer2.0设在上述引物序列所扩增片段内设计多条特异性寡核苷酸作为DNA探针,以保证所选探针与其他病毒无同源性。
1.5 HEV基因组的提取及cDNA合成
按照 TRIZOL Reagent使用说明操作。4 μ l用于PCR,剩余-20℃储存备用。
1.6 DNA微列阵的制备
用MicroGrid TAS型点样仪通过机械手臂分别以夹缝针接触式点样方式印迹于玻片上,探针的点样浓度为35 μ M,按图1阵列进行点样,其中芯片列阵中A号位点为阳性坐标探针,其序列为:5′-NH2-ggtcactcgttacgaacgc-cy3-3′,5′端使用 cy3进行荧光修饰,1/2/3/4/5/6为相对应的特异性寡核苷酸探针,0号为空白对照,芯片点样完毕后,将点样后的玻片阵列面朝下,放在盛有37℃水的湿盒中,放置12小时以上固定,进行水合及洗涤处理,完成基因芯片的制备。
1.7 样品处理和杂交
使用上述带有荧光标记的引物对提取好的cDNA进行PCR反应,制备探针。将杂交混合液加于芯片列阵上42℃杂交1 h。以上操作均在暗室中进行。芯片洗涤后以基因芯片扫描仪扫描分析,出现明显可见的杂交信号(斑点)判为阳性。
1.8 基因芯片的验证
依照上述样品处理和杂交方法,对HEV病毒、乙型肝炎病毒及丙型肝炎病毒阳性样品PCR反应,产物检测标记探针同制备的芯片进行杂交分析。杂交程序以上述规范方法进行,扫描分析后以BMP格式图像判定检测结果。
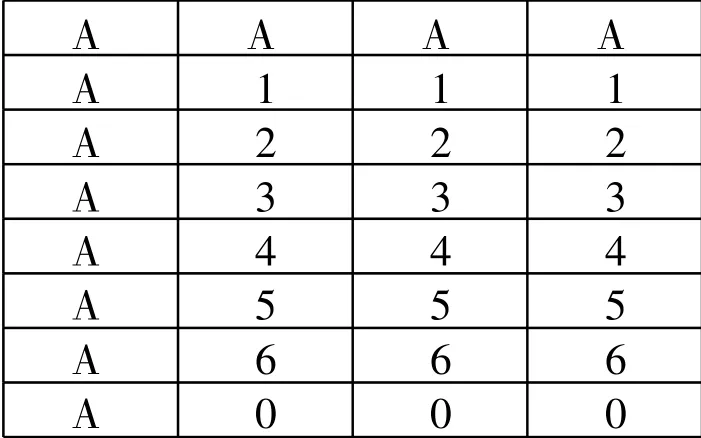
图1 探针在芯片上的点样位置
1.9 临床样品的检测及同巢式PCR的比较 分别使用本试验方法和巢式PCR方法对长春地区50份疑似HEV临床病料进行检测并进行灵敏度比较,其中巢式PCR所使用引物为以M1、M2为外引物:M3、M4为内引物;M1:5′-AAC(T)TATGCA(C)CAGTACCGGGTTG-3′(5 725-5 746);M2:5′-CCCTTATCCTGCTGAGCATTCTC-3′(6 433-6 455);M3:5′-GTC(T)ATGC(T)TGCATACATGGCT-3′(6 010-6 031);M4:5-AGCCGACGAAATC(T)AATTCTGTC-3′(6 336-6 351)。
2 结果
2.1 特异性寡核苷酸探针设计结果
根据实验设计,选择戊型肝炎病毒ORF2基因一段高度保守的序列,在此段序列内根据芯片探针设计原则,设计了6条内设计了特异性寡核苷酸探针,其中探针5′端用NH2修饰并使用T(15)作为NH2与探针之间的连接臂,探针序列见表1。
2.2 基因芯片的制备结果
本实验将经NH2修饰的寡核苷酸探针打印到醛基基片,应用阳性坐标探针对整个芯片制备过程进行监测,芯片点样后,37℃水的湿盒中,放置12小时以上固定,经过一系列洗涤,探针牢固的固定在醛基基片,芯片点样后阳性坐标探针扫描结果如图1所示。试验设计基因芯片为4×8阵列,其中阵列第一排与第一列为阳性坐标探针,第二、三、四、五、六、七排为HEV检测探针,第八排为阴性对照(图2)。
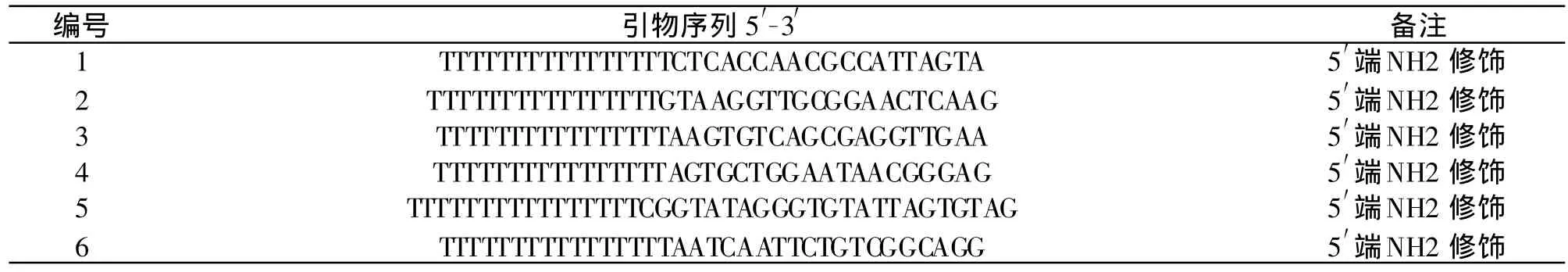
表1 实验中使用的探针
2.3 阳性样品处理和杂交
使用带有荧光标记的引物对已知阳性戊型肝炎病毒 cDNA及乙型肝炎病毒、丙型肝炎病毒进行PCR扩增,之后与芯片进行杂交,结果显示,实验所设计探针同已知阳性戊型肝炎病毒进行PCR产物可特异性杂交,信号呈阳性,而同乙型肝炎病毒、丙型肝炎病毒PCR产物进行杂交没有信号,表明制备的芯片可检测出戊型肝炎病毒ORF2基因片段(见图3)。

图2 基因芯片的制备结果

图3 阳性样品检测结果
2.4 临床样品的检测
检测了长春地区50份疑似HEV临床病料,38份阳性;而用巢式PCR法扩增HEV ORF1基因确诊为阳性的只有35份,其中巢式PCR法所检测的35份阳性病料同DNA微阵列法检测的结果为交集,结果完全符合。
3 讨论
本试验进行了DNA微列阵技术检测戊型肝炎病毒方法的研究,应用生物信息学方法针对戊型肝炎病毒ORF2基因保守片段设计了特异性寡核苷酸探针,并进行点样杂交,成功制备了HEV核酸诊断用DNA微列阵,并进行了制备DNA微列阵的检验研究;建立了DNA微列阵诊断HEV核酸的检测技术。该检测技术可以灵敏特异地对HEV病毒进行检测,具有检测灵敏性好、特异性高和芯片可重复检测的优点。
近年来,随着基因芯片技术的发展,在疫病检测中得到了越来越多的应用[2-4]。常规的基因诊断技术主要包括核酸分子杂交、聚合酶链反应、限制性酶切图谱分析、单链构象多态性分析以及DNA序列测定等技术,这些技术己广泛应用于人畜传染病的检测和诊断,如PCR技术作为当前基因诊断中最主要的技术,常与其他技术性联合,在传染性疾病的分子诊断中得到了广泛的应用,但由于PCR试验常出现假阳性、假阴性结果,严重干扰了疾病的诊断和治疗。因此高特异性自动化仍是分子生物学诊断方法需要解决问题。芯片技术可用大量核酸探针同时检测混合核酸,得出大量相关数据,通过对这些数据的计算机化处理便获得了相关病原体的基因信息,从而对病原体进行分型、亚型,以及致病性强弱、变异进行确切的判断,结果客观、灵敏性高,特异性高。实验证明,这一思路是可行的,由于DNA芯片技术则具备可以对成百上千、甚至上万个基因进行同时检测的优势,在此基础之上可以在芯片平台上不断增加探针,进而可以将甲型肝炎病毒、乙型肝炎病毒及丙型肝炎等病毒的特异性探针集成在一张芯片上,建立高通量的肝炎病毒检测方法。使临床肝炎病毒的检测变得更加方便。
[1]Schena M S D,Davis RW,BrownPO.Quantitative monitoring of gene expression patterns with a complementary DNA microarray[J].Science,1995,270:467.
[2]Richmond CS G J,Mau R,Jin H,Blattner FR.Genome-wide expression profiling inEscherichia coliK-12[J].Nucleic Acids Res,1999,27:3821.
[3]Ang S,Lee C Z,Peck K,et al.Acid-induced gene expression in Helicobacter pylori:shady in genomic scale by microarray[J].Infect Immun,2001,69(3):1679.
[4]Striebel HM B-H E,Egerer R.Virus diagnostics on microarray[J].Current Pharmaceutical Biotechnology,2003,4:401.
DNA microarray detection of hepatitis E virus Establishment and preliminaryapplication of methods
LI Zhi-shi,LIANGHaiying,YU Tian-hua.(The Affiliated Hospital of ChangchunUniversity of Traditional Chinese Medicine,Changchun130021,China)
ObjectiveTo establish a hepatitisE virus nucleic acidprobe detectionmethods,and thenexplore the method used to detect the feasibility of clinical samples.MethodsBiomedical hepatitis E virus genome database retrieval and filtering,the application of molecular biology software for sequence analysis,primer and probe design and carry outverification,establishment of hepatitis E virus nucleic acid probe detection methods.ResultsThe results of tests designed to probe the PCR product only with HEV positive hybridization with hepatitis B,hepatitis C virus,such as the control PCR product of hybridizationwere negative.Sensitivity tests have shown that this method is comparedwith the nested-PCR detection of hepatitis E virus,And Using thismethod to detect 50 copies in Changchun area of clinical disease suspected HEV material,38were positive;while nested-PCR amplification of HEV ORF1 gene diagnosed as positive,only 35 copies.ConclusionThe use of hepatitis E virus nucleic acid probe detection methods,specificity and sensitivity of strong clinical samples can be used asHEV detection methods.
hepatitis E virus;DNA probe;Detection
R512.6
A
1007-4287(2010)07-0996-03
2009-04-07)

