油茶种子总RNA提取及Cod8FAD基因的鉴定
胡 姣,谭晓风,张 琳,龙洪旭
(中南林业科技大学林学院 经济林育种与栽培国家林业局重点实验室,湖南 长沙 410004)
油茶种子总RNA提取及Cod8FAD基因的鉴定
胡 姣,谭晓风*,张 琳,龙洪旭
(中南林业科技大学林学院 经济林育种与栽培国家林业局重点实验室,湖南 长沙 410004)
油茶种子胚乳中富含脂肪、多糖和酚类公合物,要获得高质量的RNA难度较大。本研究以油茶优良无性系湘林1号近成熟种子的胚乳为实验材料,在试剂盒的基础上应用改良的CTAB法提取总RNA。结果表明,使用改良后的CTAB法配合试剂盒提取RNA操作简单,且能够有效去除油茶种子中多糖和多酚类等次生物质,从而得到高质量的RNA。以获得的RNA为模板,设计简并引物,通过RT-PCR,成功从油茶种子中鉴定出了油脂合成的关键酶基因之一△8脂肪酸脱饱和酶基因,命名为Cod8FAD(Camellia oleifera delta 8 fatty acid desaturase),该基因与茶树△8脂肪酸脱饱和酶基因相似性最高,为97%,与耧斗菜△8脂肪酸脱饱和酶基因相似性最低,为62%。
油茶;RNA提取;改良CTAB法;RT-PCR
植物组织中总RNA的提取是植物分子生物学研究的基本技术之一,高质量RNA的获得是进行RT-PCR、Northern blot、cDNA文库构建等分子生物学研究的前提条件[1]。关于从植物组织中提取RNA方法的研究报道很多,但专门针对油茶或油茶不同器官总RNA提取的有效方法报道较少,这在很大程度上制约了相关研究的深入开展。目前,提取RNA的方法主要有异硫氰酸胍法、冷酚法、热酚法、SDS/酚抽提法和TRIZOL法等[1~4]。一般说来,不同的植物材料必须经过摸索合适的取材时间和采取不同的RNA提取方法。
油茶是我国南方重要的木本油料树种,与油棕、油橄榄和椰子并称为世界四大木本食用油料树种。茶油主要由油酸、亚油酸等对人体健康有益的不饱和脂肪酸组成,而控制这些不饱和脂肪酸合成的关键酶基因主要在油茶种子中表达。但由于油茶种子中胚乳含有大量的脂肪、多糖和酚类化合物,找到一种行之有效的提取油茶种子RNA的方法至关重要,是顺利进行后续油茶油脂合成关键酶基因克隆等实验的前提条件。作者经过反复摸索将传统的CTAB法进行改良,应用于油茶种子RNA的提取,并与试剂盒提取法进行比较,发现改良的CTAB法所提取的RNA不但质量高,且简单快速,能满足基因克隆等分子生物学实验的要求。以提取的RNA为模板,成功鉴定出一条油茶油脂合成的关键酶基因——△8脂肪酸脱饱和酶基因。这对其它木本植物总RNA的提取及基因克隆等有一定的指导和借鉴意义。
1 材料与方法
1.1 实验材料
于9月底至10月初,采集油茶优良无性系湘林1号的发育中的种子为实验材料,于-70℃超低温冰箱保存。
1.2 主要实验用品及处理
由于空气中及人体普遍存在RNase,且不易变性,所以提取植物的RNA时要避免污染,防止降解。在提取前做下列准备工作:将研钵、100 mL/250 mL量筒、250 mL/100 mL容量瓶、10 mL/25 mL移液管、药匙、试剂瓶等玻璃制品均用锡纸包裹口部,置于烤箱内185℃,6 h;50 mL/1.5 mL离心管、吸头等塑料制品用1‰DEPC水浸泡过夜后,120℃高压灭菌20 min×2次;电泳槽及电泳托、梳子用3%双氧水处理10 ~ 15 min。
1.3 实验方法
1.3.1 试剂盒提取RNA法 采用的RNA提取试剂盒分别为Ambiogen公司和Invitrogen公司的植物RNA纯化试剂盒,操作方法分别按照各公司的说明书进行。
1.3.2 改良的CTAB法提取RNA
1.3.2.1 基于Ambiogen试剂盒改良的CTAB法 ①将装有研磨好的冻干样品的1.5 mL离心管从-70℃取出,立即加65℃预热好的600 µL的CTAB裂解缓冲液,涡旋混匀,65℃保温10 min,并每隔2 ~ 3 min涡旋振荡1次;②加200 µL的5 M KAC,然后加600 µL的氯仿/异戊醇(24:1),涡旋振荡5 min,冰浴10 min,使其充分裂解;③取450 µL上清液,加入450 µL的RLT溶液,高速离心(13 000 ~ 14 000 rpm)2 min,将上清转移到含收集管的过滤柱内,12 000 rpm离心1 min,将收集管里的溶液(约450 µL)转移到DEPC处理过的1.5 mL的离心管中;④加入约0.5倍体积无水乙醇,轻轻混匀;⑤后续步骤按试剂盒中说明操作。
1.3.2.2 基于Invitrogen试剂盒改良的CTAB法 ①将装有研磨好的冻干样品的1.5 mL离心管从-70℃取出,立即加65℃预热好的600 µL的CTAB裂解缓冲液,涡旋混匀,65℃保温10 min,并每隔2 ~ 3 min涡旋振荡1次;②加200 µL的5 M KAC,然后加600 µL的氯仿/异戊醇(24:1),涡旋振荡5 min,冰浴10 min,使其充分裂解;③4℃ 12 000 rpm离心2 min;④将上清液转入一DEPC水处理的1.5 mL离心管中,重复离心1次以彻底去除组织残片;⑤取450 µL上清液,加450 µL的kit提供的lysis buffer,接着加450 µL的无水乙醇,分别用枪头吸打并涡旋混匀(握于手中上下晃动3 min);⑥后续步骤按试剂盒中说明操作。
1.4 总RNA纯度及完整性检测
电泳分析:取2 µL的RNA样品与1 µL溴酚蓝混合后进行1.0%琼脂糖凝胶电泳,电压为100 V的条件下电泳30 min,在紫外凝胶成像系统上观察RNA条带的清晰度和完整性并拍照。
参照奥斯伯等的方法,取10 µL总RNA溶液,加DEPC水稀释至1 mL,分别在230、260和280 nm测吸光值。并计算A260/A280、A260/A230的比值。
RNA产量(μg)= RNA浓度(μg/mL)×VDEPC-H2O(mL)
1.5 反转录PCR(RT-PCR)
根据GenBank基因库中已公布的高等植物(向日葵、油菜、拟南芥、琉璃苣等)的△8脂肪酸脱饱和酶基因的氨基酸序列,进行序列比对,确定保守区I和保守区II,根据这两个保守区分别设计上游简并引物PF(5’-GATTGGRBBAAAGASCATCC-3’)和下游简并引物PR(GTTATGVKTCCATTTCCACCA)。以获得的油茶种子RNA为模板,使用Fementas公司的cDNA合成试剂盒反转录(RT)合成第一链cDNA,再以此第一链cDNA为模板,使用PF和PR引物组合进行PCR扩增。扩增条件为:94℃预变性4 min;94℃变性30 s;55℃退火30 s;72℃延伸90 s;35个循环;72℃延伸7 min。4℃保存备用。取5 µL扩增产物,经1.2%琼脂糖凝胶电泳,观察记录结果。
1.6 克隆、测序及序列分析
对目标PCR产物进行回收、TA克隆,DNA测序,对获得的DNA序列和推导氨基酸序列进行Blast分析,确定获得的基因类型。
2 结果与分析
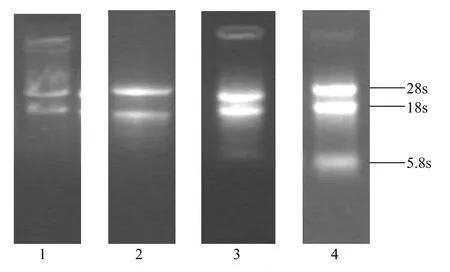
图1 不同方法提取的油茶种子总RNAFigure 1 Total RNA isolated from C. oleifra by different methods
2.1 不同方法提取的油茶种子总RNA质量分析
使用几种不同的方法,都可以从油茶种子中提取出总RNA,但通过电泳分析发现,获得的RNA条带的完整性和强度都有差别(图1)。图1-1为使用Ambiogen试剂盒提取的RNA,28 s和18 s条带模糊,无5.8 s,说明使用该试剂盒提取RNA时,样品组织中的RNA大部分降解。图1-2为使用Invitrogen试剂盒提取的RNA,28 s和18 s条带比较清晰,但亮度较弱,没有5.8 s,说明样品组织中的RNA仍有小部分降解。图1-3为使用改良的CTAB法,Ambiogen试剂盒提取的RNA三条带完整,但28 s与18 s亮度相当,5.8 s亮度较弱,28 s上方有残留DNA,有可能是由于试剂原因致使未清洗干净。图1-4显示:使用改良的CTAB法,使用Invitrogen试剂盒提取的RNA,三条带完整,带型较好,28 s的亮度是18 s的2倍左右,并且5 s亮度较弱,说明该样品完整性良好,未发生明显的降解。
上述4种方法的RNA提取结果表明,使用Invitrogen试剂盒配合改良的CTAB法所提取的RNA样品完整性好,未发生降解,即该方法适合提取高质量的油茶种子总RNA。
2.2 不同方法提取的油茶种子总RNA的纯度和得率

表1 不同方法提取的RNA的纯度及得率比较Table1 Comparison of isolated RNA purity and yield by different methods
从表1可以看出,4种方法提取的RNA的OD260 nm/OD280 nm的值均在1.8 ~ 2.0,说明没有蛋白质或酚类污染,符合要求。而OD260 nm/OD230 nm的值只有使用Invitrogen试剂盒配合改良的CTAB法为2.109,大于2.0,说明样品受离子和小分子的干扰少,多糖的去除较为干净符合要求,其余3种方法的OD260 nm/OD230 nm均小于2.0,表明有小分子和盐存在。4种方法提取的RNA产率均较大。
2.3 油茶Cod8FAD基因的鉴定
以4种不同方法所提取的油茶种子总RNA为模板进行RT-PCR检测,引物是根据不同植物△8脂肪酸脱饱和酶基因序列比对而设计的简并引物PF和PR,根据PF和PR所处基因的位置,推测其跨越的区域约为500 bp。RT-PCR结果见图2。从4种方法提取的RNA中均获得了扩增条带,且带型相同,均为2条带,大小分别为500 bp左右和300 bp左右。但4个样品中,3号和4号的条带亮度要明显强于1号和2号,反映出使用改良CTAB法提取的RNA质量要明显好于常规方法提取的RNA。根据之前的推测,500 bp的条带应该是目的片段。对目的条带进行回收、连接、转化、测序,获得了486 bp的核苷酸序列,并推导编码162个氨基酸(图3),以此氨基酸序列在Genbank基因库中进行Blast分析,结果检索到多条与其高度同源的△8脂肪酸脱饱和酶基因序列(图4)。该基因与茶树△8脂肪酸脱饱和酶基因相似性最高,为97%;与耧斗菜△8脂肪酸脱饱和酶基因相似性最低,为62%。该基因系首次从油茶中鉴定,命名为Cod8FAD(Camellia. oleifera delta 8 fatty acid desaturase)。

图2 不同方法提取的RNA的RT-PCR扩增Figure 2 RT-PCR using RNA template isolated by different methods

图3 油茶Cod8FAD基因的DNA序列和氨基酸序列Figure 3 DNA sequence and amino acid sequence of Cod8FAD

图4 RT-PCR产物的氨基酸序列Blast分析Figure 4 Blast analysis of amino acid sequence of RT-PCR product
3 讨论
不同植物或同种植物不同组织所含化学物质的成分有很大差别,因此其RNA提取的方法也不尽相同[5~6]。油茶种子含有大量的脂肪、多糖和酚类化合物,如果RNA提取过程中蛋白质去除不干净,往往造成其污染,使RNA的质量下降;由于酚类化合物的存在,RNA提取过程中极易产生褐化效应,氧化的酚类化合物(如醌类)可以与RNA不可逆地结合,导致RNA的降解及其活性的丧失,形成不溶性复合物,多糖能与RNA共沉淀或形成难溶的胶状物,还能抑制许多酶的活性[7~11];研究表明,CTAB法能够有效去除油茶胚乳中多糖、酚类物质和基因组DNA的影响,提取出高质量的总RNA。CTAB是一种阳离子强去垢剂,具有疏水性基团和亲水性基团,当CTAB透过细胞壁后,其疏水性基团十六烷基能与细胞膜中的脂质结合,而亲水性阳离子可与膜中的蛋白质结合,裂解细胞膜,打通细胞,使细胞失水,促进核酸物质释放出胞外。提取时,CTAB可与DNA和RNA结合成复合物,此类复合物在高盐下可溶,低盐下沉淀,然后用氯仿/异戊醇去除蛋白质。在提取RNA时,CTAB同时具有结合糖类的作用,此法是目前公认的提取含较多多糖和次生代谢物的植物组织RNA的较好方法[12~13]。
根据结果分析得知,无论使用何种试剂盒,最关键的是要将油茶胚乳中的多糖、酚类去除干净,得到的RNA带型好,亮度高,得率也高。使用提取的RNA,通过RT-PCR,发现RNA质量高的样品获得的PCR产物产量也高。对PCR产物进一步克隆、测序、序列分析,鉴定出了一条油茶种子中表达的△8脂肪酸脱饱和酶基因,该基因在油茶油脂不饱和脂肪酸的合成中起着重要作用,因而为油茶定向分子育种提供了技术基础。
[1] 卢圣栋. 现代分子生物学实验技术(第二版)[M ]. 北京:中国协和医科大学出版社,2001.
[2] Chomczynski P,Sacchi N. Single step method of RNA isolation by acid guanidinium thiocyanate pheno chloroform extraction[J]. Anal Biochem,1987,162(1):156-159.
[3] 王友华,卢孟柱,段留生. 棉花幼苗根总RNA提取的改进热酚法[J]. 西北植物学报,2005,5(4):723-726.
[4] VANDRIESSCHE ES,BECCKMANSS,DEJAEGERE R,et al. The antioxidant of choice for the unification of protein from phenol-rich tissues[J]. Anal. Biochem, 1984(141):184-188.
[5] Ainsworth C. Isolation of RNA from floral tissue of Rumex acetosa (sorrel) [J]. Plant Mol Biol Rep,1994,12(3):198-203.
[6] Jones CS,Iannetta PPM,Woodhead M,et al. The isolation of RNA from raspberry (Rubus idaeus) fruit[J]. Mol Biotech,1997,8(3):219-221.
[7] 王玉成,杨传平,姜静. 木本植物组织总RNA提取的要点与原理[J]. 东北林业大学学报,2002,30(2):1-4.
[8] Schneidrbauer A,Sandermann H,Jr. Ernst Jr. D. Isolation of functional RNA from plant tissues rich in phenolic compounds[J]. Anal Biochem,1991,197(1):91-95.
[9] GRAHAM G C. A method for extraction of total RNA from Pinus radiata and other conifers[J]. Plant Mol Biol Reptr,1993,11(1):32-37.
[10] Lewinsohn E,Steele C L,Croteau R. Simple isolation of functional RNA from woody stems of gymnosperms[J]. Plant Mol Biol Reptr,1994,12(1):20-25.
[11] FANG G,HAMMAR S,GRUMET R. A quick and inexpensive method for removing polysaccharides from plant genomic DNA[J]. Biotechniques,1992,13(1):52-56.
[12] 裴东,谷瑞升. 几种提取木本植物中RNA方法的改进[J]. 植物生理学通讯,2002,38(4):362-365.
[13] 肖洁凝,黄学林,黎茵,等. 富含多糖和次生物质的芒果子叶总RNA的提取[J]. 中国生物工程杂志,2003,23(11):83-86.
信息
2009年浙江省可食林产品质量安全抽查合格率达97.4%
为加强浙江省可食林产品的质量安全监管,保障可食林产品的质量安全,维护消费者的利益和社会的稳定,按照《关于开展2009年全省可食林产品质量安全抽查的通知》(浙林办科〔2009〕33号)规定,浙江省林业厅组织浙江省林产品质量检测站等质检机构对全省可食林产品质量安全进行了抽查。
2009年共抽查食用笋(春笋、鞭笋、冬笋、早笋、笋干)、干果(山核桃、香榧、白果)、山地水果(杨梅、柑橘、猕猴桃)、食用菌(香菇、黑木耳)4大类13种可食林产品1 093批次,样品按生产基地与市场比例为8:2抽取,涉及全省(嘉兴、舟山除外)9个地区。经检测,合格1 065批次,抽查合格率达97.4%。按产品分类统计,各类产品抽查合格率分别为食用笋96.7%、山地水果99.3%、干果94.1%、食用菌100%。抽查结果表明,浙江省上述可食林产品质量安全状况总体较好。
(摘自:http://www.zjly.gov.cn:8080/snyw/11951.htm)
Extraction of Total RNA and Identification of Cod8FAD Gene in Camellia oleifera seeds
HU Jiao,TAN Xiao-feng,ZHANG Lin,LONG Hong-xu(Key Lab of Non-wood Forest Products of State Forestry Administration, College of Forestry, Central South University of Forestry and Technology, Changsha 410004, China)
The isolation of high quality RNA from Camellia oleifera seeds had been very difficult because of rich polysaccharide and phenol compounds in endosperm. Using maturescent seeds of Xianglin No.1, an improved clone as test materials, the total RNA was isolated by modified CTAB combined with RNA purification Kit. Purity and concentration of isolated RNA indicated that polysaccharide and phenol compounds could be effectively eliminated in the endosperm by this method. Using the high-quality RNA as template, a key gene for lipid synthesis was successfully identified by degenerate RT-PCR, that was named Cod8FAD (C. oleifera delta 8 fatty acid desaturase), similar (97%) with delta 8 fatty acid desaturase of C. sinensis.
Camellia oleifera; RNA extraction; modified CTAB method; RT-PCR
S759.3
1001-3776(2010)02-0017-05
2009-12-09;
2010-02-17
国家“十一五”科技支撑项目(2009BADB1B02,2006BAD18B0204)
胡姣(1984-),女,湖南株洲人,硕士研究生,从事林木遗传育种研究;*通讯作者。

