乙肝病毒核酸检测方法研究进展
【摘 要】乙型肝炎病毒感染是一个重大公共卫生问题,为了避免发展成为肝硬化、肝功能衰竭和肝细胞癌,患者通常需要长期服药治疗,检测血清中乙肝病毒核酸的动态变化水平可以作为临床安全停药的重要指标。对于乙肝患者的“精准治疗”,提高乙肝病毒核酸检测的特异度和灵敏度以及对结果的科学解读都十分重要。本文简要对目前已有检测方法的运用场景及其优缺点进行总结,为将来在临床中针对不同情况选择不同方法提供参考。
【关键词】核酸检测;乙肝病毒;研究进展
【中图分类号】R373【文献标志码】A【收稿日期】2024-02-23
基金项目:国家重点研发计划项目资助基金(编号:2022YFA1303600)。
Advances in nucleic acid detection methods for hepatitis B virus
Li Yalan1,Ma Yuanyan2,Wang Deqiang1,Cai Xuefei1
(1.Key Laboratory of Molecular Biology on Infectious Diseases,Ministry of Education,Chongqing Medical University;2.Ministry of Education Key Laboratory of Clinical Laboratory Diagnostics,College of Laboratory Medicine,Chongqing Medical University)
【Abstract】Hepatitis B virus infection is a major public health problem.To avoid the development of liver cirrhosis,liver failure,and hepatocellular carcinoma,patients usually need long-term drug treatment. The dynamic change in serum hepatitis B virus nucleic acid levels can be used as an important indicator of safe drug withdrawal in clinical practice. For the precise treatment of hepatitis B,it is very important to improve the specificity and sensitivity of hepatitis B virus nucleic acid detection methods and the scientific interpreta? tion of the results. This paper summarizes the application scenarios of existing detection methods and their advantages and disadvan? tages,aiming to provide a reference for method selection according to clinical situation in the future.
【Key words】nucleic acid detection;hepatitis B virus;research progress
据世界贸易组织(World Trade Organization,WTO)数据统计显示,全球超2.9亿人感染乙型肝炎病毒(hepatitis B virus,HBV),乙型肝炎感染率高达3.5%[1]。乙肝疫苗高效使用已有40多年的历史,急性和慢性乙型肝炎病毒感染的发病率在全世界都大幅下降,特别是在儿童和年轻人中[2]。HBV传染性极强,慢性肝炎患者体内病毒难以彻底清除,目前仍是我国肝硬化和肝细胞癌的主要病因,我国每年慢性HBV感染而导致的肝癌死亡人数近40万[3]。共价闭合环状DNA (covalently closed circularDNA,cccDNA)是HBV在肝细胞内转录复制的基础。HBV感染往往是慢性感染,病毒长期贮存于肝细胞中,病毒基因组会与宿主基因组进行整合。目前的治疗药物主要包括干扰素和核苷酸类似物均无法消除cccDNA和病毒整合基因来治愈慢性乙肝患者,而针对HBV生命周期不同阶段的多类新型抗病毒药物也在持续开发中[4]。目前的临床治疗目标是实现功能性治愈,定义为血清中检测到持续的乙肝表面抗原(hepatitis B surface antigen,HBsAg)丢失和治疗24周后HBV DNA低于检测下限[5]。HBV核酸检测(包括HBV DNA和HBV RNA的检测)是近年发展起来并逐渐在临床推广应用,本文将对目前常用的几种核酸检测技术进行比较分析,为临床选择恰当乙肝核酸检测方法提供参考。
1 HBV生命周期及其标志物
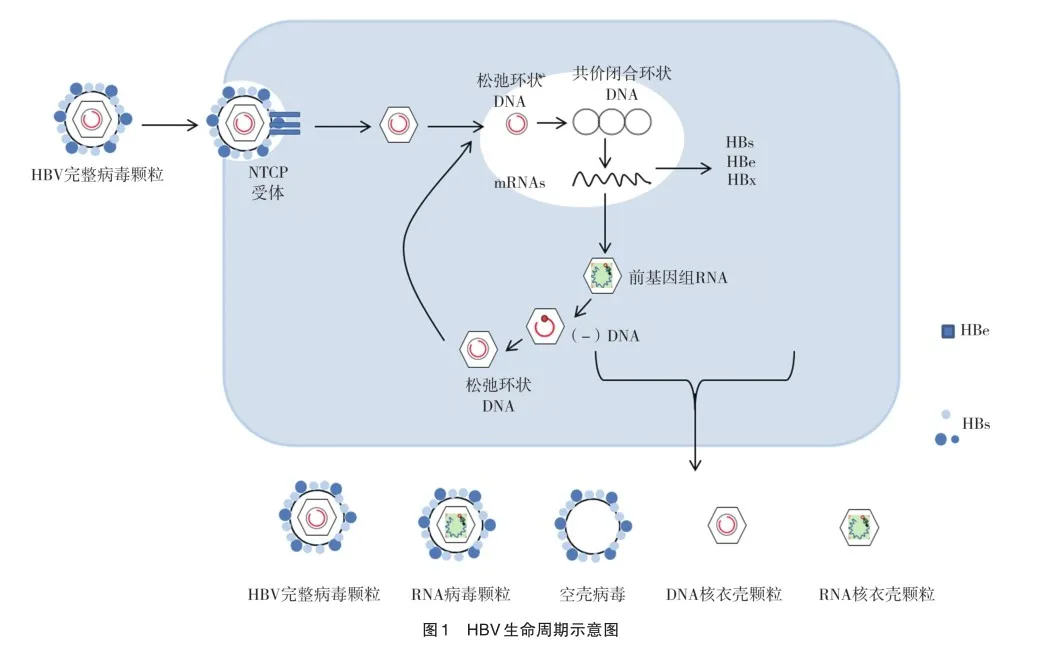
HBV通过病毒粒子包膜上的前S1区(pre-S1)与牛黄胆酸钠协同转运肽(sodium taurinecholate cotransporting poly? peptide,NTCP)受体的附着进入肝细胞,随后包膜脱落,含有基因组DNA的病毒核衣壳被运输到细胞核中,松弛环状DNA(relaxed circularDNA,rcDNA)被释放并转化为cccDNA,接着逆转录形成前基因组RNA(pgRNA)和其他mRNAs;pgRNA参与核心蛋白和逆转录酶的合成,并逆转录成病毒基因组DNA,与其他蛋白质组装形成多种形式的病毒颗粒,最终分泌入血[6-7],见图1。由此衍生出一系列HBV 标志物,间接反映cccDNA水平,比如乙肝两对半指标,核心相关抗原(HBcrAg),HBV DNA以及HBV pgRNA[8]。病毒核酸在血清中有多种存在形式,包括游离的核酸片段、整合在宿主基因组中的核酸片段、Dane颗粒中的核酸以及核衣壳中包裹的核酸。现有研究表明,Dane颗粒(完整病毒颗粒)是唯一具有感染活性的病毒颗粒,是造成患者病毒反弹的主要因素,相较于总核酸水平,其含量更能反映患者的感染状态。cccDNA是乙肝病毒转录复制的模板,是乙肝持续感染的基础,而Dane颗粒的存在,意味着病毒感染还在持续进展中,因此Dane颗粒最有希望替代cccDNA的新一代血清标志物。
2 实时荧光定量聚合酶链式反应(quantitative real-time PCR,qPCR)技术检测乙肝病毒核酸
传统的聚合酶链式反应(polymerase chain reaction,PCR)已经被用作检测包括病毒学在内的一系列科学研究中各种样本的金标准[9-10],该方法利用一对合成的引物,以双链 DNA为模板,添加的4种脱氧核糖核苷酸为原料,在DNA聚合酶的作用下通过温度变化实现扩增。传统PCR对产物的分析只能通过琼脂糖凝胶电泳,对产物进行定性或半定量分析,过程中还需要使用有毒染料对核酸进行染色,已经满足不了人们更高的实验需求[11]。因此,在1996年,Heid CA等[12]发表了一种新的定量检测核酸的方法,即qPCR。在传统PCR反应基础上加入荧光染料,利用特定的仪器可以实时检测扩增过程中荧光信号的变化,达到特定阈值荧光信号水平所需的扩增循环数称为CT值,CT值反映了初始模板浓度水平,CT值越大,初始模板数越小[13]。qPCR可分为探针法和染料法,荧光探针是一段寡核苷酸片段,能与目标序列特异性结合,含有一个荧光基团和淬灭基团,当序列发生扩增时,探针被水解,荧光基团与淬灭基团分开然后发出荧光,优点是灵敏度高,特异强,可以在一管反应中同时检测多种目的基因,但缺点是价格昂贵;目前常用的荧光染料是SYBR Green,能够与DNA双链结合,从而发出荧光,优点是使用方便,价格便宜,缺点是特异性差,无法避免一些引物二聚体和非特异扩增片段的干扰,对引物设计要求高,灵敏度相对更低。
早在1999年,Abe A等[14]就采用基于TaqMan探针的qPCR检测HBV DNA,与之前报道的定量方法相比,该检测方法的实验间和实验内变异性非常小,在定量动态范围内,变异系数<9.07%。该检测系统在慢性乙型肝炎患者中HBV DNA检出率为100%,而在HBV标志物阴性的健康志愿者中从未检测到HBV DNA。这些观察结果表明,qPCR是一种很好的标准HBV定量方法。Loeb KR等[15]也使用基于TaqMan探针的qPCR定量检测血清中的HBV DNA,文章中描述该方法重复性高,自动化程度高,可准确检测到HBV DNA浓度范围在101 copies/mL~109 copies/mL的样本,可用于准确定义HBV感染者的病毒复制水平。最新研究显示,qPCR对HBV DNA的定量检测下限可低至5 IU/mL,有助于发现低病毒载量的慢性HBV患者,对隐匿性乙型肝炎病毒感染(occult hepatitis B virus infection,OBI)人群的再激活也有监测意义[16]。在Yu GX等[17]的研究中,用RT-qPCR方法检测血清中不同形式的HBV RNA,设计了3对针对不同区域的引物以及相应的探针,分别检测全长RNA、剪接体RNA和3’端截短体RNA,检测下限为1.18×102 copies/mL,分析了不同时间点核苷酸类似物治疗患者剪接变异体和pgRNA 3’端截短体的动态变化,以及它们对未治疗和经过治疗患者血清HBV RNA定量检测的影响。
总之,qPCR是1 种灵敏度高、重复性好的检测方法,而且成本低,操作简单,已经成为检测病毒核酸的常用手段。同时也存在一些缺陷,qPCR样本容易被污染,导致扩增受到影响,尤其是在低拷贝数样品中影响明显,数据可靠性得不到保证。所以选择恰当的检测手段是保证实验数据可靠性的前提。
3 数字PCR技术检测乙肝病毒核酸
自20世纪90年代首次提出液滴数字PCR(digital drop? let PCR,ddPCR)以来[18],ddPCR越来越多地用于核酸定量[19-20],是近年来的第3代PCR技术。ddPCR技术的原理是将PCR反应体系稀释并分散至大量液滴中,使液滴成为最多只包含1个核酸分子的独立反应单元,从而产生基于泊松统计的扩增子,该扩增子完全来自1个模板,该方法可以对核酸分子进行绝对定量,具有极高的灵敏度和特异性[21]。这是1种新兴技术,与传统的qPCR相比有2个明显的区别:①在扩增之前将PCR反应划分为数千个单独反应管;②在反应终点获取数据。这些因素使得ddPCR技术优势变得明显,可以直接独立进行DNA定量,而不需要标准曲线,与qPCR相比,具有了更精确和可重复的数据,尤其是在存在可能抑制Taq聚合酶或引物扩增效率的样品污染物的情况下[22-24]。在基因表达、miRNA分析或DNA绝对定量相差2倍及以上等应用中,qPCR是具有良好统计学意义的优秀工具。但是对于低水平的样品,即使存在少量的污染物,对qPCR的结果也是巨大的,在这种情况下,ddPCR表现出极好的特异性[25]。
Caviglia GP等[26]提出了一种基于ddPCR的HBV cccDNA检测方法,定量限和检测限分别为2.4和0.8 copies/105个细胞。作者利用ddPCR确定了从抗HBc阳性肝脏捐献者中招募的OBI患者中 HBV cccDNA的流行率和数量,并评估了肝脏中病毒检测结果与血清中HBV感染标志物之间的关系。在Limothai U等[27]的研究中,比较了RT-ddPCR和RT-qPCR在HBV患者中不同HBeAg状态下检测血清HBV RNA的表现,结果显示RT-ddPCR比RT-qPCR具有更好的灵敏度和重复性,特别是在HBeAg阴性慢性乙肝患者中,RT-ddPCR对HBV RNA的检测下限低至102 copies/mL的,相比之下,RT-qPCR的检测下限约为103 copies/mL。
目前,由于ddPCR检测平台的复杂性和成本较高,该技术所需芯片结构复杂,价格昂贵,使其在推广上受到一定限制,导致在常规实验室和临床中的运用还不广泛。所以在实际运用中,应该在ddPCR和qPCR技术之间进行合理选择,在能保证数据质量的情况下,尽量减少时间成本和经济成本。
4 等温扩增技术检测乙肝病毒核酸
目前,绝大多数基因分型技术都需要先进行扩增,常用PCR或者qPCR技术,然而,这些技术需要使用特定的热循环仪器以及稳定的电源,并不是所有实验室都能开展这项技术。为了满足不同实验环境的需求,开发了等温扩增技术,无须复杂的仪器以及严苛的温度变化,在恒定的工作温度下即可实现核酸的扩增,包括基于核酸序列的扩增(nuclear acid sequence-based amplification,NASBA),链位移扩增(str and displacement amplification,SDA),滚圈扩增(rolling circle amplification,RCA),环介导的等温扩增(loop-mediated iso? thermal amplification,LAMP),解旋酶依赖性扩增(helicasedependent amplification,HDA)以及重组酶聚合酶扩增(re? combinase polymerase amplification,RPA)[28-30]。
NASBA是一种模拟逆转录病毒RNA复制的等温扩增反应,是RT-qPCR扩增RNA的替代方法,需要1对引物和3种酶(逆转录酶、RNase H和T7 DNA依赖的RNA聚合酶),最终实现靶标互补RNA链的指数扩增,产物可以通过基于杂交的方法直接检测,如横向流动试验、电化学发光等[31-32]。SDA利用限制性内切酶和链置换DNA聚合酶的活性来实现目标序列的扩增[33]。RCA是一种利用DNA置换聚合酶链在恒定低温下连续扩增环状DNA模板,产生具有串联重复序列的长DNA分子的技术[34]。LAMP使用2 组引物和1个具有链置换活性的DNA聚合酶,在60 ℃左右的温度下,在不到1 h的时间内实现靶序列的扩增,扩增结果可直接通过肉眼观察荧光[35]。RPA是一种利用重组酶、DNA聚合酶和DNA结合蛋白在低温等温条件下扩增特定DNA的方法。在37 ℃左右,重组酶-引物复合体促进引物与dsDNA靶序列的结合和非模板链的移位,移位的链通过与单链DNA结合蛋白相互作用保持稳定,引物通过链置换DNA聚合酶延伸,这2种dsDNA产物都可以复制,实现靶序列的指数扩增,然后通过观察加入的探针或者染料的荧光信号判断扩增结果[36]。RPA由于其操作简单,灵敏度高,灵活性强(有多种商业化试剂盒可供选择)以及速度快等优点受到人们的关注。RPA可用于扩增DNA,也可以通过将逆转录酶掺入RPA试剂组分来扩增RNA靶标。在一些研究中,RPA甚至可以检测到PCR无法检测到的低浓度DNA。在特异性方面,RPA可以从不同物种和标本类型的基因组DNA中识别和扩增靶基因。RPA抗干扰能力强,检测结果与PCR高度一致,已成功应用于检测多种人类病原体,包括细菌、病毒、寄生虫、真菌以及转基因生物和癌细胞中的基因突变[37-40]。
等温核酸扩增技术具有灵敏度高、特异性强、扩增效率高和操作方便等优点,并且缩短了检测周期,弥补了常规PCR对场地和仪器的依赖,有利于发展现场检测技术[41-42]。但是,等温核酸扩增技术仍存在较多技术难点,比如部分等温扩增引物设计比较复杂,没有成熟的软件和方法,部分方法在灵敏度方面依旧比不上qPCR。经过不断的优化创新,等温核酸扩增技术将有望代替qPCR成为现场快速检测的最佳选择。
5 DNA/RNA印迹检测乙肝病毒核酸
1975年,一种新的检测DNA的方法,被命名为Southern blot[43]。Southern blot是一种经典但复杂的DNA检测方法,涉及探针制备、电泳、跨膜杂交和检测等几个过程[44]。Southern印迹法被广泛应用于识别与遗传病相关的基因,大大提高了对某些疾病的诊断能力,例如亨廷顿舞蹈病、家族性生长激素缺乏症、镰状细胞贫血症等[45]。随着PCR扩增技术的发展和自动DNA片段分析仪的引入,Southern blot在实验室中的使用逐渐减少,但对于某些特定的检测项目,该方法仍是不可替代的一项技术。比如对于HBV cccDNA的检测,South? ern blot被认为是检测该项指标的“金标准”,因为它通过琼脂糖凝胶基于电泳运动性准确地分离出不同形式的HBV DNA[46-47]。cccDNA的持续存在是目前乙肝病毒感染无法完全治愈的主要原因,Southern blot只能对cccDNA进行定性或者半定量检测,而且操作复杂,灵敏度低,检测下限为105~ 106 copies,不适合高通量药物筛选,使得靶向cccDNA的药物开发受到阻碍。人们也致力于开发更灵敏、更简单的检测cccDNA的方法,但目前还未发明出大众认可的、可靠的检测方法,所以在相当长的时间内,Southern blot仍是检测cccDNA的主要方法。
Northern blot是检测RNA的传统方法,主要用于判断基因表达水平,其基本原理是利用凝胶电泳将不同大小的RNA分离,并转移到膜上使用DNA探针与互补RNA序列杂交进行检测[48-49],是1项使RNA可视化的定量检测技术。最流行的探针标记方案是结合放射性同位素(32P),然而,同位素标记通常是具有危险性的,所以其使用受到限制。Ram? kissoon SH等[50]报道了使用地高辛(digoxin,DIG)标记的探针在检测RNA时与同位素标记的探针具有相同的敏感性,且具有更长的保质期和更高的安全性。与其他技术相比,Northern blot是1种低通量、低灵敏度、相对耗时和样品要求量大的方法[51-53]。尽管如此,它仍然被广泛用作验证更新、更灵敏的检测技术数据的金标准[54]。
6 实时荧光核酸恒温扩增检测技术乙肝病毒核酸
实时荧光核酸恒温扩增检测技术(simultaneous amplifi? cation and testing,SAT)是一种RNA恒温扩增技术,原理是将目的RNA在逆转录酶的作用下合成1条含T7启动子的双链DNA,T7 RNA聚合酶以这条双链DNA为模板进行转录合成RNA,合成的RNA与分子信号结合发出荧光,利用荧光检测仪检测。以上逆转录和转录的过程能够在逆转录酶和T7 RNA聚合酶的作用下持续进行,以达到高效扩增的目的[55]。该方法的优点是在恒温条件下直接扩增RNA,减少了常规的逆转录环节,最大可能地避免了污染;操作方便,无须复杂实验步骤。在检测呼吸道病毒,肠道病毒等方面运用比较广泛。
国内有公司利用该实验技术研发成功HBV RNA检测试剂盒并获得注册,现在正在各大医院进行推广。Hu XH等[56]在研究中验证了SAT方法检测HBV RNA的性能并与RTqPCR进行了比较,结果发现2种方法检测的HBV RNA水平无差异,结果的相关性和一致性较好,SAT法对HBV RNA的检出限为50 copies/mL,线性范围为1×102~1×108 copies/mL。
总之,SAT对仪器和操作要求简单,在快速检测中具有高灵敏度、高特异性、低污染等优点。由于缺乏标准品,HBV RNA的临床结果存在多样性,不同检测方法对研究结果差异的影响仍有待探讨。
7 结 语
随着医疗的进步,大部分乙肝患者的病情得到有效控制,但乙肝病毒无法完全清除,意味着大部分患者需要长期甚至终身服药,造成一定的经济负担。本研究下一步的目标是高效准确监测病情,预测复发风险,让患者能够安全停药。目前核酸提取与检测技术逐渐成熟,临床上对于HBV DNA已有较成熟并且受大众认可的检测方法,目前主要致力于提高其检测灵敏度;HBV pgRNA作为新型HBV标志物,受到越来越多人的关注,虽已有多种检测试剂盒,但灵敏度差异度大且缺乏标准品,还未得到普遍认可。目前的检测技术存在较大的局限性,与抗原检测一样,无法区别其检测靶标是来源于病毒整合基因、核心颗粒或者完整病毒颗粒,无法反应替代cccDNA反应HBV病毒是否还具有感染活性。目前有多个团队正在研发新一代的血清标志物,希望能够提升与cccDNA的水平的相关性,进而直接/间接反应乙肝病毒的感染活性,为临床治疗提供更加精准的评价指标。
参 考 文 献
[1] Jeng WJ,Papatheodoridis GV,Lok ASF. Hepatitis B[J]. Lancet,2023,401(10381):1039-1052.
[2] Thomas DL. Global elimination of chronic hepatitis[J]. N Engl J Med,2019,380(21):2041-2050.
[3] Liu J,Liang WN,Jing WZ,et al. Countdown to 2030:eliminating hepatitisB disease,China[J]. Bull World Health Organ,2019,97(3):230-238.
[4] Wong GLH,Gane E,Lok ASF. How to achieve functional cure of HBV:stopping NUCs,adding interferon or new drug development?[J]. J Hepatol,2022,76(6):1249-1262.
[5] Ghany MG,Buti M,Lampertico P,et al. Guidance on treatment endpoints and study design for clinical trials aiming to achieve cure in chronic hepatitis B and D:report from the 2022 AASLD-EASL HBVHDV Treatment Endpoints Conference[J]. J Hepatol,2023,79(5):1254-1269.
[6] Nassal M. HBV cccDNA:viral persistence reservoir and key ob? stacle for a cure of chronic hepatitis B[J]. Gut,2015,64(12):1972-1984.
[7] Karayiannis P. Hepatitis B virus:virology,molecular biology,life cycle and intrahepatic spread[J]. Hepatol Int,2017,11(6):500-508.
[8] Inoue T,Tanaka Y. The role of hepatitis B core-related antigen[J]. Genes,2019,10(5):357.
[9] Freymuth F,Eugene G,Vabret A,et al. Detection of respiratory syncytial virus by reverse transcription-PCR and hybridization with a DNA enzyme immunoassay[J]. J Clin Microbiol,1995,33(12):3352-3355.
[10] Mullis KB,Faloona FA. Specific synthesis of DNA in vitro via a polymerase-catalyzed chain reaction[J]. Methods Enzymol,1987,155:335-350.
[11] Kidd IM,Clark DA,Emery VC. A non-radioisotopic quantitative competitive polymerase chain reaction method:application in measure? ment of human herpesvirus 7 load[J]. J Virol Methods,2000,87(1/2):177-181.
[12] Heid CA,Stevens J,Livak KJ,et al. Real time quantitative PCR[J]. Genome Res,1996,6(10):986-994.
[13] Kubista M,Andrade JM,Bengtsson M,et al. The real-time poly? merase chain reaction[J]. Mol Aspects Med,2006,27(2/3):95-125.
[14] Abe A,Inoue K,Tanaka T,et al. Quantitation of hepatitis B virus genomic DNA by real-time detection PCR[J]. J Clin Microbiol,1999,37(9):2899-2903.
[15] Loeb KR,Jerome KR,Goddard J,et al. High-throughput quanti? tative analysis of hepatitis B virus DNA in serum using the TaqMan fluo? rogenic detection system[J]. Hepatology,2000,32(3):626-629.
[16] Kusumoto S,Tanaka Y,Suzuki R,et al. Monitoring of hepatitis B virus (HBV) DNA and risk of HBV reactivation in B-cell lymphoma:a prospective observational study[J]. Clin Infect Dis,2015,61(5): 719-729.
[17] Yu GX,Chen R,Zheng SJ,et al. A standardized assay for the quantitative detection of serum HBV RNA in chronic hepatitis B pa? tients[J]. Emerg Microbes Infect,2022,11(1):775-785.
[18] Sykes PJ,Neoh SH,Brisco MJ,et al. Quantitation of targets for PCR by use of limiting dilution[J]. Biotechniques,1992,13(3):444-449.
[19] Lo YM,Lun FM,Chan KC,et al. Digital PCR for the molecular detection of fetal chromosomal aneuploidy[J]. Proc Natl Acad Sci USA,2007,104(32):13116-13121.
[20] Sanders R,Huggett JF,Bushell CA,et al. Evaluation of digital PCR for absolute DNA quantification[J]. Anal Chem,2011,83(17):6474-6484.
[21] Pomari E,Piubelli C,Perandin F,et al. Digital PCR:a new tech? nology for diagnosis of parasitic infections[J]. Clin Microbiol Infect,2019,25(12):1510-1516.
[22] Ra?ki N,Dreo T,Gutierrez-Aguirre I,et al. Reverse transcriptase droplet digital PCR shows high resilience to PCR inhibitors from plant,soil and water samples[J]. Plant Methods,2014,10(1):42.
[23] Taylor SC,Carbonneau J,Shelton DN,et al. Optimization of Drop? let Digital PCR from RNA and DNA extracts with direct comparison to RT-qPCR:clinical implications for quantification of Oseltamivirresistant subpopulations[J]. J Virol Methods,2015,224:58-66.
[24] Taylor SC,Nadeau K,Abbasi M,et al. The ultimate qPCR experi? ment:producing publication quality,reproducible data the first time[J]. Trends Biotechnol,2019,37(7):761-774.
[25] Taylor SC,Laperriere G,Germain H. Droplet Digital PCR versus qPCR for gene expression analysis with low abundant targets:from vari? able nonsense to publication quality data[J]. Sci Rep,2017,7(1):2409.
[26] Caviglia GP,Abate ML,Tandoi F,et al. Quantitation of HBV cccDNA in anti-HBc-positive liver donors by droplet digital PCR:a new tool to detect occult infection[J]. J Hepatol,2018,69(2):301-307.
[27] Limothai U,Chuaypen N,Poovorawan K,et al. Reverse transcrip? tase droplet digital PCR vs reverse transcriptase quantitative real-time PCR for serum HBV RNA quantification[J]. J Med Virol,2020,92(12):3365-3372.
[28] Deng HM,Gao ZQ. Bioanalytical applications of isothermal nucleic acid amplification techniques[J]. Anal Chim Acta,2015,853:30-45.
[29] Yan L,Zhou J,Zheng Y,et al. Isothermal amplified detection of DNA and RNA[J]. Mol Biosyst,2014,10(5):970-1003.
[30] Craw P,Balachandran W. Isothermal nucleic acid amplification technologies for point-of-care diagnostics:a critical review[J]. Lab Chip,2012,12(14):2469-2486.
[31] Connelly JT,Nugen SR,Borejsza-Wysocki W,et al. Human pathogenic Cryptosporidium species bioanalytical detection method with single oocyst detection capability[J]. Anal Bioanal Chem,2008,391(2):487-495.
[32] Asiello PJ,Baeumner AJ. Miniaturized isothermal nucleic acid amplification,a review[J]. Lab Chip,2011,11(8):1420-1430.
[33] Walker GT,Fraiser MS,Schram JL,et al. Strand displacement amplification:an isothermal,in vitro DNA amplification technique[J]. Nucleic Acids Res,1992,20(7):1691-1696.
[34] Liu DY,Daubendiek SL,Zillman MA,et al. Rolling circle DNA synthesis:small circular oligonucleotides as efficient templates for DNA polymerases[J]. J Am Chem Soc,1996,118(7):1587-1594.
[35] Notomi T,Okayama H,Masubuchi H,et al. Loop-mediated iso? thermal amplification of DNA[J]. Nucleic Acids Res,2000,28(12):E63.
[36] Piepenburg O,Williams CH,Stemple DL,et al. DNA detection using recombination proteins[J]. PLoS Biol,2006,4(7):e204.
[37] Crannell ZA,Castellanos-Gonzalez A,Irani A,et al. Nucleic acid test to diagnose cryptosporidiosis:lab assessment in animal and patient specimens[J]. Anal Chem,2014,86(5):2565-2571.
[38] Hill-Cawthorne GA,Hudson LO,El Ghany MF,et al. Recombina? tions in staphylococcal cassette chromosome mec elements compromise the molecular detection of methicillin resistance in Staphylococcus au? reus[J]. PLoS One,2014,9(6):e101419.
[39] Sakai K,Trabasso P,Moretti ML,et al. Identification of fungal pathogens by visible microarray system in combination with isothermal gene amplification[J]. Mycopathologia,2014,178(1/2):11-26.
[40] Zhang SL,Ravelonandro M,Russell P,et al. Rapid diagnostic de? tection of plum pox virus in Prunus plants by isothermal AmplifyRP(?)using reverse transcription-recombinase polymerase amplification[J]. J Virol Methods,2014,207:114-120.
[41] Daher RK,Stewart G,Boissinot M,et al. Recombinase poly? merase amplification for diagnostic applications[J]. Clin Chem,2016,62(7):947-958.
[42] Lobato IM,O’Sullivan CK. Recombinase polymerase amplifica? tion:basics,applications and recent advances[J]. Trends Analyt Chem,2018,98:19-35.
[43] Kelly KF. Southern blotting[J]. Proc Nutr Soc,1996,55(1B):591-597.
[44] Cai DW,Nie H,Yan R,et al. A southern blot assay for detection of hepatitis B virus covalently closed circular DNA from cell cultures[J]. Methods Mol Biol,2013,1030:151-161.
[45] Hayes PC,Wolf CR,Hayes JD. Blotting techniques for the study of DNA,RNA,and proteins[J]. BMJ,1989,299(6705):965-968.
[46] Li XL,Zhao JH,Yuan Q,et al. Detection of HBV covalently closed circular DNA[J]. Viruses,2017,9(6):139.
[47] Zhang H,Tu T. Approaches to quantifying hepatitis B virus cova? lently closed circular DNA[J]. Clin Mol Hepatol,2022,28(2):135-149.[48] Trayhurn P. Northern blotting[J]. Proc Nutr Soc,1996,55(1B):583-589.
[49] Hunt EA,Broyles D,Head T,et al. MicroRNA detection:current technology and research strategies[J]. Annu Rev Anal Chem,2015,8:217-237.
[50] Ramkissoon SH,Mainwaring LA,Sloand EM,et al. Nonisotopic detection of microRNA using digoxigenin labeled RNA probes[J]. Mol Cell Probes,2006,20(1):1-4.
[51] Kim SW,Li ZH,Moore PS,et al. A sensitive non-radioactive northern blot method to detect small RNAs[J]. Nucleic Acids Res,2010,38(7):e98.
[52] Pall GS,Codony-Servat C,Byrne J,et al. Carbodiimide-mediated cross-linking of RNA to nylon membranes improves the detection of siRNA,miRNA and PiRNA by northern blot[J]. Nucleic Acids Res,2007,35(8):e60.
[53] Válóczi A,Hornyik C,Varga N,et al. Sensitive and specific de? tection of microRNAs by northern blot analysis using LNA-modified oli? gonucleotide probes[J]. Nucleic Acids Res,2004,32(22):e175.
[54] Dong HF,Lei JP,Ding L,et al. MicroRNA:function,detection,and bioanalysis[J]. Chem Rev,2013,113(8):6207-6233.
[55] Jia Y, Tian C, Wang H, et al. Long non-coding RNA NORAD/ miR-224-3p/MTDH axis contributes to CDDP resistance of esophageal squamous cell carcinoma by promoting nuclear accumulation of β-catenin[J]. Mol Cancer,2021,20(1):162-182.
[56] Hu XH,Zhao LW,Ou MR,et al. Evaluation of reverse transcription-polymerase chain reaction and simultaneous amplification and testing for quantitative detection of serum hepatitis B virus RNA[J]. Heliyon,2023,9(8):e18557.
(责任编辑:周一青)
本文引用格式:
李亚兰,马圆艳,汪德强,等. 乙肝病毒核酸检测方法研究进展[J]. 重庆医科大学学报,2025,50(2):152-157.

