基于SSR标记的圆叶桉遗传多样性分析
摘 要:【目的】摸清国内圆叶桉Eucalyptus pulverulenta花卉资源遗传多样性概况,为种质资源利用及分子育种提供参考。【方法】利用多态性高的15个SSR标记,对7个来源的59株圆叶桉进行SSR基因分型和遗传多样性分析。【结果】1)59株圆叶桉在15个SSR标记共检测到83个等位片段数(NA),平均每个标记的等位片段数为5.5个,每个标记的等位片段数2~10个,平均有效等位片段数(NE)2.4个。观测杂合度(HO)和期望杂合度(HE)的平均值为0.401和0.491,多态性信息量(PIC)的平均值为0.455,具有较高的多态性。圆叶桉群体间遗传分化系数(Fst)为0.208,即有20.8%的变异存在于不同群体间;2)个体间遗传距离范围为0.033~0.698,平均为0.377,群体间亲缘关系较近,遗传背景相对单一。圆叶桉群体分为两个亚组,与其天然分布范围一致;3)圆叶桉群体间的遗传一致度为0.533~0.987,来自云南佳艺园艺和云南绿梦花卉的圆叶桉遗传一致度最高,推测二者可能为来源相同的品种。分子方差分析(AMOVA)结果表明个体间变异是方差分量的主要来源。【结论】15个SSR标记遗传多态性较高,可用于圆叶桉的遗传分化研究。个体间的分型结果表明,国内市场上常见的圆叶桉均为有性繁殖的实生苗,不同来源圆叶桉群体间的遗传分化程度较小,遗传变异主要发生在个体间。本研究对圆叶桉进行遗传多样性和遗传结构分析,对种质资源的评价和利用都具有重要意义。
关键词:圆叶桉;SSR标记;遗传多样性;遗传关系
中图分类号:S792.39 文献标志码:A 文章编号:1673-923X(2024)10-0157-08
基金项目:“十四五”国家重点研发计划项目(2022YFD2200203-2);广州市基础研究计划基础与应用基础研究项目(202102080550);广东省林业科技创新项目(2023KJCX013)。
Genetic analysis of Eucalyptus pulverulenta based on SSR markers
ZHAO Jingyi1,2, QI Xiaohui3, QU Guanzheng1, WU Youjun2, LI Zhuo2, ZHOU Changpin1,2
Abstract:【Objective】In order to find out genetic diversity condition of Eucalyptus pulverulenta resources in China, and to provide reference for germplasm resources utilization and molecular breeding.【Method】15 SSR makers with high polymorphism were used for genotyping and genetic diversity analysis of 59 E. pulverulenta from 7 sources.【Result】1) A total of 83 alleles were detected by 15 SSR markers, averaging 5.5 alleles per marker, the number of alleles per marker ranged from 2 to 10, averaging 2.4 effective alleles per marker. The average value of observed heterozygosity and expected heterozygosity were 0.401 and 0.491, respectively. The general polymorphism was high with an average polymorphic information content of 0.455, indicating these markers were suitability for studying genetic differentiation of E. pulverulenta. The genetic differentiation coefficient between populations (Fst) was 0.208, it indicated that there occurred 20.8% genetic variation among populations; 2) The genetic distance varied from 0.033 to 0.698, with an average of 0.377. The results indicated that the genetic background was relatively single and the genetic relationship was close. The cluster analysis showed that 59 samples were clustered into two subgroups, which was consistent with the natural distribution of E. pulverulenta; 3) The genetic coefficient of the populations ranged from 0.533 to 0.987. The genetic coefficient of E. pulverulenta from Yunnan Jiayi Horticulture and Yunnan Lümeng Huahui was the highest, which suggested that the two populations might be from the same source. Analysis of molecular variance showed that individual variation is the main source of variance component.【Conclusion】This study used 15 SSR markers, which had high genetic polymorphisms and could be used to study the genetic differentiation of E. pulverulenta. The results of cluster analysis among individuals showed that the common E. pulverulenta in the domestic market are generative reproduced seedlings. The extent of genetic differentiation among different populations were at a low level, and most of variations existed within individuals. The results will be great significance for the germplasm resource evaluation and utilization in E. pulverulenta.
Keywords: Eucalyptus pulverulenta; SSR marker; genetic diversity; genetic relationship
圆叶桉Eucalyptus pulverulenta又名银叶山桉,是桃金娘科Myrtaceae桉属Eucalyptus L’Hérit.蓝桉组Section Maidenaria树种,多为灌木或小乔木,幼态叶对生,圆形或心形无柄,被蓝灰色腊粉,幼茎呈蓝灰色,其自然分布于澳大利亚新南威尔士州和塔斯马尼亚州,属地中海气候区[1]。圆叶桉多为种子繁殖,大量的宿芽使其具有强大的萌蘖能力,枝条多用于鲜切花插花和花艺等,具有极高的园艺观赏价值和商业价值[2]。研究人员对引种的26种芳香植物种的观赏性状(株型、叶片)、生态适应性(抗性、花期)以及扩繁价值等进行综合评价,圆叶桉被评为“极优”[3]。圆叶桉在美国、意大利和日本等国家被广泛引种,近年来相继推出‘蓝宝贝’、‘铜钱桉’、‘冰点’和‘蓝梦’等品种[4]。当前在云南昆明、曲靖等地区大面积栽培,在浙江和福建等省份亦有少量种植,表现出一定的适应性,但在我国华南地区难以过夏,种植情况普遍不佳。
简单重复序列(Simple sequence repeat,SSR)是一组以1~6个核苷酸为重复单位串联成的重复序列,具有共显性、多态性高、标记数量多、覆盖范围广等优点[5]。SSR不仅在巨桉E.grandis、尾叶桉E.urophylla、细叶桉E.tereticornis、赤桉E.camaldulensis、柠檬桉E.citriodora、粗皮桉E.pellida和大花序桉E.cloeziana等桉树的连锁图谱构建、种质资源鉴定[6-7]、遗传多样性分析[8-9]、群体结构分析[10-11]、数量性状位点定位[12]和关联分析[13]等研究中广泛应用,而且也多用于花卉等的品种鉴定[14]。目前,对于圆叶桉的研究仍较少,且多集中在引种、苗木繁育、生理生化等方面[15],关于遗传多样性、群体遗传结构分析等分子水平的研究尚未有报道。
圆叶桉观赏价值高,市场潜力巨大。当前,国内种植的圆叶桉花卉遗传基础不明确,具有自主知识产权的新品种缺乏等问题突出,制约了圆叶桉产业的健康发展。本研究利用多态性高的15个SSR标记[16-17],对国内市场销售及不同来源的 59株圆叶桉进行遗传多样性分析,以推动圆叶桉种质资源的评价和利用。
1 材料与方法
1.1 试验材料及DNA提取
参试材料为国内在售和来自澳大利亚林木种子中心种子繁育的圆叶桉花卉,7个来源共59株(表1)。采集叶片后-80 ℃下保存,DNA提取采用改良CTAB法[18],使用NanoDrop 2000(Thermo Scientific,美国)对DNA的浓度和纯度进行检测。

1.2 SSR分型与多态性检测
利用桉树中已开发的15个多态性高的SSR标记[16-17],对59株圆叶桉样品进行PCR扩增和多态性检测。引物由上海生工公司合成,前向引物5′端采用FAM、HEX、TAM或ROX进行荧光修饰。PCR采用10 μL体系,包括:10×Buffer 1 μL、25 μmol/L dNTPs、0.5 μmol/L前向引物和后向引物、0.1 U Taq聚合酶(申能博彩生物科技有限公司)及5 ng DNA模版,ddH2O补齐至10 μL。PCR扩增程序为:94 ℃预变性4 min;35个循环:94 ℃变性30 s、60 ℃退火30 s、72 ℃延伸30 s;最终72 ℃延伸10 min,4 ℃保存。取1 μLPCR产物、9.34 μL超纯甲酰胺(Applied Biosystems,美国)、0.16 μL GeneScan 500-LIZ内标(Applied Biosystems,美国)混匀,95 ℃变性5 min,迅速冰浴冷却,利用ABI 3130XL测序仪(Applied Biosystems,美国)进行SSR标记分型,按照仪器说明进行上机操作。
1.3 数据分析
利用GeneMapper 4.0软件统计样品的荧光检测结果;使用PowerMarker 3.25软件[19]计算等位片段数(NA)、有效等位片段数(NE)、多态性信息含量(PIC)和Nei’s遗传距离;使用GenAlEx 6.5.1软件[20]计算等位片段范围(ASR)、观测杂合度(HO)、期望杂合度(HE)、香农信息指数(I)、主要等位基因频率(MAF)、遗传分化系数(Fst),进行分子方差分析(AMOVA);使用NTSYSpc2.1软件进行聚类分析并构建遗传聚类图。
2 结果与分析
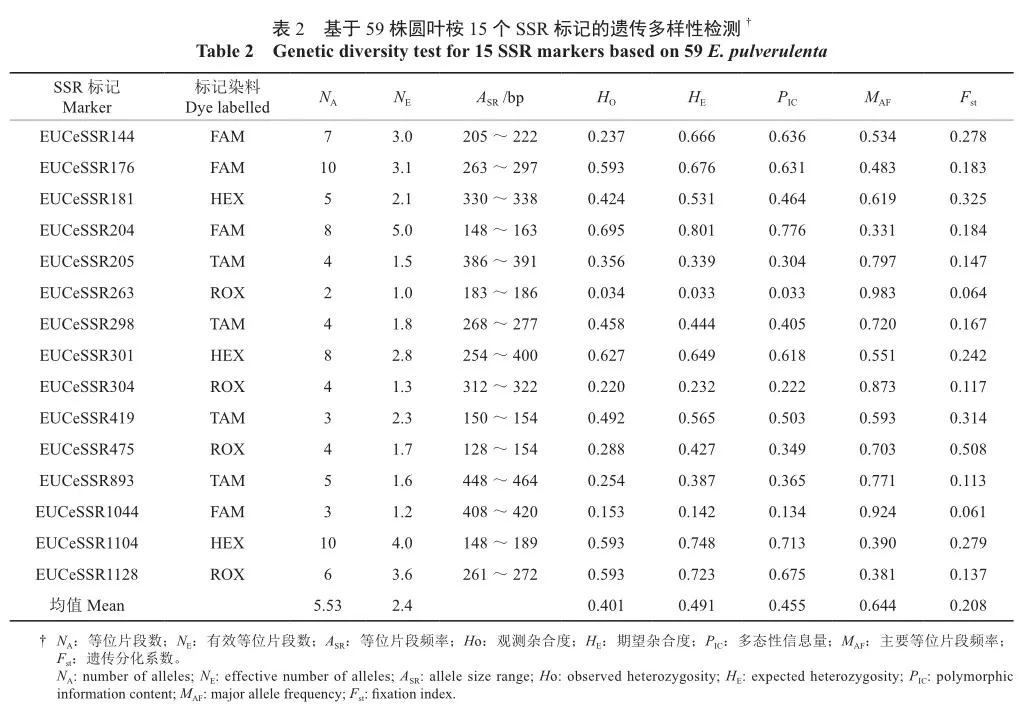
2.1 SSR标记的遗传多样性检测
本研究提取的59株圆叶桉DNA的OD260/OD280在1.80~2.00之间,符合后续试验要求。利用15个SSR荧光标记(表2)对59株圆叶桉进行了PCR扩增,并利用毛细管电泳法进行分型检测,分型结果带型清晰,易于片段统计。图1为多态性高的2个SSR(EUCeSSR204和EUCeSSR1128)标记对5个待测样品(A01、HNQS1、HNYXFF3、SHDSL1、YN01)的分型结果。15个SSR标记共检测到83个等位片段(Number of Alleles,NA),平均每个标记的等位片段数为5.5个,EUCeSSR176和EUCeSSR1044扩增出的NA最多,为10个,EUCeSSR263扩增出的NA最少,仅2个。平均每个标记的有效等位片段数(Effective number of alleles,NE)为2.4个,等位片段范围(Allele size range,ASR)为128~464 bp。观测杂合度(Observed heterozygosity,Ho)的变化范围为0.034(EUCeSSR263)~0.695(EUCeSSR204),平均值为0.401。期望杂合度(Expected Heterozygosity,HE)的变化范围为0.033~0.801(标记同HO),平均值为0.491,为中等水平。15个标记的多态性信息含量(Polymorphic Information Content,PIC)存在差异,变化范围为0.033~0.776,平均值为0.455,按PIC≥0.5为高多态性标记,0.25≤PIC<0.5为中度多态性标记,PIC<0.25为低多态性标记的标准[21],15个标记中有7个高多态性标记、5个中度多态性标记和3个低多态性标记。主要等位片段频率(Major allele frequency,MAF)的变化范围为0.331~0.983,平均值为0.644(表2)。不同SSR标记的遗传分化系数(Fst)差异较明显,变化范围为0.061(EUCeSSR1044)~0.508(EUCeSSR475),平均值为0.208,即有20.8%的变异存在于不同群体间,近79%的变异存在于圆叶桉个体间,说明各群体间的遗传分化程度较低。
2.2 圆叶桉个体间遗传关系聚类分析
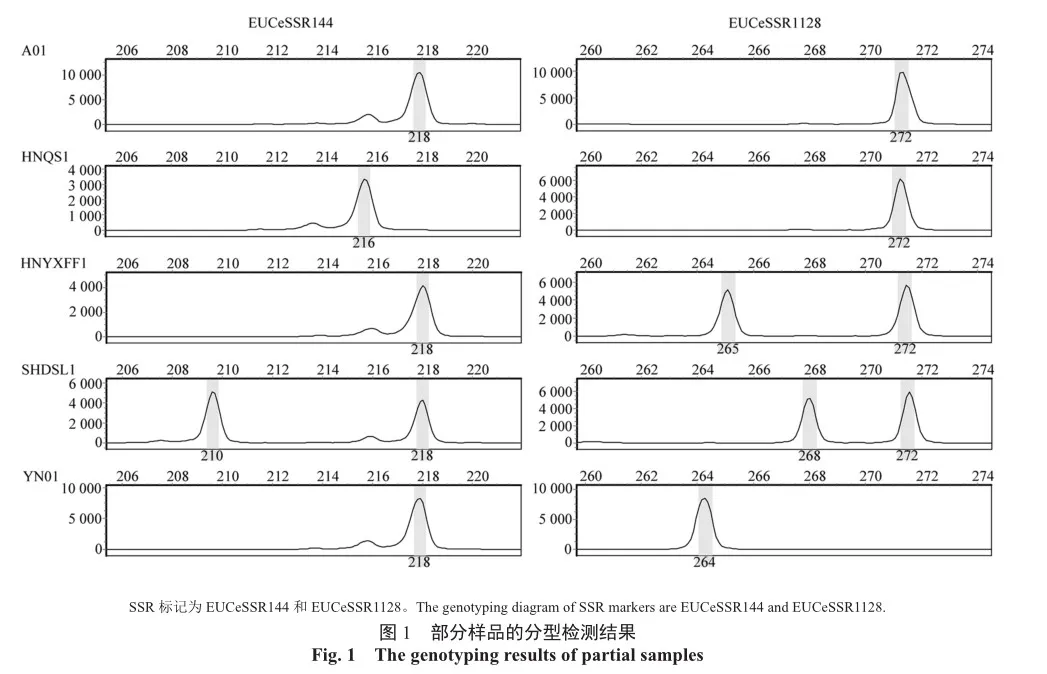
基于15个SSR标记分型检测数据计算59株圆叶桉的遗传距离,采用不加权算术平均组对方法(Unweighted pair-group method using arithmetic average,UPGMA)进行聚类分析,构建遗传聚类图(图2)。59株圆叶桉样品的遗传距离的范围为0.033(YN02和YN11)~0.698(A10和HNQS3),平均为0.377,表明不同来源的圆叶桉之间存在着一定差异。利用15个SSR标记可对59株圆叶桉进行有效的鉴定,表明当前国内圆叶桉花卉均采用种子种植的方式进行繁育。
聚类分析结果表明,在遗传距离为0.56时,59株圆叶桉可分为2个亚组,来自澳大利亚林木种子中心、云南佳艺园艺、云南绿梦花卉、湖南丘山园艺、上海大森林园艺、湖南衣想菲菲的全部圆叶桉和来自福建宏优家居的SHH1被分在第一亚组,来自福建宏优家居的4株圆叶桉(SHH2、SHH3、SHH4、SHH5)被分在第二亚组。这与圆叶桉天然分布于澳大利亚东南部的新南威尔士州和塔斯马尼亚州的温带地区2个种源的分类结果一致[1]。59株圆叶桉品种具有一定的遗传多样性,可划分为2个群体,但亲缘关系较近,遗传背景相对单一。
2.3 圆叶桉群体间遗传多样性检测
对7个不同来源的圆叶桉群体进行遗传多样性检测(表3),NA、NE、I、HO、HE参数均有差异,但相差均不大,HO与HE差距很小。其中来自云南佳艺园艺的圆叶桉NE、I、HE参数值最高,分别为2.4、0.890、0.506,遗传多样性丰富;来自澳大利亚林木种子中心的圆叶桉NA最高,为3.3;来自湖南丘山园艺的圆叶桉HO最高,为0.560。来自湖南衣想菲菲的圆叶桉参数值均为最低,说明该圆叶桉群体遗传多样性较低。
利用15个SSR标记,对7个不同来源的圆叶桉群体进行遗传多样性分析(表4),7个群体的遗传一致度为0.533~0.987,遗传一致度最小(0.533)的两个群体为来自福建宏优家居(R5)和云南佳艺园艺(R6)的圆叶桉;遗传一致度最大(0.987)的为来自云南佳艺园艺(R6)和云南绿梦花卉(R7)的圆叶桉,结合聚类分析结果(图3),推测R6和R7圆叶桉为来源相同的圆叶桉品种。7个群体间的遗传距离为0.013~0.628,平均遗传距离为0.262,表明不同来源的圆叶桉群体间遗传差异较小。
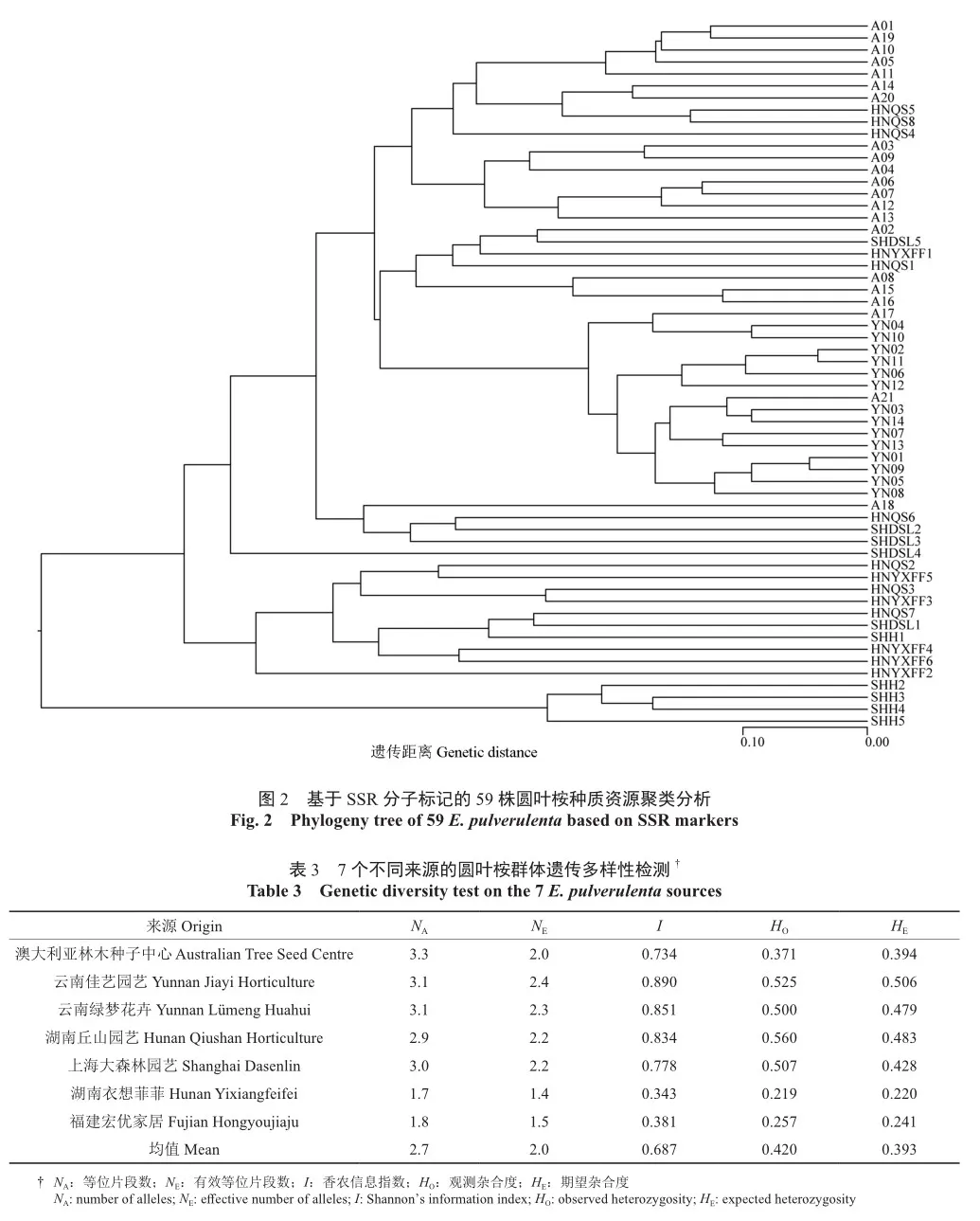
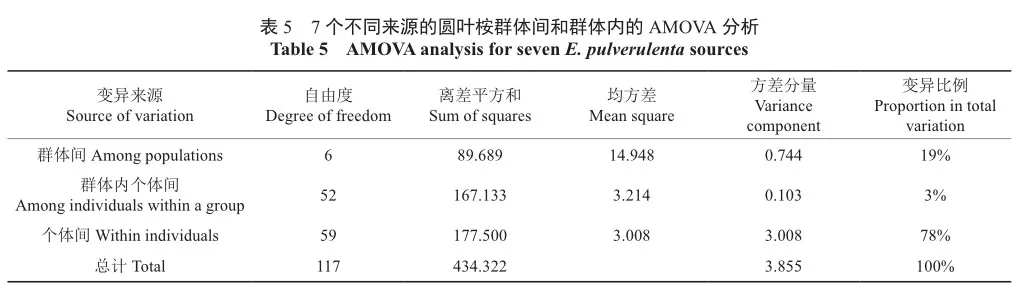
根据7个群体间的遗传相似性按照UPAMA方法进行聚类分析,构建遗传聚类图(图3)。当遗传相似性系数为0.74时,7个群体被分为两大类群,第一类群为来自澳大利亚林木种子中心(R1)、湖南丘山园艺(R2)、湖南衣想菲菲(R3)、上海大森林(R4)、云南佳艺园艺(R6)、云南绿梦花卉(R7)的圆叶桉;第二类群为来自福建宏优家居(R5)的圆叶桉,该结果与圆叶桉个体间聚类分析的结果一致。分子方差分析(Analysis of molecular variances,AMOVA)结果(表5)表明群体间变异占总方差分量的19%,与Fst遗传分化结果(20.8%)基本一致。个体间变异是方差分量的主要来源,占78%,群体内个体间变异最少,占总方差分量的3%。


3 结论与讨论
3.1 讨 论
遗传多样性一般是物种内部、群体间或种群内部个体间遗传变异的总和,林木遗传多样性的研究可以用于辅助改良品种、保存种质资源以及解释不同群体间的相关性等,是林木遗传改良的基础[22]。遗传多样性分析可通过形态学标记[23]和分子标记[24]等手段,从不同角度研究和分析林木的遗传变异,然而可直接用于鉴别的形态学标记数量有限,且多数形态学标记还受到环境和基因互作等影响,判别过程较为困难。随着分子标记技术的发展,SSR技术成为研究植物遗传多样性分析、品种鉴定、亲缘关系分析的重要工具。经筛选,本研究所用的SSR标记条带干净、整齐,便于分型,可用于遗传多样性分析,相关标记可用于今后圆叶桉品种和无性系的鉴定。所选标记在圆叶桉中均能较好地扩增,且表现出较高的多态性(中、高多态性标记占总标记的80%),表明SSR标记在不同桉树树种中呈现较高的通用性,可被较好地应用于种间遗传多样性分析、亲缘关系鉴定等相关研究。
利用分子标记开展圆叶桉群体遗传多样性分析的研究尚未见研究报道。本研究中的15个SSR标记,9个在巨桉中的PIC均值为0.529,HO均值为0.403[16];6个在尾叶桉和细叶桉中的PIC均值为0.657,HO均值为0.451[17],均为高多态性标记。圆叶桉中的PIC均值为0.455,HO均值为0.401,HE均值为0.491,这与上述2个树种的多样性水平类似,但PIC均值略低,说明相同的标记在不同的树种中的遗传多样性存在差异。前人利用等位酶分析法在4个圆叶桉群体中开展了遗传多样性分析研究,但多态性较低,其中PIC均值为0.27,HE为0.07,HO为0.05[25],说明不同的研究方法对群体多态性水平检测结果存在差异,当然这也与参试材料的来源和数量有关。基于SSR标记的圆叶桉遗传多样性水平普遍小于粗皮桉(PIC=0.718,HO=0.515,HE=0.722)[8]、大花序桉(PIC=0.771,HO=0.670,HE=0.804)[9]和巨桉(PIC=0.755,HO=0.631,HE=0.777)[13]等桉树,这可能与圆叶桉自然分布窄以及观赏性圆叶桉的人为选择有关。本研究中,7个不同来源的圆叶桉群体遗传多样性参数存在差异,其中云南佳艺园艺圆叶桉群体的NE、I、HE参数值最高,NA和HO参数值高于平均值,该群体遗传多样性较为丰富;湖南衣想菲菲圆叶桉群体的遗传多样性较低,NA、NE、HO、HE和I等参数值最低。相关研究认为,更多的标记能使品种间鉴定结果更加准确可靠,选择较多的个体数量更能有效反映群体多样性的水平[10]。本研究虽选择了遗传多样性高的SSR标记对来自澳大利亚林木种子中心种子繁育和国内在售的圆叶桉花卉进行基因分型,但并未进行大规模的标记筛选。在今后的研究中,可适当增加或选择杂合度更高的SSR标记,对不同种源/家系的圆叶桉开展遗传多样性的系统分析。
圆叶桉天然分布于澳大利亚东南部的新南威尔士州和塔斯马尼亚州[1],两州之间间隔巴斯海峡,存在一定程度的地理隔离,但地理位置上相距较近。本研究中的7个圆叶桉群体可分为2个亚组,其遗传背景相对单一,群体分化水平较低,这与圆叶桉2个种源的天然分布情况一致。AMOVA分析结果表明,圆叶桉群体间变异占总方差分量的19%,高于大花序桉的3%[10]、细叶桉的1.2%,个体间变异占总方差分量的81%。圆叶桉群体间变异水平虽高于其他桉树树种,但主要的遗传变异仍来源于个体间,这表明7个不同来源圆叶桉的分布范围和气候类型并未引起明显的遗传分化。在今后圆叶桉优良性状的遗传改良研究中,可以充分利用个体间的遗传变异。当前,国内市场主要种植‘蓝宝贝’和‘银水滴’等品种,随着圆叶桉市场规模的扩大,栽培中种植混乱、苗期优良品种表型难以分辨,利用本研究选用的SSR分子标记,亦可用于圆叶桉品种的鉴定工作。商品化圆叶桉群体聚类分析结果证实了国内圆叶桉种苗均为有性繁育,种子繁育的实生苗在叶形、叶色等方面存在遗传分化,一定程度上影响了花枝材的质量等级。当前国内仍缺乏具有自主知识产权的圆叶桉花卉新品种(http://cnpvp.flashfox. tech/),在今后的研究和生产中应加强优良品种选育,通过大范围的引种试验探索适应夏雨型气候区的种植模式,挖掘适生范围更广的新种质,以扩大圆叶桉在我国的种植范围;积极摸索圆叶桉的组培、扦插等无性扩繁方法,以期获得表现优良、性状稳定、适生范围广泛的圆叶桉花枝材。
3.2 结 论
本研究利用SSR分子标记技术对59株圆叶桉种质资源进行遗传多样性分析和聚类分析,所用的15个SSR标记在圆叶桉中均能稳定扩增,PIC均值为0.455,遗传多态性较高,可用于圆叶桉的遗传分化研究。圆叶桉个体间基因分型结果表明,国内市场上常见的圆叶桉均为有性繁殖的实生苗,聚类分析将其分为2个亚组,与圆叶桉天然分布情况一致。圆叶桉群体间的遗传分化程度较小,81%的遗传变异存在于个体间,为遗传变异的主要来源。本研究为圆叶桉种质资源保护和评价、亲缘关系分析、品种分子鉴定等提供了理论基础。
参考文献:
[1] 王豁然.桉树生物学概论[M].北京:科学出版社,2010: 83,161,266. WANG H R. A Chinese appreciation of Eucalyptus[M]. Beijing: Science Press,2010:83,161,266.
[2] 董静曦,黄甫则.桉树的园艺观赏价值[J].云南林业, 2002(6):21. DONG J X, HUANG F Z. Ornamental value of Eucalyptus in horticulture[J]. Yunnan Forestry,2002(6):21.
[3] 王希,虞超,刘慧琳,等.园林芳香植物的引种筛选及示范应用[R].四川省,成都市园林科学研究所,2014-12-31. WANG X, YU C, LIU H L, et al. Introduction and demonstration application of aromatic ornamental resources[R]. Sichuan province, Chengdu Institute of Landscape Architecture, 2014-12-31.
[4] BOLTE M L, BOWERS J, CROW W D, et al. Germination inhibitor from Eucalyptus pulverulenta[J]. Agricultural and Biological Chemistry,1984,48(2):373-376.
[5] POWELL W, MACHRAY G C, PROVAN J. Polymorphism revealed by simple sequence repeats[J]. Trends in Plant Science, 1996,1(7):215-222.
[6] 刘果,谢耀坚,吴志华,等.SSR标记在桉树树种精准鉴定中的应用分析[J].热带亚热带植物学报,2018,26(2):116-124. LIU G, XIE Y J, WU Z H, et al. Application of SSR markers in precise identification of Eucalyptus germplasms[J]. Journal of Tropical and Subtropical Botany,2018,26(2):116-124.
[7] 吕佳斌,翁启杰,李发根,等.基于SSR标记的大花序桉全同胞子代鉴定[J].中南林业科技大学学报,2021,41(10):148-155. LYU J B, WENG Q J, LI F G, et al. SSR-based identification of full-sib seedings in Eucalyptus cloeziana F.Muell[J]. Journal of Central South University of Forestry Technology,2021,41(10): 148-155.
[8] 李昌荣,熊涛,陈东林,等.粗皮桉种源遗传多样性SSR分析[J].南方农业学报,2016,47(1):7-12. LI C R, XIONG T, CHEN D L, et al. Analysis on genetic diversity of Eucalyptus pellita based on SSR markers[J]. Journal of Southern Agriculture,2016,47(1):7-12.
[9] 梁馨元,刘金炽,白天道,等.大花序桉EST-SSR标记开发及其种间通用性评价[J/OL].分子植物育种,http://kns.cnki. net/kcms/detail/46.1068.S.20220303.2202.053.html. LIANG X Y, LIU J Z, BAI T D, et al. EST-SSR marker development of Eucalyptus cloeziana and their applicability in genetic diversity and cross-species transferability[J/OL]. Molecular Plant Breeding,http://kns.cnki.net/kcms/detail/46.1068. S.20220303.2202.053.html.
[10] 王莉,李昌荣,李发根,等.大花序桉SSR位点多样性和群体结构分析[J].分子植物育种,2019,17(13):4470-4478. WANG L, LI C R, LI F G, et al. Analysis of SSR marker diversity and population structure in Eucalyptus cloeziana[J]. Molecular Plant Breeding,2019,17(13):4470-4478.
[11] SHANG X, ARNOLD J R, WU Z, et al. Combining quantitative data on growth, wood density and other traits with SSR markers to evaluate genetic diversity and structure in a planted population of Eucalyptus camaldulensis Dehn[J]. Forests,2019,10(12):1090.
[12] LI F G, ZHOU C P, WENG Q J, et al. Comparative genomics analyses reveal extensive chromosome colinearity and novel quantitative trait loci in Eucalyptus[J]. PLoS ONE, 2015,10(12):e0145144.
[13] ZHANG M M, ZHOU C P, SONG Z J, et al. The first identification of genomic loci in plants associated with resistance to galling insects: a case study in Eucalyptus L’Hér. (Myrtaceae)[J]. Scientific Reports,2018,8(1):2319.
[14] 彭继庆,曹福祥,曹基武,等.绣球花品种遗传多样性的ISSR分析及指纹图谱构建[J].中南林业科技大学学报,2016,36(12): 115-120. PENG J Q, CAO F X, CAO J W, et al. Genetic diversity and fingerprint construction of varieties of Hydrangea macrophylla by ISSR markers[J]. Journal of Central South University of Forestry Technology,2016,36(12):115-120.
[15] 陈家龙,刘辉,余宏傲,等.圆叶桉(Eucalyptus pulverulenta Sims)繁殖技术初探[J].浙江农业科学,2009(4):692-694. CHEN J L, LIU H, YU H A, et al. Exploration propagative technique of Eucalyptus pulverulenta Sims[J]. Journal of Zhejiang Agricultural Sciences,2009(4):692-694.
[16] ZHOU C P, HE X D, LI F G, et al. Development of 240 novel EST-SSRs in Eucalyptus L’He′rit[J]. Molecular Breeding, 2014,33(1):221-225.
[17] HE X D, WANG Y, LI F G, et al. Development of 198 novel EST-derived microsatellites in Eucalyptus (Myrtaceae)[J]. American Journal of Botany,2012,99(4):134-148.
[18] GAN S M, SHI J S, LI M, et al. Moderate-density molecular maps of Eucalyptus urophylla ST Blake and E. tereticornis Smith genomes based on RAPD markers[J]. Genetica,2003,118:59-67.
[19] LIU K, MUSE S V. PowerMarker: an integrated analysis environment for genetic marker analysis[J]. Bioinformatics,2005,21(9): 2128-2129.
[20] PEAKALL R O D, SMOUSE P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research[J]. Molecular Ecology Notes,2006,6(1):288-295.
[21] BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980,32(3):314.
[22] WHITE T L, ADAMS W T, NEALE D B. Forest genetics[M]. UK: CABI Publishing,2007:149-186.
[23] 傅志强,袁汕,申春晖,等.基于花器官特征的油茶遗传多样性分析[J].经济林研究,2022,40(4):12-18,28. FU Z Q, YUAN S, SHEN C H, et al. Genetic diversity analysis based on characteristics of floral organs of Camellia oleifera[J]. Non-wood Forest Research,2022,40(4):12-18,28.
[24] 高园,解元,杨自云,等.基于花期差异和ISSR-PCR分析的不同茶梅品种间亲缘关系[J].经济林研究,2022,40(4): 209-217. GAO Y, XIE Y, YANG Z Y, et al. Genetic relationship analysis of different Camellia sasanqua cultivars based on the difference of flowering season and ISSR-PCR[J]. Non-wood Forest Research, 2022,40(4):209-217.
[25] PETERS G B, LONIE J S, MORAN G F. The breeding system, genetic diversity and pollen sterility in Eucalyptus pulverulenta, a rare species with small disjunct populations[J]. Australian Journal of Botany,1990,38(6):559-570.
[本文编校:吴 彬]

