整合多芯片数据的肺癌诊断关键基因的发现与初步验证


[摘" "要]" "目的:通过GEO多芯片联合分析筛选出一组与肺癌发生密切相关的基因,作为预测肺癌的关键标志基因并进行初步验证。方法:从GEO数据库下载GSE89047、GSE108055与GSE116959肺癌表达数据集并进行合并,采用R语言中sva程序包ComBat矫正批次效应,limma程序包进行基因差异表达分析从中筛选出肺癌差异表达基因。利用String数据库结合Cytoscape 3.8.2软件构建差异表达基因蛋白质相互作用网络,并分析核心基因。运用ROC方法验证肺癌差异基因、核心基因对肺癌诊断的预测作用。通过TIMER数据库分析GPM6A基因表达及拷贝数变异与免疫细胞浸润的关系。结果:基于GEO数据库GSE89047、GSE108055与GSE116959肺癌表达数据集多芯片联合分析,筛选得到938个肺癌组织与正常肺组织间差异表达基因,以矫正的P值排序,TOP 10差异基因为GPM6A、WNT3A、SLC6A4、TMEM100、TCF21、BTNL9、HSPA12B、LIMS2、VGLL3和ITLN2。String数据库结合Cytoscape 3.8.2软件分析所得10个核心基因为CCNA2、CCNB1、CENPE、FOXM1、ITGAM、KIF11、KIF20A、KIF23、KIF2C和MMP9。ROC分析显示GPM6A的AUC(95%CI)为0.948(0.874~0.986);TOP10差异基因的AUC(95%CI)为0.961(0.886~0.992);10个核心基因的AUC(95%CI)为0.830(0.722~0.895),表明这些标志基因具有较好的肺癌预测能力。TIMER分析结果显示:肺腺癌及肺鳞癌中GPM6A表达均与巨噬细胞浸润相关性最高(肺腺癌:r=0.347,Plt;0.001;肺鳞癌:r=0.425,Plt;0.001),GPM6A基因拷贝数变异在肺腺癌中与B细胞、CD4+T细胞、巨噬细胞、中性粒细胞和树突状细胞的免疫浸润相关(均Plt;0.05),GPM6A基因拷贝数变异在肺鳞癌中与B细胞、CD8+T细胞、CD4+T细胞、巨噬细胞、中性粒细胞和树突状细胞的免疫浸润均具有较高的相关性(均Plt;0.05)。结论:通过多芯片联合分析初步开发、验证了对肺癌诊断具有较好预测能力的标志基因,并发现差异最显著的标志基因GPM6A与免疫细胞浸润关系密切。
[关键词]" "肺癌;多芯片联合;预测;开发;验证;免疫浸润
[中图分类号]" "R734.2" " " " " " " "[文献标志码]" "A" " " " " " " "[文章编号]" "1674-7887(2024)04-0307-06
Development and preliminary verification of lung cancer diagnostic marker genes
based on the joint analysis of multiple chips
[Abstract]" "Objective: To screen out a group of genes closely related to the occurrence of lung cancer through GEO multichip combined analysis, as a key marker gene for predicting lung cancer, and conduct preliminary verification. Methods: Download the GSE89047, GSE108055 and GSE116959 lung cancer expression datasets from GEO database and merge them. The sva program package ComBat in the R language corrects the batch effect, and the limma program package performs gene differential expression analysis to screen out lung cancer differentially expressed genes. String database combined with Cytoscape 3.8.2 software to construct a differentially expressed gene protein-protein interaction network and analyze core genes. The ROC method was used to verify the predictive effect of lung cancer differential genes and core genes on the diagnosis of lung cancer. TIMER database was used to analyze the relationship between GPM6A gene expression and copy number variation and immune cell infiltration. Results: Based on the multi-chip combined analysis of the GEO database GSE89047, GSE108055 and GSE116959 lung cancer expression datasets, 938 differentially expressed genes between lung cancer tissues and normal lung tissues were screened and sorted by the corrected P value. The TOP 10 differential genes were GPM6A, WNT3A, SLC6A4, TMEM100, TCF21, BTNL9, HSPA12B, LIMS2, VGLL3 and ITLN2. The 10 core genes analyzed by String database combined with Cytoscape 3.8.2 software are CCNA2, CCNB1, CENPE, FOXM1, ITGAM, KIF11, KIF20A, KIF23, KIF2C and MMP9. ROC analysis showed that the AUC(95%CI) of GPM6A was 0.948(0.874-0.986); the AUC(95%CI) of the TOP10 differential genes was 0.961(0.886-0.992); the AUC(95%CI) of the 10 core genes was 0.830(0.722-0.895), indicating that the marker genes selected in this study have good lung cancer prediction ability. TIMER analysis showed that GPM6A expression correlated highest with macrophage infiltration in both lung adenocarcinoma and lung squamous carcinoma(lung adenocarcinoma: r=0.347, Plt;0.001; lung squamous carcinoma: r=0.425, Plt;0.001), GPM6A gene copy number variation correlated with immune infiltration of B cells, CD4+T cells, macrophages, neutrophils and dendritic cells in lung adenocarcinoma(Plt;0.05), and GPM6A gene copy number variation correlated with immune(Plt;0.05), and GPM6A gene copy number variants were highly correlated with immune infiltration of B cells, CD8+T cells, CD4+T cells, macrophages, neutrophils and dendritic cells in lung squamous carcinoma(Plt;0.05). Conclusion: In this study, we initially developed and verified some marker genes with better predictive ability for lung cancer diagnosis through multi-chip combined analysis, and found that the most significant difference marker gene GPM6A is closely related to immune cell infiltration.
[Key words]" "lung cancer; multi-chip combination; prediction; development; verification; immune infiltration
肺癌是指支气管黏膜或呼吸系统腺体的恶性肿瘤。在世界范围内,肺癌是主要的公共卫生问题,它是第二大常见癌症,是癌症相关死亡的第一大原因[1]。根据组织病理学类型,肺癌可分为非小细胞肺癌和小细胞肺癌,后者被发现与吸烟密切相关[2]。非小细胞肺癌约占所有肺癌病例的85%,其主要表现为呼吸道症状和局部压迫症状。由于缺乏有效的诊断方法,大多数肺癌在中晚期才被发现。因此,肺癌的早期发现和治疗对控制其死亡率具有重要作用。
在目前的早期筛查方法中,胸片和痰细胞学是经济实用的方法。然而,它们的敏感性和特异性都不是很高。低剂量电脑断层扫描可检测肺内仅几毫米大小的小病变,敏感性高,但特异性较差。它也会给良性肿瘤结节患者造成精神和经济负担,甚至可能对身体造成不必要的创伤[3]。靶向治疗作用于肺癌基因组突变确定的关键治疗点,但仍使少数患者受益[4]。现有研究[5]表明,遗传多态性和高外显率基因等是在个体对肺癌的易感性中起重要作用的遗传因素。因此,需要新的癌症生物标志物来尽快诊断、预测和治疗肺癌。
本研究旨在通过多芯片联合分析初步开发、验证对肺癌诊断具有较好预测能力的标志基因,并分析标志基因与免疫细胞浸润的关系,以期为肺癌早期诊断提供可靠的生物学标志物。
1" "材料与方法
1.1" "肺癌组织与正常肺组织差异表达基因分析与筛选" "利用美国国立生物技术信息中心的GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)选取并下载GSE89047、GSE108055、GSE116959肺癌表达数据集的基因表达文件及注释文件。对上述3个数据集进行合并,通过R语言中sva软件包ComBat矫正批次效应,以消除不同平台的数据,同一平台不同时期的数据,同一样品不同试剂的数据,以及同一样品不同时间数据的批次效应。采用R语言limma程序包进行基因差异表达分析,以FDR(adj.P.value)lt;0.05、logFC绝对值gt;1筛选肺癌组织和正常肺组织间的差异基因。并通过R语言绘制基因表达火山图及热图。
进一步利用TCGA数据库样本对多芯片联合分析所得的差异基因进行验证。使用箱线图显示基因表达水平的分布,使用R软件Wilcoxon检验评估肿瘤和邻近正常肺组织之间差异表达的统计显著性。
1.2" "蛋白质相互作用(protein-protein interaction, PPI)网络分析及核心基因筛选" "通过String(https://www.string-db.org/)数据库进行差异基因的PPI网络分析,Cytoscape 3.8.2软件对PPI网络进行可视化分析,并通过该软件的cytoHubba模块以Degree为依据计算差异基因的核心基因(Hub基因)。
1.3" "肺癌诊断标志基因的验证" "GEO多芯片联合分析数据集纳入的肺癌患者为研究对象(GSE89047、GSE108055、GSE116959),利用正常肺组织和肺癌组织中TOP1、TOP10、Hub10基因表达数据进行ROC曲线分析,以验证本研究开发的基因对肺癌诊断的可靠性。
1.4" "肺癌诊断标志基因与免疫浸润的关系" "TIMER数据库评估GPM6A基因表达及拷贝数变异与肺癌免疫细胞浸润的关系。GPM6A基因与肺癌中免疫浸润水平的相关性通过Spearman方法进行分析。拷贝数变异与肺癌免疫细胞浸润的关系通过箱线图进行展示。双侧Wilcoxon秩和检验将每个拷贝数变异类别与正常水平进行统计分析。
2" "结" " " 果
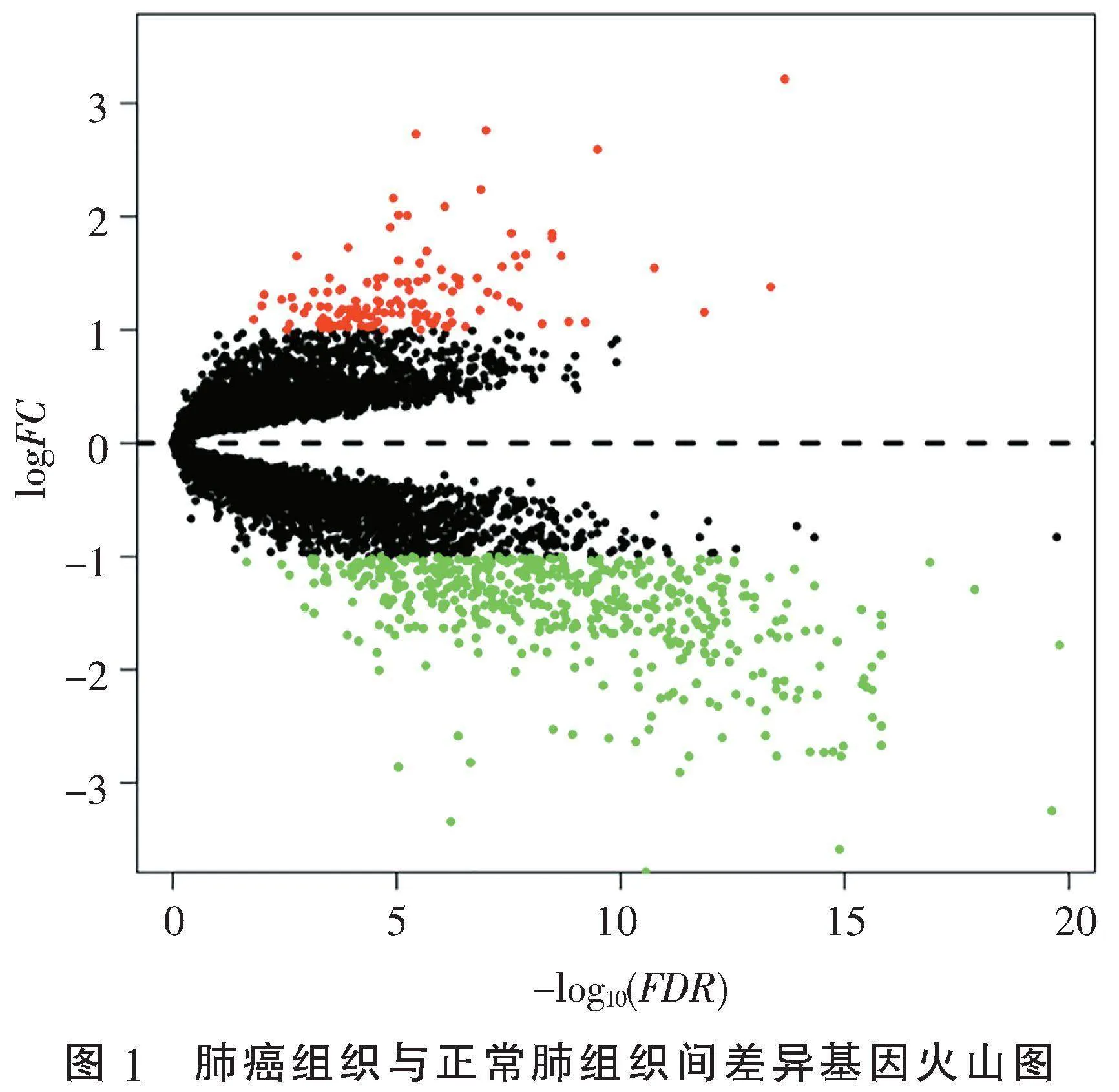
2.1" "肺癌组织与正常肺组织的差异表达基因" "基于GEO数据库的多芯片联合分析,共纳入肺癌组织119例,正常肺组织29例,其中肺癌亚型包括肺腺癌、小细胞癌,获得差异表达基因938个,包括674个在肺癌组织中上调的基因,264个在肺癌组织中下调的基因(图1)。以FDR(adj.P.value)为依据排序,差异基因TOP1为GPM6A;TOP10为GPM6A、WNT3A、SLC6A4、TMEM100、TCF21、BTNL9、HSPA12B、LIMS2、VGLL3和ITLN2。TOP100差异表达基因如图2所示。通过TCGA数据库分析显示GPM6A在多种癌症中具有显著的表达差异,在肺腺癌和肺鳞癌中均引起了显著的表达差异(Plt;0.001)(图3)。
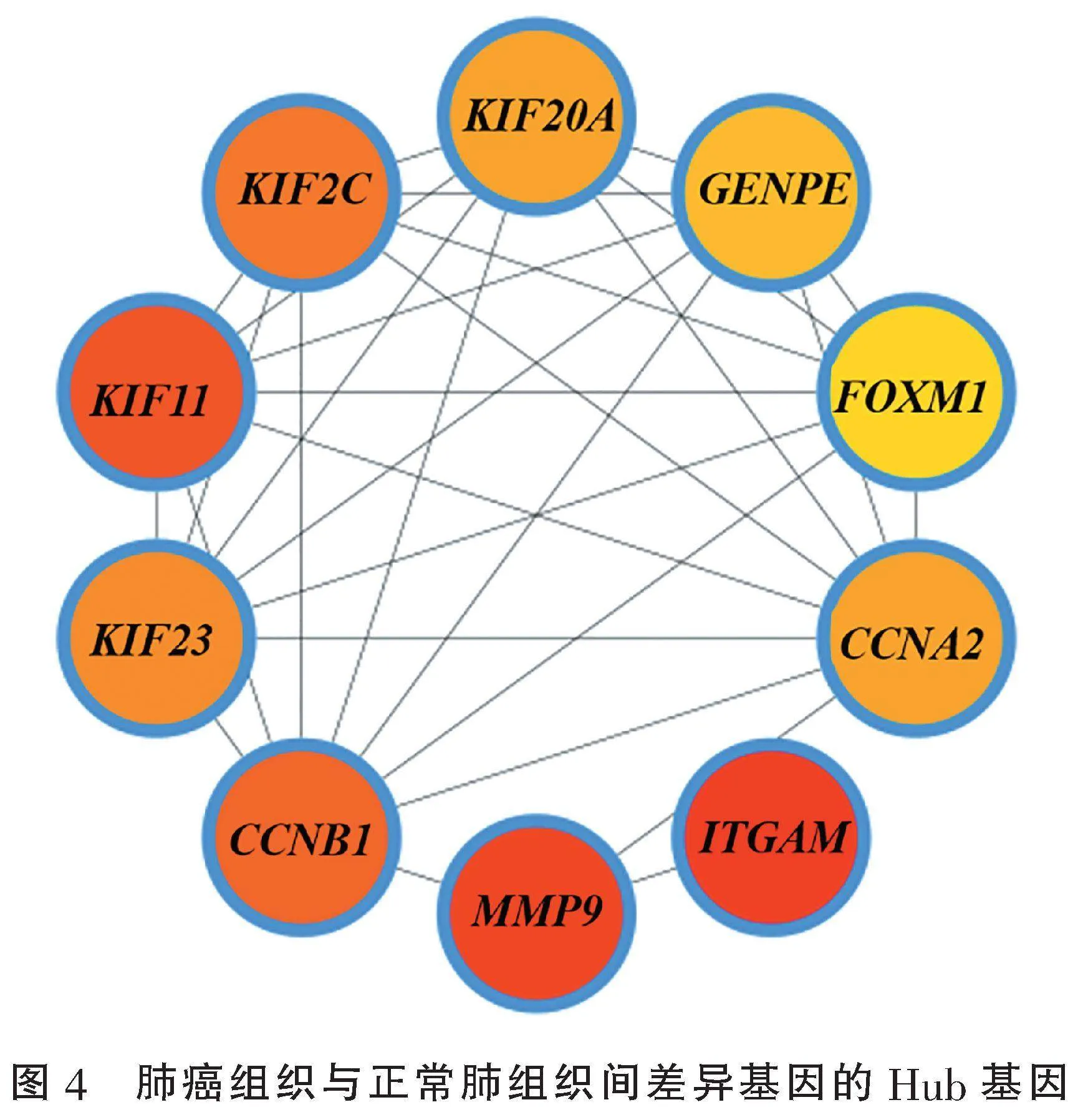
2.2" "PPI网络的建立及Hub基因的筛选" "通过String数据库与Cytoscape软件构建938个差异基因的PPI网络。在Cytoscape软件中运用cytoHubba模块以Degree为依据计算差异基因的10个Hub基因为CCNA2、CCNB1、CENPE、FOXM1、ITGAM、KIF11、KIF20A、KIF23、KIF2C和MMP9(图4)。
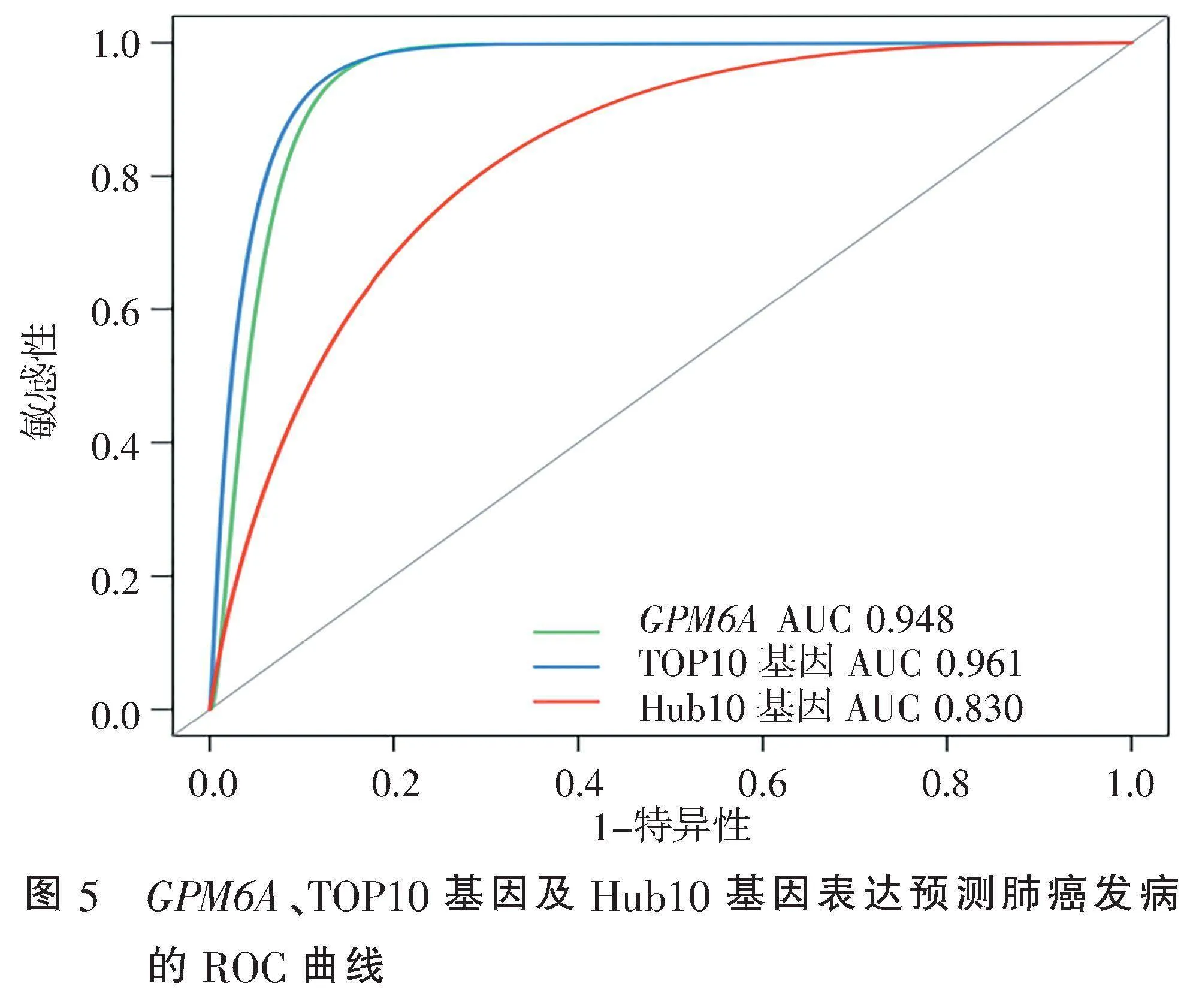
2.3" "肺癌诊断标志基因验证" "以GPM6A、TOP10基因及Hub10基因表达为依据对肺癌患者发病进行预测,绘制ROC曲线(图5)。其中GPM6A的AUC(95%CI)为0.948(0.874~0.986);TOP10基因的AUC(95%CI)为0.961(0.886~0.992);Hub10基因的AUC(95%CI)为0.830(0.722~0.895),表明本研究所选的标志基因具有较好的肺癌预测能力。
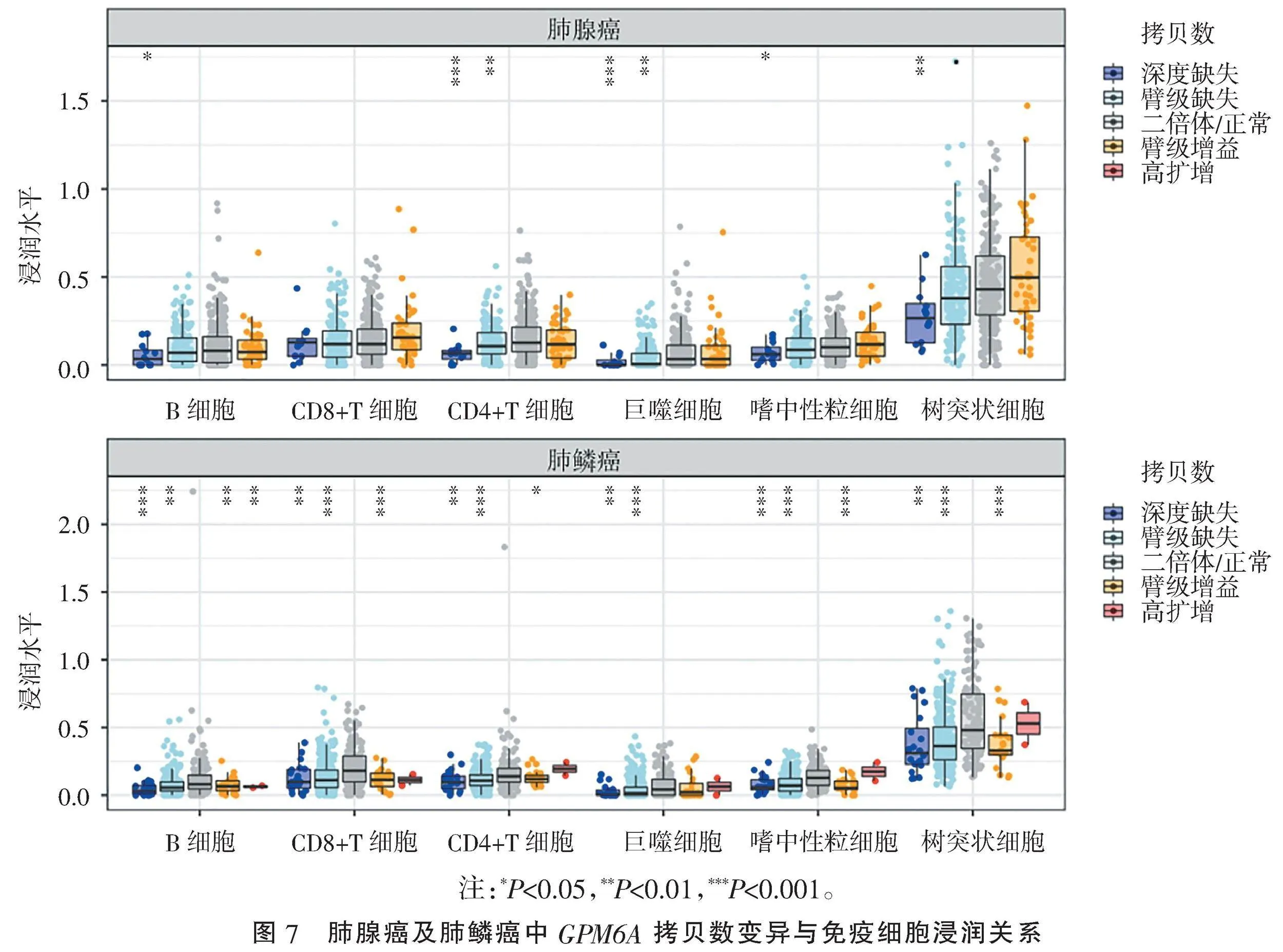
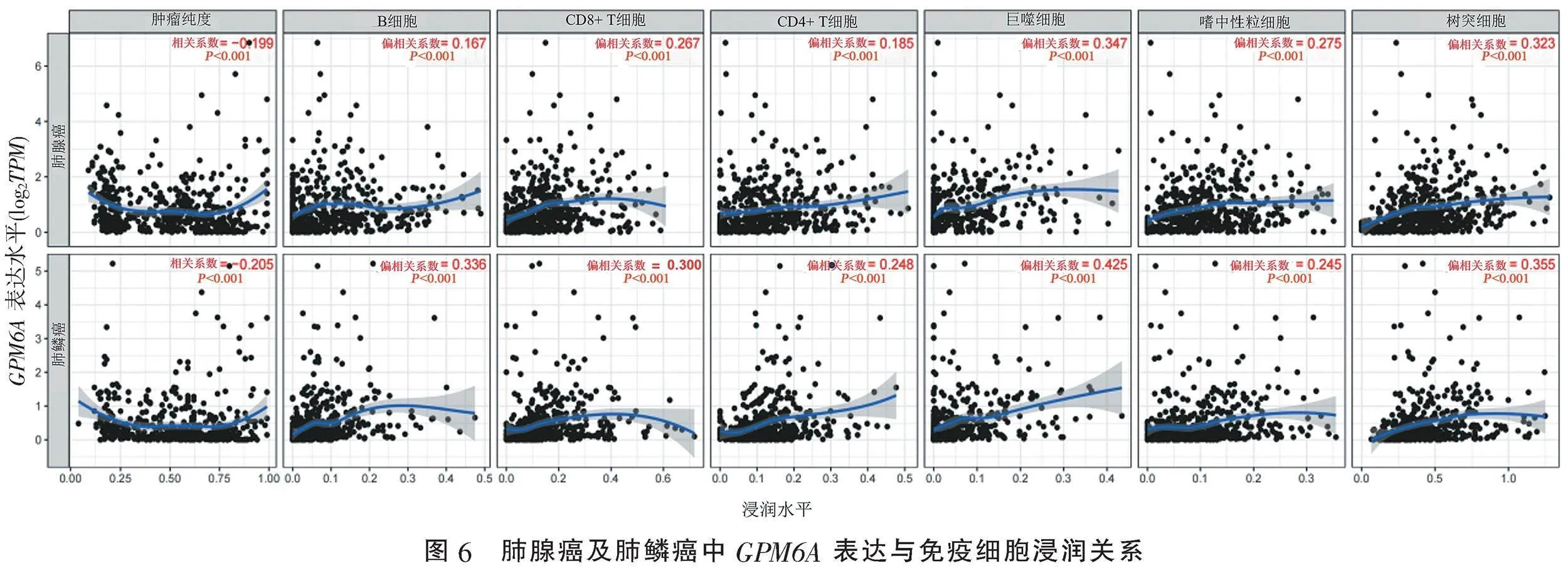
2.4" "GPM6A与免疫细胞浸润的关系" "TIMER数据库分析表明,肺腺癌及肺鳞癌中GPM6A表达均与巨噬细胞浸润相关性最高(肺腺癌:r=0.347,Plt;0.001;肺鳞癌:r=0.425,Plt;0.001)(图6)。GPM6A基因拷贝数变异在肺腺癌中与B细胞、CD4+T细胞、巨噬细胞、中性粒细胞和树突状细胞的免疫浸润相关(Plt;0.05);GPM6A基因拷贝数变异在肺鳞癌中与B细胞、CD8+T细胞、CD4+T细胞、巨噬细胞、中性粒细胞和树突状细胞的免疫浸润均具有较高的相关性(Plt;0.05)(图7)。
3" "讨" " " 论
晚期肺癌因肝转移等原因常导致治疗效果差、术后生存时间较短[6-8]。因此肺癌的早期诊断显得尤为重要。本研究通过多芯片联合分析发现了诸如GPM6A、WNT3A、SLC6A4等具有较高表达差异的肺癌相关基因。其中,GPM6A在人类B细胞恶性肿瘤的发展中发挥作用,可能充当潜在原癌基因[9]。WNT3A作为Wnt信号通路的主要成分能将癌细胞转化为侵袭性和转移性表型,通过促进上皮间质转化、调节MMP的表达和其他在细胞外基质调节中起作用的因素来促进癌症的转移进展[10-11]。SLC6A4基因单核苷酸多态性与癌症易感性及严重程度密切相关[12-14]。
肺癌组织与正常肺组织间的差异基因中核心基因的计算显示,ITGAM、KIF11、MMP9等基因的度(Degree)较大,相互作用的基因较多。ITGAM是与炎症反应发展相关的基因之一,ITGAM基因的蛋白质产物负责Ⅱ型干扰素受体的功能和炎症介质分泌的调节[15]。ITGAM基因调控区变异是头颈癌接受调强放疗患者营养不良的新预测因子[16]。KIF11作为有丝分裂相关基因影响非小细胞肺癌患者的预后[17]。MMP9的遗传变异及活性改变可影响肺癌的易感性及预后[18-19]。
以GPM6A、TOP10差异基因及Hub10基因表达为依据对肺癌患者发病进行预测的ROC曲线表明,GPM6A、TOP10差异基因作为标志基因的肺癌诊断预测方法相对更优,其AUC值均>0.900,表明运用差异基因GPM6A及TOP10差异基因进行肺癌预测的诊断准确度较高。Hub10基因的AUC值介于0.800~0.900之间,表明其肺癌预测的诊断准确度适中。
为进一步探究肺癌差异基因具有高肺癌诊断准确度的潜在原因,本研究分析了最显著的差异基因GPM6A与肺癌患者免疫细胞浸润的关系。结果显示,GPM6A表达及拷贝数变异均会影响肺癌的免疫细胞浸润水平。先前的研究[20]显示肺癌的发生和扩散不仅依赖于肿瘤细胞的特性,而且还受到与免疫系统相互作用的影响。最近,对肺癌的免疫疗法也显现出显著的生存获益[21]。
综上,本研究通过多芯片联合分析初步开发、验证了对肺癌诊断具有较好预测能力的标志基因,并发现差异最显著的标志基因GPM6A与免疫细胞浸润关系密切。
[参考文献]
[1]" "SIEGEL R L, MILLER K D, FUCHS H E, et al. Cancer statistics, 2021[J]. CA Cancer J Clin, 2021, 71(1):7-33.
[2]" "WALTER J E, HEUVELMANS M A, DE BOCK G H, et al. Relationship between the number of new nodules and lung cancer probability in incidence screening rounds of CT lung cancer screening: The NELSON study[J]. Lung Cancer, 2018, 125:103-108.
[3]" "KLUTSTEIN M, NEJMAN D, GREENFIELD R, et al. DNA methylation in cancer and aging[J]. Cancer Res, 2016, 76(12):3446-3450.
[4]" "SPENCER D H, LEY T J. Sequencing of tumor DNA to guide cancer risk assessment and therapy[J]. JAMA, 2018, 319(14):1497.
[5]" "MALHOTRA J, MALVEZZI M, NEGRI E, et al. Risk factors for lung cancer worldwide[J]. Eur Respir J, 2016, 48(3):889-902.
[6]" "ZHU R F, LIU Z H, JIAO R, et al. Updates on the patho-genesis of advanced lung cancer-induced cachexia[J]. Thorac Cancer, 2019, 10(1):8-16.
[7]" "吴海山, 邹端萍, 李建成. 影响非小细胞肺癌脑转移预后的相关因素探讨[J]. 南通大学学报(医学版), 2018, 38(1):58-62.
[8]" "张国伟, 程瑞瑞, 张国俊, 等. 有或无肝转移的晚期非小细胞肺癌应用纳武利尤单抗的疗效差异: 一项回顾性队列研究[J]. 现代肿瘤医学, 2021, 29(15):2615-2619.
[9]" "YOSHIMURA K, HANAOKA T, OHNAMI S, et al. Allele frequencies of single nucleotide polymorphisms(SNPs) in 40 candidate genes for gene-environment studies on cancer: data from population-based Japanese random samples[J]. J Hum Genet, 2003, 48(12):654-658.
[10]" "SIMMONS C P, KOINIS F, FALLON M T, et al. Prognosis in advanced lung cancer—a prospective study examining key clinicopathological factors[J]. Lung Cancer, 2015, 88(3):304-309.
[11]" "CHARFI C, EDOUARD E, RASSART E. Identification of GPM6A and GPM6B as potential new human lymphoid leukemia-associated oncogenes[J]. Cell Oncol, 2014, 37(3):179-191.
[12]" "ZHANG Q, BAI X L, CHEN W, et al. Wnt/β-catenin signaling enhances hypoxia-induced epithelial-mesenc-hymal transition in hepatocellular carcinoma via crosstalk with hif-1α signaling[J]. Carcinogenesis, 2013, 34(5):962-973.
[13]" "SAVAS S, HYDE A, STUCKLESS S N, et al. Serotonin transporter gene(SLC6A4) variations are associated with poor survival in colorectal cancer patients[J]. PLoS One, 2012, 7(7):e38953.
[14]" "LI C Y, SONG G R, ZHANG S Y, et al. Wnt3a increases the metastatic potential of non-small cell lung cancer cells in vitro in part via its upregulation of Notch3[J]. Oncol Rep, 2015, 33(3):1207-1214.
[15]" "CRISPN J C, HEDRICH C M, TSOKOS G C. Gene-function studies in systemic lupus erythematosus[J]. Nat Rev Rheumatol, 2013, 9:476-484.
[16]" "MAZUREK M, MLAK R, HOMA-MLAK I, et al. Poly-morphism of the regulatory region of the ITGAM gene(-323Ggt;A) as a novel predictor of a poor nutritional status in head and neck cancer patients subjected to intensity-modulated radiation therapy[J]. J Clin Med, 2020, 9(12):4041.
[17]" "SCHNEIDER M A, CHRISTOPOULOS P, MULEY T, et al. AURKA, DLGAP5, TPX2, KIF11 and CKAP5: five specific mitosis-associated genes correlate with poor prognosis for non-small cell lung cancer patients[J]. Int J Oncol, 2017, 50(2):365-372.
[18]" "LI W, JIA M X, WANG J H, et al. Association of MMP9-1562C/T and MMP13-77A/G polymorphisms with non-small cell lung cancer in southern Chinese population[J]. Biomol-ecules, 2019, 9(3):E107.
[19]" "DONG D D, ZHOU H, LI G. ADAM15 targets MMP9 activity to promote lung cancer cell invasion[J]. Oncol Rep, 2015, 34(5):2451-2460.
[20]" "ROSENTHAL R, CADIEUX E L, SALGADO R, et al. Neoantigen-directed immune escape in lung cancer evo-lution[J]. Nature, 2019, 567(7749):479-485.
[21]" "BRAHMER J R. Harnessing the immune system for the treatment of non-small-cell lung cancer[J]. J Clin Oncol, 2013, 31(8):1021-1028.

