环境DNA技术在长江江津段鱼类多样性监测中的应用研究





摘 要:通过环境DNA技术来研究长江江津段鱼类资源分布,为长江江津段河流管理提供重要的基础数据。于2021年6月在该江段布设6个断面,通过水样采集、环境DNA捕获及注释分析来检测鱼类的多样性组成。研究结果显示:长江江津段共检测出鱼类37种,分属于4目10科32属,包括2种国家级保护鱼类、14种长江上游特有鱼类、2种重庆市重点保护鱼类、3种外来鱼类。通过物种相对序列丰度分析发现,吻鮈(Rhinogobio typus)、子陵吻鰕虎鱼(Rhinogobius giurinus)和铜鱼(Coreius heterodon)的相对丰度较高;基于序列丰度的α和β多样性分析结果表明,各断面鱼类组成和多样性呈现出一定的差异。与最近的传统形态学调查数据相比,环境DNA技术与传统方法得到的结果相似,表明环境DNA技术可以被作为一项有效的辅助手段,用以迅速了解长江鱼种的种类数量及其地理分布情况。
关键词:环境DNA;鱼类多样性;鱼类监测;鱼类资源分布;长江江津段
中图分类号:S932.4 文献标志码:A 文章编号:1673-5072(2024)06-0579-08
鱼类资源多样性作为河流生物多样性必不可少的部分,对保持水生态系统平衡与社会可持续发展具有极其重要的意义[1]。近年来,由于人类过度地开发和利用自然资源,导致鱼类资源多样性严重降低[2]。2005年,为保护长江上游珍稀特有鱼类多样性,国务院批准设立了长江上游珍稀特有鱼类国家级自然保护区(以下简称“保护区”)[3]。江津段位于保护区下游江段,拥有显著的生态功能,不仅是保护区内珍稀特有鱼类的关键栖息地,同时也是长江下游渔业资源增殖的重要通道。江津段的重要性不仅体现在维护我国淡水鱼类的遗传资源上,同时对促进鱼类多样性具有重要意义[4]。
为保护长江流域鱼类资源多样性,农业农村部于2020年对长江流域开展“十年禁渔”。渔业资源的恢复及多样性评价需要常年持续性的监测,在当前长江大保护的背景下,传统的渔获物调查监测不仅对渔业资源有一定的干扰和损害,而且客观条件受限也难以开展[5-6]。环境DNA宏条形码(Environmental DNA Metabarcoding)技术是一种从环境样本中直接提取总DNA,利用特异性或通用引物进行聚合酶链式反应(Polymerase Chain Reaction,PCR),通过高通量测序和生物信息学分析,实现对生物多样性的群落水平鉴定[7-8]。2008年,Ficetola等[9]首次将环境DNA技术应用于淡水水生生物种群研究tAVAUVC+yHyy3NDod1mpgQ==;赵梦迪[10]通过使用Miya等[11]设计出了12S rRNA作为探针,深入探讨了黄海近海区域内的鱼类群体结构,总计发现了超过571种鱼类,其中包括许多常见的或者有历史记载的品种;为了建立一套针对长江鱼类的环境DNA检测系统,徐念和常剑波[12]在长江的中下游河段设立了共计24个取样站点,并成功识别出了15种不同类型的鱼类。这些研究不断推动着环境DNA技术在水生生态系统生物监测中的应用[13-14],为水生生物多样性研究和监测提供了一种新思路。
近年来,通过刺网、撒网等传统捕捞监测的结果表明[3-4,15-17],长江江津段渔业资源已严重衰退,一些珍稀特有鱼类种群数量急剧下降,部分经济鱼类资源趋于枯竭[18-19]。相较于传统调查方法,环境DNA宏条形码具有检测灵敏度高和对生态系统干扰低等优势,弥补了传统形态学监测的缺陷,在生物多样性评估方面具有极大的应用潜力[20]。本研究以长江江津段为调查区域,运用环境DNA技术对长江鱼类资源的多样性和群落结构进行评估,探索适用于长江鱼类资源监测和保护的新方法,为后续评估长江“十年禁渔”效果提供相关的基础数据。
1 材料与方法
1.1 采样断面设置与水样采集
长江江津段位于重庆市江津区石蟆镇与珞璜镇之间。综合长江江津段地理环境和鱼类分布的历史资料,本研究于2021年6月在该江段的三抛河、松溉、高占滩、丁家沱、綦江口和马夫沱共6个采样断面(图1)进行了1次采样。水样采集过程中,利用采水器从每个采样断面的左、中、右江段的水体共采集6 L混合后,平均分装于3个用10%漂白粉溶液消毒的聚乙烯瓶中[21]。将收集到的水样立即冷藏储存备用。
1.2 环境DNA的提取及测序
1.2.1 环境DNA的富集
在24 h内,使用真空泵将采集的水样抽滤到0.45 μm的混合纤维素滤膜(Whatman,UK)上,以富集环境DNA。每个样品在抽滤前后都需要对器材进行消毒,以防止样品之间的交叉污染。在每个样品抽滤时,同时设置一个阴性对照以评估是否存在外源DNA污染[22]。最后,将滤膜放置在-80 ℃冷冻保存备用。
1.2.2 环境DNA的提取
使用Qiagen PowerWater DNA Isolation Kits从水样滤膜提取总DNA,然后通过1%凝胶电泳质检[22]。提取的DNA样品保存在-20 ℃。
1.2.3 环境DNA扩增及建库测序
使用引物mlCOIintF[23]对样品线粒体CO I基因进行PCR扩增,扩增片段长度约为313 bp。PCR的扩增体系和反应条件参考文献[24],样本检测采用舒璐等[25]的方法,纯化后的最终产物通过Illumina NovaSeq 6000测序平台进行高通量测序。
1.3 数据分析
通过文献资料整理出长江上游淡水鱼类名录[26-27]。从NCBI(https://www.ncbi.nlm.nih.gov)和BOLD(http://www.boldsystems.org)下载长江鱼类的CO I基因序列(约3000条),构建自建数据库用于后续物种注释,然后深入研究各个样品间的种群结构差异或相似性[28]。本研究基于OTU聚类结果,进行了物种组成、α多样性和β多样性分析。在物种组成分析中,去除非鱼类数据后,筛选出identity值≥97%、E-value≤10-5的鱼类OTU,再将同一物种的OTU进行合并。在Excel中统计各样本中每种鱼的有效序列数,并完善分类学信息,最后通过R语言绘制鱼类组成柱状图。α多样性分析中,选取Chao1指数、Shannon-Wiener指数、Simpson指数及覆盖度指数,分别反映群落丰富度、群落多样性及群落覆盖度。β多样性分析基于Bray-Curtis距离矩阵进行PCoA分析,探索样本间的群落组成差异性或相似性。
2 结 果
2.1 PCR扩增及测序
对所有样本进行PCR扩增,电泳结果显示条带清晰,空白对照未出现目的条带(图2)。将18个样本进行高通量测序,共得到10 847 274条有效序列,各样本有效序列数为502 363~741 454条,平均长度为314 bp。
2.2 OTU聚类和物种注释
按序列相似性≥97%聚类,6个断面共获得33 567个代表性OTU序列。将获得的OTU在自建数据库中进行比对。
2.3 鱼类物种组成
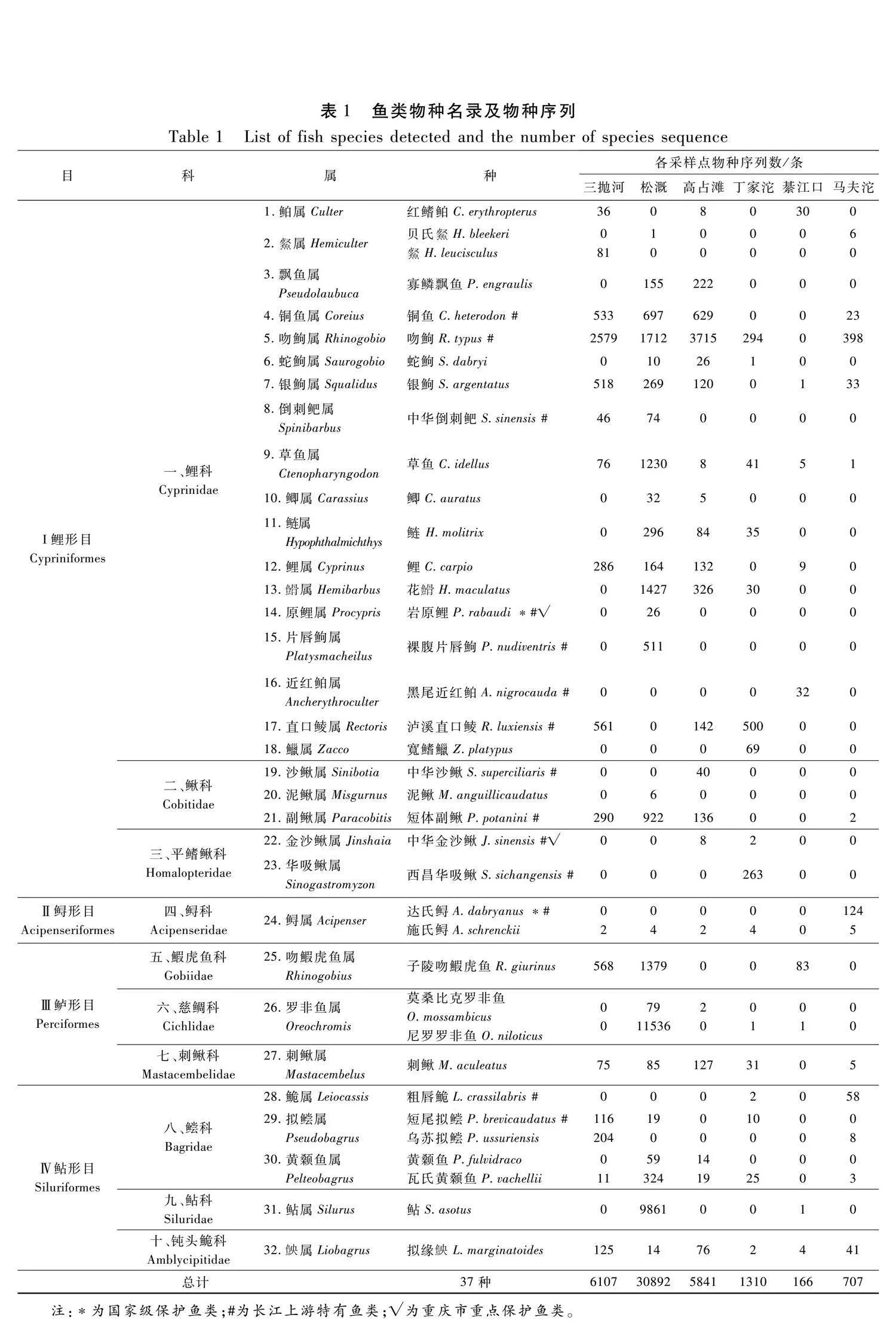
本研究基于环境DNA技术共检测出鱼类37种,隶属于4目10科32属(表1),其中包括达氏鲟(Acipenser dabryanus)和岩原鲤(Procypris rabaudi)2种国家级保护鱼类,14种长江上游特有鱼类,2种重庆市重点保护鱼类。所有鱼类的分类里,以鲤形目(Cypriniformes)的数量最为丰富,共有24种,占总数的64.5%;其次是鲇形目(Siluriformes),有7种,占18.9%;鲈形目(Perciformes)有4种,占10.8%。从科的角度来看,鲤科(Cyprinidae)鱼类最多,达19种,占51.3%;其次是鲿科(Bagridae),共6种,占16.2%;鳅科(Cobitidae)仅有3种,占8.1%;其他各个科的物种数量都相对较低(1~2种)。另外,本次调查检测出3种外来鱼类,分别是尼罗罗非鱼(O.niloticus)、莫桑比克罗非鱼(O.mossambicu)和施氏鲟(A.schrenckii)。
此外,通过表1中的物种序列数数据可知,本次调查从6个断面共获得已知物种序列45 023条,其中綦江口获得的已知物种序列数最少,为166条;而松溉获得的已知物种序列数最多,为30 892条。草鱼(Ctenopharyngodon idellus)和拟缘(Liobagrus marginatoides)在各个断面均被检测到,而其余35种鱼类仅在个别断面中被检测到。
各断面相对序列丰度的分析结果显示(图3),在属水平上,不同断面的鱼类组成结构呈现出一定的差异,其中高占滩、马夫沱和三抛河断面均为吻鮈属(Rhinogobio)相对序列丰度最高,而綦江口、松溉和丁家沱分别以吻鰕虎鱼属(Rhinogobius)、罗非鱼属(Oreochromis)和直口鲮属(Rectoris)的相对序列丰度最高。种水平上,吻鮈(Rhinogobio typus)、子陵吻鰕虎鱼(Rhinogobius giurinus)和铜鱼(Coreius heterodon)在整个研究江段呈现出较高的物种序列丰度。
2.4 鱼类多样性分析
α多样性分析结果显示,Chao1指数范围为13~65,Simpson指数范围为0.1986~0.4201,Shannon-Wiener指数范围为1.49~2.14(表2)。各断面鱼类物种α多样性指数存在一定差异,三抛河和松溉断面具有较高的Chao1指数和Shannon指数,而高占滩断面具有较高的Simpson指数。此外,各样本的覆盖度在0.981 9~1.000 0,表明测序基本上涵盖了所有的OTU数据。
基于序列丰度的PCoA分析表明,各断面间鱼类群落结构呈现一定差异性(图4)。其中高占滩和三抛河、马夫沱和丁家沱分别具有相似的鱼类多样性,而綦江口和松溉具有明显不同于其他断面的鱼类组成。
3 讨 论
3.1 基于环境DNA技术的长江江津段鱼类组成
基于文献资料整理,近十年来在江津段通过刺网、撒网等传统方法共调查到鱼类103种[3-4,15-17],其中4种国家级保护鱼类,21种长江上游特有鱼类,5种重庆市重点保护鱼类。从鱼类种类数上看,鲤科鱼类最多,共计56种,占鱼类总数的54.4%;从优势种群上看,铜鱼属(Coreius)和吻鮈属为该江段的优势属;铜鱼、圆口铜鱼(Coreius guichenonti)和长鳍吻鮈(Rhinogobio ventralis)为该江段的优势种[28]。
本研究运用环境DNA技术在长江江津段共检测出鱼类37种,因为本次环境DNA技术使用的是CO I引物,该引物的长度较长,但环境中游离的DNA大部分的片段很短,进而导致了检出率偏低[29]。同时,根据该分子标记自身特点设计的通用引物难以覆盖比较全面的物种。而且,这些引物具有一定的选择性,可能导致对特定优势种的过度扩增,从而使一些低丰度或非首选种难以被检测到[30]。与近十年来通过传统方法监测的结果相比,
优势种群相似,均显示出该江段吻鮈属为优势属。但在鱼类组成上具有一定差异,历史文献表明该江段铜鱼、圆口铜鱼和长鳍吻鮈为优势种;而本次调查中以吻鮈、子陵吻鰕虎鱼和铜鱼的相对序列丰度最高。这可能是由于文献资料调研时间跨度较长(7~10年),鱼类资源在此期间不断发生变化[3-4];也可能与调查季节有关,单季节调查无法全面反映整个江段的真实情况。
此外,本次调查检测出尼罗罗非鱼、莫桑比克罗非鱼和施氏鲟3种外来鱼类,其中尼罗罗非鱼具有较高的物种序列丰度。这可能与保护区周边养殖场缺乏必要的物种防逃设施有关[26]。尼罗罗非鱼适应能力强,生长迅速,繁殖力强,有强烈的领域和护幼行为[31],可能对保护区土著鱼类资源造成一定的生态胁迫。因此,有必要针对保护区流域的外来鱼类物种开展连续性监测以掌握其种群动态和对本地土著鱼类的影响[21]。
3.2 基于环境DNA技术的长江江津段鱼类多样性
本研究发现,长江江津段不同断面间鱼类多样性和群落结构呈现出一定的差异。綦江口断面Chao1指数值明显低于其他断面,表明其群落丰富度最低,这可能与该断面的地理环境有密切关系。吴昀晟等[32]研究表明,环境DNA得率受到水样泥沙含量和抽提操作等多种因素影响。石睿杰等[33]的相关研究表明,河流中氮质量浓度的变化可能对鱼类获取营养物质产生影响,进而对鱼类群落结构产生影响。本研究中綦江口处于长江干流与綦江的交汇口,泥沙含量较高,水样较为浑浊,且支流上方有城镇污水排入,这些自然和人为因素可能导致綦江口的群落丰富度相对较低[34-36]。其他断面的Chao1指数较为均衡,表明长江江津段鱼类群落结构较为稳定[37]。Simpson指数与Shannon-Wiener指数表明,三抛河和松溉鱼类多样性较高,这可能与样品中的鲇(Silurus asotus)、尼罗罗非鱼等个别鱼类的高序列丰富度相关。β多样性分析表明,高占滩和三抛河及马夫沱和丁家沱具有相似的鱼类多样性,而綦江口具有明显不同于其他断面的鱼类组成。这可能是因为綦江口受到自然因素和人为因素的影响,其检测出的鱼类种数明显低于其他样点。整体而言,长江江津段的各个采样区域物种分布较为聚集,这或许与采样区域之间距离较近,鱼类能够频繁进行交流有关。
3.3 环境DNA技术检测鱼类种类的适用性及局限性
已有研究表明,环境DNA技术检测到的鱼类组成和多样性与传统调查方法的结果基本一致[38-39]。与最近的传统方法调查结果相比(2018年,25种)[4],本研究检测出的鱼类虽未覆盖该江段所有鱼类种类,但检测到的鱼类组成和群落结构存在一定的相似性,表明环境DNA技术作为一种重要的补充工具在鱼类资源调查中具有一定的适用性。此外,本次调查还检测到了传统采样方法难以发现的物种,例如岩原鲤和达氏鲟等珍稀特有鱼类,这证实了环境DNA宏条形码技术应用于水生态监测的可行性。
由于环境DNA的产生和降解受到水动力条件、紫外线、水温、pH和水域底质等多种因素的影响[40-41],因而监测结果与实际情况可能存在一定的误差。目前对环境DNA序列丰度的评价方法仍需深入探讨以确定其与物种数量及生物量的关系等方面的内容。所以,有必要继续探索可能影响环境DNA生成速度和分解效率的外部环境变量,以便为其作为生物量估算等任务的基础理论提供更全面的支持[42]。另外,由于受到样本采集地点、区域规模、检测对象种类以及实验条件的多方面制约,现阶段尚无统一标准化的环境DNA技术操作规范。所以,为了环境DNA技术能更广泛地应用于水生生态监测中,未来需要对环境DNA技术的整个操作流程进行优化,建立规范统一的操作方案与流程。尽管环境DNA方法无法替代常规的户外工作及专业的分类专家所做的事务,然而它具备着高度敏感性的优点并且能以较小的影响来评估生态环境,这有助于填补传统形态学监控手段的缺陷,因而可以被视为一项有效的辅助手段,用以迅速了解长江鱼种的种类数量及其地理分布情况[8,43]。
参考文献:
[1] 董芳,方冬冬,张辉,等.长江十年禁渔后保护与发展[J].水产学报,2023,47(2):245-259.
[2] 左硕,朱峰跃,陈大庆,等.怒江西藏段鱼类群落结构及多样性研究[J].水生态学杂志,2024,45(4):73-81.
[3] 高天珩,田辉伍,叶超,等.长江上游珍稀特有鱼类国家级自然保护区干流段鱼类组成及其多样性[J].淡水渔业,2013,43(2):36-42.
[4] 李祥艳.禁渔初期长江上游保护区干流江安:重庆江段鱼类时空分布特征[D].重庆:西南大学,2023.
[5] 朱宗波,王银平,蔺丹清,等.长江近口靖江段近岸鱼类群落多样性[J].安徽农业大学学报,2021,48(4):614-619.
[6] STAT M,JOHN J,DIBATTISTA J D,et al.Combined use of eDNA metabarcoding and video surveillance for the assessment of fish biodiversity[J].Conservation Biology,2019,33(1):e13183.
[7] 郁斯贻.环境DNA技术在水生生物监测中的应用研究[J].科技视界,2019(22):78-79.
[8] DEINER K,WALSER J C,ALTERMATT F,et al.Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA[J].Biological Conservation,2015,183:53-63.
[9] FRANCESCO F G ,CLAUDE M,TABERLET P,et al.Species detection using environmental DNA from water samples[J].Biology Letters,2008,4(4):423-425.
[10]赵梦迪.利用环境DNA分析冬季中国东黄海水域的鱼类多样性[D].上海:上海海洋大学,2017.
[11]MIYA M,SATO Y,FUKUNAGA T,et al.MiFish,a set of universal PCR primers for metabarcoding environmental DNA from fishes:detection of more than 230 subtropical marine species[J].Royal Society Open Science,2015,2(7):e150088.
[12]徐念,常剑波.长江中下游干流环境DNA样本鱼类物种检测的初步研究[J].水生态学杂志,2016,37(5):49-55.
[13]GIBSON J F,SHADI S,COLIN C,et al.Large-scale biomonitoring of remote and threatened ecosystems via high-throughput sequencing[J].PLoS ONE,2015,10(10):e0138432.
[14]ANDRUSZKIEWICZ E A,STARKS H A,CHAVEZ F P,et al.Biomonitoring of marine vertebrates in Monterey Bay using eDNA metabarcoding[J].PLoS ONE,2017,12(4):e0176343.
[15]张钰,练小龙,李英文,等.重庆江津复兴河流域浮游生物群落结构、水质及鱼类资源现状[J].重庆师范大学学报(自然科学版),2021,38(4):36-47.
[16]杨志,唐会元,万力,等.三峡库区上游江津江段鱼类群落结构的年际变化[J].生态学杂志,2014,33(6):1565-1572.
[17]田辉伍,何春,刘明典,等.长江上游干流三层流刺网渔获物结构研究[J].淡水渔业,2016,46(5):3742.
[18]危起伟.长江上游珍稀特有鱼类国家级自然保护区科学考察报告[M].北京:科学出版社,2012:208-214.
[19]熊飞,刘红艳,段辛斌,等.长江上游江津江段鱼类群落结构及资源利用[J].安徽大学学报(自然科学版),2014,38(3):94-102.
[20]THOMSEN P F,WILLERSLEV E.Environmental DNA:an emerging tool in conservation for monitoring past and present biodiversity[J].Biological Conservation,2015,183:4-18.
[21]刘波,王浩,秦斌,等.基于环境DNA宏条形码技术的北京地区鱼类多样性调查和外来鱼种入侵风险评估[J].生物安全学报,2021,30(3):220-229.
[22]陈云川.环境DNA技术在六冲河上游鱼类资源调查中的应用研究[D].重庆:西南大学,2020.
[23]LIM N,TAY Y C,SRIVATHSAN A,et al.Next-generation freshwater bioassessment:eDNA metabarcoding with a conserved metazoan primer reveals species-rich and reservoir-specific communities[J].Royal Society Open Science,2016,3(11):160635.
[24]程如丽,罗杨,张玉凤,等.基于环境DNA技术的乌江干流梯级水电库区的鱼类多样性[J/OL].水产学报:1-19(2024-04-13).http://kns.cnki.net/kcms/detail/31.1283.S.20230412.1705.006.html.
[25]舒璐,林佳艳,徐源,等.基于环境DNA宏条形码的洱海鱼类多样性研究[J].水生生物学报,2020,44(5):1080-1086.
[26]巴家文,陈大庆.三峡库区的入侵鱼类及库区蓄水对外来鱼类入侵的影响初探[J].湖泊科学,2012,24(2):185-189.
[27]丁瑞华.四川鱼类志[M].成都:四川科学技术出版社,1994.
[28]王梦,杨鑫,王维,等.基于eDNA技术的长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性研究[J].水生生物学报,2022,46(1):2-16.
[29]DEAGLE B E,JARMAN S N,COISSAC E,et al.DNA metabarcoding and the cytochrome c oxidase subunit I marker:not a perfect match[J].Biology Letters,2014,10(9):20140562.
[30]ELBRECHT V,VAMOS E E,MEISSNER K,et al.Assessing strengths and weaknesses of DNA metabarcoding-based macroinvertebrate identification for routine stream monitoring[J].Methods in Ecology & Evolution,2017,8(10):1265-1275.
[31]岑忠龙,侯秀发,周江.贵州省鱼类外来种调查[J].贵州师范大学学报(自然科学版),2013,31(3):16-19.
[32]吴昀晟,唐永凯,李建林,等.环境DNA在长江江豚监测中的应用[J].中国水产科学,2019,26(1):124-132.
[33]石睿杰,唐莉华,高广东,等.长江流域鱼类多样性与流域特性关系分析[J].清华大学学报(自然科学版),2018,58(7):650-657.
[34]ERICKSON R A,MERKES C M,JACKSON C A,et al.Seasonal trends in eDNA detection and occupancy of bigheaded carps[J].Journal of Great Lakes Research,2017,43(4):762-770.
[35]刘军,曹文宣,常剑波.长江上游主要河流鱼类多样性与流域特征关系[J].吉首大学学报(自然科学版),2004,25(1):42-47.
[36]郑从奇,武玮,魏杰,等.黄河下游支流大汶河鱼类多样性及影响因子分析[J].水资源保护,2020,36(6):31-38,52.
[37]唐晟凯,钱胜峰,沈冬冬,等.应用环境DNA技术对邵伯湖浮游动物物种检测的初步研究[J].水产养殖,2021,42(3):13-20.
[38]凌建忠,姜亚洲,孙鹏,等.环境DNA技术在象山港水域鱼类多样性调查中的应用与评估[J].中国水产科学,2021,28(2):205-214.
[39]VALENTINI A,TABERLET P,MIAUD C,et al.Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding[J].Molecular Ecology,2016,25(4):929-942.
[40]BARNES M A,TURNER C R.The ecology of environmental DNA and implications for conservation genetics[J].Conservation Genetics,2015,17(1):1-17.
[41]DAVID S P,CAREN S G,ROBERT S A,et al.Factors influencing detection of eDNA from a stream-dwelling amphibian[J].Molecular Ecology Resources,2014,14(1):109-116.
[42]赵彦伟,陈家琪,董丽,等.环境DNA技术在水生态领域应用研究进展[J].农业环境科学学报,2021,40(10):2057-2065.
[43]REES H C,MADDISON B C,MIDDLEDITCH D J,et al.Review:the detection of aquatic animal species using environmental DNA-a review of eDNA as a survey tool in ecology[J].Journal of Applied Ecology,2014,51(5):1450-1459.
Application of Environmental DNA Technologyin Monitoring Fish Diversity in the Jiangjin Section of the Yangtze River
ZHANG Jia-ming1,ZHOU Xin-xin1,WANG Wei2,DUAN Cong2,
LI Ying-wen1,SHEN Yan-jun1
(1.School of Life Sciences,Chongqing Normal University,Chongqing 401331,China;
2.Chongqing Rare and Unique Fish National Nature Reserve Management Office,Chongqing 402260,China)
Abstract:Utilizing environmental DNA technology to investigate fish species distribution in the Jiangjin section of the Yangtze River provides essential fundamental data for effective river management.In June 2021,6 sampling sites were strategically positioned along this segment,employing a comprehensive approach that involved water sample collection,eDNA capture,and annotation analysis to assess fish diversity.The study identified a total of 37 fish species in the Jiangjin section,representing 4 orders,10 families,and 32 genera.Notably,the findings included 2 nationally protected fish species,14 endemic species in the upper reaches of the Yangtze River,2 key protected species in Chongqing Municipality,and 3 invasive species.Rhinogobio typus,R.giurinus,and Coreius heterodon exhibited notably higher relative abundances in this region.Furthermore,Alpha and Beta diversity analyses,based on sequence abundance,presented variations in fish composition and diversity at different sampling sites.Compared with recent traditional morphological survey data,a remarkable resemblance can be found between eDNA technology’s detection results and the traditional findings for fish species.This underscores the efficacy of eDNA technology as a valuable supplementary tool for rapidly comprehending the amounts and geographical distribution of fish species in the Yangtze River.
Keywords:environmental DNA;fish diversity;fish monitoring;distribution of fish resources;Jiangjin section of the Yangtze River

