5种山茶属植物PYL基因密码子偏好性分析





摘 要 为了更深入地了解PYL基因的遗传和进化特征,本研究针对5种山茶属植物的PYL基因密码子使用模式进行分析。基于公布的浙江红山茶、狭叶油茶、‘云抗10号’、野生大树茶、‘铁观音’基因组数据共鉴定出55个PYL基因。系统发育结果显示,PYL基因可分为4类。共线性分析表明,PYL基因在进化过程中受到的选择压力较小,主要受到纯化选择的作用。密码子偏好指数和同义密码子使用偏差分析, 59个同义密码子的使用存在显著差异。PYL基因偏好使用GCC、ATC、CTC、ACC、GTC等以G/C结尾编码疏水性氨基酸的密码子。通过ENC plot、PR2-plot、GC3-GC12分析表明,自然选择在PYL基因进化中发挥了重要作用。在异源表达时,PYL基因密码子使用频率相似性高的受体为最佳受体。本研究揭示PYL基因在山茶属植物中的密码子使用特点和进化关系,为深入理解其功能及适应性机制提供了参考依据。
关键词 山茶属;PYL基因;系统发育分析;共线性分析;密码子偏好性
山茶属植物,作为常绿灌木或乔木,主要分布于热带和亚热带地区的山地与丘陵地带,因品种多样性与显著的经济价值而受到广泛关注[1-2]。茶树、油茶和茶花等均为该属中的代表性物种。其中,茶叶含有丰富的茶多酚、咖啡碱及茶氨酸等活性成分,这些物质具有抗氧化、促进认知功能及脂肪代谢等重要作用[3-4]。油茶种子则是茶油的主要来源,因其丰富的不饱和脂肪酸和独特的健康功效被誉为“东方橄榄油”[5]。山茶花则因其丰富的花色和持久的花期,在园艺、景观设计和城市绿化等领域展现出较高的观赏价值。近年来,随着基因组学技术的飞速发展,山茶属植物的基因组研究取得了显著进展。目前,茶树、油茶和山茶等多个物种的基因组测序与组装工作已经完成,这为后续基因功能研究和遗传育种提供了坚实的物质基础[6-8]。
PYL基因家族在植物应对环境胁迫方面发挥着至关重要的作用。该基因编码的蛋白是脱落酸(ABA) 的受体,参与ABA介导的信号转导途径[9-10]。通过调控PYL-PP2C-SnRK2途径,植物能够感知ABA、传递信号以及表达应答基因,从而应对如干旱和盐碱等非生物胁迫[11]。PYL基因家族成员数量在不同植物中有所不同,从几个到几十个不等。例如,拟南芥有14个成员,鹅掌楸5个[12]、大白菜25个[13]。这些不同的基因成员在功能上出现分化,发挥不同的作用。研究表明,过表达OsPYL/RCAR7基因的玉米,通过降低水分流失和调整气孔导度来提高其在干旱环境中的生存率[14]。胡杨树中PePYL4受ABA、渗透和脱水处理诱导,且过表达PePYL4显著增加了ABA敏感性,减小了气孔孔径[15]。研究发现过表达VaPYL9基因可与VaPCMT互作增强转基因番茄的抗寒能力,过表达VaPYL9的番茄植物在冷应激条件下表现出较高的抗氧化酶活性以及增强清除活性氧的能力[16]。这些研究结果表明,PYL基因能够增强植物应对非生物胁迫的耐受力。
在分子生物学中,密码子是生物体遗传信息传递的关键组成部分,具有简并性。不同的生物体或物种在利用这些密码子时表现出一定的偏好性,密码子偏好性是由多种因素决定的,包括生物体在进化过程中基因突变及自然选择等[17-18]。研究发现,Usutu病毒的基因组构成对其密码子偏好性具有显著影响,导致其蛋白质编码序列更倾向于使用G/C末端密码子[19]。此外,病毒与宿主之间的密码子使用呈现出一种复杂的关系,既存在一致性,又存在拮抗性。病毒的密码子使用偏好可能受到多个宿主的共同影响,宿主对病毒密码子的选择和使用偏好起到重要作用[20]。这种偏好性不仅影响生物体的蛋白质合成,还在许多其他生命活动中起到关键作用,包括基因表达、蛋白质相互作用和进化等[21-22]。通过向毕赤酵母中引入额外的稀有tRNA基因拷贝,成功调整了其密码子使用偏好,进而影响了mRNA的表达水平,提高了目标蛋白的产量[23]。通过研究密码子偏好性,可以更深入地了解生物体的遗传特性和表达模式,提升目的基因的表达水平和稳定性,为基因功能和进化研究提供重要参考。
1 材料与方法
1.1 基因筛选
从茶树信息档案库(TPIA) 获取浙江红山茶(Camellia chekiangoleosa) 、狭叶油茶(Camellia lanceoleosa)、‘云抗10号’茶树(Camellia sinensis ‘Yunkang10’)、野生大树茶(Camellia sinensis ‘DASZ’)、‘铁观音’茶树(Camellia sinensis ‘Tieguanyin’) 的基因组数据。从拟南芥信息资源库(TAIR) 获取拟南芥(Arabidopsis thaliana) PYL基因序列。通过BioEdit工具构建这5个山茶属植物的BLAST数据库,利用拟南芥PYL基因作为查询序列,期望值设置为1.0×10-10,搜索出同源的PYL序列。通过序列比对去除重复的基因,通过Pfam分析去除不含PF10604结构域的基因。
1.2 序列分析
用ProtParam工具分析PYL基因序列的理化性质,包括分子质量、等电点、不稳定指数、亲水性。用codonW软件计算同义密码子第3位的T3s、C3s、A3s、G3s、GC3s含量及同义密码子相对使用度、密码子适应指数、密码子偏爱指数、有效密码子数。用EMBOSS网站的CUSP程序计算GC1、GC2、GC3含量。
1.3 系统发育关系与共线性分析分析
使用MEGA6软件,采用Neighbor-Joining算法,设置Bootstrap为1 000,构建拟南芥与这5种山茶属植物PYL基因的系统发育树。导出进化树文件后,用iTOL在线网站做进一步地修饰。
使用TBtools生物软件分别比较铁观音茶树、浙江红山茶、狭叶油茶PYL基因在基因组内与不同基因组之间的进化关系并进行可视化。使用TBtools中的Simple Ka/Ks Calculator(NG)计算基因对的Ks值和Ka/Ks比值,利用R脚本绘制Ks密度分布图,根据Ks峰值粗略估计分化时间(T = Ks/2λ,λ =6.5×10-9)。
1.4 同义密码子相对使用度(RSCU)分析
RSCU是基因样本中某个密码子使用频率与其同义密码子预期使用频率之间的比值,能够反映基因样本中密码子的使用状况[24]。如果RSCU值为1,则说明该氨基酸的密码子使用没有偏好,其选择是均等或随机的;如果RSCU值大于1,则表明其使用频率相对较高,反之亦然。PYL基因密码子RSCU值堆积图由Origin软件制作。
1.5 有效密码子数(ENC)和ENC plot分析
ENC的取值为20~61,这个数值用于衡量同义密码子的偏好程度。通过以ENC为纵坐标,GC3s为横坐标用R脚本绘制ENC-GC3s图,并生成ENC预期曲线,分析除突变外的其他因素是否参与了密码子使用模式的形成。
1.6 奇偶性(PR2)分析
为了研究随机突变和选择对基因密码子使用的影响,选择具有4个简并同义密码子的氨基酸来计算其第3个密码子的ATGC组成,并以A3/(A3 + T3)为纵坐标,G3/(G3 + C3)为横坐标用R脚本绘制PR2图[25-26]。
1.7 中性图(GC3-G12)分析
用GC3表示密码子第3位的GC含量,GC12表示密码子第1位和第2位GC含量的平均值。以GC12为纵坐标,GC3为横坐标用R脚本作图,并生成回归线。通过二者的线性关系分析突变和自然选择对密码子偏好性的影响。
1.8 密码子使用频率比较分析
从Codon Usage Database中获得拟南芥、烟草(Nicotiana tabacum)、毛白杨(Populus trichocarpa)、大肠杆菌(Escherichia coli)、酵母菌(Saccharomyces cerevisiae) 这5个物种的密码子使用频率。用TBtools中的HeatMap绘制密码子使用频率比值热图。通过比较PYL基因与这些物种之间的密码子使用频率差异,来分析其在特定物种中的最佳表达受体。
2 结果与分析
2.1 5种山茶属植物PYL基因基本性质及密码子参数分析
经过对5种山茶属植物基因组的筛选,获得了60个PYL同源序列。为确保数据的准确性和可靠性,进一步进行了序列比对,以排除潜在的重复序列。此外,经过保守结构域的筛选,最终鉴定出55个具有PF10604结构域的PYL基因(表1) 。在5种山茶属植物中,浙江红山茶共鉴定出13个PYL基因,狭叶油茶有12个,‘云抗10号’茶树有10个,野生大树茶有8个,‘铁观音’茶树鉴定出12个。55个PYL基因编码区的长度呈现出一定的变化范围,从345 bp到735 bp不等,平均长度为587 bp。根据氨基酸序列的预测,PYL蛋白的分子质量为13 018.90 ~27 621.78 u,表明PYL蛋白在分子质量上也存在较大的差异。此外,PYL蛋白的等电点为4.96~9.58,显示出其电荷特性的多样性。其中,等电点大于7的基因有18个,而等电点小于7的基因有37个。PYL蛋白的不稳定指数为32.06~53.28。这一指数反映了蛋白质序列中氨基酸残基的不稳定性,较高的不稳定指数可能意味着蛋白质在细胞内的稳定性较低。密码子适应指数(CAI) 为 0.149~0.288,表明PYL基因在密码子使用上存在一定的偏好性。较低的CAI值可能意味着这些基因在表达水平低。密码子偏爱指数(CBI) 的值为-0.182~0.266,负值表明这些PYL基因密码子偏好性较弱。PYL基因的有效密码子数(ENC) 在40.29~56.68,平均为49.63。这一数值反映了基因在编码过程中使用的密码子种类和频率,而较高的ENC值则可能表示基因在密码子使用上更为均衡。PYL蛋白的亲水性为负值,表明这些蛋白具有疏水性特性。
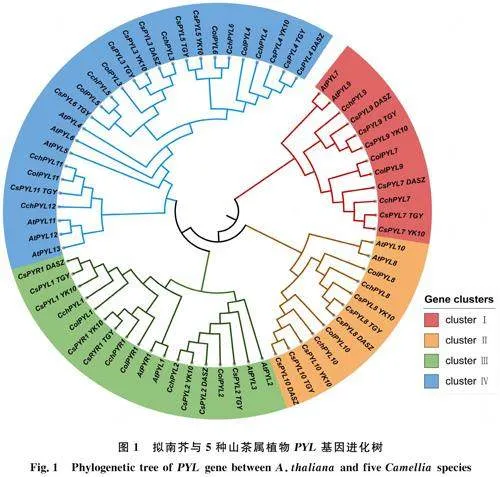
2.2 5种山茶属植物PYL基因系统发育分析
基于拟南芥、浙江红山茶、狭叶油茶、‘云抗10号’、野生大树茶、‘铁观音’的69个PYL蛋白序列使用邻接(Neighbor-Joining)法构建进化树,揭示了这些蛋白在进化过程中的亲缘关系和分类。结果显示(图1),5种山茶属植物的PYL蛋白可明确分为4类(clusterⅠ至clusterⅣ) ,这表明它们在进化过程中可能经历了不同的选择压力和功能分化。具体而言,clusterⅠ包含10个成员,clusterⅡ和clusterⅢ分别有10个和14个成员,而clusterⅣ则是最大的类群,包含21个成员。这一分布表明PYL蛋白在5种山茶属植物中的多样性和复杂性,其中clusterⅣ尤为显著,可能在该类群中发挥着重要的生物学功能。值得注意的是,进化树的结果显示亲缘关系较近的PYL蛋白序列聚集在同一簇中,表明它们在这些物种中可能具有相似的功能或结构特征。同时,不同cluster中PYL蛋白的数量差异可能与蛋白的冗余性、功能特化或丢失有关,数量较多的cluster(如clusterⅣ) 可能涉及到更多的生物或非生物响应。
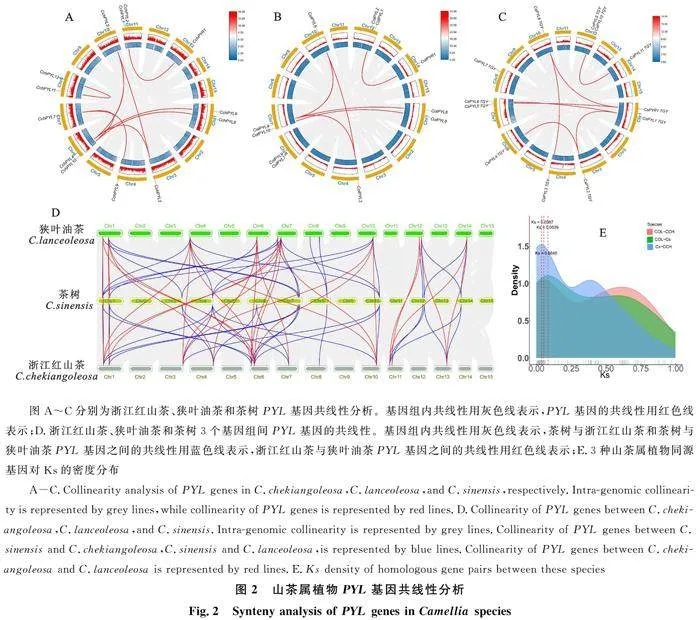
2.3 5种山茶属植物PYL基因共线性与进化 分析
以基因组组装质量较好的‘铁观音’基因组代表茶树基因组。如图2-A~2-C所示,在浙江红山茶、狭叶油茶和茶树基因组内分别有8、6和7对PYL旁系同源基因。说明PYL基因在山茶属物种中经历了不同程度的基因复制事件。3个山茶属物种的Ka/Ks均值在0.24左右,表示非同义突变的速率略高于同义突变的速率。因此可以推断,PYL基因在山茶属3个物种的进化过程中受到了纯化选择的作用。为了探究PYL基因在山茶属物种之间的进化关系,构建了浙江红山茶、茶树与油茶的共线性关系图(图2-D)。结果显示,狭叶油茶与茶树、茶树与浙江红山茶和狭叶油茶与浙江红山茶之间分别有23、24和27对PYL直系同源基因,它们的Ka/Ks均值分别为0.296、0.278和0.301,说明PYL基因在这些山茶属物种之间的进化过程中,主要受到纯化选择的影响。如图2-E所示,狭叶油茶与茶树、茶树与浙江红山茶和狭叶油茶与浙江红山茶之间的PYL直系同源基因的Ks峰值分别为0.084、 0.037和0.054,推测它们在物种中的分化时间分别发生在 6 496 881.54、2 822 213.08和 4 146 095.38年前。
2.4 5种山茶属植物PYL基因密码子RSCU分析[JP]
编码氨基酸的61个密码子中,有59个是同义密码子,意味着它们在不同的生物或基因中可能具有不同的使用频率。在本研究中,5种山茶属植物的PYL基因显示出对不同密码子的偏好性,但它们之间也存在相似的密码子使用模式(图3)。对于浙江红山茶,相对同义密码子使用频率(RSCU) 大于1时,有29个密码子被偏好使用,RSCU大于1.6时,有6个密码子(CTC、ATC、GTC、TCC、ACC、GCC) 被强烈偏好。狭叶油茶在RSCU>1时偏好使用27个密码子,在RSCU>1.6时偏好使用5个密码子(CTC、ATC、GTC、ACC、GCC以及AGG)。‘云抗10号’在RSCU>1时偏好使用28个密码子,在RSCU>1.6时偏好使用6个密码子(CTC、ATC、GTC、TCC、ACC、GCC)。野生大树茶在RSCU>1时偏好使用26个密码子,在RSCU>1.6时偏好使用6个密码子(CTC、ATC、ACC、GCC、AGA、AGG)。‘铁观音’在RSCU>1时偏好使用26个密码子,在RSCU>1.6时偏好使用5个密码子(CTC、ATC、GTC、ACC、GCC)。值得注意的是,在这些被强烈偏好的密码子中,以G/C结尾的密码子尤为显著,包括编码Alanine的GCC、编码Isoleucine的ATC、编码Leucine的CTC、编码Threonine的ACC以及编码Valine的GTC等。这可能与山茶属植物中PYL基因的特定表达模式或功能需求有关,这种偏好性也可能与转录和翻译过程中的效率、准确性或其他因素相关。
2.5 5种山茶属植物PYL基因密码子ENC plot分析
密码子ENC plot分析揭示了山茶属PYL基因在密码子使用偏好性上的重要特征。在5个不同物种中,PYL基因的ENC值在不同基因之间存在显著差异,平均值为49.63,且未发现ENC值低于35的基因,这表明PYL基因的密码子使用偏好性普遍较低(图4)。通过ENC plot分析发现,大多数基因位于弧线以下,其ENC值低于ENC期望值,可能是在进化过程中受到较强自然选择的结果。然而,少数基因如 CchPYL1、 CSPYR1-DASZ、 CSPYL1-TGY和 CSPYL1-YK10的ENC值接近或达到ENC exp曲线, 这可能表明这些特定基因在密码子使用上受到除自然选择之外的其他因素的调控,如突变、重 组等。
2.6 5种山茶属植物PYL基因密码子PR2分析
在无特定密码子偏好的情况下,碱基A/T与G/C的频率通常保持均衡状态。然而,PR2-plot分析揭示,数据点主要集中于G3 /(G3 + C3)< 0.5和A3 /(A3 + T3) < 0.5的特定区域。具体而言,G3 /(G3 + C3)的值多数位于0.21至0.58的范围内,而A3 /(A3 + T3)的值则主要分布在0.26至0.54的区间内(图5)。这说明5个山茶属物种在第3密码子的使用上存在明显的不平衡性,表现为T在第3位密码子中的出现频率高于A,而C的频率则高于G。尽管总体趋势如此,但仍存在个别基因的特例。例如,PYL2基因中碱基A的使用频率超过T,而在PYL8密码子中,第3个位置的碱基G的频率则高于C。这种碱基使用的不平衡性可能反映了自然选择在物种进化中的主导作用,同时也不能排除突变等其他潜在影响因素的存在。
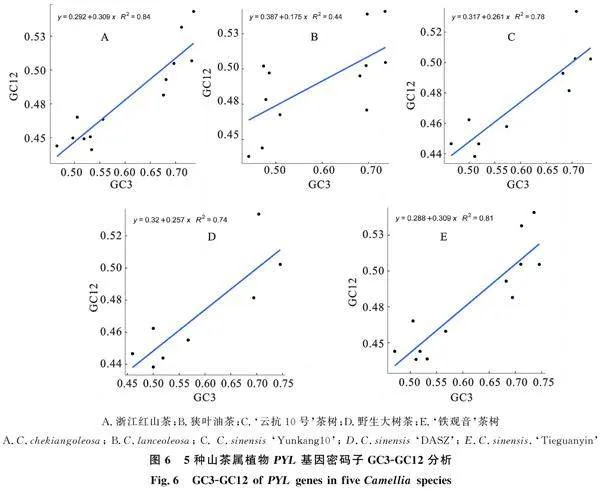
2.7 5种山茶属植物PYL基因密码子GC3-GC12分析
通过中性图分析,探讨5种山茶属植物PYL基因中GC12和GC3的关系(图6)。分析表明,GC12的含量为43.79%~54.33%,而GC3的含量则为44.14%~74.55%。这些山茶属植物PYL基因的GC12与GC3之间存在正相关关系,R2值为0.44~0.84,同时回归斜率也在 0.175~0.309。这说明在山茶属植物的密码子使用偏好中,自然选择和突变可能共同发挥作用,但自然选择的影响更为显著。
2.8 5种山茶属植物PYL基因密码子使用频率分析
对浙江红山茶、狭叶油茶、‘云抗10号’、野生大树茶以及‘铁观音’5种山茶属植物的PYL基因密码子使用频率与拟南芥、烟草、毛白杨、大肠杆菌、酵母菌进行了对比分析(图7)。结果表明,与这些常用表达受体物种相比,各山茶属植物PYL基因密码子使用模式存在差异。具体而言,浙江红山茶PYL基因密码子使用频率与参考物种的比值差异≥2.0或≤0.5的密码子分别有19、21、20、23、23个;狭叶油茶分别为21、24、19、24、23个;‘云抗10号’分别为21、25、26、24、23个;野生大树茶分别为20、22、19、24、21个;‘铁观音’分别为22、24、26、22、24个。尽管5种山茶属植物PYL基因密码子使用频率存在一定差异,但仍存在相似性。其中,差异较大的密码子主要包括GCA、CAC、CTT、CCC、CGT、CGC、CGA、CGG、AGA、ACC和GTC。这些密码子的高频或低频使用可能与山茶属植物特定的遗传特征和生理机制有关。在异源表达时,同一物种可根据密码子使用频率相似性选择最佳受体。浙江红山茶和拟南芥虽然属于不同的植物类群,但PYL基因在密码子使用模式上表现出更高的相似性,可能意味着在该表达受体转录和翻译过程中具有更高的效率[27]。当选择密码子使用频率差异较大的作为受体时,可通过对特定密码子进行优化改造,如将编码Gly的GGG改造为GGA或GGC,将编码Val的密码子改造为GTT等,以提高异源表达效率。
3 讨 论
5种山茶属植物在PYL基因家族成员数量上存在差异,其中浙江红山茶最为丰富,拥有13个成员,而野生大树茶则较为稀少,仅有8个成员。基因组的组装水平对PYL基因鉴定的数量有直接影响。相比之下,如果使用了如3代测序及Hi-C组装技术,基因组组装的质量会相对较高,这有助于更准确地鉴定PYL基因[28]。另外,在基因组注释过程中,如果设定的阈值过滤标准较低,那么基因组整体的基因数量可能会偏多。尽管山茶属植物PYL基因家族成员数量有所不同,但这种差异相对较小,可能反映了这些植物在适应不同生态环境和进化路径上的高度保守性。在遗传学中,Ka/Ks值是用来衡量非同义突变(Ka)和同义突变(Ks)的比例,它可以用来评估基因在进化过程中的选择压力[29-30]。本研究中3个山茶属物种PYL旁系同源基因和直系同源基因的Ka/Ks都小于1,这意味着PYL基因在山茶属物种间的进化过程中主要受到纯化选择的作用,保留了重要的生物学功能,且有害的非同义突变被自然选择所淘汰。旁系同源基因也反映了基因复制事件的存在为这些基因可能发生功能分化和新功能化提供了潜力。此外,3个山茶属物种的PYL基因进化过程中面临的进化压力较小且功能相对保守。山茶属最近一次的WGD事件发生在约5 748万年前[31],本研究中3个山茶属植物的PYL直系同源基因的分化时间较晚,在这次WGD事件发生之后,可能通过串联复制、逆转录等机制增加PYL基因的多样性[32]。PYL基因通常与植物的ABA信号转导相关,这是植物应对逆境(如干旱、高盐等)的一种重要机制。密码子偏好性主要反映了基因在转录和翻译过程中的使用偏好,某些密码子可能由于与tRNA的丰度匹配得更好,而被更频繁地使用,从而提高蛋白质的合成效率[33]。在山茶属植物中,如果PYL基因表现出特定的密码子偏好性,这可能意味着该基因在特定的生理或环境条件下有更高效的表达[34]。如果密码子偏好性影响了PYL基因的表达水平或调控模式,那么这可能会进一步影响山茶属植物对逆境的响应和适应能力。
本研究对5种山茶属植物的PYL基因进行RSCU分析,探讨其密码子偏好性及其在不同物种间的变化。结果揭示山茶属植物PYL基因在密码子使用模式上具有一定的保守性,多数偏好使用的密码子以G/C结尾,如GCC、ATC、CTC、ACC、GTC等。这与乌头属植物中的研究结果相契合,暗示了亲缘关系相近的物种在密码子使用模式上可能存在一定的相似性[35]。然而,在同一物种的不同基因间,密码子使用偏好性呈现出差异性,这种差异可能与基因的结构、转录和翻译机制等因素密切相关[36-37]。例如,茶树NRT1.1基因与PYL基因在密码子使用偏好性上表现出一定的相似性,但两者在具体偏好程度和密码子种类的选择上有所不同[38]。相比之下,茶树LOX基因的密码子偏好性较弱,偏好使用以A/T结尾的密码子,这可能与LOX基因作为环境诱导型基因的特性有关[39]。此外,先前关于拟南芥、油菜等植物的HSP20基因密码子偏好性的研究结果显示,这些基因的密码子偏好性相对较弱,偏好使用以A/T结尾的密码子,如AGG、CCA、GAA、GAG、GCT、GUG、UCT和UGA等[40]。这些密码子使用模式的差异可能反映了不同基因在进化过程中对环境适应性和基因功能优化的不同策略。
密码子偏好性的形成是一个复杂的过程,受到多种因素的共同影响,其中GC含量是一个关键因素。基因GC含量的差异会导致密码子使用模式的显著变化。以‘云抗10号’为例,其基因组整体的GC含量较低,分别为GC1 22.58%、GC2 40.00%、GC3 40.00%,均低于50%,因此其密码子使用更偏好于以A/T结尾的密码子[41]。然而,在其PYL基因中,GC含量分布却与之不同,具体表现为GC1 54.57%、GC2 40.71%、GC3 60.96%,相对较高。这种GC含量的差异导致了PYL基因更倾向于使用GC结尾的密码子,进而影响了其密码子偏好性。ENC(有效密码子数) 作为一个评估基因或基因组密码子使用随机性程度的指标,其值在山茶属植物的PYL基因中普遍较高。这一现象不仅在本研究的山茶属植物中发现,类似的高ENC值也在茶树NRT1.1、LOX基因[38-39],以及茄科植物番茄、辣椒的PIF基因[42]中观察到。这种较高的ENC值反映了这些基因的密码子使用偏好性相对较弱,可能与不同物种间的保守性及基因表达紧密关联[21,43]。然而,其具体的分子机制目前仍不清楚。为了深入理解密码子偏好性的形成因素,结合ENC与GC3的作图分析揭示了山茶属PYL基因在密码子使用上可能受到的自然选择、突变或其他因素的影响。PR2-plot分析和GC3-GC12分析则进一步证实了自然选择和突变在山茶属植物密码子使用偏好中的共同作用。尽管这种现象在藏红花、十字花科植物中均有报道[40,44],但具体的影响因素和机制可能因物种而异,仍需要进一步的研究和探索。
同义密码子使用频率与表达受体使用频率呈现出差异性,这种差异对基因表达水平产生显著影响。以大肠杆菌为例,其同义密码子间的翻译效率差异可高达6倍,这种差异很可能与首选密码子与同源tRNA的丰度有关[45-46]。同时,异源表达基因与最优受体物种通常展现出相似的密码子偏好,这种偏好在选择表达受体物种时具有重要作用[22,27]。因此,山茶属PYL基因可通过密码子使用频率来选择最优表达受体,或对靶基因中的罕见密码子进行修改,从而优化翻译系统并提高基因表达效率。需要指出的是,密码子优化并非简单的密码子替换过程。它需要考虑多种因素,如宿主细胞的密码子使用频率、mRNA的稳定性、启动子序列、蛋白折叠和修饰等[47]。如果其他环节存在问题,那么整体的异源表达效率仍然可能受到限制。密码子优化在实际应用中还需要考虑到成本、时间等因素。如果优化策略过于复杂或成本过高,那么其实际应用就会受到限制。为了更准确地评估和优化异源表达效率,需要针对具体宿主进行深入的试验研究和数据分析。
4 结 论
本研究对5种山茶属植物的55个PYL基因进行了系统鉴定,结果显示PYL基因在这些物种中具有高度保守性,且进化过程中主要受到纯化选择的作用。通过进一步的密码子使用分析,发现PYL基因偏好使用编码疏水性氨基酸的以G/C结尾的密码子,这种偏好可能受到自然选择的影响更为显著。此外,不同物种PYL基因在异源表达过程中可能具有不同的最佳受体。这些结果对于深入理解山茶属植物PYL基因的功能及其进化历程具有重要意义。
参考文献 Reference:
[1] 廖初琴,缪绅裕.山茶属植物传统及现代分类的研究进展[J].热带农业科学,2023,43(7):37-45.
LIAO CH Q,MIAO SH Y.Advances in the traditional and modern classification of Camellia[J].Chinese Journal of Tropical Agriculture,2023,43(7):37-45.
[2]闵天禄.山茶属山茶组植物的分类,分化和分布[J].云南植物研究,1998,20(2):127-148.
MIN T L.The classification,differentiation and distribution of the Genus Camellia Sect.Camellia[J].Acta Botanica Yunnanica,1998,20(2):127-148.
[3]TANG G,MENG X,GAN R,et al.Health functions and related molecular mechanisms of tea components:an update review[J].International Journal of Molecular Sciences,2019,20(24):6196.
[4]申 雯,黄建安,李 勤,等.茶叶主要活性成分的保健功能与作用机制研究进展[J].茶叶通讯,2016,43(1):8-13.
SHEN W,HUANG J A,LI Q,et al. The research progress of health care function and mechanism of the active ingredients in tea [J].Journal of Tea Communication,2016, 43(1):8-13.
[5]ZHU M,SHI T,CHEN Y,et al.Prediction of fatty acid composition in Camellia oil by 1H NMR combined with PLS regression[J].Food Chemistry,2019,279:339-346.
[6]GAO Q J,T W,LI F D,et al.TPIA2:an updated tea plant information archive for Camellia genomics[J].Nucleic acids research,2023,52(D1):D1661-D1667.
[7]WEN G,SHI X,LIN W,et al.Chromosome-level genome of Camellia lanceoleosa provides a valuable resource for understanding genome evolution and self-incompatibility[J].The Plant Journal :for Cell and Molecular Biology,2022,110(3):881-898.
[8]TENG S,BIN H,MENG X,et al.The reference genome of Camellia chekiangoleosa provides insights into Camellia evolution and tea oil biosynthesis[J].Horticulture Research,2022,9(1):3692-3702.
[9]PARK S Y,FUNG P,NISHIMURA N,et al.Abscisic acid inhibits PP2Cs via the PYR/PYL family of ABA-binding START proteins[J].Science,2009,324(5930):1068-1071.
[10] HIROAKI F,VISWANATHAN C,AMERICO R,et al.In vitro reconstitution of an abscisic acid signalling pathway[J].Nature,2009,462(7273):660-664.
[11]CUTLER S R,RODRIGUEZ P L,FINKELSTEIN R R, et al.Abscisic acid:emergence of a core signaling network[J].Annual Review of Plant Biology,2010,61(1):651-679.
[12]XIN W,JUN Z,XIN C, et al.PYL family genes from liriodendron chinense positively respond to multiple stresses[J].Plants(Basel,Switzerland),2023,12(14):2609.
[13]FU X X,TANG J,ZHANG C W,et al. Genome-wide characterization and expression analysis of the abscisic acid receptors PYR/PYL/RCAR(PYLs) in Chinese cabbage during abiotic stresses[J].Russian Journal of Plant Physiology,2022,69(2):29.
[14]KI H J,YEONHEE L,BEOMGI K,et al.Overexpressing OsPYL/RCAR7 improves drought tolerance of maize seedlings by reducing stomatal conductance[J].Agriculture,2022,12(12):2140.
[15]QING L,CHAO S,YUE Z,et al.PePYL4 enhances drought tolerance by modulating water use efficiency and ROS scavenging in Populus[J].Tree Physiology,2022,43(1):102-117.
[16]GUO N,GUO L,WEI M,et al.Overexpression VaPYL9 improves cold tolerance in tomato by regulating key genes in hormone signaling and antioxidant enzyme[J].BMC Plant Biology,2022,22(1):344.
[17]ADAMS M J,ANTONIW J F.Codon usage bias amongst plant viruses[J].Archives of Virology,2004,149(1):113-135.
[18]KANAYA S,YAMADA Y,KINOUCHI M,et al.Codon usage and tRNA genes in eukaryotes:correlation of codon usage diversity with translation efficiency and with CG-dinucleotide usage as assessed by multivariate analysis[J].Journal of Molecular Evolution,2001,53(4/5):290-298.
[19]JIANG Z,YA X,ZHE Z,et al.A comprehensive analysis of Usutu virus(USUV) genomes revealed lineage-specific codon usage patterns and host adaptations[J/OL].Frontiers in Microbiology,2023,13:1-15.https://doi.org/10.3389/fmicb.2022.967999.
[20]JITOBAOM K,PHAKARATSAKUL S,SIRIHONGTHONG T,et al. Codon usage similarity between viral and some host genes suggests a codon-specific translational regulation[J].Heliyon,2020,6(5):e03915.
[21]LIU X Y,LI Y,JI K K,et al.Genome-wide codon usage pattern analysis reveals the correlation between codon usage bias and gene expression in Cuscuta australis[J].Genomics,2020,112(4):2695-2702.
[22]CHANEY J L,CLARK P L.Roles for synonymous codon usage in protein biogenesis[J].Annual Review of Biophysics,2015,44(1):143-166.
[23]ZHANG S,LIN R,CUI L,et al.Alter codon bias of the P.pastoris genome to overcome a bottleneck in codon optimization strategy development and improve protein expression[J].Microbiological Research,2024,282:127629.
[24]SHARP P M,TUOHY M F,MOSURSKI K R.Codon usage in yeast:cluster analysis clearly differentiates highly and lowly expressed genes[J].Nucleic Acids Research,1986,14(13):5125-5143.
[25]SUEOKA N.Intrastrand parity rules of DNA base composition and usage biases of synonymous codons[J].Journal of Molecular Evolution,2004,40(3):318-325.
[26]SUEOKA N.Translation-coupled violation of parity rule 2 in human genes is not the cause of heterogeneity of the DNA G+C content of third codon position[J].Gene,1999,238(1):53-58.
[27]MENG Z,CHUN F T,JI S S.A preliminary analysis of synonymous codon usage in poplar species[J].Journal of Plant Physiology and Molecular Biology,2007,33(4):285-293.
[28]XIA E H,TONG W,WU Q,et al.Tea plant genomics: achievements,challenges and perspectives[J].Horticulture research,2020,7(1):7.
[29]HURST L D.The Ka/Ks ratio:diagnosing the form of sequence evolution [J].Trends Genet,2002,18(9):486.
[30]SONG J,GAO Z H,HUO X M,et al.Genome-wide identification of the auxin response factor(ARF) gene family and expression analysis of its role associated with pistil development in Japanese apricot(Prunus mume Sieb.et Zucc) [J].Acta Physiol Plant,2015,37(8):145.
[31]SHEN T F,HUANG B,XU M,et al.The reference genome of Camellia chekiangoleosa provides insights into Camellia evolution and tea oil biosynthesis[J].Horticulture Research,2022,9:uhab083.
[32]KONG H,LANDHERR L L,FROHLICH M W,et al. Patterns of gene duplication in the plant SKP1 gene family in angiosperms:evidence for multiple mechanisms of rapid gene birth[J].The Plant Journal,2007,50(5):873-885.
[33]GUSTAFSSON C,GOVINDARAJAN S,MINSHULL J.Codon bias and heterologous protein expression[J].Trends in Biotechnology,2004,22(7):346-353.
[34]SWATI T,GORAKHANATH P K,NIRANJANI G, et al.Codon usage provide insights into the adaptation of rice genes under stress condition[J].International Journal of Molecular Sciences,2023,24(2):1098.
[35]MEI Y,JIA L,WAN Y,et al. Analysis of codon usage patterns in 48 Aconitum species[J].BMC Genomics,2023,24(1):703.
[36]PRESNYAK V,ALHUSAINI N,CHEN Y H, et al.Codon optimality is a major determinant of mRNA stability[J].Cell,2015,160(6):1111-1124.
[37]STOLETZKI N,EYRE A.Synonymous codon usage in Escherichia coli:selection for translational accuracy[J].Molecular Biology & Evolution,2007,24(2):374.
[38]胡振民,万 青,李 欢,等.茶树CsNRT1.1基因密码子使用特性分析[J].江苏农业学报,2019,35(4):896-903.
HU ZH M,WAN Q,LI H,et al.Analysis of codon usage features of CsNRT1.1 gene in Camellia sinensis[J] Jiangsu Journal of Agricultural Sciences,2019,35(4):896-903.
[39]周子维,常笑君,游芳宁,等.茶树脂肪氧合酶(LOX)基因家族成员的分子进化及密码子偏好性分析[J].中国农业科技导报,2017,19(12):43-51.
3WciikGNGmHGpHSyjizM6ZQzjqh/87QZwpX9EoFqNPg=ZHOU Z W,CHANG X J,YOU F N,et al.Analysis of molecular evolution and codon bias of lipoxygenase(LOX) gene family in tea tree[J].Journal of Agricultural Science and Technology,2017,19(12):43-51.
[40]JI H,LIU J,CHEN Y,et al.Bioinformatic analysis of codon usage bias of HSP20 genes in four Cruciferous species[J].Plants,2024,13(4):468.
[41]王占军,李 豹,姜行舟,等.两种茶树全基因组数据的密码子偏好性比较分析[J].中国细胞生物学学报,2018, 40(12):2028-2039.
WANG ZH J,LI B,JIANG X ZH,et al.Comparative analysis of the codon preference patterns in two species of Camellia sinensis based on genome data[J].Chinese Journal of Cell Biology,2018,40(12):2028-2039.
[42]陈 悦,仇学文,李 钰,等.辣椒和番茄PIF基因家族密码子偏好性分析[J].分子植物育种,2023,21(17):5602-5610.
CHEN Y,QIU X W,LI Y,et al.Codon bias analysis of PIF gene family in pepper and tomato[J] Molecular Plant Breeding,2023,21(17):5602-5610.
[43]王芳平,李 宏.密码对的使用与基因组进化[J].生物物理学报,2007,23(3):176-184.
WANG F P,LI H.Codon pairs usage and genome evolution[J] Acta Biophysica Sinica,2007,23(3):176-184.
[44]SHAMSUN N,SURUCHI G,WAQAS A,et al. Environmental implications of codon usage bias in Crocus sativus and its impact on host pathogen interactions[J].Rhizosphere,2024,29:100819.
[45]FRUMKIN I,LAJOIE J,GREGG J,et al.Codon usage of highly expressed genes affects proteome-wide translation efficiency[J].Proceedings of the National Academy of Sciences of the United States of America,2018,115(21):E4940-E4949.
[46]SORENSEN M,KURLAND C,PEDERSEN S.Codon usage determines translation rate in Escherichia coli[J].Journal of Molecular Biology,1989,207(2):365-377.
[47]MERT K,FIDAN E.Effect of codon optimization and promoter choice on recombinant endo-polygalacturonase production in Pichia pastoris[J/OL].Enzyme and Microbial Technology,2020,139:1-9.https://doi.org/10.1016/j.enzmictec.2020.109589.
Codon Preference Analysis of PYL Genes in Five Camellia Species
LIU Dandan,WANG Leigang,WU Qiong,SUN Minghui,JIAO Xiaoyu,RUAN Xu and WANG Wenjie
(Tea Research Institute,Anhui Academy of Agricultural Sciences,Hefei 230001,China)
Abstract To further elucidate the genetic and evolutionary characteristics of PYL genes,this study analyzed the codon usage patterns of five Camellia species.Based on the published genome data,55 PYL genes were identified across Camellia chekiangoleosa,Camellia lanceoleosa,Camellia sinensis ‘Yunkang 10’,DASZ,and ‘Tieguanyin’.The phylogenetic analysis revealed that PYL genes could be classified into four groups,exhibiting high conservation.The collinearity analysis indicated that PYL genes were subjected to minimal selection pressure during evolution and mainly influenced by purifying selection.The analysis of codon preference index and synonymous codon usage bias demonstrated significant differences among 59 synonymous codons.The PYL genes showed a preference for codons ending with G/C,such as GCC,ATC,CTC,ACC,GTC,which were associated with encoding hydrophobic amino acids.ENC plot,PR2-plot,and GC3-GC12 analysis suggested that natural selection play an important role in the evolution of PYL genes.The receptor with high similarity in codon usage frequency of PYL genes is considered optimal for heterologous expression analysis.This study provides valuable insights into the codon usage characteristics and evolutionary relationships of PYL genes in Camellia species,contributing to a deeper understanding of their functional and adaptive mechanisms.
Key words Camellia L; PYL genes; Phylogenetic analysis; Collinearity analysis; Codon usage bias
Received 2024-03-25 Returned 2024-05-21
Foundation item National Natural Science Foundation of China(No.U23A20213); Tea Industry Technology System of Anhui Province(No.AHCYJSTX-11); Achievement Transformation of Anhui Academy of Agricultural Sciences(No.2024YL041).
First author LIU Dandan,female,master,assistant research fellow.Research area:the stress resistance mechanism of tea plants.E-mail:1653082943@qq.com
Corresponding author WANG Wenjie,male,research fellow.Research area:tea plant genetics and breeding.E-mail:391590137@qq.com
(责任编辑:史亚歌 Responsible editor:SHI Yage)
基金项目:国家自然科学基金(U23A20213);安徽省茶叶产业技术体系(AHCYJSTX-11);安徽省农业科学院成果转化项目(2024YL041)。
第一作者:刘丹丹,女,硕士,助理研究员,从事茶树抗逆机制研究。E-mail:1653082943@qq.com
通信作者:王文杰,男,研究员,从事茶树遗传育种研究。E-mail:391590137@qq.com

