大豆转录因子GmMYC2L参与植物耐盐性调控





摘要:转录因子在植物应对逆境胁迫的响应和调节过程中起着十分重要的作用。为探究大豆bHLH转录因子家族成员GmMYC2L在植物耐盐胁迫中的功能,本研究利用序列同源比对分析大豆GmMYC2L蛋白与三裂叶薯、番茄、芝麻、本氏烟草和拟南芥MYC2蛋白的保守结构域,利用半定量PCR检测盐胁迫对大豆GmMYC2L基因表达模式的诱导作用,通过烟草叶片瞬时表达分析GmMYC2L蛋白的亚细胞定位,利用浸花法获得转GmMYC2L基因拟南芥植株,并分析野生型和转基因拟南芥植株对盐胁迫的响应及机制。结果表明,大豆GmMYC2L蛋白具有bHLH家族典型的保守结构域且定位于细胞核;盐胁迫下,大豆根和叶中GmMYC2L基因表达量明显上调。盐胁迫下,GmMYC2L基因过表达拟南芥植株叶片中丙二醛含量和过氧化氢含量显著低于野生型拟南芥植株,叶片中超氧化物歧化酶(SOD)活性和过氧化物酶(POD)活性显著增加;抗氧化酶基因AtSOD和AtPOD的相对表达量亦显著上调。以上结果说明大豆GmMYC2L基因能通过抗氧化途径增强植物的耐盐性。本研究结果为大豆等作物的耐盐性遗传改良提供候选基因。
关键词:大豆;转录因子;GmMYC2L ;耐盐性
中图分类号:S565.1文献标识码:A文章编号:1000-4440(2024)07-1182-09Soybean transcription factor GmMYC2L is involved in the regulation of plant salt toleranceZHANG Bin
(Hunan University of Science and Engineering/Hunan Provincial Engineering Research Center for Ginkgo biloba, Yongzhou 425199, China)
Abstract:Transcription factors play an important role in the response and regulation of plants to adversity stress. In order to explore the function of soybean bHLH transcription factor family member GmMYC2L in plant salt tolerance, the conserved domains of soybean GmMYC2L protein and MYC2 proteins of sweet potato, tomato, sesame, Nicotiana benthamiana and Arabidopsis thaliana were analyzed by sequence homology comparison. The expression pattern of soybean GmMYC2L gene induced by salt stress was detected by semi-quantitative PCR. The subcellular localization of GmMYC2L protein was analyzed by transient expression in tobacco leaves. GmMYC2L gene transgenic Arabidopsis thaliana plants were obtained by floral dip method. The response and mechanism of wild-type and transgenic Arabidopsis thaliana plants to salt stress were analyzed. The results showed that soybean GmMYC2L protein had a typical conserved domain of the bHLH family and the protein was localized in the nucleus. Under salt stress, the expression of GmMYC2L gene in soybean roots and leaves was significantly up-regulated. Under salt stress, the contents of malondialdehyde and hydrogen peroxide in the leaves of GmMYC2L overexpressing Arabidopsis thaliana plants were significantly lower than those of wild-type Arabidopsis thaliana plants, and the superoxide dismutase (SOD) activity and peroxidase (POD) activity in the leaves were significantly increased. The relative expression levels of antioxidant enzyme genes AtSOD and AtPOD were also significantly up-regulated. The above results indicated that soybean GmMYC2L gene could enhance salt tolerance of plants through antioxidant pathway. The results of this study provide candidate genes for genetic improvement of salt tolerance in soybean and other crops.
Key words:soybean;transcription factor;GmMYC2L;salt tolerance
江苏农业学报2024年第40卷第7期张斌:大豆转录因子GmMYC2L参与植物耐盐性调控植物生长过程中容易受到不利环境因素的影响,包括极端温度、干旱和盐渍化等非生物胁迫及病虫害等生物胁迫[1]。其中,土壤盐渍化会加剧植物体内活性氧过量积累,破坏Na+和K+离子平衡,抑制光合作用,进而导致植物生长受阻甚至死亡[2]。为了在逆境下生长,植物通常会在生理、生化和分子水平上形成应对策略[3]。分子层面上转录因子是一类可以结合到下游基因启动子元件上调控基因表达水平的蛋白质家族,在植物的耐盐性调控中起重要作用[4]。目前WRKY[5]、ERF[6]、MYB[7]、NAC[8]、ARF[9]、bZIP[10]和bHLH[3]等转录因子参与植物耐盐胁迫调控已得到证实。
转录因子bHLH家族成员较多,是植物中的第二大转录因子家族,通常含有两个高度保守的结构域,即基本结构域和HLH结构域[3]。基本结构域位于N端,能够结合下游基因启动子上的G-box或E-box元件;HLH结构域主要位于C端,在蛋白质间互作时发挥作用。bHLH家族成员参与植物耐盐胁迫调控已有较多研究。拟南芥AtbHLH122基因的表达水平受盐胁迫诱导上调,拟南芥AtbHLH122突变体对盐胁迫更敏感,而AtbHLH122基因过表达植株的耐盐性明显增强[11]。番茄bHLH家族成员SlbHLH22则可通过激活抗氧化系统,清除多余活性氧,增强植株耐盐性[12]。花生AhbHLH121蛋白可以直接结合AhPOD、AhCAT和AhSOD等基因启动子的G-box/E-box区域,从而提高上述基因的表达水平;此外,AhbHLH121基因过表达的转基因花生植株在盐处理下的抗氧化酶活性显著上升,活性氧积累量明显减少,即转基因植株对盐胁迫的耐受性明显增强[13]。编码水稻bHLH家族成员OrbHLH001基因过表达则可激活K+通道基因OsAKT1的表达,进而促进盐胁迫下根和茎中的K+向叶片的运输,同时减少根对Na+的吸收,维持水稻植株Na+含量/K+含量处于正常水平,减轻盐胁迫对水稻植株造成的损伤[14]。盐诱导下,大豆bHLH家族基因GmbHLH3的表达上调,其编码蛋白质可结合到氯离子通道蛋白质基因GmCLC1的启动子上并上调其表达水平,进而介导大豆毛状根对Cl-、NO-3等离子的吸收并限制其向地上部的转运,维持GmbHLH3过表达大豆植株较低的Cl-含量/NO-3含量和Na+含量/K+含量,增强其耐盐性[15]。MYC2属于bHLH 转录因子,能够响应植物生长发育相关的多种内源和外源信号,在信号转导途径中发挥核心作用[16]。盐处理可以诱导丹参SmMYC2基因的表达,过表达该基因可以提高转基因植株体内超氧化物歧化酶(SOD)、过氧化物酶(POD)和过氧化氢酶(CAT)等抗氧化酶活性,增加脯氨酸含量,导致转基因拟南芥植株对盐胁迫的耐受性明显增强[17]。盐胁迫下,甘薯IbMYC2基因的表达水平显著上调,IbMYC2基因过表达植株的花青素含量显著增加;同时,盐胁迫还能激活抗氧化酶和脯氨酸合成酶编码基因,增强活性氧清除能力,赋予植株更强的耐盐性能[4]。
大豆(Glycine max)是中国重要的粮油作物,富含蛋白质,是食用油、植物蛋白质和动物饲料的关键原料。Xu等[18]从栽培大豆基因组中筛选鉴定出308个bHLH家族基因;其中,该家族成员GmPIB1能抑制活性氧产生的关键酶编码基因GmSPOD1的表达,减少活性氧(ROS)的积累,增强大豆疫霉菌抗性[19]。大豆GmMYC2基因参与盐胁迫适应过程调控,GmARF16转录因子可以直接激活GmMYC2的表达,GmMYC2负向调控脯氨酸的合成,增强大豆对盐胁迫的敏感性[20]。本研究团队在前期的研究中筛选到1个GmMYC2的同源基因GmMYC2L,为明确其在盐胁迫应答过程中的功能机理,本研究通过转基因、生理生化测定和分子生物学技术,分析GmMYC2L基因在盐胁迫下的表达模式及其编码蛋白质的亚细胞定位,探究野生型和转GmMYC2L基因拟南芥植株在耐盐性方面的差异及机制,为大豆的耐盐品种选育提供理论依据和重要基因资源。
1材料与方法
1.1不同植物MYC2蛋白质氨基酸序列分析
从大豆基因组参考数据库(https://soykb.org/)下载GmMYC2L氨基酸序列(登录号:Glyma.01G096600),从美国国家生物信息中心(NCBI)数据库(https://www.ncbi.nlm.nih.gov/)下载三裂叶薯(Ipomoea triloba)、番茄(Solanum lycopersicum)、芝麻(Sesamum indicum)、本氏烟草(Nicotiana tabacum)和拟南芥(Arabidopsis thaliana)的MYC2氨基酸序列,数据库登录号分别为XP_031095962.1(三裂叶薯)、XP_004244656.1(番茄)、XP_011081344.1(芝麻)、XP_016500166.1(本氏烟草)和NP_174541.1AtMYC2(拟南芥)。利用DNAMAN软件对GmMYC2L和ItMYC2、SlMYC2、SiMYC2、NtMYC2、AtMYC2的氨基酸序列进行对比。
1.2组织RNA提取和基因表达检测
大豆盆栽试验于2023年3月在湖南科技学院植物生长室进行。将大豆种子(品种为中黄13)播种于直径20 cm、高30 cm装有5 kg营养土的花盆中,每盆播种6颗种子,共12盆,于植物生长室进行培养。生长室内温度恒定20 ℃,相对湿度60%~70%,日照时间16 h/d,白天光照度为100 μmol/(m2· s)。待大豆幼苗的真叶完全展开后,使用150 mmol/L NaCl溶液浇灌,以清水浇灌作为对照(CK),各6盆,分别在0 h、3 h、6 h和12 h时取大豆根和叶各1 g,液氮速冻后在-80 ℃冰箱中保存。
在研钵中加入液氮和植物组织样品,将样品充分研磨后使用植物RNA提取试剂盒[翌圣生物科技(上海)股份有限公司产品]提取总RNA,取2 μg RNA使用反转录试剂盒[翌圣生物科技(上海)股份有限公司产品]合成cDNA。以GmUBI3为内参基因,利用2× Hieff CanacePCR Master Mix 高保真酶和PCR仪(德国Biometra公司产品)进行半定量PCR,明确大豆幼苗根和叶中GmMYC2L基因的相对表达水平。所用引物如表1所示。
1.3大豆GmMYC2L的亚细胞定位
以大豆cDNA为模板,克隆不含终止密码子的GmMYC2L基因编码序列,并连接到pCAMBIA1300-GFP表达载体(上海百风生物科技有限公司产品)35S启动子下游,获得融合质粒35S-GmMYC2L-GFP。使用农杆菌介导的方法将融合质粒35S-GmMYC2L-GFP和pCAMBIA1300-GFP载体分别转化本氏烟草叶片。其中,转化pCAMBIA1300-GFP空载体的烟草叶片为对照。转化后的烟草转移到植物生长室避光培养1 d,再按16 h/d的光照条件培养1 d,使得外源基因在烟草叶片中产生相应蛋白质。使用Ultra VIEW VoX激光共聚焦显微镜(美国PerkinElmer公司产品)观察绿色荧光在烟草叶片细胞中的位置进行GmMYC2L蛋白的亚细胞定位。
1.4载体构建和转基因植株筛选
以大豆cDNA为模板,克隆GmMYC2L基因编码序列,经1%琼脂糖凝胶电泳,切下含有目的条带的凝胶进行片段回收,然后将回收片段插入双元表达载体pFGC5941的35S启动子下游,获得重组质粒35S-GmMYC2L,并将其转入GV3101根癌农杆菌。利用浸花法转化野生型拟南芥,收获T0代种子。将T0代种子经75%乙醇消毒5 min后均匀撒在含25 mg/L潮霉素(HYG)的MS培养基上,筛选得到能正常生长的抗性幼苗。进一步提取野生型拟南芥和抗性幼苗的DNA和RNA,通过荧光定量PCR检测抗性植株中Hyg基因(所用引物见表1)是否存在以及GmMYC2L的相对表达水平,得到过表达转基因植株(OE),将转基因植株继续培养,收获相应的种子用于后续试验。荧光定量PCR所用引物见表1。
拟南芥野生型和转基因植株中Hyg基因检测方法:将能够生长的抗性幼苗移栽到营养土中继续培养,21 d后剪取植株叶片,使用植物DNA提取试剂盒[翌圣生物科技(上海)股份有限公司产品]提取DNA,利用20.0 μL PCR反应体系(2×Hieff CanacePCR Master Mix 高保真酶10.0 μL、无菌水8.0 μL、Hyg正向引物和反向引物各0.5 μL、DNA模板1.0 μL)进行扩增,即利用PCR仪(德国Biometra公司产品)进行半定量PCR,PCR产物利用1%琼脂糖凝胶电泳以检测植株中Hyg基因表达情况。
拟南芥野生型和转基因植株中GmMYC2L基因的相对表达水平检测:拟南芥野生型和抗性幼苗培养21 d后,利用拟南芥野生型和转基因植株叶片提取RNA,以AtACTIN2基因作为内参基因,2×SYBR Green Mix[翌圣生物科技(上海)股份有限公司产品]为荧光染料,参照Xu等[18]方法,利用荧光定量PCR仪(美国Bio-Rad公司产品)进行拟南芥野生型和转基因植株的GmMYC2L基因表达水平检测。
1.5拟南芥耐盐性评价和生理生化指标测定
将GmMYC2L过表达拟南芥种子(OE)和野生型拟南芥种子(WT)消毒后均匀播在常规MS培养基和含150 mmol/L NaCl的MS培养基中,每个培养皿播30粒种子,4 ℃放置3 d后转移到植物生长室[恒定温度20 ℃,相对湿度60%~70%,日照时间16 h/d,光照度100 μmol/(m2·s)]垂直放置,7 d后观察种子的萌发和根的生长情况,测量并统计平均根长。
将GmMYC2L过表达拟南芥种子(OE)和野生型拟南芥种子(WT)播种于面积36 cm2、高5 cm装有100 g营养土的塑料钵中,每钵播种6粒种子,4 ℃黑暗放置3 d后移至植物生长室[恒定温度20 ℃,相对湿度60%~70%,日照时间16 h/d,光照度为100 μmol/(m2·s)]培养。21 d后挑选长势一致的植株分成2组,各12株,以150 mmol/L NaCl水溶液浇灌作为处理,以清水浇灌作为对照,处理7 d后观察植株生长状况。参照文献[21]的方法测定不同处理下拟南芥植株叶片的丙二醛(MDA)含量,利用过氧化氢测定试剂盒(南京建成生物工程研究所有限公司产品)测定拟南芥叶片中的H2O2含量。利用总超氧化物歧化酶(T-SOD)测试盒和过氧化物酶(POD)测定试剂盒(南京建成生物工程研究所有限公司产品),按照试剂盒使用说明书进行不同处理下拟南芥植株(WT和OE)叶片中超氧化物歧化酶(SOD)、过氧化物酶(POD)的活性测定。以AtACTIN2为内参基因,2×SYBR Green Mix[翌圣生物科技(上海)股份有限公司产品]为荧光染料,参照Xu等[18]方法,利用荧光定量PCR仪(美国Bio-Rad公司产品)进行拟南芥野生型和转基因植株的AtSOD和AtPOD基因的相对表达量测定。
上述测定均设3个重复,利用SPSS v20软件进行差异显著性分析(P<0.05)。
2结果与分析
2.1大豆转录因子GmMYC2L鉴定及GmMYC2L基因表达模式大豆GmMYC2L的氨基酸序列与三裂叶薯、番茄、芝麻、本氏烟草和拟南芥的MYC氨基酸序列比对结果如图1所示。从图中可以看出,大豆GmMYC2L与其他物种MYC2的氨基酸序列均具有典型的bHLH-MYC结构域和HLH结构域。
盐胁迫及CK处理下大豆幼苗中GmMYC2L基因的表达情况如图2所示。从图可以看出,盐胁迫3 h,大豆根中GmMYC2L的表达量明显增加;6 h时表达量略有降低,但仍明显高于无盐胁迫对照;盐胁迫3 h,大豆叶中GmMYC2L的表达水平也呈上调趋势,上述结果说明GmMYC2L基因参与大豆对盐胁迫的响应过程。
2.2大豆GmMYC2L蛋白的亚细胞定位
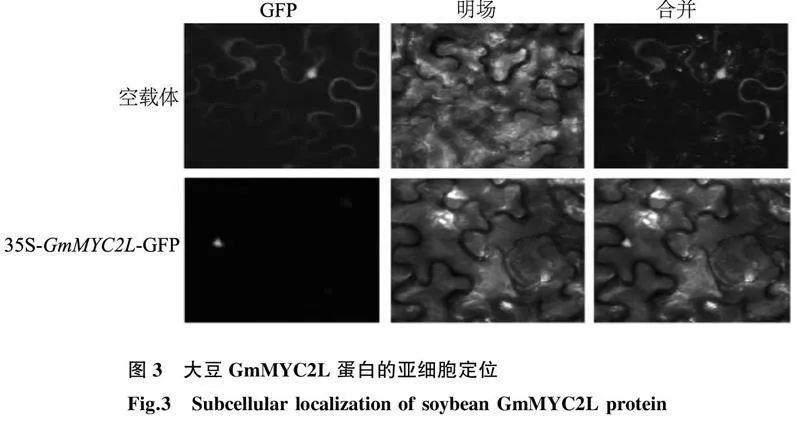
分别转化融合质粒35S-GmMYC2L-GFP和pCAMBIA1300-GFP空载体的本氏烟草叶片细胞绿色荧光特征如图3所示。从图中可以看出,转化pCAMBIA1300-GFP空载体的烟草叶片细胞细胞膜和细胞核中都能够观察到明显的绿色荧光,而转化重组质粒35S-GmMYC2L-GFP的烟草叶片细胞仅细胞核中有荧光信号分布,说明大豆GmMYC2L蛋白定位于细胞核。
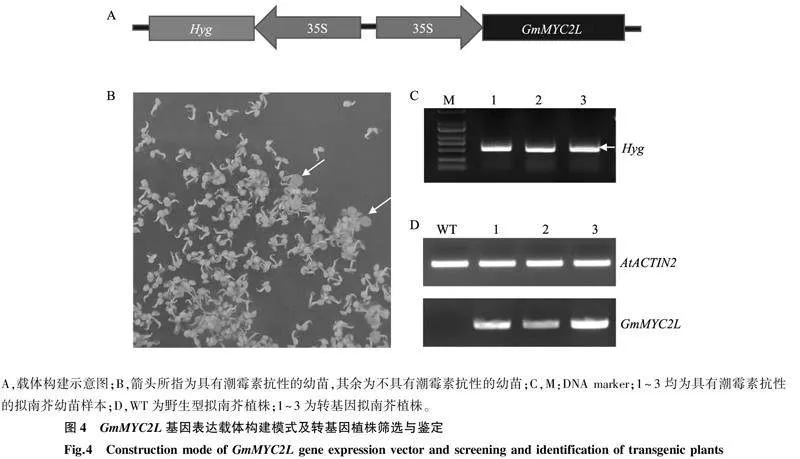
2.3外源大豆基因GmMYC2L过表达拟南芥植株的筛选鉴定克隆GmMYC2L基因全长序列,插入pFGC5941载体35S启动子下游,获得基因过表达载体35S-GmMYC2L(图4A)。含有潮霉素抗性的拟南芥幼苗能够正常生长,即子叶正常展开,不含有潮霉素抗性的幼苗则在萌发后生长受到抑制(图4B)。含有潮霉素抗性的拟南芥幼苗生长21 d后叶片PCR检测结果显示其含有外源Hyg基因(图4C)。含有潮霉素抗性的拟南芥幼苗生长21 d后叶片半定量PCR分析结果显示内参基因(AtACTIN2)在野生型拟南芥(WT)和抗性幼苗均有表达,证明RNA提取无误。野生型拟南芥中大豆GmMYC2L基因不表达,而抗性幼苗中外源大豆基因GmMYC2L正常表达(图4D),说明本研究建立的外源大豆基因GmMYC2L过表达拟南芥植株(OE)是可靠的。
2.4过表达GmMYC2L拟南芥植株的耐盐性
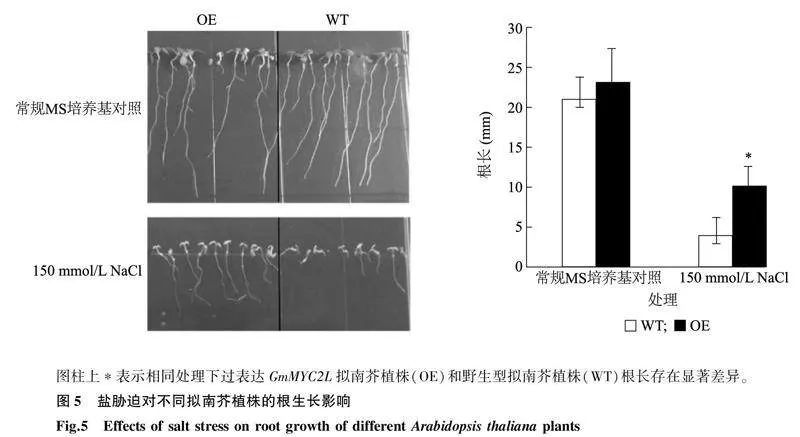
常规MS培养基上野生型拟南芥植株(WT)和过表达GmMYC2L拟南芥植株(OE)的根长无显著差异;150 mmol/L盐胁迫下,过表达GmMYC2L拟南芥植株(OE)的根长显著长于WT(图5)。
清水灌溉对照,过表达GmMYC2L拟南芥植株(OE)和野生型拟南芥植株(WT)的长势差异不大,单株鲜重无显著差异;而150 mmol/L NaCl水溶液浇灌处理,过表达GmMYC2L拟南芥植株(OE)的长势明显好于WT植株,单株鲜重显著高于WT植株(图6,表2)。同样,清水灌溉对照,过表达GmMYC2L拟南芥植株(OE)和野生型拟南芥植株(WT)叶片中MDA含量和H2O2含量亦无显著差异,而150 mmol/L NaCl水溶液浇灌处理,过表达GmMYC2L拟南芥植株(OE)和野生型拟南芥植株(WT)叶片中MDA含量和H2O2含量均显著增加,且野生型拟南芥植株(WT)叶片中MDA含量和H2O2含量显著高于过表达GmMYC2L拟南芥植株(表2),上述结果说明过表达GmMYC2L拟南芥植株对盐胁迫的耐受性增强。
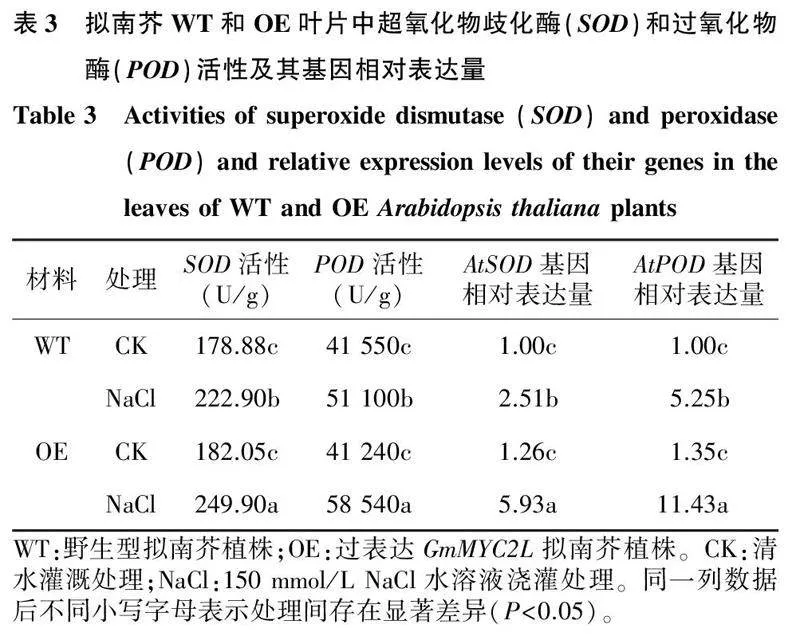
2.5GmMYC2L对抗氧化酶活性及其基因表达量的影响清水灌溉对照,拟南芥WT和OE叶片中SOD活性和POD活性均无明显差异;盐胁迫处理,OE叶片中SOD活性和POD活性高于野生型植株。与酶活性相对应,盐胁迫处理,过表达GmMYC2L拟南芥植株(OE)叶片中AtSOD基因和AtPOD基因的相对表达量显著高于野生型拟南芥植株(WT)(表3)。
3讨论
盐胁迫已成为农作物生长的主要限制因素之一[22-25],且中国盐渍化耕地面积呈扩大趋势[1]。挖掘作物耐盐基因并进行耐盐性品种选育已成为保障作物生产的重要手段。国内外学者在作物耐盐性调控的生理机理、关键基因的挖掘和分子调控网络解析方面已有较多成果[1]。转录因子在作物耐盐性调控中处于核心位置[5-10]。转录因子MYC2参与植物生长调节、信号传导、激素相互作用及多种非生物胁迫应答等过程[3,11]。茉莉酸处理条件下,拟南芥AtMYC2基因过表达,能促进拟南芥花青素的积累,但抑制根系生长[26]。IbMYC2基因的过表达能增强甘薯植株的耐盐性[4]。大豆bHLH家族基因在调节金属离子运输和提高植株耐受生物胁迫方面的作用已有报道[18-19]。本研究发现大豆GmMYC2L蛋白也包含与其他植物类似的bHLH-MYC和HLH保守结构域,无盐胁迫对照大豆根和叶中GmMYC2L基因表达水平较低,但盐胁迫处理,大豆根和叶中GmMYC2L基因表达水平均明显上调,且根中更加明显。根是作物响应盐胁迫最早的器官,GmMYC2L基因在根中迅速上调是作物根系对盐胁迫响应的具体表现。
甘蓝型油菜BnMYC2基因能调节气孔开闭,进而提高油菜的耐旱性[27]。Deng等[17]发现过表达丹参SmMYC2的转基因拟南芥和丹参毛状根的耐盐性更强,主要原因是转基因植株在盐胁迫处理下比野生型植株具有更高的抗氧化酶活性以及更低的MDA含量和活性氧(ROS)含量[17]。但拟南芥AtMYC2基因过表达则导致转基因植株耐盐性明显降低[16]。因此,不同植物的MYC2基因功能是不保守的。本研究通过构建GmMYC2L基因过表达载体并获得转GmMYC2L基因拟南芥植株(OE),发现GmMYC2L基因过表达不影响拟南芥植株的正常生长,在培养基和营养土上拟南芥野生型和转基因植株的生长状况无明显差异;而在盐胁迫处理下,转基因植株的幼苗根长显著长于野生型植株,拟南芥幼苗地上部的单株鲜重亦明显优于野生型植株,说明GmMYC2L基因过表达赋予拟南芥植株更强的盐胁迫适应能力。但是,Wang等[20]研究显示大豆GmMYC2负调控植物耐盐性,分析发现其报道的GmMYC2基因(Glyma.08G271900)与本研究基因GmMYC2L均被注释为MYC2,二者相似性为87.37%,进一步证明植物MYC2同源基因功能的多样性。通常非生物胁迫会导致活性氧(ROS)在植株中过量积累,造成细胞的氧化伤害,进而降低酶活性,抑制植物生长[28]。植物可以通过激活抗氧化酶(如SOD和POD)活性,维持细胞ROS稳态,减少对植株的损害[29]。脯氨酸是重要的渗透调节物质,可降低逆境下细胞的渗透电位,增强细胞吸水量,减轻逆境对植物生长的影响[30]。本研究发现大豆GmMYC2L调节植物耐盐性的生理机制与丹参SmMYC2类似,过表达GmMYC2L植株在盐胁迫下积累更少的MDA和H2O2,同时SOD和POD活性增强;对应的基因AtSOD、AtPOD也在盐胁迫处理的OE中表现出更高的表达水平。即大豆bHLH家族转录因子GmMYC2L能够通过激活抗氧化途径,提高抗氧化酶活性,清除盐胁迫导致的过量活性氧,从而增强植物的耐盐性。本研究结果为大豆和其他作物耐盐性遗传改良提供依据和基因资源。
参考文献:
[1]ZHU J K. Abiotic stress signaling and responses in plants[J]. Cell,2016,167:313-324.
[2]TAYLOR N L, TAN Y F, JACOBY R P, et al. Abiotic environmental stress induced changes in the Arabidopsis thaliana chloroplast, mitochondria and peroxisome proteomes[J]. Journal of Proteonomics,2009,72(3):367-378.
[3]LIU Y J, JI X Y, NIE X G, et al. Arabidopsis AtbHLH112 regulates the expression of genes involved in abiotic stress tolerance by binding to their E-box and GCG-box motifs[J]. New Phytologist,2015,207:692-709.
[4]HU Y F, ZHAO H Y, XUE L Y, et al. IbMYC2 contributes to salt and drought stress tolerance via modulating anthocyanin accumulation and ROS-scavenging system in sweet potato[J]. International Journal of Molecular Sciences,2024,25(4):2096.
[5]PHUKAN U J, JEENA G S, SHUKLA R K. WRKY transcription factors:molecular regulation and stress responses in plants[J]. Frontiers in Plant Science,2016,7:760.
[6]ZHANG G, CHEN M, LI L, et al. Overexpression of the soybean GmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought, and diseases in transgenic tobacco[J]. Journal of Experimental Botany,2009,60:3781-3796.
[7]CUI M H, YOO K S, HYOUNG S J, et al. An Arabidopsis R2R3-MYB transcription factor, AtMYB20, negatively regulates type 2C serine/threonine protein phosphatases to enhance salt tolerance[J]. FEBS Lett,2013,587(12):1773-1778.
[8]WANG Y X, LIU Z W, WU Z J, et al. Transcriptome-wide identification and expression analysis of the NAC gene family in tea plant [Camellia sinensis (L.) O. Kuntze][J]. PLoS One,2016,11:e0166727.
[9]ZHANG M J, XUE Y Y, XU S, et al. Identification of ARF genes in Cucurbita pepo L. and analysis of expression patterns,and functional analysis of CpARF22 under drought,salt stress[J]. BMC Genomics,2024,25(1):112.
[10]YANG Y, YU T F, MA J, et al. The soybean bZIP transcription factor gene GmbZIP2 confers drought and salt resistances in transgenic plants[J]. International Journal of Molecular Sciences,2020,21(2):670.
[11]LIU W W, TAI H H, LI S S, et al. bHLH122 is important for drought and osmotic stress resistance in Arabidopsis and in the repression of ABA catabolism[J]. New Phytologist,2014,201(4):1192-1204.
[12]WASEEM M, RONG X Y, LI Z G. Dissecting the role of a basic Helix-Loop-Helix transcription factor, SlbHLH22, under salt and drought stresses in transgenic Solanum lycopersicum L. [J]. Frontiers in Plant Science,2019,10:734.
[13]ZHAO X B, WANG Q, YAN C X, et al. The bHLH transcription factor AhbHLH121 improves salt tolerance in peanut[J]. International Journal of Biological Macromolecules,2024,256(2):128492.
[14]CHEN Y, LI F, MA Y, et al. Overexpression of OrbHLH001, a putative helix-loop-helix transcription factor, causes increased expression of AKT1 and maintains ionic balance under salt stress in rice[J]. Journal of Plant Physiology,2013,170(1):93-100.
[15]LIU X, PI B Y, DU Z Y, et al. The transcription factor GmbHLH3 confers Cl-/salt tolerance to soybean by upregulating GmCLC1 expression for maintenance of anion homeostasis[J]. Environmental and Experimental Botany,2022,194:104755.
[16]VERMA D, JALMI S K, BHAGAT P K, et al. A bHLH transcription factor, MYC2, imparts salt intolerance by regulating proline biosynthesis in Arabidopsis[J]. FEBS Journal,2020,287(12):2560-2576.
[17]DENG H Y, QI L, CAO R Z, et al. Overexpression of SmMYC2 enhances salt resistance in Arabidopsis thaliana and Salvia miltiorrhiza hairy roots[J]. Journal of Plant Physiology,2023,280:153862.
[18]XU Z L, LIU X Q, HE X L, et al. The soybean basic Helix-Loop-Helix transcription factor ORG3-like enhances cadmium tolerance via increased iron and reduced cadmium uptake and transport from roots to shoots[J]. Frontiers in Plant Science,2017,8:1098.
[19]CHENG Q, DONG L D, GAO T J, et al. The bHLH transcription factor GmPIB1 facilitates resistance to Phytophthora sojae in Glycine max[J]. Journal of Experimental Botany,2018,69(10):2527-2541.
[20]WANG C F, LI X M, ZHUANG Y B, et al. A novel miR160a-GmARF16-GmMYC2 module determines soybean salt tolerance and adaptation[J]. New Phytologist,2024,241(5):2176-2192.
[21]LIU X, YANG X X, ZHANG B. Transcriptome analysis and functional identification of GmMYB46 in soybean seedlings under salt stress[J]. PeerJ,2021,9:e12492.
[22]赵欣,卢海峰,钱程,等. 紫花苜蓿叶面积和叶解剖结构对盐胁迫的响应[J]. 江苏农业科学,2023,51(19):145-152.
[23]徐文清,李少楠,方啸宇,等. 盐胁迫对2个葡萄品种扦插苗光合特性及抗氧化能力的影响[J]. 南方农业学报,2023,54(10):3000-3009.
[24]陈星星,刘新社,王盛荣. 腐殖酸对盐胁迫下土壤理化性质、微环境及苦瓜生长的影响[J]. 江苏农业科学,2023,51(17):138-144.
[25]陈亚辉,张师,杨庆山,等. 多枝柽柳叶片响应NaCl胁迫的转录组分析[J]. 江苏农业学报,2022,38(5):1188-1202.
[26]LORENZO O, CHICO J M, SNCHEZ-SERRNO J J, et al. Jasmonate-insensitive1 encodes a MYC transcription factor essential to discriminate between different jasmonate-regulated defense responses in Arabidopsis[J]. Plant Cell,2004,16(7):1938-1950.
[27]WANG W, SHI X, CHEN D, et al. The Brassica napus MYC2 regulates drought tolerance by monitoring stomatal closure[J]. European Journal of Horticultural Science,2020,85:226-231.
[28]ZHU J K. Plant salt tolerance[J]. Trends in Plant Science,2001,6:66-71.
[29]AI HASSAN M, CHAURA J, DONAT-TORRES M P, et al. Antioxidant responses under salinity and drought in three closely related wild monocots with different ecological optima[J]. AoB Plants,2017,9(2):plx009.
[30]KRASENSKY J, JONAK C. Drought, salt, and temperature stress-induced metabolic rearrangements and regulatory networks[J]. Journal of Experimental Botany,2012,63(4):1593-1608.
(责任编辑:石春林)

