青钱柳全基因组SSR位点分析及多态性引物开发


摘要:【目的】通过对青钱柳全基因组序列分析,开发基因组SSR分子标记;尝试构建19个青钱柳优良药用无性系的DNA分子身份证,为后续种质资源评价、遗传多样性和种质鉴定提供技术支撑。【方法】利用MISA(microsatellite identification tool)软件对青钱柳全基因组进行SSR位点搜寻、筛选、识别及富集分析,采用Primer 3.0进行SSR引物设计;用重复性和稳定性高的SSR标记构建青钱柳无性系的识别系统。【结果】①从全基因组中共检测出89 741个SSR位点,SSR位点的发生频率为62.07%。②基因组SSR位点中单核苷酸重复单元比例最高,占总SSR位点的62.67%;六核苷酸重复单元比例最低,占0.15%;SSR位点的重复基序大多以(A/T)n为主。③单核苷酸和二核苷酸重复类型的SSR位点基序重复次数集中在6~16次;随重复次数增加,各SSR位点重复类型出现频率均呈下降趋势。④基因组SSR序列长度介于10~476 bp,不同类型重复单元的SSR序列长度存在变异性;随着重复次数的增加,SSR序列出现的频率整体呈下降趋势。⑤利用Primer 3.0成功设计出78 285对SSR引物;合成的377对中有75对引物可扩增出多态性条带;用5对单碱基重复的多态性SSR引物分析19个药用无性系,构建出无性系的二维码DNA分子身份证。【结论】青钱柳基因组SSR位点出现频率高,位点种类丰富,可为种质资源评价及指纹图谱的构建提供丰富的候选分子标记。
关键词:青钱柳;简单重复序列(SSR);全基因组;DNA分子身份证
中图分类号:S722;Q756""" 文献标志码:A开放科学(资源服务)标识码(OSID):
文章编号:1000-2006(2024)04-0067-09
Analysis of SSR locus based on the whole genome" sequences of Cyclocarya paliurus and" the development of polymorphic primers
LIU Li1,2, QU Yinquan1, YU Yanhao1, WANG Qian1, FU Xiangxiang1*
(1.College of Forestry and Grassland, Nanjing Forestry University," Nanjing 210037, China;2.Shanghai Pudong Forestry Station, Shanghai"" 200120, China)
Abstract: 【Objective】 Genomic simple repeat sequence (SSR) loci were analyzed by screening the whole genome of Cyclocarya paliurus. DNA molecular ID cards of 19 excellent medicinal clones of C. paliurus were constructed based on the newly-developed SSR primers. These genomic SSR markers could support further research, such as the evaluation of the germplasm resource, analysis of genetic diversity, and"" identification of cultivars/clones. 【Method】 The SSR loci were screened along with the whole genome of C. paliurus and were"" enriched and analyzed using MISA software. Subsequently, SSR primers were designed using Primer 3.0. Furthermore, a system for identifying clones of C. paliurus was constructed based on selected SSR markers with high reproducibility and stability. 【Result】 (1) We detected 89 741 SSR loci from the whole genome, with an occurrence frequency of 62.07%. (2) Among all SSR loci, the proportion of SSRs with a mononucleotide motif was the highest (62.67%) and a hexa-nucleotide repeat was the lowest (0.15%). Most of the repeated motifs in the SSR loci were dominated by (A/T)n. (3) The repeat number of mono-nucleotide and di-nucleotide motifs ranged from 6 to 16. With the increase in the repeat number, the frequencies of various SSR repetition types displayed a downward trend. (4) The length of the SSR sequences varied from 10 to 476 bp," and this length variation existed in different repetitive motifs. Additionally, the frequency of SSR occurrence tended to decrease as the repeat number increased. (5) We successfully designed 78 285 pairs of SSR primers using Primer 3.0. A total of 377 primer pairs were randomly synthesized for amplifying polymorphic SSR fragments, among which 75 pairs primers were successful. Moreover, quick response code DNA molecular ID cards for 19 medical-use clones of C. paliurus were constructed by five pairs of polymorphic SSR primers with a mono-nucleotide motif. 【Conclusion】 The frequency of genomic SSR loci was high, and there was variability in the type of SSR loci. Simple repeat sequences developed from the whole genome of C. paliurus could be effective candidate molecular markers with further applications in germplasm resource evaluation and fingerprint construction for multi-use clones of C. paliurus.
Keywords:Cyclocarya paliurus; simple sequence repeat (SSR); whole genome; DNA molecular ID card
青钱柳(Cyclocarya paliurus),又名青钱李、甜茶树、摇钱树等,是我国胡桃科(Juglandaceae)特有的单属种植物[1]。广泛分布于江西、浙江、江苏等海拔420~1 100 m(东部)或2 500 m(西部)的山区、溪谷或石灰岩山地。青钱柳集药用、材用和观赏功能于一体[1-2]。其叶中富含黄酮、三萜、多糖、酚酸等生物活性物质,具有降血糖、降血脂、抗氧化、抗菌、抗癌等功效[3-7]。
青钱柳具有显著的药用价值,因此广受学者们的关注。目前青钱柳的开发利用主要源于天然林资源[8],而天然资源存在显著的地域特点且资源有限[9],其药用品质难以保证,严重制约了青钱柳的产业化发展。研究还发现,来自青钱柳不同种源、家系和个体内主要的生物活性物质含量差异显著[7,10-11],为药用优良种植材料的筛选提供了条件,但仅依据表型选择的单株,其药用品质并不十分稳定[12]。基于此,有必要开展分子辅助选择,将有效的分子标记结合表型进行选择,提高选择效率。此外,利用分子标记还可对青钱柳天然资源进行遗传多样性分析、种质资源鉴定和评价、构建优良品系的指纹图谱等,可为进一步的开发利用提供技术支撑。
随着分子标记技术的发展,分子标记已成为种质资源鉴定和保护、遗传多样性分析等的有效手段[13]。其中,由于SSR(简单重复序列,simple sequence repeat)标记技术具有快速、共显性、特异性和重复性好的特点,广泛应用于种质资源评价、遗传多样性分析及品种鉴定等方面的研究[14-16]。随着高通量测序技术的发展,许多生物都已完成了全基因组测序(whole genome sequencing,WGS),为SSR位点的开发和应用提供了条件。如鹅掌楸(Liriodendron chinense)、黄檀(Dalbergia hupeana)、茶树(Camellia sinensis)等木本植物基于全基因组序列进行了SSR标记的开发和应用[17-19]。
本研究基于青钱柳全基因组测序结果,分析了基因组水平上的SSR位点数量、SSR重复基序类型和重复次数、序列长度等。在此基础上设计引物并按一定比例随机合成,筛选出多态性SSR引物,构建19个优良药用无性系的DNA分子身份证,为后续无性系的推广栽培和多态性引物构建身份识别系统提供技术支撑和方法初探。青钱柳SSR多态性位点的开发可为核心种质资源库的构建、种质资源的遗传多样性评价和鉴定提供有效的研究手段和方法。
1 材料与方法
1.1 供试材料
引物验证及DNA分子身份证构建所用材料为根据青钱柳表型特征筛选出的19个药用无性系[12],分别种植于南京林业大学白马教学科研基地(江苏南京溧水区白马镇,119°09′E,31°35′N)及安徽省池州市石台县青钱柳人工林试验基地(117°17′E, 30°14′N)。无性系来自浙江千岛湖、湖南张家界、云南、安徽清凉峰和舒城、湖北五峰和鹤峰、贵州黎平和剑河的种源和家系。采集幼嫩叶片,-40 ℃冰箱保存备用。经细胞流式仪测定及细胞学分析,除S4号为二倍体外,其余均为四倍体。
1.2 DNA提取
采用改良CTAB法提取植物基因组DNA。1%(质量分数)的琼脂糖凝胶电泳检测DNA质量,微量紫外分光光度计检测DNA浓度和吸收峰,将电泳条带明亮清晰、吸光度(A260/A280)值为1.8~2.0的DNA样品浓度稀释到50 ng/μL,-20 ℃保存备用。
1.3 全基因组测序
通过single-molecule real-time (SMRT) 技术对青钱柳二倍体植株进行了全基因组测序和组装,获得35 221条序列(已去除冗余,无重复),总基因组大小为553.87 Mb。
1.4 SSR位点识别及引物设计
利用软件MISA(microsatellite identification tool)搜索全基因组序列中的SSR位点。为保证较高的位点检出率和引物设计成功率[20],具体查找标准为:单、二、三、四、五、六核苷酸的最低重复次数为10、6、5、5、5、5次,SSR侧翼序列长度 ≥ 50 bp;当相邻SSR之间的间隔区域长度小于100 bp时,两个微卫星组成一个微卫星,也称复合型微卫星(C型)。
根据MISA查找SSR位点两端的保守序列,利用Primer 3.0(V 2.3.7; http://primer3.source-forge. net/)进行引物设计[21],各项参数设置为默认值[22]。
1.5 PCR扩增及SSR检测
PCR反应体系10" μL,包括250 U Taq酶0.1" μL(TaKaRa,中国),上下游引物各0.6" μL(10" μmol/L),2.5 mmol/L Mg2+ 0.8" μL,2.5 mmol/L dNTPs 2" μL,10×buffer 1" μL,50 ng/μL的模板DNA 1" μL,剩余用3.9" μL dd H2O补足。普通引物和毛细管电泳检测的荧光引物(3′端添加荧光修饰,FAM、HEX、TAMRA和ROX)均由南京擎科生物技术有限公司合成。
PCR反应程序:95 ℃预变性2 min,39个循环包括:94 ℃变性45 s;55~62 ℃退火,复性45 s,72 ℃延伸30 s;72 ℃延伸10 min。扩增产物用8%(质量分数)的变性聚丙烯酰胺凝胶电泳检测,进行引物初筛;对多态性SSR位点,用荧光引物进行避光扩增,扩增产物在+ABI-3730XL基因分析仪(Applied Biosystems, Foster City, CA, USA)上进行毛细管电泳检测。
1.6 DNA分子身份证构建方法
统计毛细管电泳检测位点,有效位点采用数字和英文字母编码。将每对引物的扩增条带按分子量由大到小排序,编码为A—F,构建数字与字母结合的字符串形式的DNA分子身份证。将每个无性系的来源、种质类型、特性和唯一的DNA分子身份证编码信息导入二维码生成软件(https://cli.im/),生成可供扫描的二维码DNA分子身份证[23]。
1.7 数据处理
利用Excel对SSR位点的数量、重复基元类型、重复次数、SSR序列长度及其变异进行统计。
2 结果与分析
2.1 青钱柳全基因组中SSR位点特性
2.1.1 SSR位点的分布特点
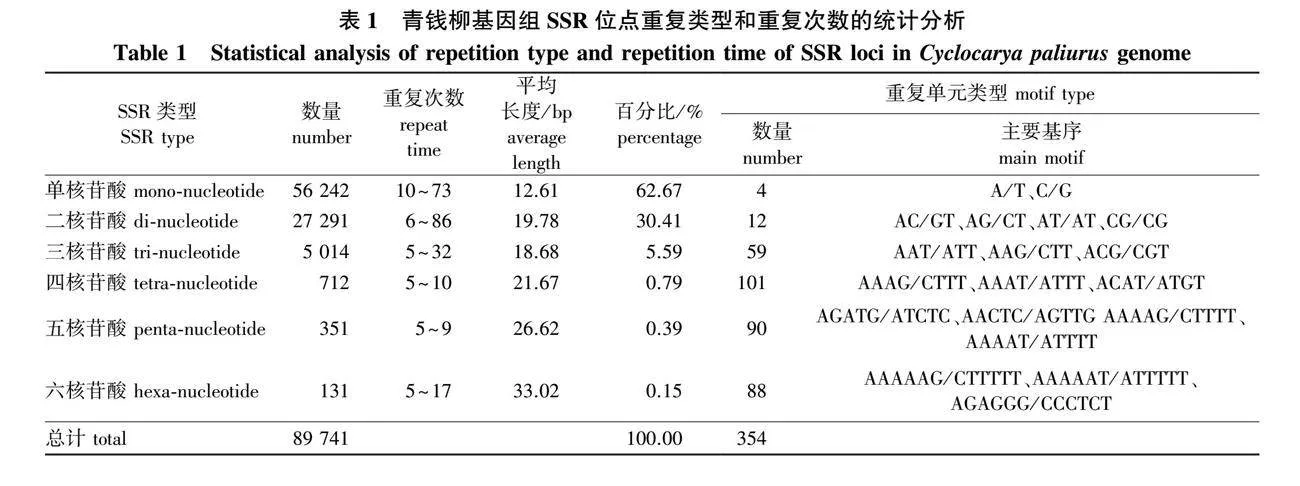
MISA搜索基因组35 221条序列发现,有21 863条序列含有共89 741个SSR位点,SSR的发生频率为62.07%。其中包含1个以上SSR位点的序列有16 234条,占发生SSR位点序列的74.25%;5 629条序列包含1个SSR位点,占包含SSR位点序列的25.75%。所有SSR位点中共包含11 445个复合型SSR(C型SSR),占SSR位点总数的12.75%。平均每1.81 kb含有1个SSR位点。
2.1.2 SSR重复类型
分析发现,青钱柳基因组SSR核苷酸重复基元种类丰富,共存在354种类型,重复基序的碱基变化为1~6(表1)。单核苷酸重复基元有A/T和C/G两种类型,其中以A/T为主,共有50 610个位点,占单核苷酸重复基序的89.99%。二核苷酸重复基元有AC/GT、AT/AT、CG/CG和AG/CT4种重复类型,其中以AC/GT为主,有13 779个,占二核苷酸重复基元的50.49%;AT/AT重复基元最少,仅有108个,占二核苷酸重复基元的0.003%。三核苷酸重复基元共有59种类型,其中以AAT/ATT为主要重复基元,其位点数为1 697个,占三核苷酸重复基元的33.85%;其次为AAG/CTT,有1 479个,占三核苷酸重复基元的29.50%;ACG/CGT重复基元最少,仅有65个,占三核苷酸重复基元的1.30%。四核苷酸、五核苷酸和六核苷酸重复类型较多,分别有101种、90种和88种重复类型,但数量较少, 仅占总SSR位点的1.33%(表1)。
2.1.3 SSR位点基序重复次数
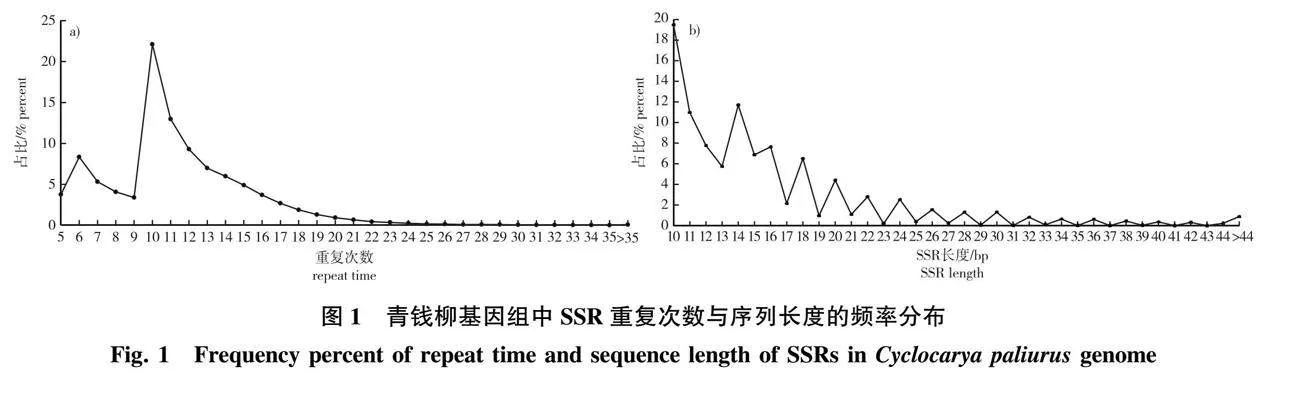
从核苷酸重复次数来看,核苷酸基元重复次数主要集中在5~18次。其中以重复10次最为常见,有19 843个位点,占总数的22.11%;重复5、6、7、8、9、11、12次的SSR位点分别有3 377、7 485、4 767、3 654、3 040、11 636、8 338个;其他重复频率较少(图1)。
单核苷酸重复次数主要集中在10~15次,以重复10次最为常见,有17 485个位点,占单核苷酸总数的31.09%;重复11、12、13、14次的SSR位点分别有9 861个(17.53%)、6 978个(12.41%)、5 155个(9.17%)、4 433个(7.88%)。二核苷酸重复次数主要集中在6~11次,以重复6次最为常见,有6 070个位点,占二核苷酸总数的22.24%;重复7、8、9、10、11次的SSR位点分别有4 123个(15.11%)、3 351个(12.28%)、2 857 个(10.47%)、2 270个(8.32%)、1729(6.34%)个。三核苷酸重复基元主要集中在5~9次,以重复5次最为常见,有2 491个位点,占三核苷酸总数的49.48%;重复6、7、8、9次的SSR位点分别有1 201个(23.95%)、593个(11.83%)、279个(5.56%)、171个(3.41%)。四、五、六核苷酸重复次数主要集中在5~6次,其他重复次数见表1。
2.1.4 SSR序列长度及变异分析
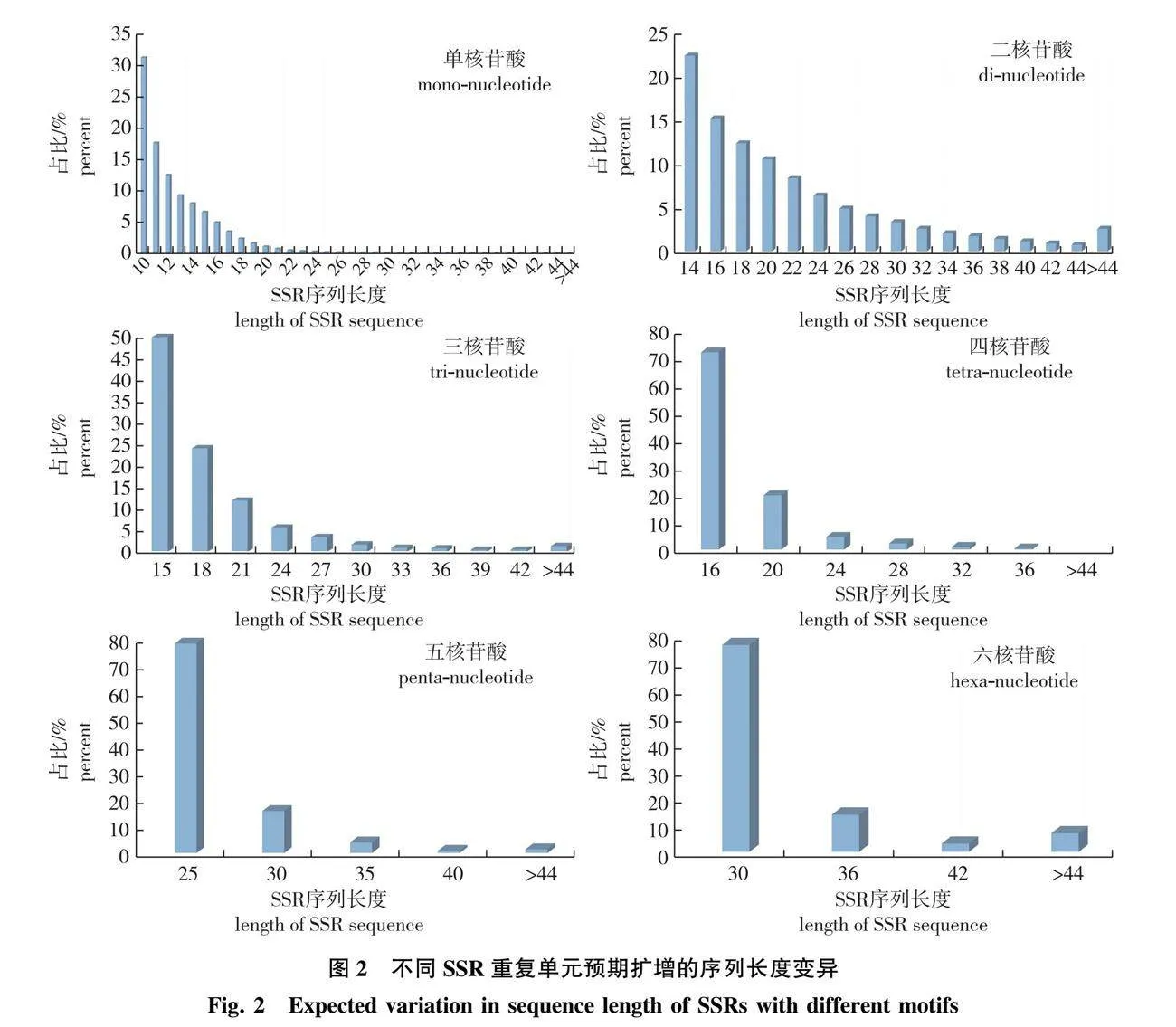
分析发现,SSR序列长度分布于10~476 bp,平均长度为15.29 bp。不同重复单元类型的SSR序列长度具有多态性。随着SSR序列长度的增加,其出现的频次整体呈逐渐下降的趋势(图2)。单核苷酸重复基序最丰富,共有56 242个,占总数的62.67%;平均长度为12.61 bp,其中(C/G)n序列的平均长度为14.84 bp,(A/T)n 平均长度为12.36 bp。其次为二核苷酸重复,有27 291个,占总数的30.41%;平均长度为19.78 bp,其中(AT/AT)n序列的平均长度最大,为21.04 bp,(CG/GC)n平均长度最小,仅13.33 bp。三核苷酸重复的位点共5 014个,占总数的5.59%;平均长度为18.68 bp。其他重复类型较少,分别为四核苷酸重复712个(0.79%),五核苷酸重复351个(0.39%),六核苷酸重复131个(0.15%),平均长度分别为21.67、26.62、33.02 bp(表1)。其中C型(复合型)SSR位点的平均长度为21.52 bp。
此外,不考虑SSR重复基元类型,序列长度为10 bp的SSR位点数量最多,为17 485条,占总位点数的19.48%;其次为长度14 bp的位点,为10 503条,占总位点数的11.70%;其他长度数量及所占频率见图2。
2.2 青钱柳基因组SSR位点的验证和应用
2.2.1 SSR引物设计和验证
应用Primer 3.0对筛选获得的89 741个SSR位点进行引物设计,共设计78 285对SSR引物,引物长度为18~25 bp。其中,从78 296个P型(完全重复型)SSR位点中共设计出68 109对引物,设计成功率为86.99%,占总引物数的87.00%;从11 445个C型SSR位点中共设计出10 176对引物,设计成功率为88.91%,占总数的13.00%。预测扩增产物长度为100~280 bp(引物长度及预计扩增产物长度的数据均来源于Primer设计结果)。在78 285对SSR引物中,预期扩增产物的核心序列为单核苷酸、二核苷酸和三核苷酸重复的SSR引物数分别为44 242对(56.51%)、19 100对(24.40%)和3 843对(4.91%)(图2)。
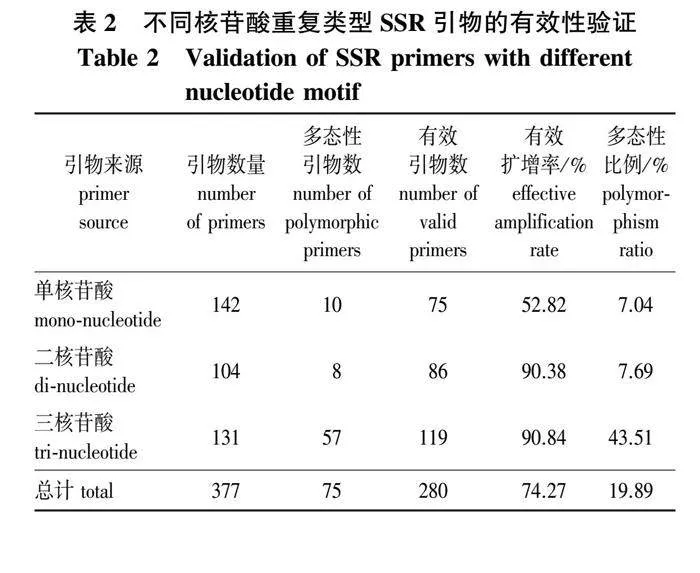
将设计的引物按核心序列的核苷酸重复类型分组,每组随机进行选取,由于单核苷酸重复基序引物占比较大,每组随机选择60对,其他基序重复类型每组随机选择10对(不足10对全部选取),则单碱基和双核苷酸重复共选取引物246对,从19个青钱柳无性系样本提取的DNA中随机选择两个对引物的有效性进行验证。经变性聚丙烯酰胺凝胶电泳检测,其中161对引物能扩增出清晰、重复性好且在预期片段长度范围内的产物,有效扩增率为65.45%;其余85对引物的扩增结果为非目的条带、无扩增产物或条带模糊。三核苷酸重复类型共选出131对SSR引物,用以上方法筛选后结果显示,其中119对引物的扩增条带为清晰、重复性好的目的产物,有效扩增率为90.84%;其余12对引物的扩增结果为非目的条带、无扩增产物或条带模糊(表2)。总体有效扩增率为74.27%,多态性比例为19.89%。
2.2.2 青钱柳优良药用无性系的SSR分子鉴定
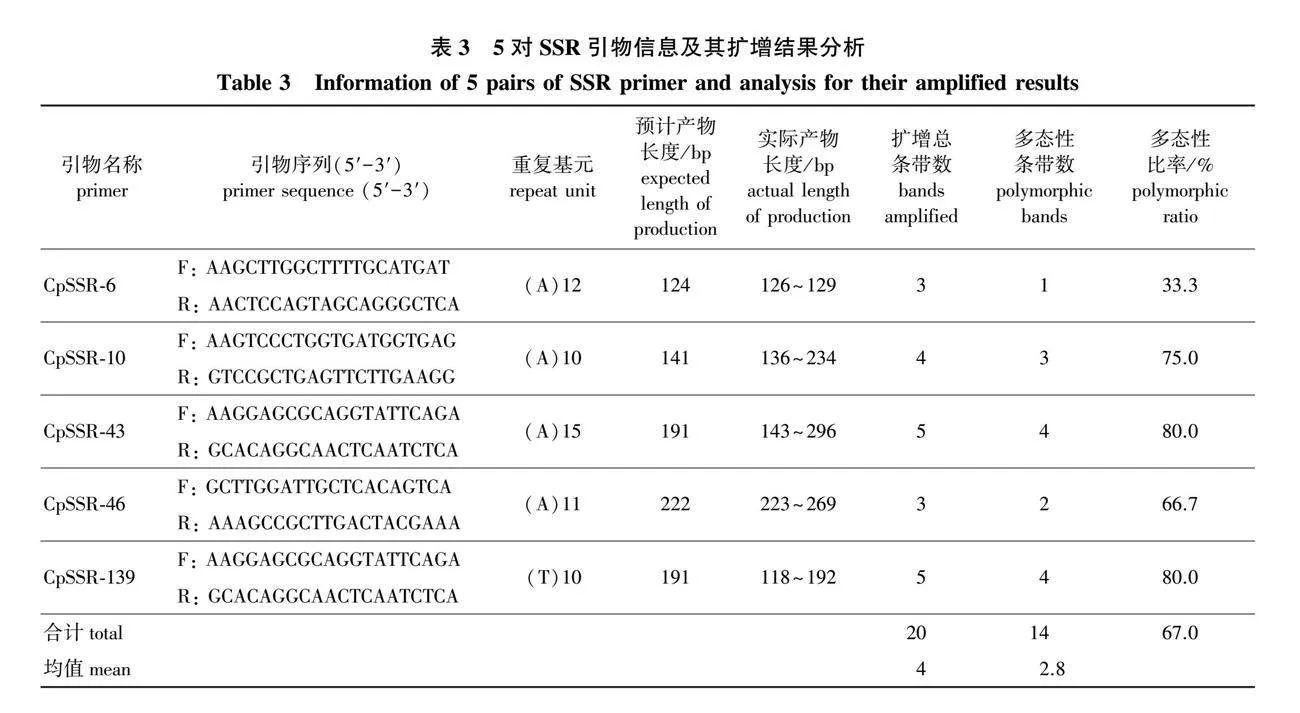
1) SSR引物的有效性和多态性。经变性聚丙烯酰胺凝胶电泳复筛,以19个优良无性系DNA为样本,161对引物中有18对SSR引物的扩增结果具有多态性。从单核苷酸重复类型的多态性引物中选取5对条带清晰、多态性高、稳定好的引物进行毛细管电泳检测。5对SSR引物的扩增产物长度均与预计产物长度吻合。5对引物共扩增出20条条带,其中14条具有多态性,多态性比率为67.0%(表3);引物扩增条带数为3~5条,平均每对引物扩增出4条条带,实际扩增片段大小在118~296 bp。
2) 无性系DNA分子身份证构建。5对多态性SSR引物对19个无性系的扩增产物进行毛细管电泳的统计分析,分别检测到5、5、6、4、2种特征带型(表4)。
利用多态性位点及编码信息构建DNA分子身份证,编码方法为引物名称+片段编码;片段编码为字母格式,每对引物中的A—F分别对应于不同的扩增产物。如LC1样本的分子身份证编码为“CpSSR139ACpSSR10ACpSSR43ACpSSR46A CpSS-R6A”。将各无性系的来源、种质特性和DNA分子身份证编码导入二维码生成软件,获得每个无性系唯一的二维码分子身份证(图3)。
3 讨 论
由于SSR标记的多态性、重复性及稳定性好,长期以来SSR标记在各物种的开发应用广受关注。随着高通量测序技术的不断发展,各物种的基因组信息逐渐被破译,大大加快了SSR标记位点的开发和应用[16,18,24-26]。例如,Lee等[19]通过应用茶树基因组SSR进行种群遗传多样性分析。青钱柳作为中国特有树种,因其独特的药用价值成为各领域的研究热点。但其研究基础薄弱,缺乏基因组序列信息;分子方面的研究也刚起步[27]。在标记位点的研究方面,陈秀娟等[28] 和周一旸等[29]分别利用ISSR和SRAP分子标记分析了青钱柳种质资源亲缘关系及其多样性;Fan等[30]基于AFLP技术开发了少数SSR标记,但其应用效果并不十分理想[15]。而青钱柳基因组测序工作的完成,为SSR标记的开发和应用奠定基础。
本研究从青钱柳基因组中挖掘出89 741个SSR位点,核心序列1~6核苷酸重复均有发生,位点发生频率为62.07%,平均每1.81 kb序列中含有一个SSR位点,此分布频率高于玉米(Zea mays)的1/15.48 kb、短莛飞蓬(Erigeron breviscapus)的1/5.25 kb[31-32]。研究认为,物种基因组越小,单核苷酸重复序列的SSR位点比例越高;随着基因组的增大,其他重复序列的SSR位点会逐渐增多[33-34]。如灯盏花(Erigeron breviscapus)[35]基因组序列全长是青钱柳基因序列全长的8.79倍,但其单核苷酸(32.72%)和二核苷酸(47.14%)重复的SSR序列数量显著少于青钱柳中单核苷酸(62.67%)和二核苷酸(30.41%)重复数量占比;而其他种类重复序列则显著大于青钱柳。
一般来说,随着重复次数的增多,SSR出现的频率会呈指数趋势下降,且SSR标记数量随着重复基元长度的增加而减少,这一现象是由于长微卫星更倾向于突变为短微卫星造成的[33,36]。对青钱柳基因组的分析发现,总SSR的长度分布在10~476 bp,平均为15.29 bp,其中71.09%集中在10~18 bp。由此可见,青钱柳基因组中微卫星重复基元变异较活跃,微卫星发生变异的概率较高。
分析还发现,青钱柳全基因组SSR序列中1~6核苷酸共有354种重复单元变异类型,不同类型SSR重复单元均显示出明显的碱基偏好性。总体上以A/T为主要基序,而以G/C为基序的相对较少,这一研究结果与龙眼(Dimocarpus longan)[33]、四倍体野花生(Arachis monticola)[37]、毛果杨(Populus trichocarpa)[26]和鹅掌楸[17]的研究结果相似。Liu等[38]研究认为,重复比例高的核苷酸序列,可能与重复基元转录翻译合成的蛋白质呈正相关关系。另有研究表明,G/C基序的重复单元少可能是基因组DNA中CpG岛的甲基化造成的,通过脱氨基作用,甲基化的胞嘧啶C很容易转变成胸腺嘧啶T[39]。
为保证SSR标记位点的稳定性和可靠性,研究中一般选择预计扩增产物大小在150~250 bp的完全重复型SSR引物(P型)。本研究发现1~3核苷酸重复的P型引物总多态性比率为19.89%;其中,三核苷酸SSRs的多态性比例(43.51%)远高于单核苷酸(7.04%)和二核苷酸(7.69%)SSRs。可能因为是单/二核苷酸的等位基因差异小,易产生误读或混淆,因此多态性比例较低[40-41],这与Beghè等[42]和Cregan等[43]在核心基序重复数量和多态性比例间相关性的研究结果一致。在青钱柳基因组中三核苷酸以上重复的SSR位点占总SSR的6.92%,占比虽小,但相较单核苷酸和双核苷酸重复基序的位点,更具潜在的开发利用价值。
本研究通过对青钱柳全基因组的扫描,对大量具有潜在应用价值的SSR位点进行查找。利用筛选出的5对多态性SSR引物初步尝试了对19个青钱柳药用无性系的DNA分子身份证构建,此方法不仅可使各个无性系被唯一识别,还可以充分了解其起源和无性系特点。在后续更多多态性引物得到开发的基础上,可针对不断发掘的优良无性系,建立二维码识别系统,通过更多的SSR特异性位点加以识别,形成动态和可准确识别的无性系库,为青钱柳无性系的规模化推广和应用提供技术保障。
参考文献(reference):
[1]方升佐,洑香香.青钱柳资源培育与开发利用的研究进展[J].南京林业大学学报(自然科学版),2007,31(1):95-100.FANG S Z,FU X X.Progress and prospects on silviculture and utilization of Cyclocarya paliurus resources[J].J Nanjing For Univ (Nat Sci Ed),2007,31(1):95-100.DOI: 10.3969/j.issn.1000-2006.2007.01.023.
[2]孙戴妍,尚旭岚,洑香香,等.青钱柳胸径生长和木材密度的地理变异规律[J].南京林业大学学报(自然科学版),2017,41(4):1-5.SUN D Y,SHANG X L,FU X X,et al.Regularity on geographic variation in DBH growth and wood density of Cyclocarya paliurus[J].J Nanjing For Univ (Nat Sci Ed),2017,41(4):1-5.DOI: 10.3969/j.issn.1000-2006.201610005.
[3]侯小利,刘晓霞,王硕,等.青钱柳叶总黄酮对自发性高血压大鼠的影响[J].中药药理与临床,2014,30(2):62-69.HOU X L,LIU X X,WANG S,et al.Effect of the flavonoids from" Cyclocarya paliurus on spontaneous hypertension rats[J].Pharmacol Clin Chin Mater Med,2014,30(2):62-69.DOI: 10.13412/j.cnki.zyyl.2014.02.021.
[4]WU Z F,MENG F C,CAO L J,et al.Triterpenoids from Cyclocarya paliurus and their inhibitory effect on the secretion of apoliprotein B48 in Caco-2 cells[J].Phytochemistry,2017,142:76-84.DOI: 10.1016/j.phytochem.2017.06.015.
[5]YANG Z W,WANG J,LI J G,et al.Antihyperlipidemic and hepatoprotective activities of polysaccharide fraction from Cyclocarya paliurus in high-fat emulsion-induced hyperlipidaemic mice[J].Carbohydr Polym,2018,183:11-20.DOI: 10.1016/j.carbpol.2017.11.033.
[6]郑观涛,殷志琦.药用植物青钱柳的开发研究进展[J].世界最新医学信息文摘,2019,19(43):123-124.ZHENG G T,YIN Z Q.Research progress on development in Cyclocarya paliurus[J].World Latest Med Inf,2019,19(43):123-124.DOI: 10.19613/j.cnki.1671-3141.2019.43.058.
[7]林源,陈培,周明明,等.天然居群青钱柳叶主要生物活性物质及抗氧化活性研究[J].南京林业大学学报(自然科学版),2020,44(2):10-16.LIN Y,CHEN P,ZHOU M M,et al.Key bioactive substances and their antioxidant activities in Cyclocarya paliurus (Batal.) Iljinskaja leaves collected from natural populations[J].J Nanjing For Univ (Nat Sci Ed),2020,44(2):10-16.DOI: 10.3969/j.issn.1000-2006.201901045.
[8]周永晟,徐子恒,袁发银,等.亚热带3个地点青钱柳群落特征比较[J].南京林业大学学报(自然科学版),2021,45(1):29-35.ZHOU Y S,XU Z H,YUAN F Y,et al.Comparisons of community characteristics among three natural forests of Cyclocarya paliurus in the subtropical region of China[J].J Nanjing For Univ (Nat Sci Ed),2021,45(1):29-35.DOI: 10.12302/j.issn.1000-2006.202005017.
[9]SUN C W,ZHOU Y S,FANG S Z,et al.Ecological gradient analysis and environmental interpretation of Cyclocarya paliurus communities[J].Forests,2021,12(2):146.DOI: 10.3390/f12020146.
[10]SUN C W,SHANG X L,DING H F,et al.Natural variations in flavonoids and triterpenoids of Cyclocarya paliurus leaves[J].J For Res,2021,32(2):805-814.DOI: 10.1007/s11676-020-01139-1.
[11]ZHOU M M,QUEK S Y,SHANG X L,et al.Geographical variations of triterpenoid contents in Cyclocarya paliurus leaves and their inhibitory effects on HeLa cells[J].Ind Crops Prod,2021,162:113314.DOI: 10.1016/j.indcrop.2021.113314.
[12]田力,徐骋炜,尚旭岚,等.青钱柳药用优良单株评价与选择[J].南京林业大学学报(自然科学版),2021,45(1):21-28.TIAN L,XU C W,SHANG X L,et al.Evaluation and selection on superior individuals for medicinal use of Cyclocarya paliurus[J].J Nanjing For Univ (Nat Sci Ed),2021,45(1):21-28.DOI: 10.12302/j.issn.1000-2006.202002018.
[13]王久利,朱明星,徐明行,等.基于RAD-seq技术的异型花SSR信息分析[J].植物研究,2017,37(3):447-452,460.WANG J L,ZHU M X,XU M H,et al.Analysis on SSR in Sinoswertia tetraptera base on RAD-seq[J].Bull Bot Res,2017,37(3):447-452,460.DOI: 10.7525/j.issn.1673-5102.2017.03.016.
[14]GONZAGA Z J,ASLAM K,SEPTININGSIH E M,et al.Evaluation of SSR and SNP markers for molecular breeding in rice[J].Plant Breed Biotech,2015,3(2):139-152.DOI: 10.9787/pbb.2015.3.2.139.
[15]LI X C,FU X X,SHANG X L,et al.Natural population structure and genetic differentiation for heterodicogamous plant:Cyclocarya paliurus (Batal.) Iljinskaja (Juglandaceae)[J].Tree Genet Genomes,2017,13(4):80.DOI: 10.1007/s11295-017-1157-5.
[16]SAVIC' A,PIPAN B,VASIC' M,et al.Genetic diversity of common bean (Phaseolus vulgaris L.) germplasm from Serbia,as revealed by single sequence repeats (SSR)[J].Sci Hortic,2021,288:110405.DOI: 10.1016/j.scienta.2021.110405.
[17]LI B,LIN F R,HUANG P,et al.Development of nuclear SSR and chloroplast genome markers in diverse Liriodendron chinense germplasm based on low-coverage whole genome sequencing[J].Biol Res,2020,53(1):21.DOI: 10.1186/s40659-020-00289-0.
[18]LI C H,ZHENG Y Q,LIU Y,et al.Development of genomic SSR for the subtropical hardwood tree Dalbergia hupeana and assessment of their transferability to other related species[J].Forests,2021,12(6):804.DOI: 10.3390/f12060804.
[19]LEE K J,LEE J R,SEBASTIN R,et al.Assessment of genetic diversity of tea germplasm for its management and sustainable use in Korea genebank[J].Forests,2019,10(9):780.DOI: 10.3390/f10090780.
[20]王希,陈丽,赵春雷.利用MISA工具对不同类型序列进行SSR标记位点挖掘的探讨[J].中国农学通报,2016,32(10):150-156.WANG X,CHEN L,ZHAO C L.Mining SSR molecular marker sites with MISA tool for different types of sequences[J].Chin Agric Sci Bull,2016,32(10):150-156.
[21]UNTERGASSER A,CUTCUTACHE I,KORESSAAR T,et al.Primer3:new capabilities and interfaces[J].Nucleic Acids Res,2012,40(15):e115.DOI: 10.1093/nar/gks596.
[22]乔舒婷,董文其,胡齐赞,等.基于丝瓜全基因组序列SSR分子标记开发[J].分子植物育种,2023,21(6):1937-1947.QIAO S T,DONG W Q,HU Q Z,et al.Development of SSR molecular markers based on whole genome sequences of sponge gourd[J].Mol Plant Breed,2023,21(6):1937-1947.DOI: 10.13271/j.mpb.021.001937.
[23]郭艳春,张力岚,陈思远,等.黄麻应用核心种质的DNA分子身份证构建[J].作物学报,2021,47(1):80-93.GUO Y C,ZHANG L L,CHEN S Y,et al.Establishment of DNA molecular fingerprint of applied core germplasm in jute (Corchorus spp.)[J].Acta Agron Sin,2021,47(1):80-93.DOI: 10.3724/SP.J.1006.2021.04022.
[24]XIA E H,ZHANG H B,SHENG J,et al.The tea tree genome provides insights into tea flavor and independent evolution of caffeine biosynthesis[J].Mol Plant,2017,10(6):866-877.DOI: 10.1016/j.molp.2017.04.002.
[25]宋立肖,李国旗,靳长青,等.大麻状罗布麻的全基因组分析和SSR标记开发[J].植物遗传资源学报,2019,20(5):1309-1316.SONG L X,LI G Q,JIN C Q,et al.Whole genome sequencing and development of SSR markers in Apocynum cannabinum[J].J Plant Genet Resour,2019,20(5):1309-1316.DOI: 10.13430/j.cnki.jpgr.20181218002.
[26]崔哲,左力辉,韩坤瑾,等.毛果杨(Populus trichocarpa)全基因组SSR位点分布规律[J].分子植物育种,2020,18(11):3683-3692.CUI Z,ZUO L H,HAN K J,et al.Distribution rule of SSR loci in whole genome of Populus trichocarpa[J].Mol Plant Breed,2020,18(11):3683-3692.DOI: 10.13271/j.mpb.018.003683.
[27]蒋向辉,苑静,王翔.青钱柳叶片转录组数据组装及基因功能注释[J].华中师范大学学报(自然科学版),2018,52(6):822-831.JIANG X H,YUAN J,WANG X.De novo transcriptome assembly and annotation of the leaves of Cyclocarya paliurus[J].J Cent China Norm Univ (Nat Sci),2018,52(6):822-831.DOI: 10.19603/j.cnki.1000-1190.2018.06.012.
[28]陈秀娟,柏明娥,王丽玲,等.青钱柳种质资源亲缘关系的ISSR分析评价[J].中国林副特产,2016(4):6-10.CHEN X J,BAI M E,WANG L L,et al.ISSR analysis and evaluation of genetic relationship of Cyclocarya paliurus germplasm resources[J].For Prod Speciality China,2016(4):6-10.DOI: 10.13268/j.cnki.fbsic.2016.04.002.
[29]周一旸,洑香香,尚旭岚,等.青钱柳种质资源多样性SRAP初步分析[J].基因组学与应用生物学,2011,30(1):40-46.ZHOU Y Y,FU X X,SHANG X L,et al.Preliminary study on the genetic diversity of germplasm for Cyclocarya paliurus revealed by SRAP markers[J].Genom Appl Biol,2011,30(1):40-46.DOI: 10.3969/gab.030.000040.
[30]FAN D M,YE L J,LUO Y,et al.Development of microsatellite loci for Cyclocarya paliurus (Juglandaceae),a monotypic species in subtropical China[J].Appl Plant Sci,2013,1(6):apps.1200524.DOI: 10.3732/apps.1200524.
[31]XU J,LIU L,XU Y B,et al.Development and characterization of simple sequence repeat markers providing genome-wide coverage and high resolution in maize[J].DNA Res,2013,20(5):497-509.DOI: 10.1093/dnares/dst026.
[32]HE S M,DONG X,ZHANG G H,et al.High quality genome of Erigeron breviscapus provides a reference for herbal plants in Asteraceae[J].Mol Ecol Resour,2021,21(1):153-169.DOI: 10.1111/1755-0998.13257.
[33]林恩文,林榕榕,陈钦常,等.龙眼全基因组和转录本序列SSR位点的鉴定[J].福建农林大学学报(自然科学版),2022,51(4):493-501.LIN E W,LIN R R,CHEN Q C,et al.SSR loci analysis in genome and transcriptome of Longan[J].J Fujian Agric For Univ (Nat Sci Ed),2022,51(4):493-501.DOI: 10.13323/j.cnki.j.fafu(nat.sci.).2022.04.007.
[34]KARAOGLU H,LEE C M Y,MEYER W.Survey of simple sequence repeats in completed fungal genomes[J].Mol Biol Evol,2005,22(3):639-649.DOI: 10.1093/molbev/msi057.
[35]刘松卫,卢迎春,宋婉玲,等.基于灯盏花全基因组SSR位点分析及多态性引物开发[J].分子植物育种,2018,16(12):4003-4009.LIU S W,LU Y C,SONG W L,et al.SSR loci analysis based on Erigeron breviscapus genome and polymorphism primers development[J].Mol Plant Breed,2018,16(12):4003-4009.DOI: 10.13271/j.mpb.016.004003.
[36]CARDLE L,RAMSAY L,MILBOURNE D,et al.Computational and experimental characterization of physically clustered simple sequence repeats in plants[J].Genetics,2000,156(2):847-854.DOI: 10.1093/genetics/156.2.847.
[37]王玉龙,黄冰艳,王思雨,等.四倍体野生种花生(A.monticola)全基因组SSR的开发与特征分析[J].中国农业科学,2019,52(15):2567-2585.WANG Y L,HUANG B Y,WANG S Y,et al.Development and characterization of whole genome SSR in tetraploid wild peanut(Arachis monticola)[J].Sci Agric Sin,2019,52(15):2567-2585.DOI: 10.3864/j.issn.0578-1752.2019.15.002.
[38]LIU S R,LI W Y,LONG D,et al.Development and characterization of genomic and expressed SSRs in citrus by genome-wide analysis[J].PLoS One,2013,8(10):e75149.DOI: 10.1371/journal.pone.0075149.
[39]SCHORDERET D F,GARTLER S M.Analysis of CpG suppression in methylated and nonmethylated species[J].Proc Natl Acad Sci USA,1992,89(3):957-961.DOI: 10.1073/pnas.89.3.957.
[40]宋莎,冯建文,吴亚维,等.基于RAD-seq技术的花红SSR信息分析[J].贵州农业科学,2019,47(11):103-106.SONG S,FENG J W,WU Y W,et al.Analysis on SSR in Malus asiatica Nakai.base on RAD sequencing[J].Guizhou Agric Sci,2019,47(11):103-106.DOI: 10.3969/j.issn.1001-3601.2019.11.021.
[41]周晓君,王海亮,李方玲,等.基于RAD-seq技术开发灵宝杜鹃多态性SSR标记[J].农业生物技术学报,2019,27(1):55-62.ZHOU X J,WANG H L,LI F L,et al.Development of polymorphic SSR markers in Rhododendron henanense subsp.lingbaoense based on RAD-seq[J].J Agric Biotechnol,2019,27(1):55-62.DOI: 10.3969/j.issn.1674-7968.2019.01.006.
[42]BEGH D,MOLANO J F G,FABBRI A,et al.Olive biodiversity in Colombia.A molecular study of local germplasm[J].Sci Hortic,2015,189:122-131.DOI: 10.1016/j.scienta.2015.04.003.
[43]CREGAN P B,JARVIK T,BUSH A L,et al.An integrated genetic linkage map of the soybean genome[J].Crop Sci,1999,39(5):1464-1490.DOI: 10.2135/cropsci1999.3951464x.
(责任编辑 吴祝华)
收稿日期Received:2022-06-01""" 修回日期Accepted:2022-09-05
基金项目:国家自然科学基金项目(31971642);中央财政林业科技推广示范项目(皖[2023]TG13号)。
第一作者:刘莉(943444013@qq.com)。
*通信作者:(xxfu@njfu.edu.cn),洑香香,教授。
引文格式:刘莉,瞿印权,余延浩,等. 青钱柳全基因组SSR位点分析及多态性引物开发[J]. 南京林业大学学报(自然科学版),2024,48(4):67-75.
LIU L, QU Y Q, YU Y H, et al. Analysis of SSR locus based on the whole genome sequences of Cyclocarya paliurus and the" development of polymorphic primers[J]. Journal of Nanjing Forestry University (Natural Sciences Edition),2024,48(4):67-75.
DOI:10.12302/j.issn.1000-2006.202206001.

