3D UNeXt:轻量级快速脑提取网络
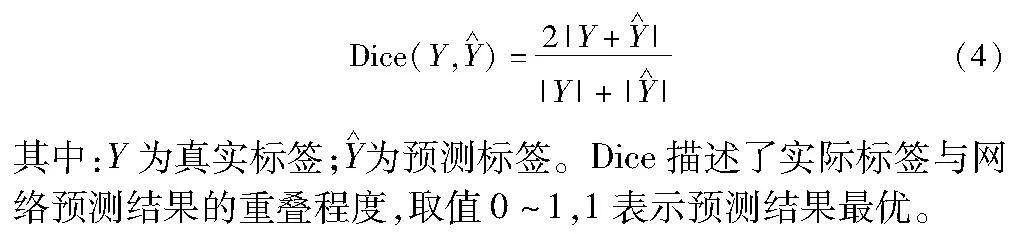



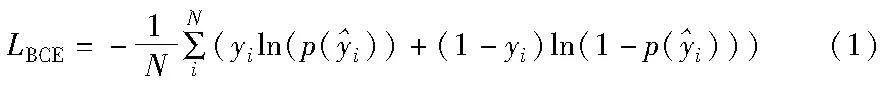

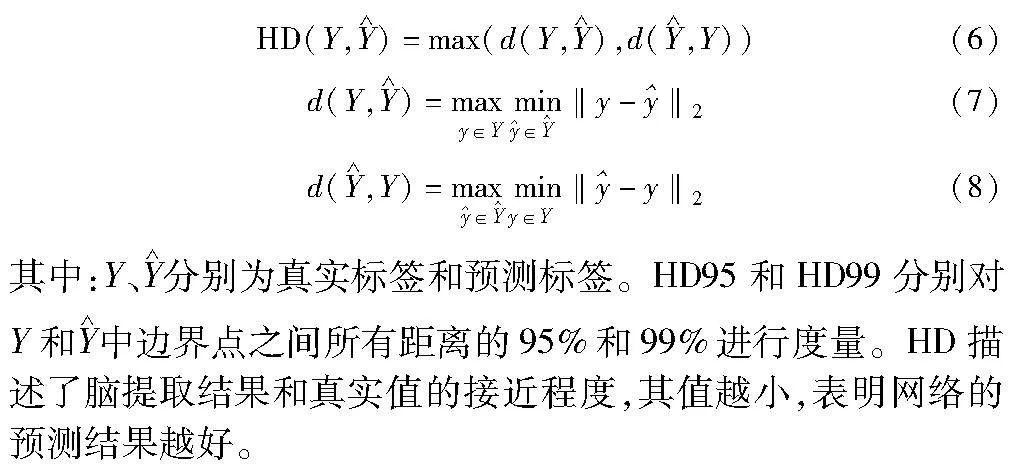

摘 要:为了解决现有脑提取网络结构复杂、参数量大且推理速度不高的问题,受UNeXt启发,提出一种基于3D卷积、3D多层感知机(multilayer perception,MLP)和多尺度特征融合的轻量级快速脑提取网络3D UNeXt,极大地减少了参数和浮点运算量,取得了令人满意的结果。3D UNeXt以U-Net为基本架构,在编码阶段使用3D卷积模块获取局部特征;在瓶颈阶段通过3D MLP模块获取全局特征和特征之间的远程依赖;在解码阶段借助多尺度特征融合模块高效融合浅层特征和深层特征。特别地,3D MLP模块在三个不同特征轴向进行线性移位操作,以获取不同维度特征的全局感受野并建立它们之间的远程依赖。在IBSR、NFBS和HTU-BrainMask三个数据集上进行实验,以和先进网络进行对比。实验结果表明,3D UNeXt在网络参数、浮点运算量、推理精度和速度等方面显著优于现有模型。
关键词:脑提取; 深度神经网络; U-Net; 多尺度特征融合; 3D MLP
中图分类号:TP391 文献标志码:A
文章编号:1001-3695(2024)06-040-1876-06
doi:10.19734/j.issn.1001-3695.2023.09.0405
3D UNeXt: lightweight and efficient network for effective brain extraction
Abstract:In order to solve the drawbacks of existing brain extraction network, i.e., complex network structure, large amounts of parameters and low inference speed, this paper proposed a novel network 3D UNeXt for fast and effective brain extraction. 3D UNeXt greatly reduced parameters and the number of floating point operators(FLOPs), and achieved promising results with the combination of 3D convolution, 3D MLP and multi-scale feature fusion. 3D UNeXt used U-Net as the basic architecture and employed 3D convolutional modules to obtain local features in encoding stage. Specifically, the proposed 3D MLP module at the bottleneck stage enhanced the extraction of global features and long-range dependencies among them. In decoding stage, this paper designed a lightweight multiscale feature fusion module to effectively fuse multiscale low-level features and high-level counterparts. In detail, the 3D MLP module performed linear shift operations in three different axes to obtain global receptive fields from different dimension features and establish long-range dependencies among them. This paper compared 3D UNeXt with other counterparts on three datasets:IBSR, NFBS, and HTU-BrainMask. Experimental results show that the 3D UNeXt is superior over other baselines in terms of network parameters, FLOPs, inference accuracy, and inference speed.
Key words:brain extraction; deep neural network; U-Net; multi-scale feature fusion; 3D MLP
0 引言
脑提取是脑神经影像临床分析和诊断的基础,为脑图像配准[1]、脑区分割、脑体积测量和脑神经外科手术方案制定等提供诊断和决策依据[2,3]。脑提取的精度和速度决定着脑神经影像临床分析和诊断的效果。脑提取的临床金标准由影像科医生以半手工方式得到:他们首先使用FSL[4,5]等软件自动得到人脑的初始提取掩码,之后再手工对掩码进行修正。一名有经验的影像科医生完成一例脑提取操作通常需要花费30~40 min。显然该过程存在着劳动强度大、依赖专家和效率低下等问题。
随着计算机视觉和深度学习研究的进展,深度神经网络被广泛应用到医学图像分类和分割等领域[6]。以卷积神经网络(convolutional neural network,CNN)为代表的深度神经网络[7],使用卷积提取图像特征,能够从大量训练样本中学习到高层语义。这使得CNN成为早期应用于脑提取的首选网络架构[8,9]。
传统CNN的网络结构复杂、参数量大,在网络训练和推理阶段需要消耗大量的计算资源,并且随着网络深度的增加,CNN容易丢失浅层网络的信息。这影响了CNN的分割效率和精度。U-Net[10]设计了编码器和解码器架构,并在对应的编码单元和解码单元之间引入跳跃连接。编码单元和解码单元的卷积层分别进行下采样和上采样,以提取医学图像的低级特征和高级特征;跳跃连接将对应的低级特征和高级特征进行残差连接,以降低梯度消失对网络性能的影响,并实现特征融合。
近年来,基于U-Net的多种网络变体被相继提出。Ibtehaz等人[11]设计了MultiResUNet,它将带残差连接的MultiRes模块与U-Net结合,表现出较好的分割性能。Wang等人[12]提出的Non-local U-Net将注意力机制、编码器和解码器相结合,以获取更大的感受野,解决卷积操作存在的全局信息缺失问题。3D U-Net[13]是U-Net在3D体数据上的拓展,它使用3D卷积和反卷积分别替换2D卷积和反卷积。3D U-Net借助较小的3D卷积核学习3D体数据的局部特征,通过多层卷积的叠加实现对全局特征的提取。王磐等人[14]在3D U-Net的基础上提出DA 3D-UNet,在解码器的最后两层加上空间和通道双注意力模块以学习更大范围的全局特征,提高分割精度。Jin等人[15]为3D U-Net引入残差注意力模块[16],提出了混合残差和注意机制的RA-UNet。该网络使用堆叠的残差模块,在增加网络深度并细化对图像细节关注的同时,解决梯度消失的影响。
为获得特征之间的远程依赖,基于注意力机制的Transformer[17]被集成到U-Net,Chen等人[18]提出的TransUNet融合了Transformer和U-Net,该网络使用Transformer对输入信息进行编码,以保留图像分割结果的细节。Cao等人[19]提出的SwinUNet将Transformer和U-Net架构结合,以学习特征之间的远程依赖。Xu等人[20]在LeViT[21]的基础上提出LeViT-UNet,将Transformer和2D卷积的优势结合,在提高分割精度的同时不显著消耗计算资源。Wang 等人[22]设计一种新的注意力模块Mixed Transformer,以同时学习单个训练样本和所有训练样本之间的关联信息,提高U-Net的分割精度。然而,Transformer的复杂运算导致这些网络的参数量和浮点操作(floating points operators,FLOPs)数量很大,在训练和推理阶段消耗更多的计算资源。这使得这些方法很难被部署在计算资源受限的临床环境[23,24]。
最近,MLP[25]开始被视觉领域关注和研究。MLP通过全连接层和非线性激活函数学习不同位置特征之间的远程依赖;其中的线性操作消耗更少的计算资源。Lian等人[26]设计了包含一种轴向移位策略的AS-MLP,以学习不同位置特征之间的远程依赖。Valanarasu等人[27]提出的UNeXt融合了2D卷积和MLP;在网络浅层使用2D卷积学习局部特征,在网络深lRS0Y4MGAMBoANVfyBIkuSeDLF6YpkbvpCBl+LL6KYU=层使用MLP学习特征间的远程依赖。Yu等人[28]提出仅包含MLP模块的S2-MLP,该网络仅在通道方向对特征进行MLP操作,以实现图像块之间的信息交互并减少计算开销。Pang等人[29]提出混合Transformer和MLP的3D Shuffle-Mixer。该网络使用Transformer和MLP分别学习空间上下文信息和全局信息。Tu等人[30]提出多轴门控MLP将输入特征分为两路,使用MLP分别提取它们蕴涵的局部特征和全局特征。MLP有效减少了UNeXt的参数数量,克服了现有基于CNN和Transformer的2D网络参数过多和计算复杂度过高的不足。
以U-Net为骨干的卷积网络使用多层卷积,扩大感受野以学习高层语义信息。然而随着卷积层的叠加,这类网络的参数量不断增加、计算复杂度显著提高。将Transformer引入U-Net架构,虽然能够提高其对特征远程依赖的学习能力,但是带来了更多的计算开销。UNeXt将CNN和MLP集成到U-Net框架,实现了对2D医学图像的快速和准确分割。但是,脑提取的处理对象为3D体数据,使用UNeXt直接进行脑提取时只能将3D体数据划分为2D切片送入网络。这存在两个问题:a)2D切片的划分破坏了原始3D体数据包含的空间信息,造成脑提取精度下降;b)UNeXt仅使用跳跃连接融合同一尺度的浅层特征和深层特征,不能融合蕴涵不同语义级别信息的多尺度特征,也不能进一步提高脑提取的精度。
针对上述问题,本文对UNeXt进行改进,设计一种新的面向3D体数据的轻量级快速脑提取网络3D UNeXt。其主要贡献如下:a)提出一种新的3D MLP模块,该模块在网络深层,沿宽度、高度和深度三个不同轴向对特征张量进行移位,以线性计算开销融合不同位置的特征,之后使用尺寸较小的全连接层获取特征之间的远程依赖;b)设计一种融合不同尺度和不同层次特征的多尺度特征融合模块,该模块通过3D卷积和三线性插值实现不同尺度特征之间的尺寸对齐,之后通过小尺寸3D卷积实现特征之间的高效融合;c)提出一个轻量级快速脑提取网络3D UNeXt,并在公有和私有数据集上进行了充分实验,对比3D UNeXt和其他主流方法用于脑提取时的性能,结果表明,3D UNeXt的脑提取精度不低于当前先进网络,其计算复杂度也显著优于这些网络。
1 3D UNeXt
1.1 网络结构
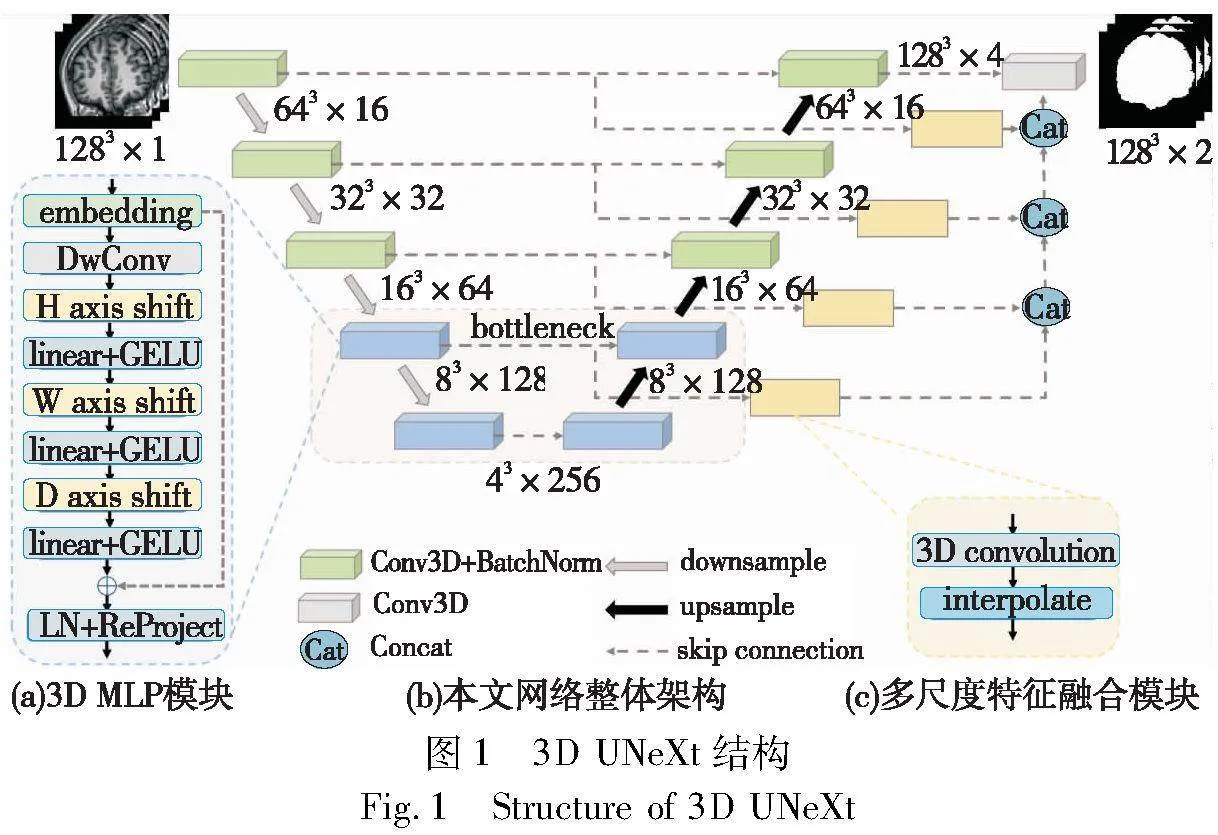
图1展示了3D UNeXt的基本架构和关键模块。该网络包括编码器、网络瓶颈和解码器三部分。编码器使用3D卷积提取医学影像体数据的局部特征;网络瓶颈使用3D MLP学习特征间的远程依赖;解码器使用多尺度特征融合模块对不同尺度的特征进行融合。
编码器包含五个模块:从上到下,前三个模块均由一个3D卷积层、一个批次规范化层(batch normalization, BN)和一个最大池化层(max pooling)组成。后两个模块均由一个3D卷积层、一个3D MLP层和一个规范化层(layer normalization,LN)组成。输入的3D 医学影像体数据经过前三个模块进行下采样,得到局部特征表达。接着,这些特征通过后两个模块以提取它们之间的远程依赖。最大池化层的引入提高了模型的泛化能力,并缩小了特征图的尺寸,从而减小计算开销。每经过编码器的一个模块,特征图的尺寸减少一半、通道数增加一倍。
解码器由五个模块组成,并与编码器的五个模块一一对应。从下至上,前两个模块均由一个3D卷积层、一个BN层、一个三线性插值层、一个3D MLP模块和一个LN层组成。后三个模块均由一个3D卷积层、一个BN层和一个三线性插值层组成。上述各模块增加了修正线性单元(rectified linear unit, ReLU)以增强解码器的泛化性能。每经过一个模块,特征图的尺寸增加一倍、通道数减少一半。
网络瓶颈包括编码器底部和解码器底部的各两个模块。如前所述,网络瓶颈的每一个模块由一个3D卷积层、一个3D MLP层和一个LN层组成。相比于输入图像的原始尺寸,网络瓶颈的输入特征图尺寸显著减少,并包含更多的全局信息和高层语义信息。其中3D卷积层用于平滑特征图的噪声并提取全局特征,3D MLP层则通过线性移位操作和全连接进一步挖掘特征间的远程依赖。
图1(c)展示了本文设计的特征融合模块。为高效融合不同尺度的浅层特征和深层特征且不增加网络复杂度,该模块被放置到编码模块和对应的解码模块之间。对前四个编码模块进行输出,该特征融合模块通过一个3×3×3的3D卷积和三线性插值,将这些输出恢复到输入图像原始尺寸;之后,将恢复的特征图与解码模块输出的特征图进行拼接;最后,拼接后的特征图经过一个1×1×1的3D卷积进行融合。
1.2 3D MLP
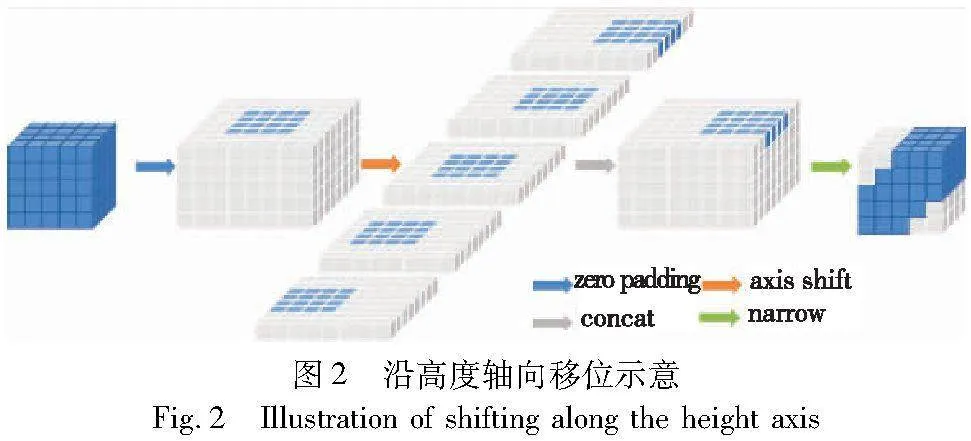
如图1(a)所示,3D MLP模块包含五部分,即嵌入层(embedding)、深度卷积层(depth-wise convolution,DWConv)、移位层(shift)、线性层(linear)和高斯误差线性单元(Gaussian error linear units,GELU)[31]。嵌入层使用核为3×3×3的3D卷积提取特征并对其进行规范化。对于所得特征的每一个通道,DWConv层分别使用一个独立的2D卷积提取单通道的深层语义信息。与通常的3D卷积相比,这种方式显著减少了参数数量和计算开销。移位层分别沿高度、宽度和深度三个轴向对特征图进行移位。图2显示了在高度轴向上的移位操作:首先将特征图平均切分为5个分区;之后将每个分区沿高度轴方向分别移动-2、-1、0、1、2个位置;最后拼接移动后的分区并得到新的特征图。拼接后的特征图经过线性层获取不同空间位置特征之间的远程依赖。GELU层为3D MLP模块引入非线性激活机制,以提高模型的泛化能力。
3D MLP模块的末端包含一个残差连接、一个LN层和ReProject层。残差连接将原始输入特征和末端输出的特征进行相加以对抗梯度消失;LN层对相加特征进行归一化;ReProject层将特征图从底维空间投影至原始高维空间,以恢复其蕴涵的空间信息。
1.3 损失函数
二元交叉熵(binary cross entropy,BCE)和骰子损失(dice loss,DL)被用作损失函数。BCE损失函数的计算式为
DL损失函数的计算式为
总损失函数L为LBCE与LDL的加权和为
L=LBCE+αLDL(3)
其中:α>0为超参数,本文实验取α=0.4。
1.4 脑提取流程
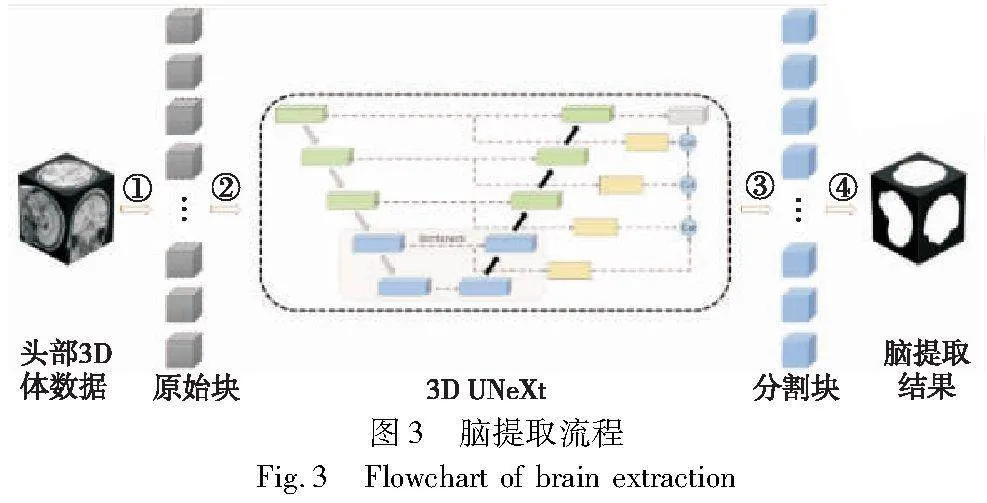
对于一例头部3D体数据,本文提出如图3所示的脑提取流程,共包含四个步骤:a)原始块划分,将头部3D体数据均匀划分为大小相等的体数据块,本文取体数据块的大小为128×128×128;b)逐块分割,将原始块送入3D UNeXt,分别进行脑提取;c)得到初始结果,得到各个图像块的脑提取结果;d)结果合并,将各个图像块的脑提取结果对齐到原始头部3D影像,得到最终的脑提取结果。
2 实验
2.1 数据集
实验使用三个数据集:IBSR(https://www.nitrc.org/projects/ibsr)、NFBS(http://preprocessed-connectomes-project.org/NFB_skullstripped)和HTU-BrainMask。
IBSR包含18例T1加权的头部3D医学影像及对应的人脑标签。其中每例影像的尺寸为256×256×128、分辨率为1 mm×1 mm×2 mm。NFBS包含125例T1加权的头部3D医学影像及其对应的人脑标签。其中每例影像的尺寸为192×256×256、分辨率为1 mm×1 mm×1 mm。HTU-BrainMask是本文收集的私有数据集,包含482例T1加权的头部3D医学影像,这些影像从IXI数据集(https://brain-development.org/ixi-dataset)选取。其中每例影像的尺寸为256×256×150、分辨率为0.9 mm×0.9 mm×1.2 mm。首先使用FSL对这些影像进行脑提取,之后由放射科医师手工修正提取结果以得到最终的人脑标签。
2.2 数据预处理
使用FSL将原始数据投影到MNI(montreal neurological institute)标准空间[32],并将其分辨率转换为1 mm×1 mm×1 mm。在训练阶段,使用随机裁剪进行数据增强:对同一训练批次的每例影像,一个128×128×128的块被随机裁出并送入网络。在推理阶段,使用滑动窗口将每例影像划分为多个128×128×128的重叠块并依次送入网络。之后组合所有重叠块的分割结果以得到最终结果。
2.3 实验环境
使用PyTorch实现本文网络,在一台配有一块NVIDIA A100 GPU、一块Intel Xeon Gold 6348 @ 2.60 GHz CPU的计算机上进行实验。使用Adam优化器,初始学习率为0.003,权重衰减系数为2E-6,训练批次为4。每50轮验证一次,若网络连续500轮不优化则结束训练。
2.4 评价指标
使用Dice、MSD、HD95和HD99对不同方法的脑提取结果进行评价。
Dice的计算式为
MSD的计算式为
HD的计算式为
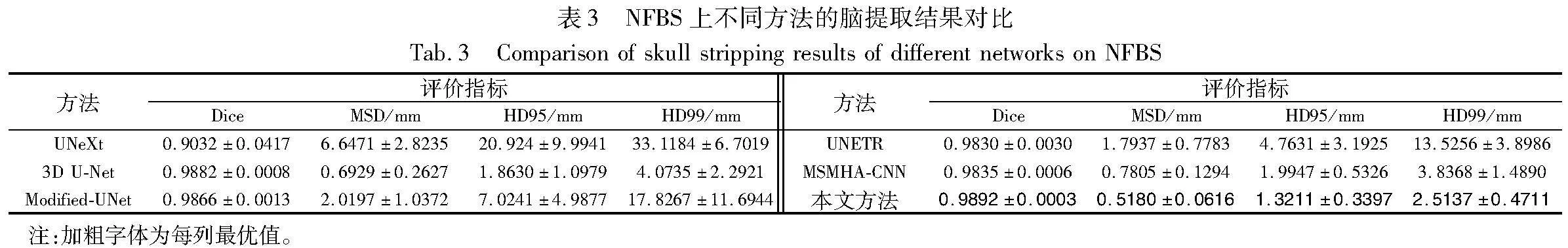
2.5 结果对比
在上述三个数据集上分别进行三折交叉验证,对比本文方法与其他主流网络的性能。
2.5.1 计算复杂度
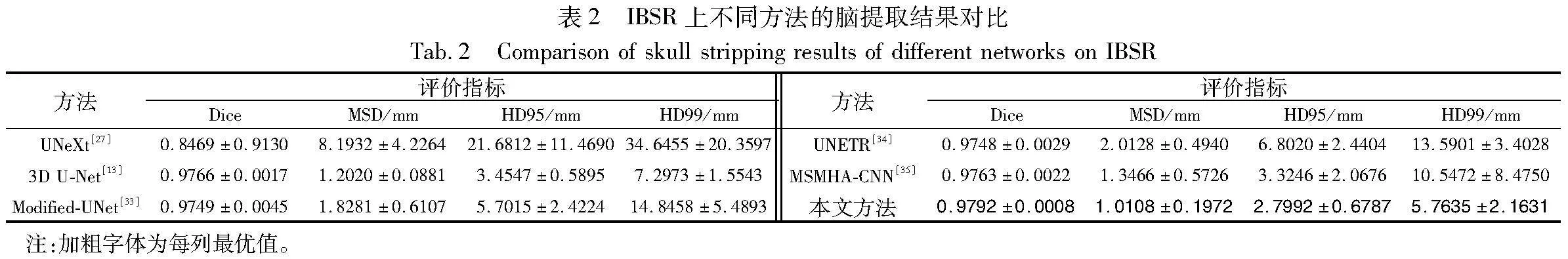
表1比较了3D UNeXt与其他主流网络的参数量、FLOPs和平均CPU推理时间。对于UNeXt,本文将3D体数据划分为2D切片,之后将这些切片逐个送入网络。从表中可以看出,UNeXt的训练参数、FLOPs和CPU推理时间均最小,分别为1.47 M、0.31 G和2.22 s。本文网络的FLOPs和CPU推理时间次之,分别为8.77 G和2.54 s。这是因为UNeXt使用了2D卷积和2D MLP。因为过多使用3D卷积或Transformer模块,3D U-Net、Modified-UNet、UNETR和MSMHA-CNN的FLOPs和CPU推理时间均明显大于本文网络。相反,本文网络使用较少的3D卷积,代之以3D MLP模块,从而显著降低了FLOPs和CPU推理时间。特别地,与经典的3D U-Net相比,本文网络的FLOPs下降98.58%、CPU推理时间缩短91.5%。
2.5.2 定量结果
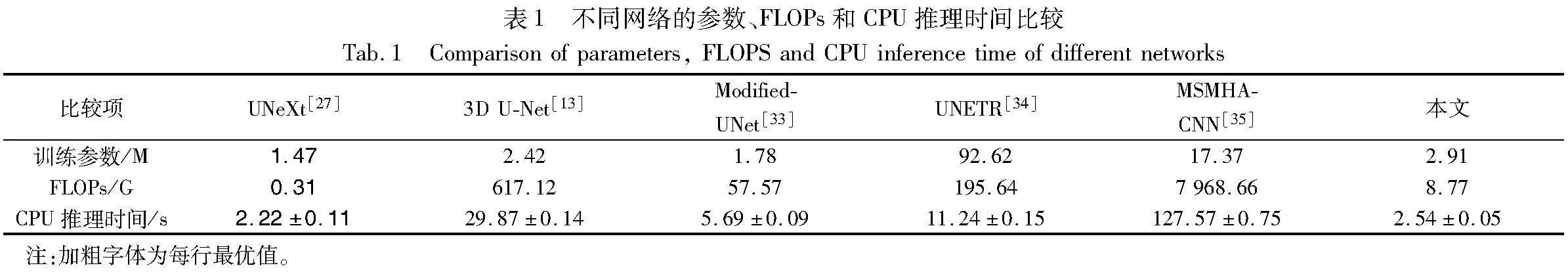
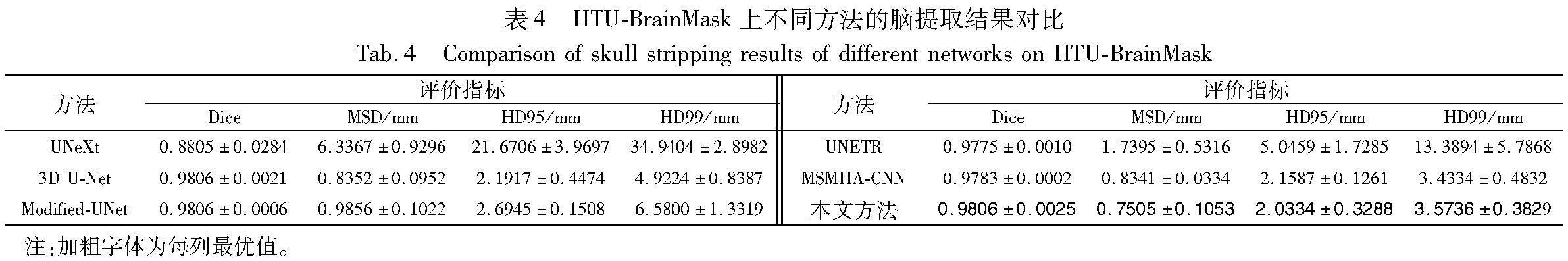
表2~4分别定量比较了3D UNeXt和其他主流方法在三个不同数据集上的脑提取精度。在IBSR数据集上,本文网络的Dice、MSD、HD95和HD99分别为0.979 2、1.010 8 mm、2.799 2 mm和5.763 5 mm,均优于其他方法。在NFBS数据集上,本文网络的Dice、MSD、HD95和HD99分别为0.989 2、0.518 0 mm、1.321 1 mm和2.513 7 mm,在所有方法中均为最优。在HTU-BrainMask数据集上,本文网络的Dice、MSD、HD95和HD99分别为0.980 6、0.750 5mm、2.033 4 mm和3.573 6 mm,均接近或优于其他网络。这是因为本文网络在网络深层使用3D MLP模块获取特征之间的远程依赖,在解码阶段使用多尺度特征融合模块实现浅层特征和深层特征的高效融合,从而显著提高了脑提取精度。UNeXt的输入为2D切片,丢弃了3D体数据蕴涵的空间信息,这导致其颅脑剥离效果最差,虽然其计算复杂度最低。3D U-Net、Modified-UNet、UNETR和MSMHA-CNN采用3D卷积或Transformer获取局部特征或特征间的远程依赖,取得了较好的剥离效果,但是这些方法并没有充分获取上述信息,并且它们的FLOPs和平均CPU推理时间都显著大于本文网络。
2.5.3 定性结果
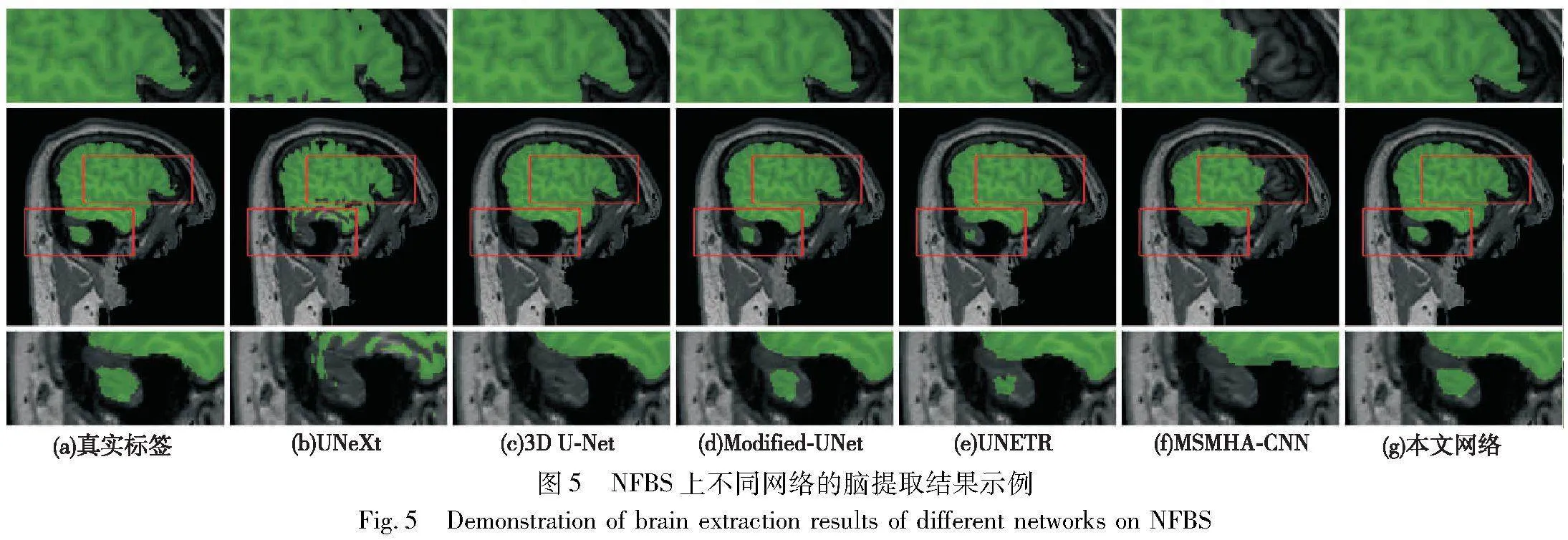
图4~6分别展示了3D UNeXt和其他先进网络在IBSR、NFBS和HTU-BrainMask数据集上的脑提取可视化结果。从中可以看出,本文网络的剥离结果没有明显噪声且脑边缘平滑程度最好,脑提取结果最接近于真实标签。这是因为3D MLP模块和多尺度特征融合模块能够充分学习特征之间的远程依赖并充分融合浅层特征与深层特征。UNeXt的剥离结果存在明显空洞,因为其丢失了3D体数据包含的空间信息和远程依赖信息。3D U-Net、Modified-UNet、UNTER和MSMHA-CNN的剥离结果存在边缘噪声且提取出的脑边缘平滑程度较差。这是因为这些方法虽然能够获取丰富的局部细节信息,但不能充分学习特征之间的远程依赖以剔除非脑部区域产生的噪声。
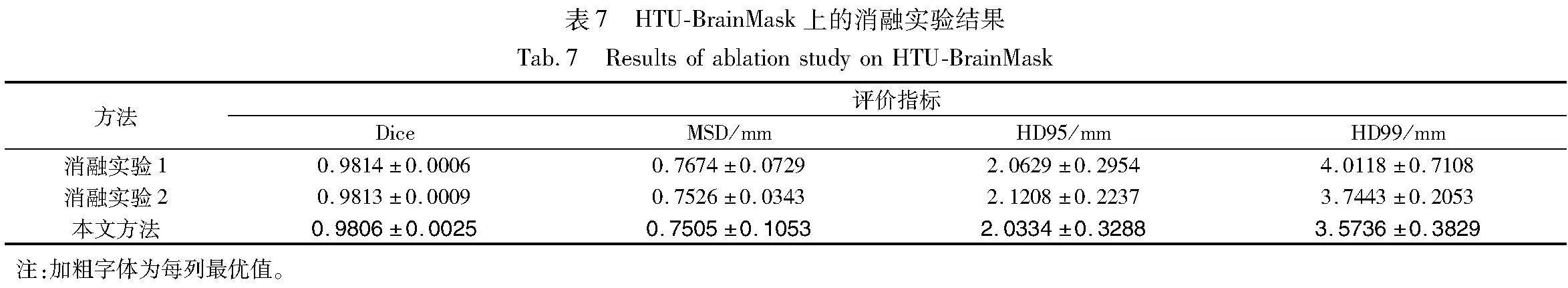
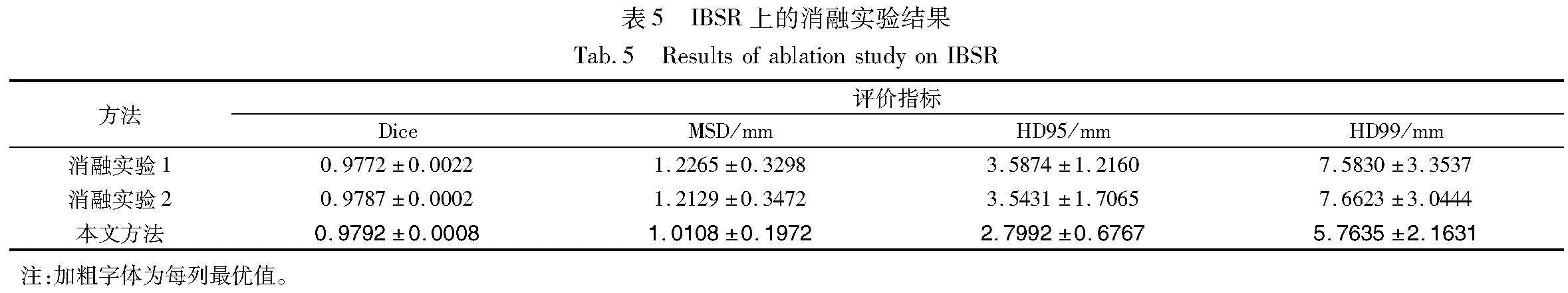
2.6 消融实验
为验证3D MLP模块和多尺度特征融合模块的有效性,本文进行以下两组消融实验。消融实验1,移除3D MLP模块;消融实验2,移除多尺度特征融合模块。在三个数据集上的消融实验结果如表5~zwQKeKwOuYSLVRLpiQ8HtA==7所示。从中可以看出,3D MLP模块和多尺度特征融合模块在3D UNeXt中具有重要作用。
3 结束语
脑提取的结果对实验室分析和临床诊断产生重要影响,因此研究准确和高效脑提取的模型具有重要的理论意义和应用价值。本文提出的3D UNeXt是一种全新的用于脑提取的轻量级网络。该网络能够在普通CPU上快速运行,有效解决了现有主流网络参数量大和推理速度慢的问题。3D UNeXt在网络浅层使用3D卷积提取丰富的局部特征信息,在网络深层使用3D MLP模块,借助移位操作在不同轴向实现特征间的交互、表征间的远程依赖。移位操作为线性操作,从而有效降低了3D UNeXt的计算复杂度并提高了网络的推理速度。解码阶段的多尺度特征融合模块有效融合了不同尺度的浅层特征和深层特征,进一步提高了网络的脑提取精度。
本文仅在三个数据集上进行实验,这些数据集的样本数量相对较少并且成像模态较为单一。这可能产生一定的统计误差,不能非常全面地评估3D UNeXt的泛化能力。后期可以在更多数据集上进行实验,充分验证本文网络。
参考文献:
[1]田梨梨, 程欣宇, 唐堃, 等. 集成注意力增强和双重相似性引导的多模态脑部图像配准[J]. 中国图象图形学报, 2021, 26(9): 2219-2232. (Tian Lili, Cheng Xinyu, Tang Kun, et al. Multimodal brain image registration with integrated attention augmentation and dual similarity guidance[J]. Journal of Image and Graphics, 2021, 26(9): 2219-2232.)
[2]Isensee F, Schell M, Pflueger I, et al. Automated brain extraction of multisequence MRI using artificial neural networks[J]. Human Brain Mapping, 2019,40(17): 4952-4964.
[3]Monteiro M, Newcombe V F, Mathieu F, et al. Multiclass semantic segmentation and quantification of traumatic brain injury lesions on head CT using deep learning: an algorithm development and multicentre validation study[J]. The Lancet Digital Health, 2020,2(6): e314-e322.
[4]Woolrich M W, Jbabdi S, Patenaude B, et al. Bayesian analysis of neuroimaging data in FSL[J]. NeuroImage, 2009,45(1): S173-S186.
[5]Jenkinson M, Beckmann C F, Behrens T E, et al. FSL[J]. NeuroImage, 2012, 62(2): 782-790.
[6]Litjens G, Kooi T, Bejnordi B E, et Xon81kjJ8Wf8AGzvoPXL4OR/EaUC7nf4yLhvavdMyDA=al. A survey on deep learning in medical image analysis[J]. Medical Image Analysis, 2017,42: 60-88.
[7]Shin H C, Roth H R, Gao Mingchen, et al. Deep convolutional neural networks for computer-aided detection: CNN architectures, dataset characteristics and transfer learning[J]. IEEE Trans on Medical Imaging, 2016,35(5): 1285-1298.
[8]Dou Qi, Chen Hao, Jin Yueming, et al. 3D deeply supervised network for automatic liver segmentation from CT volumes[C]//Proc of the 19th International Conference on Medical Image Computing and Computer-Assisted Intervention. Berlin: Springer, 2016: 149-157.
[9]Kleesiek J, Urban G, Hubert A, et al. Deep MRI brain extraction: a 3D convolutional neural network for skull stripping[J]. Neuro-Image, 2016,129: 460-469.
[10]Ronneberger O, Fischer P, Brox T. U-Net: convolutional networks for biomedical image segmentation[C]//Proc of the 18th International Conference on Medical Image Computing and Computer-Assisted Intervention. Berlin: Springer, 2015: 234-241.
[11]Ibtehaz N, Rahman M S. MultiResUNet: rethinking the U-Net architecture for multimodal biomedical image segmentation[J]. Neural Networks, 2020,121: 74-87.
[12]Wang Zhengyang, Zou Na, Shen Dinggang, et al. Non-local U-Nets for biomedical image segmentation[C]//Proc of the 34th AAAI Conference on Artificial Intelligence. Palo Alto,CA: AAAI Press, 2020: 6315-6322.
[13]iek , Abdulkadir A, Lienkamp S S, et al. 3D U-Net: learning dense volumetric segmentation from sparse annotation[C]//Proc of the 19th International Conference on Medical Image Computing and Computer-Assisted Intervention. Berlin: Springer, 2016: 424-432.
[14]王磐, 强彦, 杨晓棠, 等. 基于双注意力3D-UNet的肺结节分割网络模型[J]. 计算机工程, 2021, 47(2): 307-313. (Wang Pan, Qiang Yan, Yang Xiaotang, et al. Network model for lung nodule segmentation based on double attention 3D-UNet[J]. Engineering with Computers, 2021, 47(2): 307-313.)
[15]Jin Qiangguo, Meng Zhaopeng, Sun Changming, et al. RA-UNet: a hybrid deep attention-aware network to extract liver and tumor in CT scans[J]. Frontiers in Bioengineering and Biotechnology, 2020, 8: 605132.
[16]Wang Fei, Jiang Mengqing, Qian Chen, et al. Residual attention network for image classification[C]//Proc of the 30th IEEE Confe-rence on Computer Vision and Pattern Recognition. Piscataway, NJ: IEEE Press, 2017: 3156-3164.
[17]Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16×16 words: transformers for image recognition at scale[EB/OL]. (2021-06-03) . https://doi. org/10.48550/arXiv.2010.11929.
[18]Chen Jieneng, Lu Yongyi, Yu Qihang, et al. TransUNet: transfor-mers make strong encoders for medical image segmentation[EB/OL]. (2021-02-08) . https://doi.org/10.48550/arXiv.2102.04306.
[19]Cao Hu, Wang Yueyue, Chen J, et al. Swin-UNet: UNet-like pure transformer for medical image segmentation[C]//Proc of European Conference on Computer Vision. Cham: Springer, 2022: 205-218.
[20]Xu Guoping, Zhang Xuan, He Xinwei et al. LeViT-UNet: make faster encoders with transformer for medical image segmentation[C]//Proc of Chinese Conference on Pattern Recognition and Computer Vision. Singapore: Spinger Natide Singapore, 2023:42-53.
[21]Graham B, El-Nouby A, Touvron H, et al. LeViT: a vision transformer in convnet’s clothing for faster inference[C]//Proc of IEEE/CVF International Conference on Computer Vision. Piscataway, NJ: IEEE Press, 2021: 12259-12269.
[22]Wang Hongyi, Xie Shiao, Lin Lanfen, et al. Mixed Transformer U-Net for medical image segmentation[C]//Proc of the 47th IEEE International Conference on Acoustics, Speech and Signal Processing. Pisca-taway, NJ: IEEE Press, 2022: 2390-2394.
[23]张航, 王雅萍, 耿秀娟, 等. 等强度婴儿脑MR图像分割的深度学习方法综述[J]. 中国图象图形学报, 2020, 25(10): 2068-2078. (Zhang Hang, Wang Yaping, Geng Xiujuan, et al. Review of deep learning methods for isointense infant brain MR image segmentation[J]. Journal of Image and Graphics, 2020, 25(10): 2068-2078.)
[24]何康辉, 肖志勇. LRUNet: 轻量级脑肿瘤快速语义分割网络[J]. 中国图象图形学报, 2021, 26(9): 2233-2242. (He Kanghui, Xiao Zhiyong. LRUNet: a lightweight rapid semantic segmentation network for brain tumors[J]. Journal of Image and Graphics, 2021,26(9): 2233-2242.)
[25]Pinkus A. Approximation theory of the MLP model in neural networks[J]. Acta Numerica, 1999, 8: 143-195.
[26]Lian Dongze, Yu Zehao, Sun Xing, et al. AS-MLP: an axial shifted MLP architecture for vision[EB/OL]. (2022-03-17) . https://doi.org/10.48550/arXiv.2107.08391.
[27]Valanarasu JMJ, Patel VM. UNeXt: MLP-based rapid medical image segmentation network[C]//Proc of the 25th International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2022: 23-33.
[28]Yu Tan, Li Xu, Cai Yunfeng, et al. S2-MLP: spatial-shift MLP architecture for vision[C]//Proc of the 22nd IEEE/CVF Winter Conference on Applications of Computer Vision. Piscataway, NJ: IEEE Press, 2022: 3615-3624.
[29]Pang Jianye, Jiang Cheng, Chen Yihao, et al. 3D Shuffle-Mixer: an efficient context-aware vision learner of transformer-MLP paradigm for dense prediction in medical volume[J]. IEEE Trans on Medical Imaging, 2023, 42(5): 1241-1253.
[30]Tu Zhengzhong, Talebi H, Zhang Han, et al. MAXIM: multi-axis MLP for image processing[C]//Proc of IEEE/CVF Conference on Computer Vision and Pattern Recognition. Piscataway, NJ: IEEE Press, 2022: 5769-5780.
[31]Hendrycks D, Gimpel K. Gaussian error linear units(GELUs)[EB/OL]. (2023-06-06). http://doi.org/10.48550/arXiv.1606.08415.
[32]Feindel W. The montreal neurological institute[J]. Journal of Neurosurgery, 1991,75(5): 821-822.
[33]Isensee F, Kickingereder P, Wick W, et al. Brain tumor segmentation and radiomics survival prediction: contribution to the brats 2017 challenge[C]//Proc of the 20th International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Sprin-ger, 2018: 287-297.
[34]Hatamizadeh A, Tang Y, Nath V, et al. UNETR: transformers for 3D medical image segmentation[C]//Proc of the 22nd IEEE/CVF Winter Conference on Applications of Computer Vision. Piscataway, NJ: IEEE Press, 2022: 574-584.
[35]Sun Liang, Shao Wei, Zhu Qi, et al. Multi-scale multi-hierarchy attention convolutional neural network for fetal brain extraction[J]. Pattern Recognition, 2023, 133: 109029.

