植物基因编辑技术及其在果树上的应用
张春渝 许小琼 赖钟雄

收稿日期:2024-01-06
基金项目:国家自然科学基金项目(31572088);国家重点研发计划(2019YFD1000901);国家现代农业产业技术体系(香蕉)专
项资金(GARS-31-15);福建省高原学科建设经费(102/71201801101)
作者简介:*为通讯作者:赖钟雄(1966-),男,研究员,博士,博士生导师,主要从事园艺植物生物技术研究工作,E-mail:
laizx01@163.com。张春渝(1999-),女,在读博士生,主要从事果树生物技术研究,E-mail:zcynhba@163.com
摘要:基因编辑技术不同于传统转基因技术,可以在不引入外源DNA片段的情况下快速、精确地对生物体基因组进行改造,具有较高的安全性。基因编辑技术在水稻、玉米、马铃薯等农作物以及拟南芥等模式植物中已经进行了大量的研究,近年来在果树上也取得了一定成就,在果树育种上显示出巨大的应用潜力。据此介绍了植物基因编辑技术的种类及基本原理,总结了果树基因编辑技术研究进展,分析了存在的问题,并对果树基因编辑技术的应用前景进行了展望。
关键词:果树;基因编辑;现状;展望
中图分类号:S667 文献标识码:A 文章编号:2095-5774(2024)01-0001-16
Plant Gene Editing Technology and its Applications in Fruit Trees
Zhang Chunyu,Xu Xiaoqiong,Lai Zhongxiong*
(Institute of Horticultural Biotechnology/Institute of Subtropical Fruits,Fujian Agriculture and Forestry University,
Fuzhou,Fujian 350002,China)
Abstract:Compared with traditional transgenic technology,gene editing technology can rapidly and precisely modify the genome of an organism without introducing exogenous DNA fragments,and presents higher security. In recent years,gene editing technology has been widely used in many crops,such as rice,maize and potatoes and model plant Arabidopsis thaliana,which has also been successfully applied in fruit trees,showing great potential in fruit tree breeding. In this paper,we introduced the types and basic principles of plant gene editing technology,summarized the research progress of this technology in fruit trees,analysed the existing problems and looked forward to the prospect of the applications of gene editing technology in fruit trees.
Key words:Fruit tree;Gene editing;Current situation;Prospect
基因編辑技术根据序列特异性核酸酶的不同,可以分为3类:锌指核酸酶(Zinc-finger nucleases,ZFNs)技术[1]、转录激活因子样效应因子核酸酶(Transcription activator like effector nucleases,TALENs)技术[2]以及CRISPR(Clustered regularly interspaced short palindromic repeats)技术。基因编辑技术的原理是利用特定的酶和RNA序列,精确地识别和切割目标DNA序列,触发DNA双链断裂(DNA double-strand break,DSB),随后激活细胞自身的修复系统来诱导目标基因的失活或突变,或导入外源基因。细胞自身的修复系统主要包括非同源末端连接(Non-homologous end joining,NHEJ)修复与同源定向修复(Homology directed repair,HDR)途径。在大多数情况下,NHEJ能够引起随机插入或删除(indels),如果损伤发生在基因的编码区,则可能导致移码突变,从而实现基因敲除。当有与DSB周围序列具有同源区域的模板时,DNA损伤可以被HDR修复,利用这一机制可以实现精确的基因修饰或基因插入[3]。总的来说,ZFNs、TALENs和CRISPR技术都能够对生物体基因组特定目标进行修饰,这些技术的发展为植物功能基因组学研究和遗传改良提供了技术支撑。
植物基因编辑技术能够精确定向改良植物性状,可操作性强,安全性高,已经在植物中得到广泛的应用。本文在介绍基因编辑技术的种类及基本原理的基础上,总结了果树基因编辑技术应用情况,分析了存在的问题,并对果树基因编辑技术的应用前景进行了展望,以期为果树基因编辑的研究与应用提供参考。
1. 植物基因编辑技术及其工作原理
1.1 ZFNs技术
1996年,第一代基因编辑技术ZFNs技术问世[4],这是一种人工改造的核酸内切酶,由负责识别的锌指模块组成的DNA结合结构域和负责切割DNA链的FokI核酸酶结构域组成[5]。二者结合能够造成DNA在特定位点断裂,进一步诱发细胞自身的修复方式,从而实现基因编辑。锌指DNA结合域一般包含3个独立的锌指重复结构,每个锌指结构能够识别3个碱基,也就是说一个锌指DNA结合域可以识别9 bp长度的特异性序列(ZFN二聚体包含6个锌指,可以识别18bp长度的特异性序列)[6],这说明增加锌指的数量可以扩大ZFN特异性识别DNA序列的长度,从而获得更强的序列特异性。一般通过模块化组合单个ZF,来获得特异性识别足够长的DNA序列的锌指DNA结合域。目前,ZFNs技术在拟南芥[7]、玉米[8]等多种植物中都得到了应用。但是ZFNs技术也存在局限性,包括有限的锌指蛋白类别只可识别有限的DNA序列导致可编辑的靶基因位点有限,编辑的效率低,操作复杂,成本高,容易脱靶等。
1.2 TALENs技术
继ZFNs技术之后,TALENs技术又一次点燃了基因编辑革命[9]。典型的TALEN由一个包含核定位信号(Nuclear localization signal,NLS)的N端结构域、一个包含可识别特定 DNA序列的典型串联TALE重复序列的中央结构域,以及一个具有FokI核酸内切酶功能的C端结构域组成。TALEs作为一个重要的转录激活子样效应因子核酸酶,最初在黄单胞菌中发现的,其一旦进入植物细胞,就能够定位到细胞核,与目标启动子结合并诱导基因表达。TALEs的N端结构域包含细菌分泌信号(Type III secretion signal,T3SS)和4个非典型的重复序列(Non-canonical repeats,NCR)[10-12],它们的C端包含一个植物轉录因子TFIIA的相互结合位点(Transcription factor binding site,TFB)、两个功能性核定位信号(Nuclear localization signals,NLS)和一个酸性激活结构域(Acidic activation domain,AD)[13-15](图 1)。TALEN的DNA识别域是由一些非常保守的重复氨基酸序列模块组成,每个模块由33~35 aa串联重复的氨基酸组成,其中第12和13位的氨基酸种类为可变的(Repeat-variable di-residues,RVDs)[16],这些RVD中有些能特异性识别单个核苷酸序列,有些则可以识别2~4个核苷酸序列(其中NG可以识别T,NI可以识别A,NN可以识别G或A,HD可以识别C,NS可以识别A或G或C或T),如果重新排列这些重复序列,TALEs和TALEN的DNA结合特异性可以随意改变[9]。TALENs技术的工作原理是将识别特异DNA序列的TALE与内切核酸酶FokI偶联,然后通过DNA识别模块将TALEN元件靶向特异性的DNA位点并结合,两个邻近的FokI形成二聚体,发挥非特异性内切酶活性,于两个靶位点之间打断目标基因[17]。诱发DNA损伤修复机制,借助于细胞内固有HDR或NHEJ修复机制来完成对基因的修饰过程。
1.3 CRISPR-Cas系统
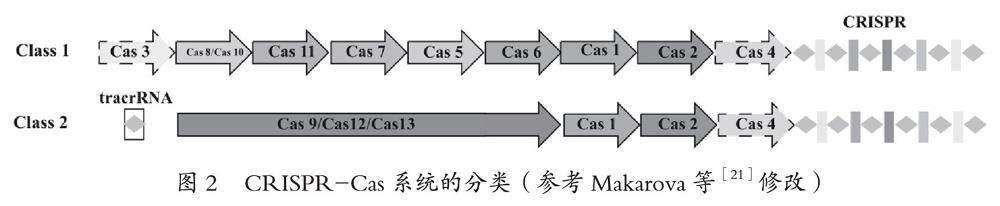
1987年,人们在大肠杆菌的基因组中首次发现了一个特殊的重复间隔序列——聚集规律间隔短回文重复(CRISPR)序列[18]。CRISPR是大多数细菌和古细菌基因组的一个显著特征,被认为与噬菌体的抗性相关[19-20]。CRISPR-Cas系统是CRISPR序列与Cas蛋白组合形成的一个以RNA为指导的适应性免疫系统,其中CRISPR基因座主要由前导区(leader)、重复序列区(repeat)和间隔区(spacer)构成。Cas基因则位于CRISPR基因附近或分散于基因组其他地方,编码的蛋白包括Cas1-Cas10。CRISPR-Cas系统能够根据干涉阶段切割靶标所用 Cas 效应物为单体酶还是多亚基复合物分为2类[21](图2):第1类CRISPR-Cas系统中具有由多个Cas蛋白组成的效应模块,它们形成一个crRNA结合复合体,共同作用于靶标的结合和加工,该类包括Ⅰ型、Ⅲ型和Ⅳ型;第2类CRISPR-Cas系统具有一个单一的、多结构域的crRNA结合蛋白,该类包括Ⅱ型、Ⅴ型和Ⅵ型[21]。目前,我们使用的CRISPR-Cas9基因编辑技术属于第2类,第 2 类 CRISPR-Cas 系统是目前植物基因编辑应用最广泛的系统,还包含了CRISPR-Cas12和CRISPR-Cas13编辑技术。虽然它们都属于第2类CRISPR-Cas系统,但是CRISPR-Cas9(II型)与CRISPR-Cas12(V型)以及CRISPR-Cas13(VI型)技术还存在许多不同之处。CRISPR-Cas9系统利用sgRNA(single-guide RNA,由crRNA和tracrRNA 组成)特异性靶向dsDNA[22]。Cas9一旦感知到正确的碱基配对,就能够结合到与PAM(Protospacer adjacent motif)相邻的sgRNA间隔序列互补的DNA序列上[23],从而激活RuvC和HNH核酸酶来切割非靶标和靶标DNA链,造成靶位点DNA双链断裂[22]。CRISPR-Cas9系统切割类型为平末端,大小为1000~1600个氨基酸。CRISPR-Cas12a系统也被称为CRISPR-Cpf1系统,Cas12a能够由crRNA引导靶向ssDNA和dsDNA。Cas12a结合到与crRNA间隔序列互补的DNA序列上,通过正确的碱基配对来激活其RuvC核酸酶以获得一般的ssDNA酶活性,从而切割非靶DNA链和靶DNA链以及反式ssDNA底物[24]。CRISPR-Cas13a系统也被称为CRISPR-C2c2,能够靶向切割单链RNA,其由crRNA引导Cas13a,让其与crRNA间隔序列互补的ssRNA序列结合。一旦Cas13a进行了正确的碱基配对就能够激活HEPN核酸酶,从而获得ssRNase活性来实现对单链RNA的切割过程[25-26]。总的来说,CRISPR-Cas9能够切割dsDNA,CRISPR-Cas12a能够切割ssDNA和dsDNA,CRISPR-Cas13a能够实现对ssRNA的切割,各有优点。目前应用较为广泛的是CRISPR-Cas9和CRISPR-Cas12a基因编辑技术。
1.3.1 CRISPR-Cas9系统
作为第三代基因编辑技术,CRISPR-Cas9相较于ZFNs、TALENs等基因编辑技术具有效率高、简便、成本低、容易上手等优点,因此成为当今最主流的基因编辑系统。在众多天然CRISPR-Cas系统中,第2类(class 2)系统只需要一个RNA指导的Cas核酸酶就能够完成对靶点的切割[21]。其中Cas9核酸酶成为了第一个被用于基因组编辑的Cas效应子[22]。
CRISPR-Cas9系统由Cas9、crRNA、tracrRNA三部分组成。其中,crRNA和tracrRNA通过局部碱基配对组成gRNA(guide RNA),gRNA与Cas9蛋白结合后引导Cas9蛋白识别和切割目标DNA序列[22]。2012年,为了方便实验设计并提高gRNA的稳定性,Doudna和Charpentier团队将crRNA和tracrRNA融合成一条RNA,并把其称为sgRNA[27]。改造之后的CRISPR-Cas9系统成为了基因编辑研究的首选工具。一般来说,CRISPR-Cas9系统能够实现的一切,原则上也可以使用ZFNs或TALENs技术来实现。然而,相对于ZFNs、TALENs技术来说,CRISPR-Cas9具有简单性、高效性、成本低等多方面的明显优势。CRISPR-Cas9系统因为不涉及蛋白质工程,所以构建起来十分快速。在此过程中,通过使用两个互补的退火寡核苷酸进行体外转录,可以产生任意数量的gRNA。并且可以将多个sgRNA串联对一个基因的多个位点进行编辑或同时对多个基因进行编辑[28]。然而,CRISPR-Cas9存在较为严重的脱靶效应,可能会在错位的基因位点切割DNA双链,从而导致潜在风险。与ZFNs和TALENs复杂的二聚体结构不同,因为CRISPR-Cas9系统有别于ZFNs和TALENs技术形成复杂的二聚体,其拥有更简单的单体结构,可通过碱基配对识别同源位点,因此在识别和切割目标DNA位点方面特异性低。因此,如何降低CRISPR-Cas9系统的脱靶概率,提高编辑效率是基因编辑技术发展面临的一个重要挑战。2014年,研究者提出gRNA与Cas9蛋白可以预组装成RNP(核糖核蛋白),RGENRNPs以高达79%的频率诱导位点靶向编辑,同时减少与质粒转染相关的脱靶编辑,RGEN RNPs几乎在转染后立即切割染色体DNA,并在细胞中迅速降解,减少了脱靶效应[29]。2021年,斯坦福大学亓磊团队找到了一种分子量大小不到目前使用的CRISPR系统(例如Cas9或Cas12a)一半的新型Cas效应蛋白(V-F Cas12f),并通过sgRNA设计和蛋白质工程对Cas12f进行了优化和修补,构建出一个高效的微型Cas系統(CasMINI),其更容易传递到哺乳动物细胞中来应用于CRISPR基因编辑的临床治疗[30]。2022年,研究者首先发现了CRISPR-Cas9基因编辑系统中脱靶背后的结构机制,在此基础上重新设计了Cas9蛋白——SuperFi-Cas9,其脱靶概率降低了数千倍,且编辑效率与原始版本的Cas9蛋白相同[31]。
1.3.2 CRISPR-Cas12a(Cpf1)系统
基于Cas9的II型CRISPR系统被认为是最简单的CRISPR系统,最容易适应基因组编辑,但V型Cpf1驱动系统的引入为CRISPR编辑系统提供了另一种选择。2015年,Zetsche等从酸胺球菌和毛螺科中鉴定出两个候选Cpf1酶,发现它们在人类细胞中具有高效的基因组编辑活性[24]。CRISPR- Cpf1被认为是第2类CRISPR系统,但不同于CRISPR-Cas9系统,它被归类为V型。与Cas9核酸酶一样,Cpf1家族成员都含有一个RuvC-like核酸内切酶结构域,但缺少Cas9的第二个HNH核酸内切酶结构域。在II型CRISPR-Cas9系统中,crRNA的成熟是由宿主内护蛋白RNase III和与pre-crRNA碱基配对的tracrRNA共同完成的[32-33]。相反,Cas12a不需要tracrRNA,能够将自身的pre-crRNA加工成成熟的crRNA[34]:当pre-crRNA转录完成后,Cas12a在CRISPR重复序列形成的发夹结构上游4 nt处将其切割,产生中间crRNA分子,这些中间crRNA分子在体内进一步加工成成熟的crRNA。相较于Cas9,Cpf1可能更适合用于基因组编辑,CRISPR-Cpf1被称为是新一代的基因组编辑系统。
Cpf1最显著的特征在于它是由一个单crRNA引导的内切酶。Cas9需要tracrRNA加工crRNA阵列,并且需要crRNA和tracrRNA共同介导干扰[33],Cpf1则不需要tracrRNA来加工crRNA,Cpf1-crRNA复合物能够单独切割靶DNA分子,不需要任何额外的RNA种类。Cpf1能够在5'端进行交错切割,产生一个粘性末端,与Cas9产生的平末端形成对比,这可能更有利于以精确方向整合DNA序列[35]。如果能对粘性末端的精确序列进行编程,就能将设计的DNA插入物以正确的方向整合到基因组中。在哺乳动物中证明这种裂解产物的结构有利于促进基于非同源末端连接NHEJ的基因插入哺乳动物基因组[36]。此外,Cpf1还可能提高HDR效率,主要体现在Cas9仅在PAM位点上游切割3 bp,因此NHEJ途径导致indel突变会破坏识别序列,阻止了后续的编辑。相比之下,由于Cpf1的切割位置相对远离PAM(通常切割PAM位点下游18~23 bp的DNA),对识别序列并没有造成破坏。识别序列的存在可能会赋予Cpf1再次切割的能力,并可能介导HDR,增加了进行所需基因组编辑的机会[24]。
Cpf1能够识别富含T的PAM 序列,能够在具有丰富AT基因组的生物体中进行基因组编辑,例如恶性疟原虫这种富含AT的结构域控制的细菌基因组[37],或对AT富集感兴趣的领域(支架/基质附着区和着丝粒DNA的难以延伸的DNA片段)。所以说与Cas9富含G的PAM要求相比,CRISPR- Cpf1系统为基因组目标基因的选择扩大了范围。
1.3.3 基于CRISPR的碱基编辑器
碱基编辑器包括腺嘌呤碱基编辑器(Adenine base editor,ABE)、胞嘧啶碱基编辑器(Cytosine base editor,CBE)和鸟嘌呤碱基编辑器(Guanine base editor,GBE),这些技术是在不产生DSB的情况下,为精确的基因组编辑提供了选择,以此来避免基因组不稳定和DNA修复导致的不可预测结果的风险。2016年,DavidR. Liu实验室首先提出CBE与ABE[38-39],两者都是依托于CRISPR的DNA定位能力,将碱基进行替换的过程。其中,CBE技术能够在PAM 位点附近内将胞嘧啶C转化为尿嘧啶U,然后在DNA复制的过程中被当作T,实现C到T转换(相反链上的G到A的转变);ABE技术则是实现A到G的转变(相反链上的T到C的转变)。2020年,GBE碱基编辑器技术出现在大家的视野中[40-41],这项技术能够实现碱基C到G的突变。GBE与CBE技术的原理十分接近,都是将C先转化为U,只不过GBE接下来直接用融合在Cas9蛋白另一端的Ugn酶将U碱基去除,随后引发单碱基缺失修复反应,提供C碱基向其它碱基突变的机会,但C会更倾向于突变为G。
2植物基因编辑技术在果树上的应用
在园艺领域中,CRISPR-Cas9基因编辑技术已经较为成功地被应用于模式作物番茄的研究中[42]。D Ambrosi等[43]通过编辑2个类胡萝卜素生物合成基因,编辑效率达到84%,说明CRISPR-Cas9系统是一种可以在番茄中产生有用突变的有效且快速的方法。目前,运用CRISPR-Cas9技术揭示了许多基因在番茄中的分子机制[44-45],并通过编辑不同的基因来提高番茄产量和品质[46];提高番茄果实GABA[47-48]、番茄红素的含量[49];提高番茄对白粉病真菌[50]、果斑病[51]、黄卷叶病毒[52]以及灰葡萄孢[53]的抗性。还能使用CRISPR-Cas9技术加速番茄的进化[54],实现从头驯化[55],获得矮化[56]、抗除草剂的番茄[57-58],并能重新设计番茄果实形状[59]。2021年,富含GABA的基因编辑番茄进入市场[60];2022年,能够积累维生素D3前体的基因编辑番茄被开发[61]。CRISPR-Cas9技术在番茄中的成功应用可以为许多果树基因编辑的研究提供参考。自2013年以来,基因编辑技术被广泛应用于许多果树的遗传改良,在柑橘[62-72]、香蕉[73-77]、草莓[78-82]、苹果[83-87]、葡萄[88-94]、猕猴桃[95-100]等果树中,均有基因编辑的相关报道。基因编辑技术能够改良果树的植株形态、开花结果特性、果实品质,调控果实成熟,提高抗病性和抗逆性等。
2.1柑橘
2014年,研究者发现将柑橘黄单胞菌亚种接种于柑橘叶片能够显著促进GUS的瞬时表达,并在技术基础上通过Cas9/sgRNA成功对CsPDS进行了编辑[62]。2019年,邓秀新院士团队建立了山金柑CRISPR-Cas9基因编辑系统,通过编辑PDS基因,导致叶片的白化表型,并通过编辑类胡萝卜素裂解基因DIOXYGENASE 4(CCD4),获得了两个携带目标突变的阳性T0苗[63];同年,王年团队在柑橘中首次建立CRISPR-Cas12a基因编辑系统[64];Wang等[65]发现突变CsWRKY22基因能够降低万金橙对溃疡病菌的敏感性;Yang等[66]证实内含子多顺反子 tRNA-gRNA (inPTG) 基因组编辑技术在柑橘的原生质体瞬时表达系统和上胚轴稳定转化系统中适用。研究者们在山金柑中实现了使用双位点CRISPR-Cas9基因编辑系统进行基因组大片段高效敲除[67]。在多年生植物和无性繁殖的植物中,T0代植物的无转基因基因组编辑是理想的,但具有挑战性。核糖核蛋白复合物(RNP)常被用于生产无转基因植物。研究者通过使用Cas12a/crRNA核糖核蛋白转化胚性原生质体来编辑溃疡易感性基因CsLOB1,在10个月内开发出T0代的无转基因耐溃疡柑橘品系[68],目前无转基因抗溃疡病柑橘品系已获得美国农业部动植物检疫局的监管批准,不受美国环保局的监管,这项研究为柑橘溃疡病的控制提供了有效的解决方案以及有效的无转基因基因组编辑策略。2023年3月,一种不依赖于RNP技术也被用于柑橘的基因编辑中,研究者利用CBE/gRNA对ALS基因进行碱基编辑,使植株具有抗除草剂表型的选择标记;使用Cas12a/crRNA编辑目标基因;GFP用于检测是否为转基因植株,并运用此技术在番茄、烟草、马铃薯和柑橘中有效获得T0代无转基因基因编辑植物[69]。此外,碱基编辑器技术也被应用在柑橘中。2022年,研究者利用ABE技术编辑了甜橙LOB1基因启动子区的TATA-box,編辑过的植株对溃疡病病原体柑橘黄单胞菌亚种具有抗性,还运用CBE技术编辑柑橘中的ALS基因,编辑植株对除草剂氯磺隆具有抗性,并且这种转基因不会遗传给后代[70]。基因编辑技术也被用于柑橘品质的改良,2023年徐强团队通过基因编辑技术发现,CitPH4基因的敲除能够显著地降低柠檬酸的积累,表明CitPH4能够促进柠檬酸的积累[71];张飞等[72]发现CsLMI1基因具有能够调控油胞发育的功能。
2.2香蕉
2017年广东农科院胡春华建立了香蕉CRISPR-Cas9基因编辑技术体系,定点敲除八氢番茄红素脱氢酶MuPDS[73]。Tripathi等[74]利用基于CRISPR-Cas的基因编辑技术控制香蕉的细菌性病害香蕉黄单胞菌枯萎病。2023 年8月,Hu等[75]报道了香蕉高效非转基因的REG-2启动子驱动的基因删除系统;同年12月,研究人员发现香蕉早期结节蛋白样3(MusaENODL3)基因的靶向敲除,能够使香蕉获得抗黄单胞菌枯萎病功能。在基因编辑香蕉产业应用上[76],2023年5月菲律宾同意推广抗褐变的基因编辑香蕉[77]。
2.3草莓
商业化草莓是八倍体,其基因编辑是巨大挑战。Xing等[78]报道CRISPR-Cas9编辑靶向FvPDS、FvUF3GT(Anthocyanidin 3-O-glucosyltransferase)、FvF3H(Naringenin,2-oxoglutarate 3-dioxygenase)、和FvLDOX(Leucoanthocyanidin dioxygenase)在野生草莓(Fragaria vesca)中产生的编辑效率高达10%。Zhou等[79]在野生草莓中,利用CRISPR-Cas9和两个gRNA编辑辅助素生物合成和信号相关基因FveTAA1和FveARF8,编辑成功的部分原因可能是使用了U6启动子,该启动子能增强引导序列的表达并提高突变率。Martín-Pizarro等[80]针对八倍体草莓(Fragaria × ananassa Duch. cv. Camarosa)进行花发育调节因子FaTM6的编辑,证明在八倍体草莓的编辑也是可行的;并使用CRISPR-Cas9系统瞬时表达,敲除R2R3 MYB转录因子(FvMYB10)和FvCHS基因,造成花青素积累的减少。此外,CRISPR-Cas9编辑草莓FaPG1基因能够提高草莓果实硬度[81]。产业应用上,Rasa Aridi报道J R Simplot 植物科学公司宣布利用基因编辑技术开发更耐贮藏的草莓[82]。
2.4苹果
在苹果的基因编辑研究中,最早是使用CRISPR-Cas9系统突变了PDS基因[83]。2018年,在苹果中报道了CRISPR-Cas9能够直接递送CRISPR-Cas9 RNPs的方法,用于获得无外源DNA的编辑方法[84]。TFL1是一个参与花期抑制子,CRISPR介导的基因敲除TFL1会导致93%的编辑苹果提前开花[85]。此外,CRISPR-Cas9技术还被用于提高苹果的抗性,由Erwinia amylovora细菌引起的火疫病是商业苹果园的主要病害之一。通过CRISPR-Cas9介导敲除嘎啦和金美中的类激酶受体DIPM 4,可显著降低火疫病的易感性,编辑过的植株中坏死组织减少了50%[86]。2023年,研究报道了国产苹果利用CRISPR-Cas9编辑AGAMOUS-like基因[87]。
2.5葡萄
目前还没有关于 TALENs或 ZFNs介导的葡萄基因编辑的报道,可能是由于许多栽培品种转化困难。葡萄基因组有多达3500万个靶位点可用,意味着有多达3500万个目标位点可用于基因编辑,这使得使用单个引导基因有可能同时针对多个性状[88]。在‘霞多丽(chardonnay)中,使用CRISPR-Cas9 编辑 L-亚酮酸脱氢酶基因(L-IdnDH),基因编辑效率约为100%,这是一个很有希望的发现[89]。葡萄的基因编辑效率受到多种因素的影响,据报道,通过靶向编辑PDS基因获得了白化叶片再生植株,与新出现的上部叶片相比,较低、较老的叶片中突变细胞的比例更高[90],此外,GC含量较高的gRNA可以提高编辑效率[91]。2018年,Wang等[92]对葡萄转录因子VvWRKY52进行编辑,结果产生了15个双等位基因品系和7个杂合品系,编辑效率高达64%。2021年,Ren等[93]证明VvU3/U6和UBQ2启动子可以通过改善sgRNA的表达,显著提高葡萄的编辑效率。此外,还使用此系统对TMT1和TMT2进行编辑,总体编辑效率高于10%,并导致糖分水平降低,这表明这2个基因在葡萄糖分积累中的作用。2023年,Ren等[94]使用CRISPR-LbCas12a编辑系统,在葡萄中敲除了TMT1和DFR1基因,此外,基因编辑技术还被应用于研究葡萄的花青素积累。
2.6猕猴桃
目前,CRISPR-Cas9基因编辑技术已经成功运用于猕猴桃的基因功能研究,加速了猕猴桃的育种进程。在中华猕猴桃中,CEN和CEN4的双等位基因突变产生了快速开花的雄性植物,CEN4和SyGl的双编辑植株产生了快速开花的雌雄同体植株,且雌蕊和雄蕊均可育[95]。此外,利用CRISPR-Cas9技术,发现AcNAC1基因的突变,能够导致猕猴桃果实中的柠檬酸水平显著下降,利用这些材料,通过自交选育有望获得非转基因的低酸猕猴桃[96]。编辑AcBFT2基因有可能减少植物休眠,而不会对开花产生不利影响,这为选育更适合气候变化的品种提供可能[97]。AcFLCL基因编辑株系在4周后仍处于休眠状态,说明AcFLCL的正确表达对猕猴桃腋芽的正常萌发至关重要[98]。此外,研究人员还发现了一种比CRISPR-Cas9系统更适用于猕猴桃基因编辑的技术——Polycistronic tRNA-gRNA(PGT)-Cas9基因编辑技术[99],相比与传统CRISPR-Cas9系统其具有更高的突变效率。在猕猴桃毛状根中利用PTG系统编辑已报道的CEN4基因和AeCBL3基因,编辑效率都可以达到50%以上,其中纯合突变率达20%以上[100]。
2.7基因编辑技术在其他果树中的应用
除了上述几种果树之外,基因编辑技术也被应用于其他多种果树的遗传改良。2022年,研究人员以CRISPR-Act3.0为基本骨架,在梨中构建了基于CRISPR-dCas9的基因轉录激活系统,对PybZIPa、PyMYB114、PyMYB10、PybHLH3、PyDFR、PyANS和PyUFGT这些花青苷生物合成的重要基因进行了单基因或多基因激活,证明它们能够显著促进花青苷积累[101]。在蓝莓中,利用CRISPR-Cas技术通过敲除PDS基因[102]、gusA基因[103]来评估敲除效率。在可可中利用农杆菌介导的瞬时转化将CRISPR-Cas9组分导入可可叶片和子叶细胞,通过编辑防御反应的抑制因子TcNPR3,增强对病原菌热带疫霉感染的抵抗力[104]。在石榴中利用CRISPR-Cas9技术靶向两种udp依赖性糖基转移酶:PgUGT84A23和PgUGT84A24,在ugt84a23 ugt84a24双编辑系里中观察到没食子酸3-O和4-O糖苷的特异性积累[105]。2022年,欧洲板栗中也报道了CRISPR-Cas9技术能够以RNP的方式对原生质体进行转化[106]。CRISPR-Cas9介导的PDS基因编辑使核桃芽出现了白化现象[107];WOX11的编辑则调控了核桃树中的不定根和营养生长[108]。此外,最近许小琼基于龙眼胚性愈伤组织体胚发生系统的CRISPR-Cas9技术编辑BX1基因,BX1表达量显著下降(尚未发表)。
3存在问题与展望
3.1 存在问题
虽然基因编辑技术已经在果树研究中得到了广泛的应用,但是还存在着许多问题。1)木本植物的再生体系难建立,转化系统不够完善。2)一些果树遗传背景不够清楚,性状复杂,控制性状的基因和调控机制不够明确。3)获得无外源DNA的编辑植株比较困难。多数木本果树存在原生质体再生难等技术问题,难以采用RNP介导的Cas9和sgRNA的传递方法;传统的CRISPR-Cas9组分导入细胞往往借助DNA形式,常见的有将编码Cas9和sgRNA的质粒转导到细胞内,再培养成苗,如此获得的编辑植株带有外源DNA。4)基因编辑系统本身的局限性,编辑效率不够高。目前最常用的CRISPR-Cas9系统虽然应用很多,但该系统本身也存在一些局限性,比如PAM位点选择有限、脱靶问题等。5)缺乏高质量参考基因组。高质量的基因组信息是设计精准的sgRNA靶标位点和评估脱靶效应的重要遗传基础,目前多数果树缺乏高质量参考基因组。6)脱靶是基因编辑的一个常见问题,需要开发新的技术来提高果树基因编辑的特异性。7)实现全基因组编辑的碱基编辑器技术有待于开发,才能真正做到分子设计育种。
8)利用CRISPR-Cas9技术进行大片段序列精准敲入技术还有待于开发,该技术对于研究果树基因功能有重要意义。
3.2 展望
随着越来越多高质量参考基因组的发表,比如香蕉[109]、苹果[110]、猕猴桃[111]、龙眼[112,113]等,为基因编辑设计精准的靶位点提供了高质量的基因组信息,果树的精准分子设计育种将逐步实现。当然,在后基因组时代,基因编辑系统也同样需要进一步优化:1)扩展基因编辑系统的多功能性,实现基因的敲除、敲入、基因的转录调控(基因敲低和转录激活、表观遗传修饰等)。2)开发大片段序列精准敲入技术有助于研究果树基因的功能。3)建立多基因编辑系统。中国农业大学宗媛教授团队在小麦上实现同时编辑8个基因的编辑系统[114],但在果树上还有待于开发。4)开发碱基编辑工具,实现更精准的基因编辑。要提高果树基因编辑的特异性,特别是要开发实现全基因组编辑的碱基编辑器技术,才能真正做到果树分子设计育种。5)开发无外源DNA的基因编辑技术,关键是要建立果树的高效原生质体再生体系。植物DNA-free的基因编辑方法包括瞬时表达CRISPR-Cas9质粒DNA/RNA、体外转录CRISPR-Cas9以及由纯化好的Cas9蛋白和sgRNAs组成的预组装核糖核酸复合物RNP递送,相比于前面二者,RNP具有简单易行、成本低等优点,不仅明显降低脱靶效应,还能成功避免了外源DNA片段整合到基因组中的潜在风险。
CRISPR-Cas基因编辑是一种有效的全基因组筛选工具。构建大型的CRISPR文库已成为正向(forword)遗传筛选的重要手段,它可以在基因组范围内高精度地引入突变[115];依赖CRISPR的碱基编辑技术能够鉴定具有单碱基分辨率的功能元件[116-117]。此外,CRISPR筛选可与单细胞测序相结合,为解析基因功能提供新的方法[118]。目前在番茄上已开发了全基因组CRISPR突变体文库[119]。CRISPR突变体文库的开发利用将成为果树功能基因组学研究的有力手段,加速果树的育种进程。
随着AI技术的不断发展,应该考虑将基因编辑技术与AI相结合:碱基编辑技术的出现让基因编辑变得更加可操作性,但编辑部位碱基的准确性,以及编辑部位上下文序列对编辑的影响,一直难以预测。2021年,苏黎世大学建立了一种深度学习算法BE-DICT,该算法能够高精度地预测碱基编辑结果[120]。同年,研究人员开发了一个C-to-G碱基编辑器,其修改的目标序列的上下文可通过机器学习方法进行预测;同时还开发了一个深度学习模型,可以准确预测具有特定序列上下文的目标位点的OPTI-CGBE编辑系统,该方法有助于开发果树的碱基编辑器[121]。
參考文献:
[1]Takatsuji H. Zinc-finger proteins: the classical zinc finger emerges in contemporary plant science [J]. Plant Molecular Biology,1999,39(6): 1073-1078.
[2]Li T,Huang S,Jiang WZ,et al. TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and FokI DNA-cleavage domain [J]. Nucleic Acids Research,2011,39(1): 359-372.
[3]Bortesi L,Fischer R. The CRISPR/Cas9 system for plant genome editing and beyond [J]. Biotechnology Advances,2015,33(1): 41-52.
[4]Kim YG,Cha J,Chandrasegaran S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain [J]. Proceedings of the National Academy of Sciences of the United States of America,1996,93(3): 1156-1160.
[5]Jo YI.,Kim H,Ramakrishna S. Recent developments and clinical studies utilizing engineered zinc finger nuclease technology [J]. Cellular and Molecular Life Sciences,2015,72(20): 3819-3830.
[6]Pavletich NP,Pabo CO. Zinc finger-DNA recognition: crystal structure of a Zif268-DNA complex at 2.1 A [J]. Science,1991,252(5007): 809.
[7]Zhang F,Voytas DF. Targeted mutagenesis in Arabidopsis using zinc-finger nucleases [J]. Methods in Molecular Biology,2011,701: 167-177.
[8]Shukla VK,Doyon Y,Miller JC,et al. Precise genome modification in the crop species Zea mays using zinc-finger nucleases [J]. Nature,2009,459(7245):437-441.
[9]Becker S,Boch J. TALE and TALEN genome editing technologies [J]. Gene and Genome Editing,2021,2: 100007.
[10]Szurek B,Rossier O,Hause G,et al. Type III-dependent translocation of the Xanthomonas AvrBs3 protein into the plant cell [J]. Molecular Microbiology,2002,46(1): 13-23.
[11]Kay S,Hahn S,Marois E,et al. Bacterial effector acts as a plant transcription factor and induces a cell size regulator [J]. Science,2007,318(5850): 648-651.
[12]Gao H,Wu X,Chai J,et al. Crystal structure of a TALE protein reveals an extended N-terminal DNA binding region [J]. Cell Research,2012,22(12): 1716-1720.
[13]Ackerveken GVD,Marois E,Bonas U. Recognition of the bacterial avirulence protein AvrBs3 occurs inside the host plant cell [J]. Cell,1996,87(7): 1307-1316.
[14]Zhu W,Yang B,Chittoor JM,et al. AvrXa10 contains an acidic transcriptional activation domain in the functionally conserved C terminus [J]. Molecular Plant-Microbe Interactions,1998,11(8): 824-832.
[15]Yuan M,Ke Y,Huang R,et al. A host basal transcription factor is a key component for infection of rice by TALE-carrying bacteria [J]. eLife,2016,5: e19605.
[16]Sakuma T,Ochiai H,Kaneko T,et al. Repeating pattern of non-RVD variations in DNA-binding modules enhances TALEN activity [J]. Scientific Reports,2013,3: 3379.
[17]Nemudryi AA,Valetdinova KR,Medvedev SP,et al. TALEN and CRISPR/Cas genome editing systems: tools of discovery [J]. Acta Naturae,2014,6(3): 19-40.
[18]Ishino Y,Shinagawa H,Makino K,et al. Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion in Escherichia coli,and identification of the gene product [J]. Journal of Bacteriology,1987,169(12): 5429-5433.
[19]Barrangou R,Fremaux C,Deveau H,et al. CRISPR provides acquired resistance against viruses in prokaryotes [J]. Science,2007,315(5819): 1709-1712.
[20]Marraffini LA,Sontheimer EJ. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA [J]. Science,2008,322(5909): 1843-1845.
[21]Makarova KS,Wolf YI,Iranzo J,et al. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants [J]. Nature Reviews Microbiology,2020,18(2): 67-83.
[22]Knott GJ,Doudna JA. CRISPR-Cas guides the future of genetic engineering [J]. Science,2018,361: 866-869.
[23]Marraffini LA.,Sontheimer EJ. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea [J]. Nature Reviews Genetics,2010,11(3): 181-190.
[24]Zetsche B,Gootenberg JS,Abudayyeh OO,et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system [J]. Cell,2015,163(3): 759-771.
[25]Abudayyeh OO,Gootenberg JS,Konermann S,et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector [J]. Science,2016,353(6299): aaf5573.
[26]East-Seletsky A,O'Connell MR,Knight SC,et al. Two distinct RNase activities of CRISPR-C2c2 enable guide-RNA processing and RNA detection [J]. Nature,2016,538(7624): 270-273.
[27]Jinek M,Chylinski K,Fonfara I,et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science,2012,337(6096): 816-821.
[28]O'Connell MR,Oakes BL,Sternberg SH,et al. Programmable RNA recognition and cleavage by CRISPR/Cas9 [J]. Nature,2014,516(7530):263-266.
[29]Kim S,Kim D,Cho S,et al. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins [J]. Genome Research,2014,24(6): 1012-1019.
[30]Xu,X.,Chemparathy,A.,Zeng,L.,et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing [J]. Molecular Cell,2021,81(20): 4333-4345.
[31]Bravo JPK,Liu MS,Hibshman GN,et al. Structural basis for mismatch surveillance by CRISPR/Cas9 [J]. Nature,2022,603(7900):343–347.
[32]Charpentier E,Richter H,van der Oost J,et al. Biogenesis pathways of RNA guides in archaeal and bacterial CRISPR-Cas adaptive immunity [J]. Fems Microbiology Reviews,2015,(3): 428-441.
[33]Deltcheva E,Chylinski K,Sharma CM,et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III [J]. Nature,2011,471(7340): 602-607.
[34]Fonfara I,Richter H,BratoviM,et al. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA [J]. Nature,2016,532(7600): 517.
[35]Barrangou R,Horvath P. A decade of discovery: CRISPR functions and applications [J]. Nature microbiology,2017,2: 17092.
[36]Maresca M,Lin VG,Guo N,et al. Obligate ligation-gated recombination (ObLiGaRe): Custom-designed nuclease-mediated targeted integration through nonhomologous end joining [J]. Genome Research,2013,23(3): 539-546.
[37]Gardner MJ,Shallom SJ,Carlton JM,et al. Sequence of Plasmodium falciparum chromosomes 2,10,11 and 14 [J]. Nature,2002,419(6906): 531-534.
[38]Komor AC,Kim YB,Packer MS,et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage [J]. Nature,2016,533(7603): 420-424.
[39]Gaudelli NM,Komor AC,Rees HA,et al. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage [J]. Nature,2017,551(7681): 464-471.
[40]Kurt IC,Zhou R,Iyer S,et al. CRISPR C-to-G base editors for inducing targeted DNA transversions in human cells [J]. Nature Biotechnology,2021,39(1): 41-46.
[41]Zhao D,Li J,Li S,et al. Glycosylase base editors enable C-to-A and C-to-G base changes [J]. Nature Biotechnology,2021,39(1): 35-40.
[42]Brooks C,Nekrasov V,Lippman ZB,et al. Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR-associated9 system [J]. Plant Physiology,2014,166(3): 1292-1297.
[43]D'Ambrosio C,Stigliani AL,Giorio G. CRISPR/Cas9 editing of carotenoid genes in tomato [J]. Transgenic Research,2018,27(4): 367-378.
[44]Li R,Zhang L,Wang L,et al. Reduction of tomato-plant chilling tolerance by CRISPR-Cas9-mediated SlCBF1 mutagenesis [J]. Journal of Agricultural and Food Chemistry,2018,66(34): 9042-9051.
[45]Yang Q,Wan X,Wang J,et al. The loss of function of HEL,which encodes a cellulose synthase interactive protein,causes helical and vine-like growth of tomato [J]. Horticulture Research,2020,7(1): 180.
[46]ermák T,Baltes NJ,egan R,et al. High-frequency,precise modification of the tomato genome [J]. Genome Biology,2015,16: 232.
[47]Li R,Li R,Li X,et al. Multiplexed CRISPR/Cas9-mediated metabolic engineering of γ-aminobutyric acid levels in Solanum lycopersicum [J]. Plant Biotechnol Journal,2018,16(2): 415-427.
[48]Nonaka S,Arai C,Takayama M,et al. Efficient increase of -aminobutyric acid (GABA) content in tomato fruits by targeted mutagenesis [J]. Scientific Reports,2017,7(1): 7057.
[49]Li X,Wang Y,Chen S,et al. Lycopene is enriched in tomato fruit by CRISPR/Cas9-mediated multiplex genome editing [J]. Frontiers in Plant Science,2018,9: 559.
[50]Nekrasov V,Wang C,Win J,et al. Rapid generation of a transgene-free powdery mildew resistant tomato by genome deletion [J]. Scientific Reports,2017,7(1):482.
[51]Ortigosa A,Gimenez-Ibanez S,Leonhardt N,et al. Design of a bacterial speck resistant tomato by CRISPR/Cas9-mediated editing of SlJAZ2 [J]. Plant Biotechnology Journal,2018,17(3): 665-673.
[52]Tashkandi M,Ali Z,Aljedaani F,et al. Engineering resistance against tomato yellow leaf curl virus via the CRISPR/Cas9 system in tomato [J]. Plant Signaling & Behavior,2018,13(10): e1525996.
[53]Jeon JE,Kim JG,Fischer CR,et al. Pathogen-responsive gene cluster for highly modified fatty acids in tomato [J]. Cell,2020,180(1): 176-187.e19.
[54]Li T,YangX,Yu Y,et al. Domestication of wild tomato is accelerated by genome editing [J]. Nature Biotechnology,2018,36(12): 1161-1163.
[55]Zsgn A,ermák T,Naves ER,et al. De novo domestication of wild tomato using genome editing [J]. Nature Biotechnology,2018,36: 1211-1216.
[56]Tomlinson L,Yang Y,Emenecker R,et al. Using CRISPR/Cas9 genome editing in tomato to create a gibberellin-responsive dominant dwarf DELLA allele [J]. Plant Biotechnol Journal,2019,17(1): 132-140.
[57]Danilo B,Perrot L,Mara K,et al. Efficient and transgene-free gene targeting using Agrobacterium-mediated delivery of the CRISPR/Cas9 system in tomato [J]. Plant Cell Reports,2019,38(4): 459-462.
[58]Veillet F,Perrot L,Chauvin L,et al. Transgene-free genome editing in tomato and potato plants using Agrobacterium-mediated delivery of a CRISPR/Cas9 cytidine base editor [J]. International Journal of Molecular Sciences,2019,20(2): 402.
[59]Zhu Q,Deng L,Chen J,et al. Redesigning the tomato fruit shape for mechanized production [J]. Nature Plants,2023,9(10): 1659-1674.
[60]Waltz E. GABA-enriched tomato is first CRISPR-edited food to enter market [J]. Nature Biotechnology,2022,40(1): 9-11.
[61]Li J,Scarano A,Gonzalez NM,et al. Biofortified tomatoes provide a new route to vitamin D sufficiency [J]. Nature Plants,2022,8(6): 611-616.
[62]Jia H,Wang N. Targeted genome editing of sweet orange using Cas9/sgRNA [J]. PLoS One,2014,9(4): e93806.
[63]Zhu C,Zheng X,Huang Y,et al. Genome sequencing and CRISPR/Cas9 gene editing of an early flowering mini citrus (Fortunella hindsii) [J]. Plant Biotechnology Journal,2019,17(11): 2199–2210.
[64]Jia H,Orbovi V,Wang N. CRISPR-LbCas12a-mediated modification of citrus [J]. Plant Biotechnol Journal,2019,17(10): 1928-1937.
[65]Wang L,Chen S,Peng A,et al. CRISPR/Cas9-mediated editing of CsWRKY22 reduces susceptibility to Xanthomonas citri subsp. citri in Wanjincheng orange (Citrus sinensis (L.) Osbeck) [J]. Plant Biotechnology Reports,2019,13(5): 501-510.
[66]Yang W,Ren J,Liu W,et al. An efficient transient gene expression system for protein subcellular localization assay and genome editing in citrus protoplasts [J]. Horticultural Plant Journal,2023,9(3): 425-436.
[67]Xu Y,Zhang L,Lu L,et al. An efficient CRISPR/Cas9 system for simultaneous editing two target sites in Fortunella hindsii [J]. Horticulture Research,2022,9: uhac064.
[68]Su H,Wang Y,Xu J,et al. Generation of the transgene-free canker-resistant Citrus sinensis using Cas12a/crRNA ribonucleoprotein in the T0 generation [J]. Nature Communications,2023,14(1): 3957.
[69]Huang X,Jia H,Xu J,et al. Efficient transgene-free genome editing in plants in the T0 generation based on a co-editing strategy [J/OL]. BioRxiv,2023,https://doi.org/10.1101/2023.03.02.530790.
[70]Huang X,Wang Y,Wang N. Base editors for citrus gene editing [J]. Frontiers in Genome Editing,2022,4: 852867.
[71]Huang Y,He J,Xu Y,et al. Pangenome analysis provides insight into the evolution of the orange subfamily and a key gene for citric acid accumulation in citrus fruits [J]. Nature Genetics,2023,55(11): 1964-1975.
[72]Wang H,Ren J,Zhou S,et al. Molecular regulation of oil gland development and biosynthesis of essential oils in Citrus spp. [J]. Science,2024,383(6683): 659-666.
[73]胡春華,邓贵明,孙晓玄,等. 香蕉CRISPR/Cas9基因编辑技术体系的建立[J]. 中国农业科学,2017,50(7): 1294-1301.
[74]Tripathi JN,Ntui VO,Shah T,et al. CRISPR/Cas9-mediated editing of DMR6 orthologue in banana (Musa spp.) confers enhanced resistance to bacterial disease [J]. Plant Biotechnology Journal,2021,19(7): 1291-1293.
[75]Hu C,Liu F,Sheng O,et al. Efficient and transgene-free genome editing in banana using a REG-2promoter-driven gene-deletion system [J]. Molecular Horticulture,2023,3(1): 16.
[76]Ntui VO,Tripathi JN,Shah T,et al. Targeted knockout of early nodulin-like 3 (MusaENODL3) gene in banana reveals its function in resistance to Xanthomonas wilt disease [J]. Plant Biotechnology Journal,2023: 1-12.
[77]Macaulay T. Browning bananas under threat as Philippines approves gene-edited version.[EB/OL]. 2023[2024-01-02]. https://thenextweb.com/news/phillipines-approves-gene-editing-browning-bananas-tropic-biosciences.
[78]Xing S,Jia M,Wei L,et al. CRISPR/Cas9-introduced single and multiple mutagenesis in strawberry [J]. Journal of Genetics and Genomics,2018,45(12): 685-687.
[79]Zhou J,Wang G,Liu Z. Efficient genome editing of wild strawberry genes,vector development and validation [J]. Plant Biotechnol Journal,2018,16(11): 1868-1877.
[80]Martín-Pizarro C,Trivio JC,Posé D. Functional analysis of the TM6 MADS-box gene in the octoploid strawberry by CRISPR/Cas9-directed mutagenesis [J]. Journal of Experimental Botany,2019,70(3): 885-895.
[81]López-Casado G,Sánchez-Raya C,Ric-Varas PD,et al. CRISPR/Cas9 editing of the polygalacturonase FaPG1 gene improves strawberry fruit firmness [J]. Horticulture Research,2023,10(3): uhad011.
[82]AridiR. Using CRISPR technology,scientists plan to grow a more durable strawberry[EB/OL]. 2021[2024-01-02]. https://www.smithsonianmag.com/smart-news/using-crispr-technology-scientists-plan-to-grow-a-more-durable-strawberry-180978981/.
[83]Nishitani C,Hirai N,Komori S,et al. Efficient genome editing in apple using a CRISPR/Cas9 system [J]. Scientific Reports,2016,6:31481.
[84]Osakabe Y,Liang Z,Ren C,et al. CRISPR-Cas9-mediated genome editing in apple and grapevine [J]. Nature Protocols,2018,13(12),2844–2863.
[85]Charrier A,Vergne E,Dousset N,et al. Efficient targeted mutagenesis in apple and first time edition of pear using the CRISPR-Cas9 system [J]. Frontiers in Plant Science,2019,10: 40.
[86]Pompili V,Dalla Costa L,Piazza S,et al. Reduced fire blight susceptibility in apple cultivars using a high-efficiency CRISPR/Cas9-FLP/FRT-based gene editing system [J]. Plant Biotechnol Journal,2020,18(3): 845-858.
[87]Jacobson S,Bondarchuk N,Nguyen TA,et al. Apple CRISPR-Cas9-A recipe for successful targeting of AGAMOUS-like genes in domestic apple [J]. Plants,2023,12(21): 3693.
[88]Wang Y,Liu X,Ren C,et al. Identification of genomic sites for CRISPR/Cas9-based genome editing in the Vitis vinifera genome [J]. BMC Plant Biology,2016,16(1): 96.
[89]Ren C,Liu X,Zhang Z,et al. CRISPR/Cas9-mediated efficient targeted mutagenesis in Chardonnay (Vitis vinifera L.) [J]. Scientific Reports,2016,6: 32289.
[90]Nakajima I,Ban Y,Azuma A,et al. CRISPR/Cas9-mediated targeted mutagenesis in grape [J]. PLoS One,2017,12(5): e0177966.
[91]Ren F,Ren C,Zhang Z,et al. Efficiency optimization of CRISPR/Cas9-mediated targeted mutagenesis in grape [J]. Frontiers in Plant Science,2019,10:612.
[92]Wang X,Tu M,Wang D,et al. CRISPR/Cas9-mediated efficient targeted mutagenesis in grape in the first generation [J]. Plant Biotechnology Journal,2017,16(4):844–855.
[93]Ren C,Liu Y,Guo Y,et al. Optimizing the CRISPR/Cas9 system for genome editing in grape by using grape promoters [J]. Horticulture Research,2021,8(1): 52.
[94]Ren C,Gathunga EK,Li X,et al. Efficient genome editing in grapevine using CRISPR/LbCas12a system [J]. Molecular Horticulture,2023,3(1): 21.
[95]Varkonyi-Gasic E,Wang T,Cooney J,et al. Shy Girl,a kiwifruit suppressor of feminization,restricts gynoecium development via regulation of cytokinin metabolism and signalling [J]. New Phytologist,2021,230(4): 1461–1475.
[96]Fu BL,Wang WQ,Li X,et al. A dramatic decline in fruit citrate induced by mutagenesis of a NAC transcription factor,AcNAC1 [J]. Plant Biotechnology Journal,2023,21(8): 1695-1706.
[97]Herath D,Voogd C,Mayo-Smith M,et al. CRISPR-Cas9-mediated mutagenesis of kiwifruit BFT genes results in an evergrowing but not early flowering phenotype [J]. Plant Biotechnology Journal,2022,20(11): 2064-2076.
[98]Voogd C,Brian LA,Wu R,et al. A MADS-box gene with similarity to FLC is induced by cold and correlated with epigenetic changes to control budbreak in kiwifruit [J].New Phytologist,2022,233(5):2111-2126.
[99]Wang Z,Wang S,Li D,et al. Optimized paired-sgRNA/Cas9 cloning and expression cassette triggers high-efficiency multiplex genome editing in kiwifruit [J]. Plant Biotechnology Journal,2018,16(8): 1424-1433.
[100]Li P,Zhang Y,Liang J,et al. Agrobacterium rhizogenes-mediated marker-free transformation and gene editing system revealed that AeCBL3 mediates the formation of calcium oxalate crystal in kiwifruit [J]. Molecular Horticulture,2024,4(1): 1.
[101]Ming M,Long H,Ye Z,et al. Highly efficient CRISPR systems for loss-of-function and gain-of-function research in pear calli [J]. Horticulture Research,2022,9: uhac148.
[102]Vaia G,Pavese V,Moglia A,et al. Knockout of phytoene desaturase gene using CRISPR/Cas9 in highbush blueberry [J]. Frontiers in Plant Science,2022,13: 1074541.
[103]Han X,Yang Y,Han X,et al. CRISPR Cas9- and Cas12a-mediated gusA editing in transgenic blueberry [J]. Plant Cell,Tissue and Organ Culture,2022,148(2): 217-229.
[104]Fister AS,Landherr L,Maximova SN,et al. Transient expression of CRISPR/Cas9 machinery targeting TcNPR3 enhances defense response in Theobromacacao[J]. Frontiers in Plant Science,2018,9: 268.
[105]Chang L,Wu S,Tian L. Effective genome editing and identification of a regiospecific gallic acid 4-O-glycosyltransferase in pomegranate(Punica granatum L.) [J]. Horticulture Research,2019,6: 123.
[106]Pavese V,Moglia A,Abbà S,et al. First report on genome editing via ribonucleoprotein (RNP) in Castanea sativa Mill[J]. International Journal of Molecular Sciences,2022,23(10): 5762.
[107]Walawage SL,Zaini PA,Mubarik MS,et al. Deploying genome editing tools for dissecting the biology of nut trees [J]. Frontiers in Sustainable Food Systems,2019,3:100.
[108]Chang Y,Song X,Zhang Q,et al. Robust CRISPR/Cas9 mediated gene editing of JrWOX11 manipulated adventitious rooting and vegetative growth in a nut tree species of walnut [J]. Scientia Horticulturae,2022,303:111199.
[109]Li X,Yu S,Cheng Z,et al. Origin and evolution of the triploid cultivated banana genome [J]. Nature Genetics,2024,56(1): 136-142.
[110]Zhang L,Hu J,Han X,et al. A high-quality apple genome assembly reveals the association of a retrotransposon and red fruit colour [J]. Nature Communications,2019,10(1): 1494.
[111]Huang S,Ding J,Deng D,et al. Draft genome of the kiwifruit Actinidia chinensis [J]. Nature Communications,2013,4: 2640.
[112]Lin Y,Min J,Lai R,et al. Genome-wide sequencing of longan (Dimocarpus longan Lour.) provides insights into molecular basis of its polyphenol-rich characteristics [J]. GigaScience,2017,6(5): 1-14.
[113]Chen Y,Xie D,Ma X,et al. Genome-wide high-throughput chromosome conformation capture analysis reveals hierarchical chromatin interactions during early somatic embryogenesis [J]. Plant Physiology,2023,193(1): 555-577.
[114]Ni P,Zhao Y,Zhou X,et al. Efficient and versatile multiplex prime editing in hexaploid wheat [J]. Genome Biology,2023,24(1): 156.
[115]Smith I,Greenside G,Natoli T,et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map [J]. PLoS Biology,2017,15(11): e2003213.
[116]Cuella-Martin R,Hayward SB,Fan X,et al. Functional interrogation of DNA damage response variants with base editing screens [J]. Cell,2021,184(4): 1081-1097.
[117]Hanna RE,Hegde M,Fagre CR,et al. Massively parallel assessment of human variants with base editor screen [J]. Cell,2021,184(4): 1064-1080.
[118]Replogle JM,Norman TM,Xu A,et al. Combinatorial single-cell CRISPR screens by direct guide RNA capture and targeted sequencing [J]. Nature Biotechnology,2020,38(8): 954-961.
[119]Jacobs TB,Zhang N,Patel D,et al. Generation of a collection of mutant tomato lines using pooled CRISPR libraries [J]. Plant Physiology,2017,174(4): 2023-2037.
[120]Marquart KF,Allam A,Janjuha S,et al. Predicting base editing outcomes with an attention-based deep learning algorithm trained on high-throughput target library screens [J]. Nature Communications,2021,12(1): 5114.
[121]Yuan T,Yan N,Fei T,et al. Optimization of C-to-G base editors with sequence context preference predictable by machine learning methods [J]. Nature Communications,2021,12(1): 4902.
(責任编辑:赖瑞联)