不同产蛋数岩鸽卵巢转录组的SNP分析
毛楠楠 孙勇胜 陈辉 周荣艳 籍颖
摘要:为探索岩鸽(Columba livia)产蛋数相关基因及其遗传标记,利用STAR对高产蛋数和低产蛋数岩鸽卵巢组织转录组数据高质量序列与参考基因组进行比对,去除重复序列后利用GATK进行单核苷酸多态性(SNP)筛选,利用VCFtools对SNP质控后进行主成分分析、进化树构建和Fst计算,利用KEGG富集分析SNP所在基因。结果显示,高产蛋组共有SNP数为298 957个,低产蛋组共有SNP数为296 137个,高产蛋组、低产蛋组共有SNP数为254 118个,KEGG分析结果发现,SNP所在基因显著富集在孕酮调节的卵母细胞成熟、卵母细胞减数分裂和代谢等信号通路;高产蛋组和低产蛋组的102个特异SNP分布在14个注释基因上(LOC110357454、KDM5A、PIGS、BNC2、GBF1、SLAIN2、FGD4、DIAPH2、KIF3B、NFIB、EPG5、OSTF1、EMB、ST3GAL3),且富集的信号通路可能与其产蛋能力密切相关。
关键词:岩鸽(Columba livia);卵巢转录组;SNP;产蛋数
中图分类号:S836 文献标识码:A
文章编号:0439-8114(2024)04-0185-06
SNP analysis of the ovarian transcriptome of Columba livia with
different egg production numbers
Abstract: In order to explore the genes and genetic markers related to egg production in Columba livia, STAR was used to compare high-quality transcriptome sequences of ovarian tissue transcriptome data from high and low egg production Columba livia with the reference genome,after removing duplicate sequences, single nucleotide polymorphism (SNP) screening was performed using GATK. After controlling SNP quality using VCFtools, principal component analysis, evolutionary tree construction, and Fst calculation were performed. KEGG enrichment analysis was used to identify the gene where the SNP was located. The results showed that the high-yielding egg group had a total of 298 957 SNPs, the low yielding egg group had a total of 296 137 SNPs, and the high-yielding and low-yielding egg groups had a total of 254 118 SNPs. The KEGG analysis results showed that the SNP gene was significantly enriched in the progesterone regulated signaling pathways of oocyte maturation, meiosis, and metabolism;102 specific SNPs were distributed on 14 annotation genes(LOC110357454, KDM5A, PIGS, BNC2, GBF1, SLAIN2, FGD4, DIAPH2, KIF3B, NFIB, EPG5, OSTF1, EMB, and ST3GAL3) in the high and low egg production groups, and the enriched signaling pathways may be closely related to their egg production ability.
Key words: Columba livia(Columba livia); ovarian transcriptome; SNP; egg production numbers
随着生活水平的不断提高,鸽蛋和鸽肉产品越来越受到人们青睐。但由于鸽具有一夫一妻制且需要经过孵化、哺喂乳鸽等特性,提高其繁殖能力是生产效率提升的关键因素之一。蛋鸽的产蛋间隔一般为9~12 d,肉种鸽在哺喂乳鸽的情况下,产蛋间隔一般为29~35 d,受外界因素的影响,会发生产蛋异常,使产蛋间隔延长而影响产蛋数量。由于繁殖性状属于低遗传力性状,通过表型选择的遗传进展慢,筛选产蛋数相关的主效基因及其遗传变异,能够为加快产蛋鸽的选育提供依据。
单核苷酸多态性(SNP)主要是指在基因組水平上由单个核苷酸的变异所引起的DNA序列多态性,其在基因组广泛分布,数量极其丰富,是挖掘重要经济性状遗传基础的遗传标记。转录组是在特定时期、特定组织中的基因表达情况,其基因种类可能随时空改变发生变化。基于转录组数据进行SNP的分析是一种高效筛选转录区域DNA变异位点的方法[1,2]。因此,本研究基于产蛋数存在显著差异的岩鸽卵巢RNAseq数据挖掘岩鸽产蛋数相关基因及其变异位点,为岩鸽产蛋能力、繁殖能力的提升提供理论依据。
1 数据与方法
1.1 试验数据
从NCBI SRA数据库下载6只岩鸽(Columba livia)卵巢RNAseq数据[3],其中3只岩鸽年产蛋数平均为(38.67±3.40)枚(SRR13218846、SRR13218847、SRR13218848),3只岩鸽年产蛋数平均为(10.00±1.63)枚(SRR13218849、SRR13218850、SRR13218851)。
1.2 SNP检测方法
利用FastQC软件对下载的数据进行质控,获得高质量序列(Clean reads),利用STAR(Spliced transcripts alignment to a reference)对高质量序列与参考基因组(GCA_001887795.1)进行比对,去除重复序列后利用GATK(Genome analysis tool kit)进行SNP筛选,利用VCFtools进行SNP质控后注释、进化树分析和Fst计算。
2 结果与分析
2.1 SNP统计
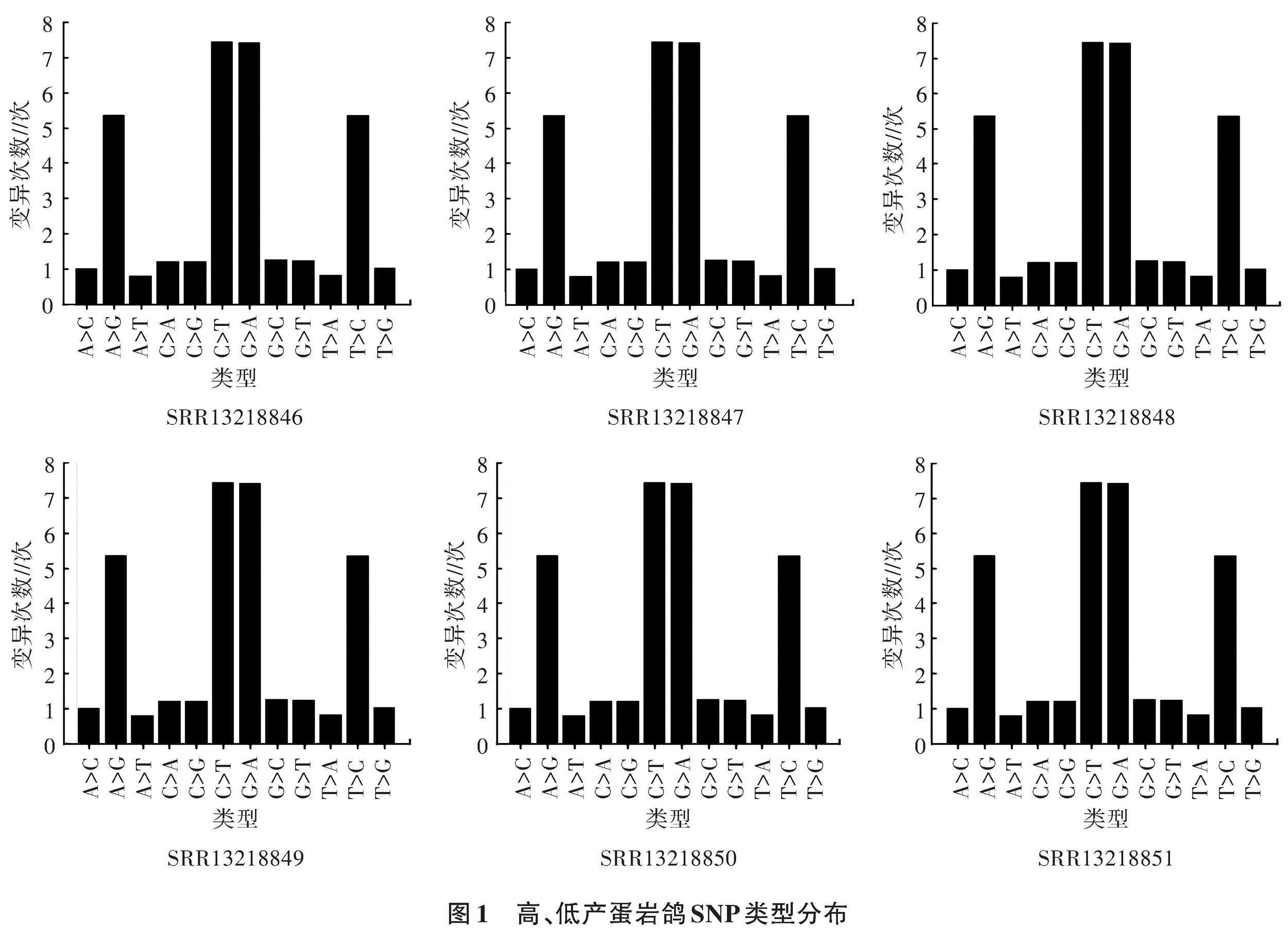
利用 Samtools 将经过排序、去PCR 重复后的文件同参考序列进行比对,6个样本共有的SNP为340 975个。根据变异的统计结果,对高产蛋数和低产蛋数个体的SNP突变每种变异类型分布进行统计(图1),结果表明,6种单核苷酸变异中,C>T和G>A变异次数较多。
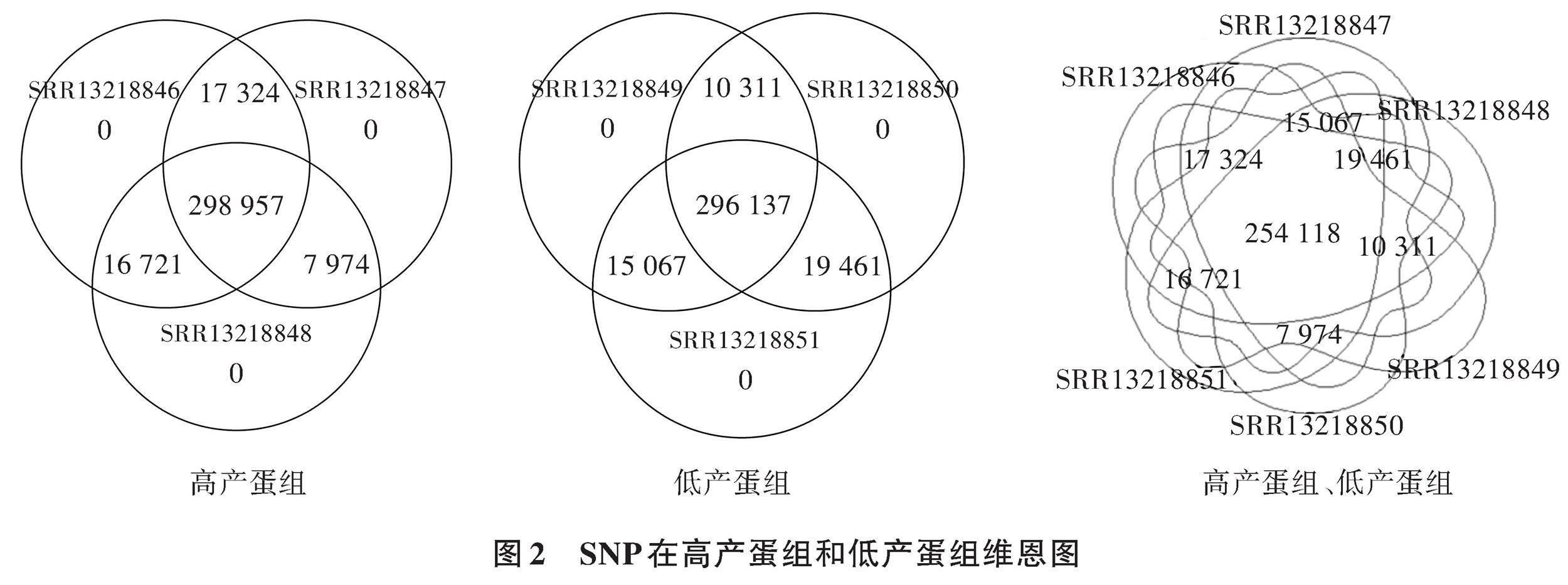
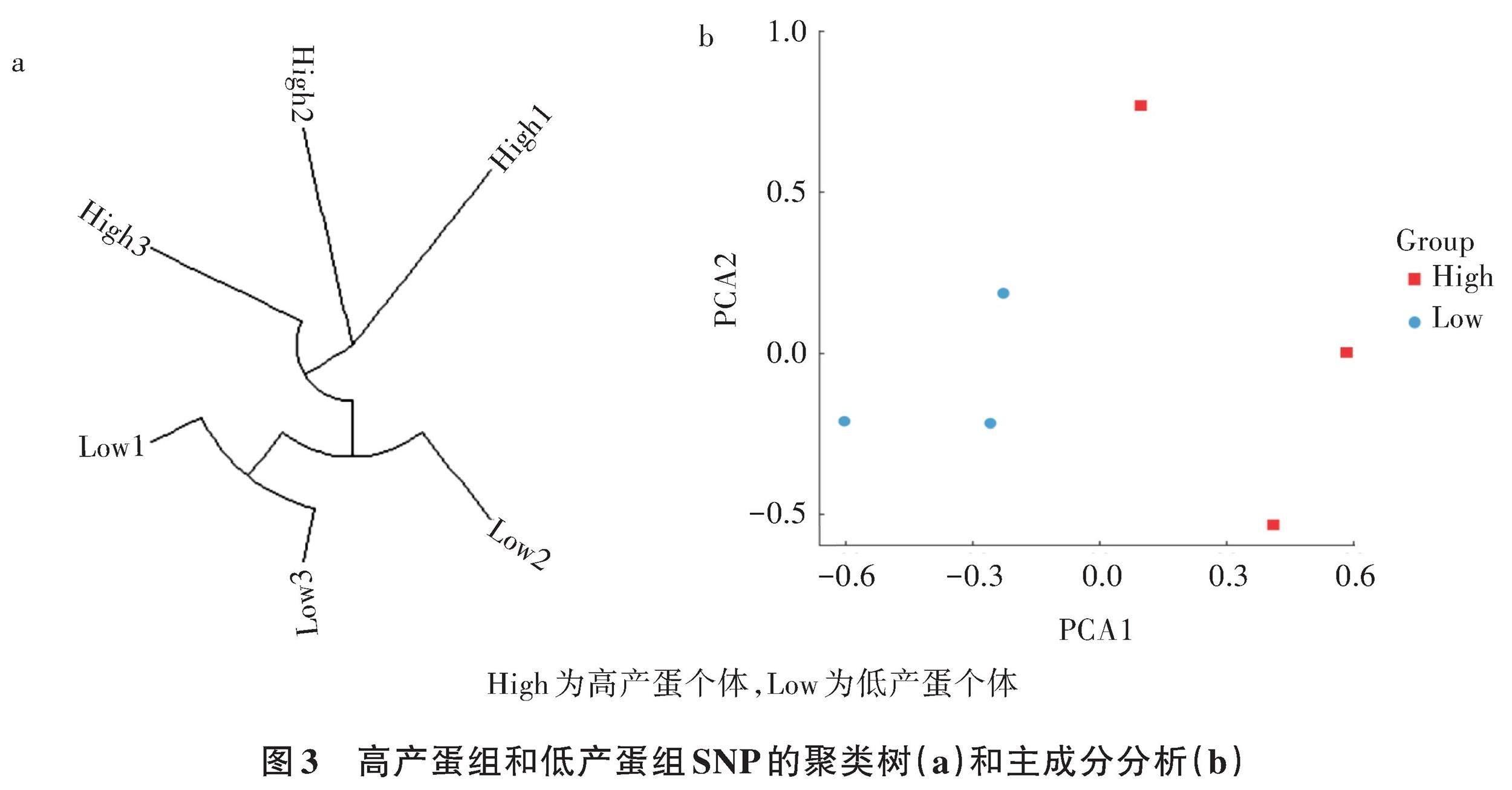
高产蛋组共有SNP数为298 957个,低产蛋组共有SNP数为296 137个,高低产蛋组共有SNP数为254 118个(图2)。通过高产蛋组、低产蛋组共有SNP构建的聚类树(图3a)可以看出,高产蛋个体和低产蛋个体分别聚类,共有SNP的主成分分析结果(图3b)表明第一主成分可以明确区分高产蛋和低产蛋个体。因此,选取的254 118个SNP可用于岩鸽产蛋数差异的遗传变异筛选。
2.2 SNP所在基因的注释
利用KOBAS(http://kobas.cbi.pku.edu.cn/)对SNP所在基因进行KEGG富集分析,发现共有5 854个基因注释到152个通路上。统计发现 SNP所在基因显著富集在孕酮调节的卵母细胞成熟、卵母细胞减数分裂和代谢等信号通路(图4和表1)。
2.3 两个群体特异SNP位点基因的生物信息分析
对高产蛋组和低产蛋组基因型进行分析,共有102个位点Fst值等于1,其中89个位于基因间隔区,2个位于内含子区,5个位于外显子区,6个位于3′UTR区。高产蛋组和低产蛋组的102个特异SNP分布在14个注释基因上(LOC110357454、KDM5A、PIGS、BNC2、GBF1、SLAIN2、FGD4、DIAPH2、KIF3B、NFIB、EPG5、OSTF1、EMB、ST3GAL3),可作为影响岩鸽产蛋数的候选基因(表2)。KEGG注释结果发现,半乳糖苷α-2,3-唾液酸转移酶3(ST3GAL3)主要参与其他类型O-糖苷生物合成,磷脂酰肌醇聚糖锚生物合成s类(PIGS)主要参与GPI锚定的生物合成(表3),推测这2个信号通路与岩鸽产蛋数相关。
3 讨论
利用转录组数据进行SNP分析,有利于快速筛选出外显子和非翻译区存在的变异位点,通过对岩鸽卵巢转录组数据进行SNP分析,发现在高产蛋组和低产蛋组存在多个变异位点,确定了群体特异SNP分布在14个基因上,半乳糖苷α-2,3-唾液酸转移酶3(ST3GAL3)主要参与其他类型O-糖苷生物合成,磷脂酰肌醇聚糖锚生物合成s类(PIGS)主要参与GPI锚定的生物合成,推测这2个信号通路与岩鸽产蛋数相关。
鸽属于晚成鸟,与鸡、鸭等其他家禽区别较大,每窝只能产2枚蛋,2窝间需要间隔10~20 d,年产蛋量较低。因此提高鸽繁殖能力,缩短产蛋间隔,可加快鸽产业发展[4]。不同品种鸽的繁殖能力存在差异,但同一品种同一鸽舍内产蛋数也存在明显差异,为研究鸽产蛋数差异的遗传基础,?对高、低产蛋组岩鸽卵巢转录组数据进行分析,鉴定出34 346个mRNA和24 601个lncRNA,其中811个mRNA和148个lncRNA(??P??<0.05)为显著差异表达[3],本研究利用该转录组数据进行SNP分析,筛选出高产蛋组和低产蛋组存在多个变异位点以及群体差异位点,为鸽产蛋数的分子育种提供依据。
通过对高产蛋组和低产蛋组SNP筛选发现高产蛋组和低产蛋组的102个特异SNP分布在14个注释基因上。OSTF1基因为破骨细胞刺激因子1,该基因的敲除能够增加小鼠小梁骨量[5]。?FGD4是一种F-肌动蛋白结合蛋白,具有GTP/GDP交换活性,对CDC42具有特异性,参与肌动蛋白细胞骨架的重组[6]。研究发现肌动蛋白解聚或聚合在卵泡发育过程中发挥重要作用[7,8]。??EPG5是一种Rab7效应子,自噬体与晚期内体/溶酶体的融合特异性与EPG5活性丧失导致自噬体与各种内吞囊泡的异常融合[9],自噬体通过自噬作用循环细胞内源物,自噬参与原发性卵泡发育和闭锁的调节[10]。EMB(ZOV3)基因?编码跨膜糖蛋白,是免疫球蛋白超家族的成员,可以通过介导细胞和细胞外基质之间的相互作用来参与细胞生长和发育,主要?在鸡卵巢卵泡的类固醇生成细胞和胚胎性腺中表达[11],也是鸽高、低产蛋数个体卵巢差异表达基因[3],可作为鸽产蛋数差异的候选基因。SLAIN2连接微管和末端跟踪蛋白并控制有丝分裂间期微管生长[12]。驱动蛋白??超家族??蛋白??(KIFs)????是??沿??微管??运输??膜状??细胞器??和??大分子??的蛋白,KIF3A和KIF3B作用于甲壳类动物的精子发生过程[13],鸟嘌呤核苷酸因子1(GBF1)通过在顺式高尔基体和内质网之间的循环来调节囊泡运输和内质网到高尔基体的转运,而在鸽的1个产蛋周期内,血清氨基酸水平升高为蛋白形成提供物质基础,蛋白合成和转运相关基因表达水平发生显著变化[14,15],内质网到高尔基体的转运是蛋白分泌途径中的第一步,推测蛋白转运相关蛋白可能在蛋形成过程中起重要调控作用。
高产蛋组和低产蛋组102个特异SNP所在的13个注释基因主要富集在糖胺聚糖生物合成/糖苷键合成和糖基磷脂酰肌醇(Glycosyl phosphatidyl inositol,GPI)锚定蛋白质的生物合成。蛋白质糖基化(N-和O-聚糖或糖胺聚糖)主要开始于内质网,并在高尔基小室100多个糖基转移酶的作用下完成。高尔基聚糖在动物卵子发生中发挥重要作用[16]。缺乏O-聚糖的卵母细胞通过增加卵泡FSH敏感性、减少凋亡和修改GDF9、BMP15表达来改变卵泡发育和提高生育能力[17]。DPAGT1介导的蛋白质??N??-糖基化对于小鼠卵母细胞和卵泡发育不可或缺?[18]。ST3β半乳糖苷α-2,3-唾液酸转移酶3(ST3GAL3)基因编码的蛋白质是一种II型膜蛋白,可催化唾液酸从CMP-唾液酸转移到含半乳糖的底物,存在于高尔基体中,是糖基转移酶家族29的成员,可以将靶蛋白α-2,3-唾液酸化[19],推测其在卵泡发育过程中发挥重要调节作用。卵泡液总唾液酸水平与生发泡卵母细胞和中期I卵母细胞呈正相关[20]。糖基磷脂酰肌醇锚定蛋白的生物合成是一种广泛存在于所有真核生物中的蛋白质翻译后修饰类型,GPI锚定蛋白主要参与细胞与细胞、细胞与环境的作用,在精子发生、发育、精子获能等方面具有重要作用[21]。?卵母细胞特异性敲除GPI锚定蛋白的雌性小鼠表现出不孕症?[22]。 PIGS是催化GPI锚定蛋白生物合成途径中的关键酶S亚基,推测其在卵母细胞发育过程中发挥重要作用。因此,ST3GAL3和PIGS可能在鸽卵泡发育和成熟中发挥重要作用,但其功能需要进行深入研究。
4 小结
利用高、低产蛋数岩鸽的卵巢转录组数据筛选岩鸽产蛋数相关SNP,发现在2个群体中共有的SNP为340 975个,C>T和G>A变异次数较多。高产蛋组和低产蛋组的102个特异SNP分布在14个注释基因上(LOC110357454、KDM5A、PIGS、BNC2、GBF1、SLAIN2、FGD4、DIAPH2、KIF3B、NFIB、EPG5、OSTF1、EMB、ST3GAL3),且富集的信号通路可能与其产蛋能力密切相关。
参考文献:
[1] JEHL F, DEGALEZ F, BERNARD M, et al. RNA-Seq data for reliable SNP detection and genotype calling: Interest for coding variant characterization and cis-regulation analysis by allele-specific expression in livestock species[J]. Frontiers in genetics, 2021, 12: 655707.
[2] ZHAO Y,WANG K,WANG W L,et al. A high-throughput SNP discovery strategy for RNA-seq data[J]. BMC genomics,2019,20(1):160.
[3] MAO H G, XU X L, CAO H Y, et al. Comparative transcriptome profiling of mRNA and lncRNA of ovaries in high and low ggg production performance in domestic pigeons (Columba livia)[J]. Frontiers in genetics, 2021, 12: 571325.
[4] 徐小钦, 董丽艳, 陶争荣, 等. 白羽王鸽与泰平王鸽种鸽繁殖性能比较[J]. 养禽与禽病防治, 2017(4): 4-5.
[5] VERMEREN M, LYRAKI R, WANI S, et al. Osteoclast stimulation factor 1 (Ostf1) KNOCKOUT increases trabecular bone mass in mice[J]. Mammalian genome, 2017, 28(11-12): 498-514.
[6] BOSSAN A, OTTMAN R, ANDL T, et al. Expression of FGD4 positively correlates with the aggressive phenotype of prostate cancer[J]. BMC cancer, 2018, 18(1): 1257.
[7] 王清泉. 肌動蛋白解聚因子在小鼠卵泡的发育过程中的表达与调节[D]. 哈尔滨: 东北农业大学,2009.
[8] CHENG Y, FENG Y, JANSSON L, et al. Actin polymerization-enhancing drugs promote ovarian follicle growth mediated by the Hippo signaling effector YAP[J]. Official publication of the federation of American societies for experimental biology,2015,29(6): 2423-2430.
[9] WANG Z, MIAO G Y, XUE X, et al. The Vici syndrome protein EPG5 Is a Rab7 effector that determines the fusion specificity of autophagosomes with late endosomes/lysosomes[J]. Molecular cell, 2016, 63(5): 781-795.
[10] ZHOU J W, PENG X W, MEI S Q. Autophagy in ovarian follicular development and atresia[J]. International journal of biological sciences, 2019, 15(4): 726-737.
[11] KUNITA R,NAKABAYASHI O,KIKUCHI T, et al. Predominant expression of a Z-chromosome-linked immunoglobulin superfamily gene, ZOV3, in steroidogenic cells of ovarian follicles and in embryonic gonads of chickens[J]. Differentiation, 1997, 62(2): 63-70.
[12] VAART B, MANATSCHAL C, GRIGORIEV I, et al. SLAIN2 links microtubule plus end-tracking proteins and controls microtubule growth in interphase[J]. The journal of cell biology, 2011, 193(6): 1083-1099.
[13] LU Y,WANG Q,WANG D H, et al. Functional analysis of KIF3A and KIF3B during spermiogenesis of Chinese mitten crab Eriocheir sinensis[J]. PLoS one, 2014, 9(5): e97645.
[14] AKINTAYO A, STANLEY P. Roles for golgi glycans in oogenesis and spermatogenesis[J]. Frontiers in cell and developmental biology, 2019, 7: 98.
[15] REN Y, LI X T, HAN G F, et al. Dynamic variations in serum amino acid and the related gene expression in liver, ovary, and oviduct of pigeon during one egg-laying cycle[J]. Poultry science, 2021, 100(7): 101184.
[16] LU L Z, XU X Q, DU X, et al. Transcriptome analyses to reveal the dynamic change mechanism of pigeon magnum during one egg-laying cycle[J]. Molecular reproduction and development, 2020, 87(11): 1141-1151.
[17] GRASA P, PLOUTARCHOU P, WILLIAMS S A. Oocytes lacking O-glycans alter follicle development and increase fertility by increasing follicle FSH sensitivity, decreasing apoptosis, and modifying GDF9:BMP15 expression[J]. Official publication of the federation of American societies for experimental biology, 2015, 29(2): 525-539.
[18] LI H, YOU L J TIAN Y F, et al. DPAGT1-Mediated protein N-glycosylation is indispensable for oocyte and follicle development in mice[J]. Advanced science, 2020, 7(14): 2000531.
[19] QI F,ISAJI T,DUAN C,et al. ST3GAL3, ST3GAL4, and ST3GAL6 differ in their regulation of biological functions via the specificities for the α2,3-sialylation of target proteins[J]. Official publication of the federation of American societies for experimental biology, 2020, 34(1): 881-897.
[20] CETIN B A, OCAL P, IREZ T, et al. The association between follicular fluid sialic acid levels, oocyte quality, and pregnancy rates[J]. Reproductive sciences, 2022, 29(2): 633-638.
[21] WATANABE H, KONDOH G. Mouse sperm undergo GPI-anchored protein release associated with lipid raft reorganization and acrosome reaction to acquire fertility[J]. Journal of cell science, 2011, 124(15): 2573-2581.
[22] ALFIERI J A, MARTIN A D, TAKEDA J, et al. Infertility in female mice with an oocyte-specific knockout of GPI-anchored proteins[J]. Journal of cell science, 2003, 116(11): 2149-2155.

