基于BLI技术的BamAβ-barrel结合化合物检测方法的建立
郑怡凡?朱小红?陈明华?卢宇 ?魏元娟? 时文静? 司书毅?李妍


摘要:目的 以革蘭阴性菌外膜蛋白折叠辅助因子关键蛋白BamA为靶蛋白,基于生物膜干涉(Biolayer interferometry,BLI)技术建立化合物与BamA蛋白β折叠结构域(BamAβ-barrel)结合活性的评价方法,为建立靶向BamA蛋白的抗革兰阴性菌先导物奠定基础。方法 应用BLI方法检测BamAβ-barrel与已知的阳性化合物darobactin的结合活性。原核表达并纯化带有His标签的大肠埃希菌BamAβ-barrel蛋白,使用表面活性剂LDAO对其进行复性和折叠;使用生物素标记折叠和未折叠蛋白,并结合到超级链霉亲和素(super streptavidin,SSA)生物传感器,然后检测蛋白与不同浓度的darobactin结合信号的变化,同时做无蛋白或darobactin稀释液对照;空白对照采用未结合生物素化的BamAβ-barrel蛋白的传感器,检测上述系列稀释样品。相应信号采用Steady state analysis方式拟合分析,计算平衡常数(KD)值。结果 成功获得高纯度的折叠状态BamAβ-barrel蛋白,通过BLI技术检测到折叠状态的BamAβ-barrel与阳性化合物darobactin具有良好结合活性且呈现浓度依赖性,R2为0.9998,KD值为(2.2E-06±8.0E-08)M。结论 基于BLI技术成功建立了折叠状态的BamAβ-barrel-化合物结合活性的评价方法,为后续BamA蛋白靶向性抗革兰阴性菌抗生素的发现建立基础。
关键词:革兰阴性菌;Darobactin;BamAβ-barrel;生物膜干涉技术
中图分类号:R978.1文献标志码:A
Establishment of a method to detect the binding activity of compounds and BamA β-barrel based on BLI assay
Zheng Yifan, Zhu Xiaohong, Chen Minghua, Lu yu, Wei Yuanjuan, Shi Wenjing, Si Shuyi, and Li yan
(Beijing Key Laboratory of Antimicrobial Agents, Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100050)
Abstract Objective Using the key protein BamA of the Gram-negative bacterial outer membrane protein folding cofactor as the target protein, this study established a method to detect the binding activity between BamAβ-barrel and compounds based on biolayer interferometry (BLI) technology and laid the foundation for screening antibiotics against Gram-negative bacteria targeting the BamA protein. Methods Double subtraction BLI was used to detect the interaction of BamAβ-barrel and the positive compound darobactin. E. coli BamAβ-barrel was expressed and purified and then it was refolded with LDAO in vitro. The refolded BamAβ-barrel or unfolded BamAβ-barrel were labeled with biotin and bound to the Super Streptavidin (SSA) biosensor. The binding of darobactin to BamAβ-barrel was detected based on the changes in binding signals. To identify the diluted samples, a blank control was employed, which involved employing unbound biotinylated BamAβ-barrel protein sensors. This control was performed simultaneously with the protein-free or darobactin dilution control. The signal that corresponded to it was fitted and evaluated using steady-state analysis in order to get the value of the equilibrium constant (KD). Results High-purity folded BamAβ-barrel was successfully obtained. The folded BamAβ-barrel bound to the sensor showed good binding activity with darobactin with R2=0.9998 and KD=2.2E-06 ± 8.0E-08 M. Conclusion An optimized BLI method for detecting the interaction between compounds and BamAβ-barrel had been established, which could be used for antibiotics against Gram-negative bacteria targeting BamAβ-barrel.
Key words Gram-negative bacteria; Darobaction; BamAβ-barrel; Biolayer interferometry
抗生素耐藥菌导致的感染性疾病是21世纪人类健康和公共卫生领域面临的巨大威胁之一。相对于革兰阳性(Gram-positive bacteria,G+)菌,革兰阴性菌(Gram-negative bacteria,G-)耐药性感染的治疗迫切性更为突出。中国CHINET 2005—2022年的临床监测数据显示G-耐药菌(大肠埃希菌、肺炎克雷伯菌、鲍曼不动杆菌)的检出率则仍呈现相当程度的增长趋势[1]。2017年由世界卫生组织(World Health Organization,WHO)发布的亟须推动新抗生素研发的重点病原体清单中,列为1类重点、迫切性最高的3类病原:耐碳青霉烯类的鲍曼不动杆菌、铜绿假单胞菌、肠杆菌科细菌均为G-菌[2]。但在药物研发方面,相较于G+菌,抗G-菌新药研发的进展明显缓慢。因此新型抗G-菌抗生素的发现具有重要的意义。
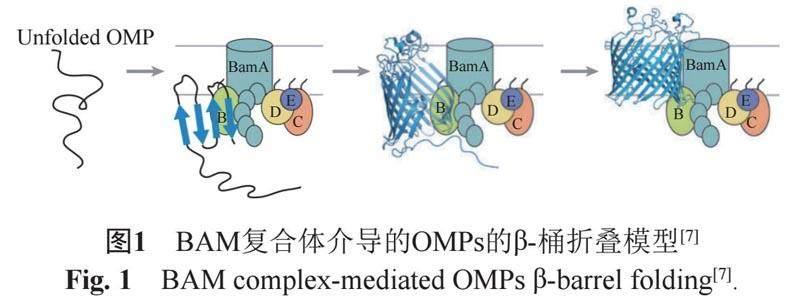
相较G+菌,G-菌有独特的非对称外膜结构(outer membrane,OM),由脂多糖(lipopolysaccharide,LPS)和磷脂组成基本结构,外膜蛋白(outer membrane proteins,OMPs)镶嵌其中。LPS组成的厚达8~10 nm的生物学屏障可保护G-菌免受菌体外环境中的有害分子的侵害,同时也是造成G-菌固有耐药的重要因素和内毒素形成的物质基础。外膜受损时,菌细胞会出现外膜稳定性下降、抗生素敏感性增加、易被免疫细胞清除等现象,对细菌造成致死性伤害。因此,抑制G-菌外膜生物合成是广受认可的抗G-菌药物研发策略[3-4]。OMPs是一大类功能各异的蛋白,镶嵌于LPS层中,在LPS层的形成、OM的稳定以及致病菌毒力方面发挥着重要作用。OMPs在外膜中主要以反向平行的β-折叠(β-Strands)通过相邻的氢键结合形成的β桶状(β-barrel)结构存在,这种结构是OMPs发挥正常功能的基础。OMPs通常在细胞质中合成,借助N端信号肽结合Sec和Sur伴侣蛋白完成跨膜的转运,然后通过BAM(β-barrel assembly machine)复合体在外膜中进行折叠组装[5-7]。G-菌的BAM复合体通常包括BamA、B、C、D和E 5个蛋白,其中BamA是OMPs折叠和组装于LPS层的核心场所(图1)[7]。BamA本身也是β折叠的外膜蛋白,其C端β桶结构域定位于外膜,是OMPs折叠和插入外膜的场所,其涉及到的β桶组装机制在G-菌中是高度保守的[8-10]。BamA蛋白的N端由5个多肽转运结构域(POTRA)组成,存在于周质空间,与其他BAM复合体相互作用,完成OMPs的转运且维持BAM复合体的稳定。BamA是G-菌的必需蛋白,其编码基因的敲除会导致外膜合成的损伤,并最终导致细胞死亡;同时BamA蛋白定位于外膜,其靶向性抗生素可能不需要跨外膜发挥抗菌活性,能够克服外膜带来的固有耐药,增强其他抗生素的敏感性。目前已经发现多个具有抗G-菌活性的先导物具有潜在的BamA抑制活性[11],证实了BamA蛋白作为新型抗G-菌抗生素研发靶标的潜能,但是目前尚未有以BamA尤其是BamA折叠结构域为靶标的抗G-菌抗生素的高通量筛选的报道。
生物膜干涉(biolayer interferometry,BLI)技术是一种无须使用酶、荧光或者放射性元素对待测样本进行标记就能够实时检测和表征分子相互作用的一项技术,在BLI分析中获得的亲和常数(KD值)是抗体、适配体、肽等生物分子质量的良好指标,近年来被广泛应用于蛋白与小分子结合、抗体筛选等多个研究领域。本研究通过体外折叠获得大量BamA活性蛋白,以已经报道的具有明确BamA蛋白结合活性的darobactin为探针,基于BLI技术建立评价检测BamA蛋白β桶结构域与化合物结合活性的方法,为高通量筛选靶向BamA蛋白的抗G-菌抗生素奠定了基础。
1 材料与方法
1.1 材料
1.1.1 试剂
Darobactin由中国医学科学院医药生物技术研究所国家新药(微生物) 筛选实验室制备;pET-15b表达载体由本实验室保存;蛋白标准Maker(BioRad);蛋白胨、酵母粉、琼脂粉(Oxoid);氨苄西林、4×非变性蛋白上样缓冲液、异丙基-β-D硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)、牛血清白蛋白(bovine serum albumin,BSA)(索莱宝); HisTrapTM 层析柱、HiTrap Q HP阴离子交换柱(GE Healthcare);5×蛋白上样缓冲液(碧云天);考马斯亮蓝染色液(普利莱);超滤浓缩管(Millipore);PBS磷酸盐缓冲液(中杉金桥);Tween-20(Sigma);生物素化标记试剂 NHS-LC-LC-Biotin、Zeba desalting spin columns脱盐柱(Thermo);96孔板(Corning Costar);SSA传感器(ForteBio);其他试剂均为国产分析纯。
1.1.2 缓冲液
缓冲液A1:8 mol/L尿素,50 mmol/L Tris,
300 mmol/L NaCl,pH 8.0;缓冲液A2:20 mmol/L Tris,LDAO 0.1%,pH 8.0;洗脱液B1:8 mol/L尿素,50 mmol/L Tris,300 mmol/L NaCl,300 mmol/L咪唑,pH 8.0;洗脱液B2:20 mmol/L Tris,LDAO 0.1%,500 mmol/L NaCl,pH 8.0;复性缓冲液:
50 mmol/L Tris,300 mmol/L NaCl,500 mmol/L精氨酸,0.5% LDAO,10 mmol/L DTT,pH 8.0。
1.1.3 仪器
超净工作台、生化培养箱(哈东联);恒温摇床(北京沃德创技贸有限公司);多功能微孔板检测仪(PerkinElmer);高速冷冻离心机(Sigma);高压细胞破碎仪(Constant Systems);加热制冷型金属浴(Thermo);蛋白质电泳仪、AKTA层析系统(GE Healthcare);微量分光光度计(Life Real);分子相互作用检测仪器(Octet Red96)。
1.2 方法
1.2.1 BamAβ-barrel蛋白的原核表达
全基因合成大肠埃希菌(E.coli)BamA蛋白β-桶折叠结构域(421aa-810aa)编码基因序列(Gene ID:944870),在其5'端和3'端分别插入NdeI和Xhol酶切位点。构建重组质粒pET-15b-bamAβ-barrel(N端带有6×His标签),通过热激法转化至大肠埃希菌E. coli BL21感受态细胞,通过氨苄西林(Amp)抗性筛选获得阳性克隆。阳性克隆接种至8 mL含有100 μg/mL Amp的LB液体培养基,37 ℃以180 r/min的转速震荡培养至对数期(A600=0.6),IPTG(0.5、1、1.5和2 mmol/L)诱导表达(110 r/min,18 ℃,12 h)。蛋白表达量通过10% SDS-PAGE和考马斯亮蓝染色进行检测。
1.2.2 BamAβ-barrel蛋白的纯化
终浓度为0.5 mmol/L 的IPTG加入到2 L培养至对数期的菌液,18 ℃培养12 h。5000 r/min离心20 min收集菌体,而后重悬于缓冲液A1。反复3次高压破碎(38 kpsi)后收集破碎液,5000 r/min离心(4 ℃)30 min以除去细胞碎片。上清经0.22 μm滤膜过滤后,在4 ℃环境下加载到1 mL HisTrapTM亲和层析色谱柱上,使用AKTA系统对蛋白进行纯化:五倍柱体积的缓冲液A1平衡色谱柱,然后使用洗脱液B1进行梯度洗脱(10%、40%、60%和100%),对280 nm处的UV峰值进行监测并收集蛋白,10% SDS-PAGE检测蛋白纯度。使用10kD超滤离心管离心(4 ℃,5000 r/min)浓缩蛋白,微量分光光度计定量后调整蛋白浓度为1 mg/mL,-80 ℃冷冻保存。
1.2.3 BamAβ-barrel蛋白的复性折叠与纯化
蛋白的复性和折叠按照Hartmann等[11]的描述进行。使用复性缓冲液稀释1 mg/mL的蛋白储液至
0.1 mg/mL,4 ℃缓慢搅拌12 h。然后使用10 kD的透析袋在20 mmol/L Tris (pH 8.0)缓冲液中透析
12 h,全程保持4 ℃低温。透析后的蛋白溶液加载到HiTrap Q HP离子交换柱上,使用5倍体积的缓冲液A2平衡后,以洗脱液B2进行梯度洗脱(15%、25%、35%、45%、55%、65%、75%、85%和100%),对280 nm处的UV峰值进行监测,收集洗脱液B2含量为45%~85%的洗脱液。
1.2.4 BamAβ-barrel蛋白的测定(Semi-SDS-PAGE)
蛋白样品加入4×蛋白非变性缓冲液(无SDS),混合均匀后直接进行10% SDS-PAGE电泳。电泳全程80V恒压,冰浴。
1.2.5 生物膜干涉技术检测darobactin与BamAβ-barrel蛋白间相互作用
参照说明书对BamAβ-barrel蛋白(Folded BamAβ-barrel)和仅得到复性但仍处于非折叠状态的BamAβ-barrel蛋白(Unfolded BamAβ-barrel)进行生物素化,然后使用PBST缓冲液稀释至200 μg/mL。4根SSA传感器分别置于传感器支架第一、二列,每列两根,第一列结合生物素化蛋白,第二列为空白对照。使用PBST-BSA(含0.2% BSA的PBST)缓冲液将darobactin自2 μg/mL起梯度稀释至0.125 μg/mL,保持每孔DMSO浓度均为1%。在96孔板中按照表1所示加入样品,A行为待测样品,B行为空白对照,将样品孔替换为等量的Reference缓冲液,其余各孔不变,每孔总体积均为200 μL。在Data Acquisition 9.0软件中按照表2所示的检测条件进行设置,检测不同样品孔与传感器的结合。使用Data analysis 9.0软件对数据进行分析。选择“双参比”模式,以对应的未结合蛋白的传感器及Reference缓冲液作为对照进行扣除,以稳态分析(Steady state analysis)方式拟合,计算响应值与KD值。
2 结果与分析
2.1 BamAβ-barrel原核表达质粒的构建与鉴定
重组质粒pET-15b-bamAβ-barrel轉化入大肠埃希菌BL21细胞并涂布于Amp抗性LB平板,随机挑取阳性菌落培养,提取质粒后,进行双酶切鉴定,琼脂糖凝胶电泳显示出预期为1185 bp的目的基因和5.7 kbp的载体片段(图2)。对同一阳性菌落bamAβ-barrel表达片段测序,比对分析显示,重组质粒基因序列与预期构建序列相似率达100%,表明成功构建了重组质粒pET-15b-bamAβ-barrel。
2.2 BamAβ-barrel原核表达
Amp抗性筛选得到的含有重组质粒的菌液在0.5、1、1.5和2 mmol/L的IPTG浓度下低温诱导蛋白表达,经由10% SDS-PAGE进行检测,结果如图3A所示,与空载体菌相比,在37~50 kDa之间,IPTG诱导的全菌和包涵体中均存在与目的蛋白分子量
(43 kDa)相符的明显条带。IPTG浓度对蛋白表达量无明显影响。图3B显示,0.5 mmol/L IPTG诱导后的全菌和包涵体中在37~50 kDa出现明显条带,破碎后上清和培养基中无明显条带,与文献报道一致[11]。
2.3 BamAβ-barrel蛋白的纯化
由于BamAβ-barrel蛋白大量表达后以不溶性包涵体的形式存在,因此使用含有高浓度变性剂(8 mol/L尿素)的缓冲液对蛋白进行处理[12],然后通过亲和层析法进行纯化。结果发现在第54 min左右,洗脱液B1含量为10%~40%时,UV280 nm处出现峰值(图4A)。对流穿液和洗脱液B1含量为10%、40%和60%时的洗脱液进行10% SDS-PAGE检测,结果显示在43 kDa处出现单一条带(图4B)。
2.4 BamAβ-barrel的复性折叠与分离纯化
纯化后获得的蛋白在含有高浓度变性剂(8 mol/L尿素)的缓冲液中以非折叠状态的形式存在,并不具备生物活性,因此需要对蛋白进行复性,进一步使之重新折叠。蛋白折叠后立体结构发生变化,具有折叠结构的蛋白未经变性处理时在电泳中的移动速率比线性结构的非折叠蛋白更为迅速,因此可以借此区分折叠和非折叠蛋白[13]。在含有0.5%表面活性剂LDAO的复性缓冲液中透析12 h后,通过10% Semi-SDS-PAGE检测蛋白的折叠状态,结果如图5A所示,仅经过复性缓冲液稀释未经透析的蛋白在43 kDa出現单一条带,仍处于非折叠状态,而经过透析处理后的样品在43 kDa与约30 kDa处呈现两个条带,这与文献报道相符,证明获得了折叠状态的蛋白[11]。
为了去除未折叠的BamAβ-barrel蛋白,应用离子交换柱对折叠后的蛋白样品进行纯化。发现在第70 min左右,洗脱液B2含量为55%时,UV280nm处出现峰值(图5B)。收集洗脱液B2含量为45%~85%时的流出液进行10% Semi-SDS-PAGE,结果显示浓度为55%时洗脱下来的蛋白为复性折叠的蛋白,在30 kDa附近呈现单一条带,具有较高的纯度(图5C)。因为后续BLI检测方法中使用的缓冲液体系有所改变,因此对该条件(PBST+0.2% BSA,pH 6.0)下蛋白的稳定性进行了进一步检测,结果依旧仅在30 kDa附近发现单一蛋白条带,说明该缓冲液对折叠蛋白未产生影响(图5D)。
2.5 生物膜干涉技术检测蛋白与小分子相互作用
Darobactin是已知的与折叠状态的BamAβ-barrel特异性结合的化合物。为了验证BLI技术是否能应用于检测化合物与BamAβ-barrel的结合,本研究以darobactin为阳性化合物对该方法进行了评价[14]。同时以未折叠BamAβ-barrel蛋白(Unfolded BamAβ-barrel)为对照。为了排除darobactin与传感器的结合造成的假阴性,首先检测了darobactin与空白传感器的结合,结果如图6A所示,darobactin与空白传感器具有明显的非特异性结合。因此在后续检测中使用封闭剂Tween 20与BSA对缓冲液体系进行优化,得到能够较好消除darobactin与空白传感器非特异性结合影响的缓冲液体系(PBST+0.2% BSA,pH 6.0)(图6B)。如图6C所示, darobactin能与Folded BamAβ-barrel结合,并且随着化合物浓度增加,响应值明显增强,具有浓度依赖性,R2=0.9998,KD=2.2E-06±8.0E-08M;同时,并未检测到darobactin与Unfolded BamA β-barrel蛋白存在明显的相互作用(图6D)。
3 讨论
OM是G-菌特有的结构,外膜蛋白是近年来抗G-菌药物研发中受到广泛关注的潜在靶标,主要集中于LPS的合成和转运过程[15-17]。随着OMPs在外膜中的重要功能和结构的解析,OMPs靶向性抗生素的发现也受到越来越多的关注。外膜蛋白LptD是LPS在外膜组装的场所,是必需蛋白。主蛋白OmpA在外膜稳定性中起着重要的作用,同时也是细菌毒力因子,介导生物被膜的形成。OMPs中的孔道蛋白OmpF、OmpC、phoE和Maltoporin等是营养物质和部分抗生素进入菌体内的通道。此外,有些OMPs还具有蛋白酶、黏附因子和外排泵等功能,是重要的致病性病原菌的毒力因子和耐药因素。OMPs的缺失会导致LPS层的缺损和菌体死亡;毒力因子下调,病原菌致病性降低。因此,靶向OMPs生物合成的抗生素具有独特的抗菌优势。首先,抗生素本身具有抑菌、杀菌和降低病原菌毒力的作用;其次,此类抗生素导致的外膜损伤能够增加跨膜困难的抗生素对GNB的抗菌活性,从而发挥增敏作用,克服GNB固有耐药。因此,OMPs生物合成的调控因子是目前抗GNB药物研发的热点靶标之一。
作为OMPs折叠辅助因子的BAM复合体,其核心组成部分BamA蛋白既是外膜蛋白折叠的场所,在外膜的组装和稳定中发挥重要作用,是细菌的必需蛋白,同时作为外膜蛋白,其靶向性抗生素无须跨膜即可发挥抗菌活性。因此BamA蛋白在抗G-菌抗生素的研发中可能具有良好的成靶性。目前已经报道多种化合物的抗G-菌活性与BamA抑制相关[18]。JB-95是一种新型β发卡环肽,可与包括BamA在内的多种外膜蛋白结合,选择性地破坏大肠埃希菌外膜[19];单克隆抗体MAB1与细胞外BamA表位直接结合,抑制其β桶折叠活性,诱导周质应激,破坏外膜完整性并杀死细菌[20];由变形杆菌分泌的凝集素样细菌素(LlpAs)可选择性识别BamA蛋白结合到靶细胞表面,诱发BAM复合体机制受损,实现对革兰阴性菌的杀伤作用[21];分离自线虫共生菌的天然环肽darobactin可以选择性与BamA折叠结构域结合并抑制其活性,抑制外膜形成,并且在体内外均表现出良好的抗G-菌活性,是目前最为明确且活性最好的BamA靶向性抑制剂[22]。
Darobactin是目前研究得最为深入的BamA抑制剂,其与BamA蛋白的结合模式已经得到较为全面的解析。BamA蛋白在细胞质中合成后以线性的结构存在,然后转运到外膜进行C端的β-折叠与外膜组装。与多数非常稳定的β折叠蛋白不同,折叠状态BamA蛋白的β桶状结构域存在一个不稳定且呈现开放状态的“侧门”,以利于OMPs的折叠和在外膜中的组装。Darobactin通过模仿β-链的结合方式,以高亲和力与BamA侧门结合,稳定BamA的侧向封闭构象阻止其打开,抑制同源新生外膜蛋白与BamA的结合,进而抑制外膜形成,发挥抗菌活性[14,23]。在本研究中,darobactin与折叠的BamA蛋白具有明显结合,这再次确证了darobactin能够与折叠状态的BamA结合发挥其抗菌活性这一论点。此外,darobactin与未折叠的BamA蛋白之间不存在明显的相互作用,这可能与darobactin和BamA的结合特性相关。darobactin自身的反向平行的β折叠构象与BamA的Gly424-Ile430配对,形成主链氢键以发挥竞争性抑制剂作用[24]。线性的BamA蛋白不存在折叠构象;同时,与未折叠的BamA相比,形成桶状折叠结构域的BamA蛋白的氨基酸之间(比如Gly424和Ile430)在空间距离上发生了很大的变化,这些都会造成其与darobactin亲和力的改变。这一结果说明本研究建立的检测方法较好地还原了天然状态下BamA蛋白与darobactin的结合方式,能够应用于筛选靶向折叠BamA蛋白的特异性抑制剂。
目前已知的BamA靶向性抗G-菌先导物大部分都是在机制研究中偶然发现的。Li等[25]以酵母双杂交技术建立BamA/BamD蛋白相互作用模型,高通量筛选相互作用阻断剂,发现了IMB-H4能够与未折叠BamA结合,阻断其与BamD蛋白结合活性,并具有抗G-菌活性,这可能与影响BamA蛋白自身的折叠有关[26]。相对于未折叠的BamA蛋白,位于外膜的折叠状态的BamA蛋白可能具有更好的成靶性,但是目前尚未有以折叠状态的BamA蛋白为靶向的高通量筛选的报道。本研究是首次以BLI技术建立具有折叠BamA蛋白结合活性的化合物的评价方法,发现经过生物素化的折叠BamAβ-barrel蛋白可以很好地与传感器结合,同时以darobactin为阳性化合物也能检测到其与折叠BamAβ-barrel蛋白的结合。这证明该BLI检测方法能够应用于BamAβ-barrel结合化合物的结合活性检测,这也为后续高通量筛选靶向BamA蛋白的抗革兰阴性菌抗生素的发现奠定了的基础。
参 考 文 獻
CHINET 2023上半年中国细菌耐药监测结果[EB/OL]. http://www.chinets.com/Document.
WHO publishes list of bacteria for which new antibiotics are urgently needed[EB/OL]. https://www.who.int/en/news-room/detail/27-02-2017-who-publishes-list-of-bacteria-for-which-new-antibiotics-are-urgently-needed.
Pagès J M. Antibiotic transport and membrane permeability: new insights to fight bacterial resistance[J]. Biologie aujourdhui, 2017, 211(2): 149-154.
MacNair C R, Tsai C N, Brown E D. Creative targeting of the Gram-negative outer membrane in antibiotic discovery[J]. Ann N Y Acad Sci, 2020, 1459(1): 69-85.
Rapoport T A. Protein translocation across the eukaryotic endoplasmic reticulum and bacterial plasma membranes[J]. Nature, 2007, 450(7170): 663-669.
Driessen A J M, Nouwen N. Protein translocation across the bacterial cytoplasmic membrane[J]. Annu Rev Biochem, 2008, 77(1): 643-667.
Hagan C L, Silhavy T J, Kahne D. β-Barrel membrane protein assembly by the Bam complex[J]. Annu Rev Biochem, 2011, 80(1): 189-210.
Hagan C L, Westwood D B, Kahne D. Bam lipoproteins assemble BamA in vitro[J]. Biochemistry, 2013, 52(35): 6108-6113.
Konovalova A, Kahne D E, Silhavy T J. Outer membrane biogenesis[J]. Annu Rev Microbiol, 2017, 71(1): 539-556.
Doyle M T, Bernstein H D. BamA forms a translocation channel for polypeptide export across the bacterial outer membrane[J]. Mol Cell, 2021, 81(9): 2000-2012.e3.
Hartmann J B, Zahn M, Burmann I M, et al. Sequence-specific solution NMR Assignments of the β-barrel insertase bama to monitor its conformational ensemble at the atomic level[J]. J Am Chem Soc, 2018, 140(36): 11252-11260.
Das A, Mukhopadhyay C. Urea-mediated protein denaturation: A consensus view[J]. J Phys Chem B, 2009, 113(38):12816-12824.
Opdam L V, Polanco E A, de Regt B, et al. A screening method for binding synthetic metallo-complexes to haem proteins[J]. Anal Biochem, 2022, 653: 114788.
Ritzmann N, Manioglu S, Hiller S, et al. Monitoring the antibiotic darobactin modulating the β-barrel assembly factor BamA[J]. Structure, 2022, 30(3): 350-359.e3.
Simpson B W, Trent M S. Pushing the envelope: LPS modifications and their consequences[J]. Nat Rev Microbiol, 2019, 17(7): 403-416.
Ho H, Miu A, Alexander M K, et al. Structural basis for dual-mode inhibition of the ABC transporter MsbA[J]. Nature, 2018, 557(7704): 196-201.
代晓伟, 朱小红, 司书毅, 等. 基于BLI技术的脂多糖转运蛋白LptA/LptC相互作用检测方法的建立[J]. 生物工程学报, 2021, 37(9): 3300-3309.
朱小红, 郑怡凡, 张晶, 等. BAM复合体及其靶向性抗革兰阴性菌药物研究进展[J]. 中国抗生素杂志, 2023, 48(6): 617-624.
Urfer M, Bogdanovic J, Lo Monte F, et al. A peptidomimetic antibiotic targets outer membrane proteins and disrupts selectively the outer membrane in Escherichia coli[J]. J Biol Chem, 2016, 291(4): 1921-1932.
Storek K M, Auerbach M R, Shi H, et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal[J]. Proc Natl Acad Sci USA, 2018, 115(14): 3692-3697.
Ghequire M G K, Swings T, Michiels J, et al. Hitting with a BAM: Selective killing by lectin-like bacteriocins[J]. mBio, 2018, 9(2): e02138-17.
Hart E M, Mitchell A M, Konovalova A, et al. A small-molecule inhibitor of BamA impervious to efflux and the outer membrane permeability barrier[J]. Proc Natl Acad Sci USA, 2019, 116(43): 21748-21757.
Doyle M T, Jimah J R, Dowdy T, et al. Cryo-EM structures reveal multiple stages of bacterial outer membrane protein folding[J]. Cell, 2022, 185(7): 1143-1156.e13.
Kaur H, Jakob R P, Marzinek J K, et al. The antibiotic darobactin mimics a β-strand to inhibit outer membrane insertase[J]. Nature, 2021, 593(7857): 125-129.
Li Y, Zhu X, Zhang J, et al. Identification of a compound that inhibits the growth of Gram-negative bacteria by blocking BamA-BamD interaction[J]. Front Microbiol, 2020, 11: 1252.
Tomasek D, Rawson S, Lee J, et al. Structure of a nascent membrane protein as it folds on the BAM complex[J]. Nature, 2020, 583(7816): 473-478.
基金項目:国家自然基金(No. 32141003);中国医学科学院医学与健康科技创新工程(No. 2021-I2M-1-070)
作者简介:郑怡凡,女,生于1999年,在读硕士研究生,主要研究方向为抗感染药物的发现和作用机理研究,E-mail: zhengyifan@imb.pumc.edu.cn
*通信作者,E-mail: liyan@imb.pumc.edu.cn

