土壤固碳微生物的绝对定量检测实验设计
付小花, 陈 皓, 张 华, 周 磊, 唐贤春
(同济大学a.环境科学与工程学院;b.污染控制与资源化研究国家重点实验室,上海 200092)
0 引 言
在全球变暖及我国“双碳”目标提出的背景下,自养微生物CO2的固定同化已成为研究热点[1-3]。对特定环境,如湿地、森林、农田等土壤中的固碳微生物种类及数量进行监测,有助于近似估算这些区域的固碳潜能,对我国实现“碳中和碳达峰”目标有积极意义[4-5]。目前关于固碳微生物的分析有培养法和不需培养的分子生物学方法,培养法费时费力,而且结果中缺失不可培养的微生物信息,而不需培养的分子生物学方法在分析微生物群落时具有快速、高效、信息全面等特点[6]。常用的基于16SrRNA基因的测序分析,得到的是所有微生物的类群信息,难以获得确切的固碳微生物数量信息。以卡尔文循环的关键酶——核酮糖-1,5-二磷酸羧化酶/加氧酶(Ribulose-1,5-bisphosphate carboxylase/oxygenase,Rubisco)的大亚基编码基因cbbL作为靶标表征固碳微生物是科学研究中常用的方法,被广泛应用于固碳微生物分析[7-8]。
目前主要运用实时荧光定量聚合酶链式反应(PCR)法进行以cbbL基因为靶标的固碳微生物定量检测[9],该定量方法依赖于Ct值(Ct指反应管内的荧光信号达到设定的阈值时所经历的循环数),需要用标准品制备标准曲线,此过程较为费时费力,而且环境样品中抑制剂的存在会改变样品的Ct值,使得定量结果不准确。微滴数字聚合酶链式反应(ddPCR)是近年来新兴的一种靶基因绝对定量实验技术,该方法不依赖于Ct值和标准曲线就可以实现对核酸分子数目的绝对定量,检测灵敏度高,理论上可检出20 μL反应体系中的一个目的DNA分子[10]。分液技术可稀释背景序列干扰,结果判读时仅判断“有”或“无”2 种扩增状态,因此ddPCR对PCR反应抑制剂的耐受能力大大提高[11]。ddPCR技术在检测低拷贝基因时有优势,特别适合定量检测复杂环境样品中微量/痕量目的基因,目前已应用于医疗、食品、养殖业、环境等领域[12-15],尚无应用于固碳微生物检测的文献。建立了以cbbL基因为检测靶标、绝对定量土壤固碳微生物的ddPCR技术,为土壤固碳微生物的测定及碳汇能力评估提供技术支撑。
1 材料与方法
1.1 实验仪器与试剂
(1)实验仪器。PCR仪(BioRad T100),定量PCR仪(BioRad CFX96),ddPCR 系统(BioRad QX200),台式高速离心机(Thermo Pico21),生物样品均质仪(MP FastPrep24-5G),单道可调微量移液器(Eppendorf,Rainin),超微量紫外/可见光分光光度计(NanoDrop2000),水浴锅,恒温摇床,隔水式恒温培养箱。
(2)主要试剂。土壤DNA提取试剂盒(DNeasy®PowerSoil Pro Kit)购自Qiagen 公司;ddPCR Supermix for Probes(No dUTP)、微滴生成油和微滴读取油均购自Bio-Rad公司;Premix Ex TaqTM(Probe qPCR)购自TaKaRa公司;引物、探针由生工生物工程(上海)股份有限公司合成。
1.2 引物、探针的选择
通过文献资料调研,以编码卡尔文循环关键酶大亚基的cbbL基因表征固碳微生物,选择引物cbbLR1F(5′-AAG GAY GAC GAG AAC ATC-3′)、cbbLR1intR(5′-TGC AGS ATC ATG TCR TT-3′)以及探针cbbLRpro(5′-CAT GCA YTG GCG CGA CCG-3′),其中,S = C/G,R = A/G,Y = C/T,扩增产物大小约275 bp[16]。
1.3 土壤总DNA提取
土壤样品充分混合,称取0.25 g 土壤,采用Qiagen公司的DNeasy® PowerSoil Pro Kit试剂盒按操作说明提取DNA,最后溶于60 μL无菌水,-20 °C 保存备用。
1.4 含cbbL基因质粒的构建
用cbbLR1F和cbbLR1intR 扩增提取得到的土壤样品DNA,扩增条件为:95 ℃、2 min;95 ℃、15 s,55℃、30 s,72 ℃、30 s,35 个循环。PCR 产物连接pMD18T载体,转化大肠杆菌感受态细胞,涂布于含60 μg/mL氨苄青霉素的LB 固体平板上,37 ℃倒置培养过夜。筛选阳性转化子并测序,序列比对正确的转化子保存菌种并提取质粒,获得阳性质粒,命名为pMDcbbL,NanoDrop2000 超微量紫外/可见光分光光度计测定质粒浓度。
1.5 定量PCR检测引物与探针的适用性
将pMD-cbbL质粒的质量浓度根据分子量换算成拷贝数浓度2.31010copies/μL,10 倍稀释,取拷贝数浓度2.3100~2.3106copies/μL的pMD-cbbL为模板,以引物cbbLR1F、cbbLR1intR及探针cbbLRpro进行实时荧光定量PCR实验,以评估引物探针的适用性。实时荧光定量PCR 反应程序为:95 ℃、5 min;95 ℃、15 s;60 ℃、1 min(收集荧光),40 个循环。
1.6 ddPCR实验条件的确定
(1)ddPCR检测cbbL基因。参考BioRad 公司的ddPCR操作使用指南配制反应体系(见表1)。混合均匀,离心除气泡。将微滴生成卡DG8 放入支架中,用Rainin的20 μL移液器和20 μL吸头吸取20 μL反应液并加入DG8 中间一排孔中,在DG8 下面一排孔中分别加入70 μL微滴生成油,盖上专用胶垫,放入微滴生成仪生成微滴。待微滴生成后,在微滴发生卡的上排能看见云雾状的溶液,这就是生成的油包水微滴。缓慢吸取微滴(约40 μL),转移到专用的96 孔板中。盖上铝膜,用预热好的封膜仪180 °C、5 s 封膜,放入PCR 仪扩增,扩增程序如表1 所示,全程升降温速率均小于等于2 °C/s。扩增完成后,读取微滴信号,用QuantaSoft软件分析数据。

表1 ddPCR反应体系和反应程序
(2)ddPCR反应条件的优化。选用pMD-cbbL质粒为模板,对ddPCR 反应条件进行优化,包括退火温度、探针浓度和引物浓度。优化的关键因素是最大限度地提高阴性和阳性微滴分区之间的荧光振幅,并最小化具有中等荧光强度的微滴数量。退火温度设为62.0、61.2、60.0、58.1、55.8、53.9、52.7、52.0 ℃。在优化退火温度的基础上,设置4 组探针浓度进行ddPCR 反应,分别为200、250、300、350 nmol/L,每组设3 个重复。最优退火温度和探针浓度确定后,设定上下游引物终浓度600、750、900 nmol/L 进行ddPCR 反应,每组设3 个重复。
(3)线性范围和灵敏度实验。以pMD-cbbL 质粒为标准品,10 倍稀释,取拷贝数浓度2.3 ×100~2.3 ×106copies/μL 的pMD-cbbL 为模板,采用优化的退火温度、引物浓度和探针浓度进行实验,评估ddPCR 检测cbbL基因的线性范围和检测灵敏度。
(4)重复性实验。将提取的湿地土壤稀释10 倍后作为模板,以优化的反应条件检测cbbL基因拷贝数,设置21 个重复,计算变异系数,以评估该方法的可重复性。
(5)特异性检测。以优化的反应条件检测pMDcbbL质粒、湿地土壤、大肠杆菌、产碱芽孢杆菌、产气肠杆菌、真菌18SrDNA,以双蒸水代替模板为空白对照,评估ddPCR方法的特异性。
2 结果与分析
2.1 引物与探针的适用性
用引物cbbLR1F、cbbLR1intR及探针cbbLRpro 实时荧光定量PCR 检测质粒pMD-cbbL,结果如图1 所示。从图1 可以看出,10 倍稀释的质粒样品间呈现明显的线性关系,R2= 0.996,扩增效率为94.2%,说明实验中选用的引物和探针能有效检测cbbL基因。
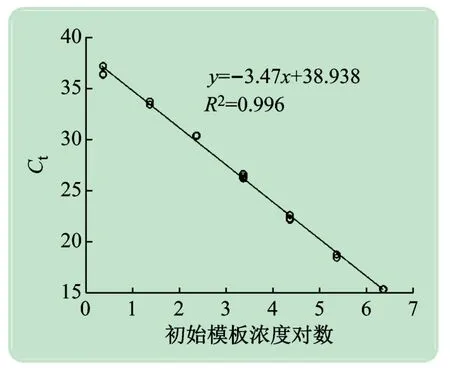
图1 定量PCR检测cbbL基因的标准曲线
2.2 ddPCR测定cbbL 基因方法的建立和反应条件优化
(1)ddPCR 方法的微滴数量控制。ddPCR 的数据分析是用QuanatSoft软件统计阴阳性微滴数,计算阳性微滴的占比p,由泊松分布计算出每个微滴中所含有的靶基因拷贝数为-ln(1 -p),再根据理论微滴总数计算出靶基因总拷贝数[10]。为了获得可靠数据,一般微滴总数需超过10 000,微滴总数低于10 000 的结果舍弃。本文中涉及的所有实验,扩增的微滴总数均达到10 000 个以上,说明实验结果均成立,后续不再赘述。图2 所示为梯度稀释实验中16 个反应(编号1 ~16)的微滴总数。
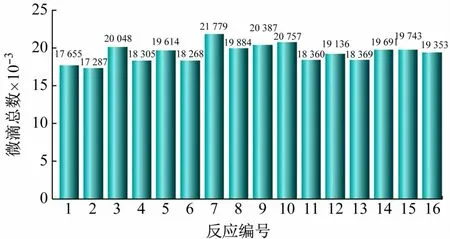
图2 ddPCR检测cbbL基因生成的微滴总数
(2)ddPCR反应条件的优化。不同退火温度扩增结果如表2、图3 所示。各温度间测得的质粒分子数有差异,其中52.7 ℃测定的质粒分子数最大,其次是55.8 ℃。PCR 实验中,退火温度越高,反应的特异性越好。当温度为55.8 ℃时,荧光振幅最大,阳性微滴和阴性微滴分布界限最为明显。因此,确定反应的最佳退火温度为55.8 ℃。
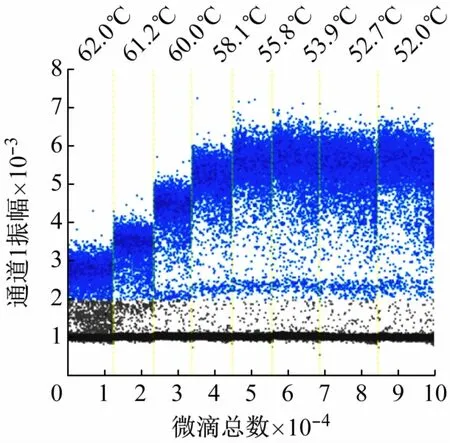
图3 退火温度优化cbbL-ddPCR测定结果
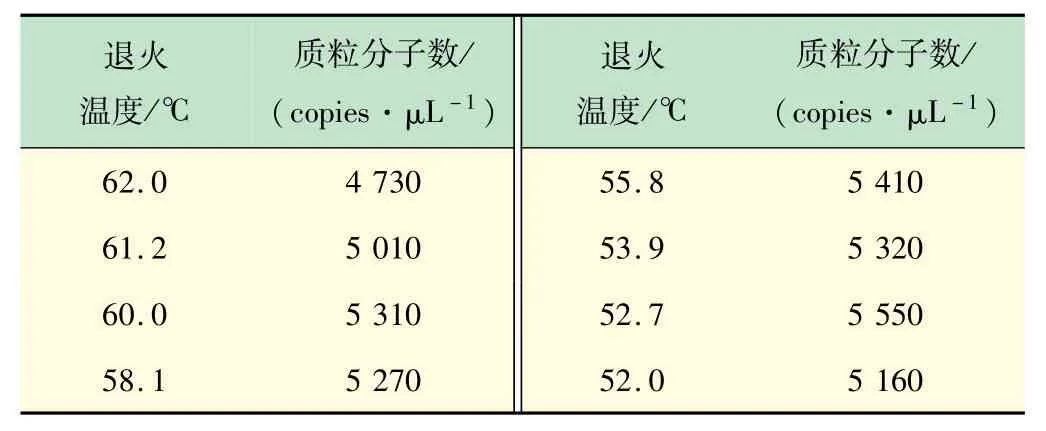
表2 不同退火温度下ddPCR检测的质粒分子数
设置A05 ~C05、D05 ~F05、G05 ~A06、B06 ~D06的探针浓度分别为200、250、300、350 nmol/L,cbbLddPCR测定结果如图4 所示。随着探针浓度的增加,ddPCR测定的质粒浓度逐渐增加,但不同探针浓度测得的质粒拷贝数差异不显著(P>0.05)。200、250、300、350 nmol/L 探针浓度下,ddPCR 检测的质粒分子数分别为(618. 00 ± 24.17)、(627. 33 ± 22.17)、(629.33 ±50.76)、(634. 67 ±14.08)copies/μL。当探针浓度为350 nmol/L 时,荧光振幅最高,阴阳微滴区分最为明显。因此,确定反应的最佳探针浓度为350 nmol/L。
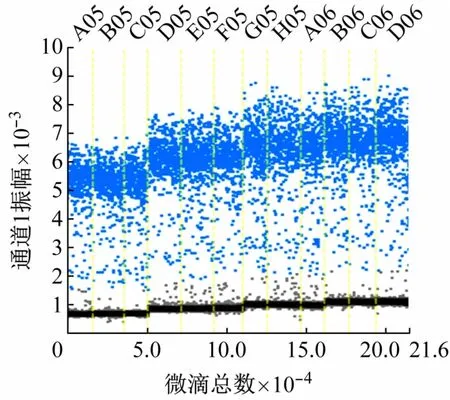
图4 探针浓度优化cbbL-ddPCR测定结果
当探针浓度为350 nmol/L 时,设置A07 ~C07、D07 ~F07、G07 ~A08 的引物浓度分别为900、750、600 nmol/L,cbbL-ddPCR 测定结果如图5 所示。可见,在所选定的引物浓度范围内,随着引物浓度降低,ddPCR测定的质粒浓度逐渐增加,但差异不显著(P>0.05)。900、750、600 nmol/L引物浓度下,ddPCR 检测的质粒分子数分别为(604.67 ±10.87)、(614.67 ±41.71)、(644. 67 ± 21.97)copies/μL。当引物浓度为600 nmol/L时,测得的质粒分子数最高,但荧光振幅低于900、750 nmol/L时。当引物浓度为750 nmol/L时,测得的质粒浓度较高,荧光振幅最高,阳性微滴和阴性微滴区分最为明显。因此,确定反应的最佳引物浓度为750 nmol/L。
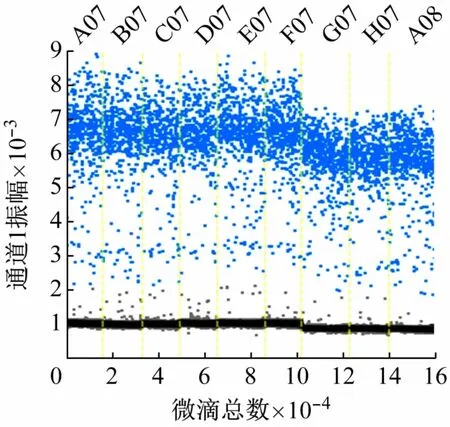
图5 引物浓度优化cbbL-ddPCR测定结果
2.3 ddPCR方法的线性范围和灵敏度
以拷贝数浓度为2.3 ×100~2.3 ×106copies/μL的pMD-cbbL质粒为模板,每个浓度设置3 个平行实验,用优化后的反应条件进行ddPCR扩增,结果如图6所示。从图6(a)可以看出,拷贝数浓度为2.3 ×106copies/μL的样品,所有微滴都显示为阳性微滴,无阴性微滴,检测结果不可靠,舍去。由图6(b)可见,其余6 个稀释度的质粒浓度与ddPCR检测的拷贝数呈线性关系,曲线方程为y= 0.107 7x- 95.562,R2=0.999 7,线性关系良好,说明建立的cbbL-ddPCR 检测方法定量结果可信。该方法的最高定量限为2.3 ×105copies/μL-DNA,最低定量限为2.3 copies/μLDNA。ddPCR实验灵敏度极高,在20 μL 反应液中只要有1 个cbbL基因分子该技术就能检测出来,实验中20 μL 反应液中加入2 μL DNA模板,所以该方法的检出限或者灵敏度为0.5 copy/μL-DNA。
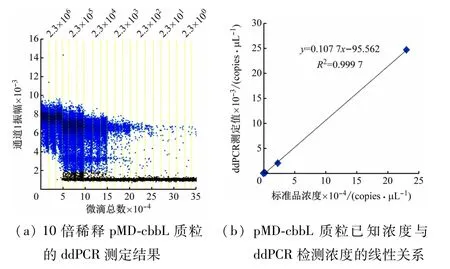
图6 ddPCR检测cbbL基因的线性范围
2.4 ddPCR方法的重复性
以优化的反应条件检测稀释10 倍的湿地土壤DNA的cbbL基因拷贝数,设置21 个重复(编号1 ~21)。结果显示,21 个重复的cbbL基因拷贝数浓度范围为2 080 ~2 380 copies/μL,组内变异系数为3.92%,说明建立的cbbL-ddPCR检测方法具有极好的重复性(见表3、图7)。
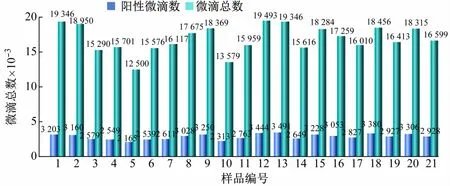
图7 ddPCR方法的组内重复性实验
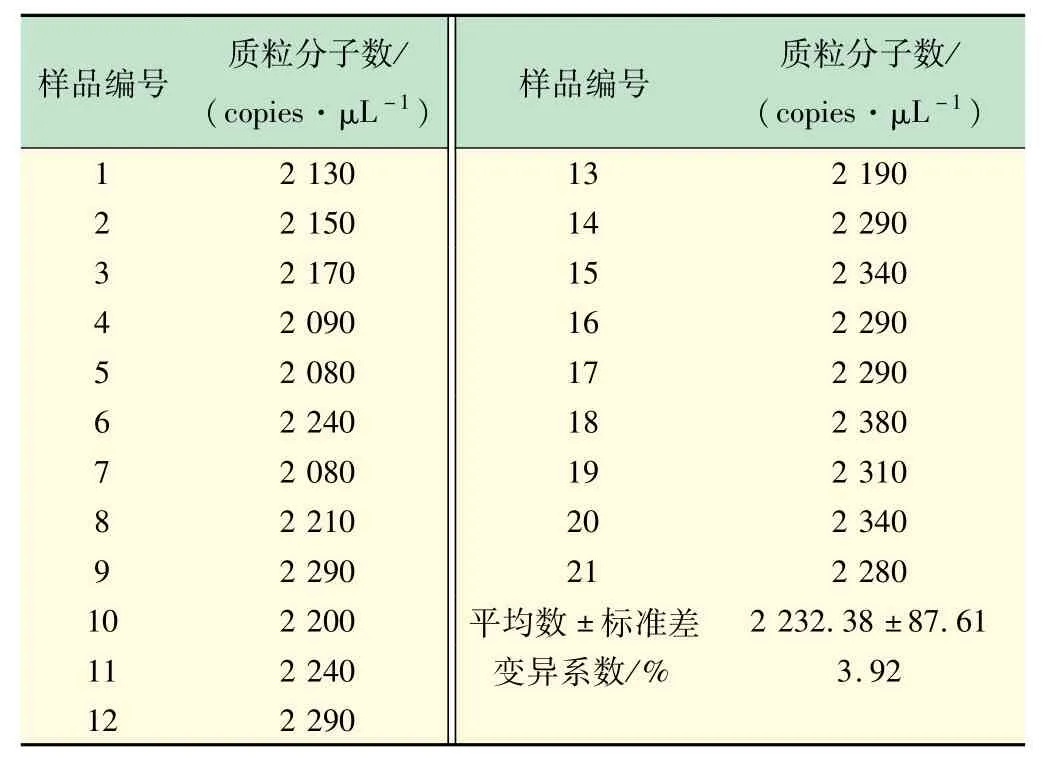
表3 ddPCR方法检测湿地土壤DNA的重复性实验
2.5 ddPCR方法的特异性
用建立的cbbL-ddPCR方法分别对大肠杆菌、产碱芽孢杆菌、产气肠杆菌和真菌18SrDNA 进行检测,测试该方法的特异性。结果显示,除了pMD-cbbL 质粒和湿地土壤样本出现了阳性微滴(分别为5 967 和5 238 个),其他4 个DNA样本和空白对照均未出现阳性微滴,说明建立的ddPCR 检测方法对含有cbbL基因的固碳微生物具有较好的特异性(见图8)。
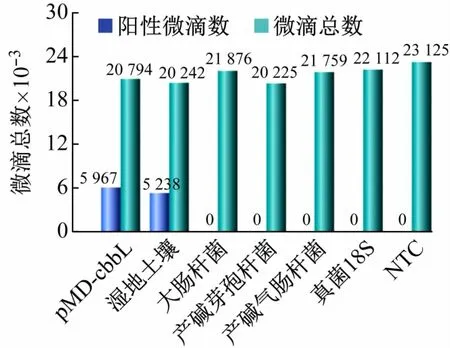
图8 ddPCR方法的特异性实验
3 结 语
建立了以cbbL基因为靶标的固碳微生物ddPCR检测方法。通过优化得到最佳退火温度为55.8 ℃,最佳引物与探针浓度分别为750 nmol/L和350 nmol/L。测试结果表明,该方法灵敏度和重复性高、特异性好,实现了真正意义上的绝对定量,可用于测定土壤固碳微生物数量,从而评估土壤碳汇能力。

