小麦及其祖先物种GRF转录因子家族鉴定与表达分析
王添宁 冯雅岚 琚吉浩 吴 毅 张 均 马 超,*
小麦及其祖先物种GRF转录因子家族鉴定与表达分析
王添宁1冯雅岚2琚吉浩1吴 毅1张 均1马 超1,*
1河南科技大学农学院, 河南洛阳 471000;2武昌理工学院生命科学学院, 湖北武汉 430000
生长调控因子(growth-regulating factor, GRF)在植物的生长发育、逆境响应和激素信号转导中起着重要的调控作用。系统分析小麦及其祖先物种GRF转录因子家族成员在基因组上的分布、结构、进化以及表达特性, 对于深入研究GRF家族的生物学功能和小麦的进化具有重要的意义。本研究利用生物信息学方法, 对乌拉尔图小麦、拟斯卑尔脱山羊草、粗山羊草、栽培二粒小麦和普通小麦5个物种的GRF成员进行全基因组鉴定, 并对其理化性质、系统发育关系、基因结构、启动子顺式作用元件以及表达特性进行了分析。结果表明, 乌拉尔图小麦、拟斯卑尔脱山羊草、粗山羊草、栽培二粒小麦和普通小麦中分别有15、12、19、29和53个GRF成员, 通过种间共线性分析发现,分别有18个和29个成员与和具有共线性,分别有36个和37个成员与和具有共线性。启动子顺式作用元件预测发现基因具有基本的转录元件以及一些与生长发育和逆境响应的结合元件。RT-qPCR分析表明, 多数基因在外源IAA、GA和干旱胁迫条件下呈上调表达趋势, 而在高温胁迫条件下呈下调表达趋势, 表明GRF家族成员在响应激素和逆境胁迫中有重要作用。系统进化分析表明, 小麦与其祖先物种之间的GRF成员存在保守且复杂的进化关系。上述结果为GRF转录因子家族的进化及其功能研究提供了理论基础。
小麦; 祖先物种; GRF; 生物信息学; 进化分析
转录因子是一类与RNA聚合酶II形成转录起始复合体, 从而参与调控基因表达的一大类基因, 在生物的生长发育、代谢繁殖、细胞分化和胁迫响应等多种生物学过程中起重要作用[1-3]。人们目前已经在植物中发现多种转录因子家族, 其中生长调控因子(growth-regulating factor, GRF)是植物所特有的一类转录因子, 并在其生长发育中起着重要的调控作用[4-5]。基因首次在水稻中被鉴定到, 命名为, 并发现该基因编码一种可以参与赤霉素信号通路从而诱导水稻茎伸长的蛋白质[6]。近年来, 随着参考基因组的不断发展, GRF转录因子家族在越来越多的物种被鉴定, 其中包括已经在拟南芥中鉴定到的9个成员[7], 在玉米中鉴定到的14个成员[8], 以及在水稻中鉴定到的12个成员[9]。
GRF转录因子的N端区域中包含QLQ (Gln、Leu、Gln)和WRC (Trp、Arg、Cys)两个保守结构域[10-12]。QLQ结构域具有蛋白互作特性, 可以与GRF互作因子GIF (GRF interacting factors)相互作用形成转录激活因子[13]; WRC结构域包含一个核定位信号NLS (nuclear localization signal)区域和一个DNA结合基序(锌指结构), 可以与其靶基因的顺式作用元件结合并调节下游基因的表达[14]。基因在幼嫩的植物组织中高度表达, 如: 根尖、花芽和幼叶等, 而在成熟的组织中表达相对较弱[15-16]。另外基因在植物组织或器官的形成过程以及对环境胁迫的应答反应中发挥着重要的调节作用, 例如有研究发现拟南芥突变体的植株矮化、叶片更小[7]; 水稻中表达被抑制也会导致植株矮化和花序发育延迟[17], 而的过量表达则会导致植株穗长、粒长和粒重的增加[18]。另外王平等在人参中鉴定到20个基因成员, 这些成员可能与赤霉素诱导以及响应高温胁迫密切相关[19]。还有研究发现基因在植物的氮代谢中起到重要作用, 大白菜中基因通过调控侧根数量和参与氮吸收及同化基因的表达, 加速植物生长和硝态氮同化[20]。近期有研究人员提出可以利用生长因子GRF和GIF、BBM来提高基因组编辑植物的再生效率[21]。也有较多的学者提出, miRNA396- GRF-GIF作用网络在作物育种和改良中具有潜在的应用价值[22-23]。
小麦()是由3个亚基因组(A、B、D)整合形成的异源六倍体(AABBDD)植物[24-25], 其具有庞大且复杂的基因组, 大小约为16.23 GB[26]。普通小麦是由二倍体植物乌拉尔图小麦()、拟斯卑尔脱山羊草()和粗山羊草()以及四倍体植物栽培二粒小麦()进化而来的[27-28]。由于小麦种植面积占世界谷类作物的三分之一, 是最重要的粮食作物之一。尽管GRF转录因子家族已经在玉米[8]、水稻[9]、大豆[29]和谷子[30]等多种植物中被鉴定, 但对于基因在小麦及其祖先物种间的基因扩展机制以及特定的进化关系还知之甚少。因此, 本研究利用生物信息学, 对小麦及其祖先物种GRF转录因子家族进行鉴定, 并对上述5个物种的系统发育关系、种内和种间差异进行比较分析, 旨在从物种进化的角度进一步深入研究小麦GRF转录因子的功能提供理论依据。
1 材料与方法
1.1 小麦及其祖先物种GRF转录因子家族成员鉴定
从NCBI数据库(https://www.ncbi.nlm.nih.gov/)上获取拟南芥、乌拉尔图小麦(, A基因组供体, GCF_003073215.2)、拟斯卑尔脱山羊草(, B基因组可能的供体, GCA_ 021437245.1)、粗山羊草(, D基因组供体, GCF_002575655.2)、栽培二粒小麦(, 染色体组型AABB, GCA_900231445.1)和普通小麦(, 染色体组型AABBDD, GCF_018294505.1)的基因组序列、氨基酸序列以及基因注释文件(gff文件)。以拟南芥AtGRF氨基酸序列为参考进行BLASTP比对(-value<1e–5)获得同源序列。登录Pfam数据库(http://pfam.xfam.org/)下载GRF家族的隐马尔科夫模型文件QLQ结构域(PF08880)和WRC结构域(PF08897), 利用HMM3.0进行保守结构域比对, 同时利用Pfam搜索、SMART(http://samrt.embl-heidelberg.de/)和HMMER (https://www.ebi.ac.uk/Tools/hmmer/search/hmscan)验证候选蛋白保守结构域。
1.2 小麦及其祖先物种GRF转录因子家族蛋白理化性质、蛋白结构和亚细胞定位分析
利用ExPASy-ProtParam (https://web.expasy.org/ protparam/)计算GRF蛋白的氨基酸长度和分子量等基本理化性质。利用软件ProtComp 9.0 (https://linux1. Softbe-rry.com/berry.phtml)预测GRF的亚细胞定位。在NCBI数据库中获取GRF基因组序列, 并利用网站NPS-SOPMA (https://npsaprabi.ibcp.fr/cgi-bin/secpred_sopma.pl)进行GRF的二级结构预测以及利用网站SWISS-MODEL (https://swissmodel.expasy.org/)进行GRF的三级结构预测。
1.3 小麦及其祖先物种GRF转录因子家族的系统进化分析
利用Clustal W工具对5个物种GRF蛋白序列进行多重比对, 利用MEGA11[31]工具采用邻接法 (Neighbor-Jointing, NJ)构建5个物种的系统发育进化树(bootstrap, 1000), 利用TBtools[32]软件进行可视化分析。使用贝叶斯方法, 利用MrBayes v3.1 (Ronquist和Huelsenbeck 2003)设置4链(chains, 4), 一百万代(ngen, 1,000,000), 每100代进行取样建树(samplefreq=100), 舍弃25万代运行程序[33]。
1.4 小麦及其祖先物种GRF转录因子家族的染色体定位、基因结构和保守结构域分析
在NCBI数据库中获取基因结构注释文件, 使用TBtools软件对其进行染色体定位和基因结构分析可视化, 利用MEME[34](https://meme-suite.org/ meme/)基于Motif分析家族成员序列保守特性, 并使用TBtools软件对其进行可视化。
1.5 小麦及其祖先物种GRF转录因子家族启动子顺式作用元件预测
在NCBI数据库中获取基因的核酸序列, 使用TBtools软件获取5个物种基因上游2000 bp的启动子序列, 提交至PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行启动子顺式作用元件预测, 并使用TBtools软件对其进行可视化分析。
1.6 小麦及其祖先物种GRF转录因子家族共线性分析和Ka/Ks分析
在NCBI数据库中获取5个物种基因序列, 利用MCScanX[35]进行共线性分析(-value<1e–5), Advance Circos进行可视化分析, 并利用MCScanX的默认参数分析栽培二粒小麦与乌拉尔图小麦、拟斯卑尔脱山羊草之间的共线性关系, 以及普通小麦与栽培二粒小麦、粗山羊草之间的共线性关系, 并获得了他们之间的共线性基因对。在NCBI数据库中获取基因的核酸序列, 并使用TBtools软件的a/sCalculator程序计算非同义替换(non- synonymous substitution,a)、同义替换(synonymous substitution,s)及a/s值, 进行选择压力分析。
1.7 小麦及其祖先物种GRF转录因子家族部分成员表达特性分析
选用乌拉尔图小麦(PI428198)、拟斯卑尔脱山羊草(611)、粗山羊草(HN2203)、栽培二粒小麦(PI79899)和普通小麦 (中国春)为试验材料, 选择籽粒饱满大小一致的种子, 经过蒸馏水冲洗后, 进行营养钵种植, 在室温下待种子发芽后置于光照培养箱中, 在22℃、光/暗周期为16 h/8 h的条件下培养。待其生长至二叶一心期时进行外源激素和逆境胁迫处理, 外源激素包括: 生长素(100 μmol L–1IAA, 叶面喷施50 mL)、赤霉素(100 μmol L–1GA, 叶面喷施50 ml)、脱落酸(100 μmol L–1ABA, 叶面喷施50 mL)、玉米素(100 μmol L–1ZT, 叶面喷施50 mL); 逆境胁迫包括: 盐胁迫(200 mmol L–1NaCl, 浇灌100 mL)、低温(培养箱4℃)、高温 (培养箱38℃)、干旱(20% PEG-6000, 浇灌100 mL)处理, 以正常生长为对照(CK), 所有处理重复3次。在处理后12 h, 分别取不同物种及不同处理的幼叶, 立即置于液氮保存, 用于实时荧光定量PCR (Quantitative Real-time PCR, RT-qPCR)分析。使用TRIZOL (TaKaRa, 9108Q)试剂提取RNA, 反转录成cDNA (TaKaRa, RR047Q)。利用软件Primer Premier 5设计引物,()和()为内参基因, 各基因引物序列(见表1)。其中反应体系与程序参考ChamQ Universal SYBR qPCR masier Mix (Vazyme)试剂盒说明书, 采用2–ΔΔCt法计算相对表达量。
2 结果与分析
2.1 小麦及其祖先物种GRF转录因子家族成员鉴定
根据拟南芥GRF的蛋白序列, 利用BLASTP和HMM进行比对搜索, 在5个物种中总共得到342个GRF家族成员, 通过合并去重后得到209个GRF候选成员, 利用NCBI CD-search去除结构域不完整成员, 最终5个物种共获得128个GRF家族成员, 其中乌拉尔图小麦、拟斯卑尔脱山羊草、粗山羊草、栽培二粒小麦和普通小麦中分别有15、12、19、29和53个GRF家族成员。根据NCBI基因注释将其命名为“物种名-基因编号-染色体位置”。使用TBtools软件对5个物种GRF蛋白的理化性质进行分析发现(附表1), 5个物种GRF蛋白序列长度为107~627个氨基酸, 分子量为12.19~67.05 kD。GRF蛋白理论等电点在4.25~10.83, 其中40个位于酸性位置, 1个位于中性位置, 其余均位于碱性位置。蛋白质不稳定指数在39.77~77.10, 其中只有TuGRF8- UN (XM_048695887.1)不稳定指数小于40, 属于稳定蛋白。脂肪系数范围为44.09~87.25。平均疏水指数范围为–0.983~ –0.274, 依据平均疏水指数是正值为疏水蛋白、负值为亲水蛋白、介于–0.5~0.5之间为两性蛋白的原则, 其中有33个是两性蛋白, 其他为亲水蛋白。
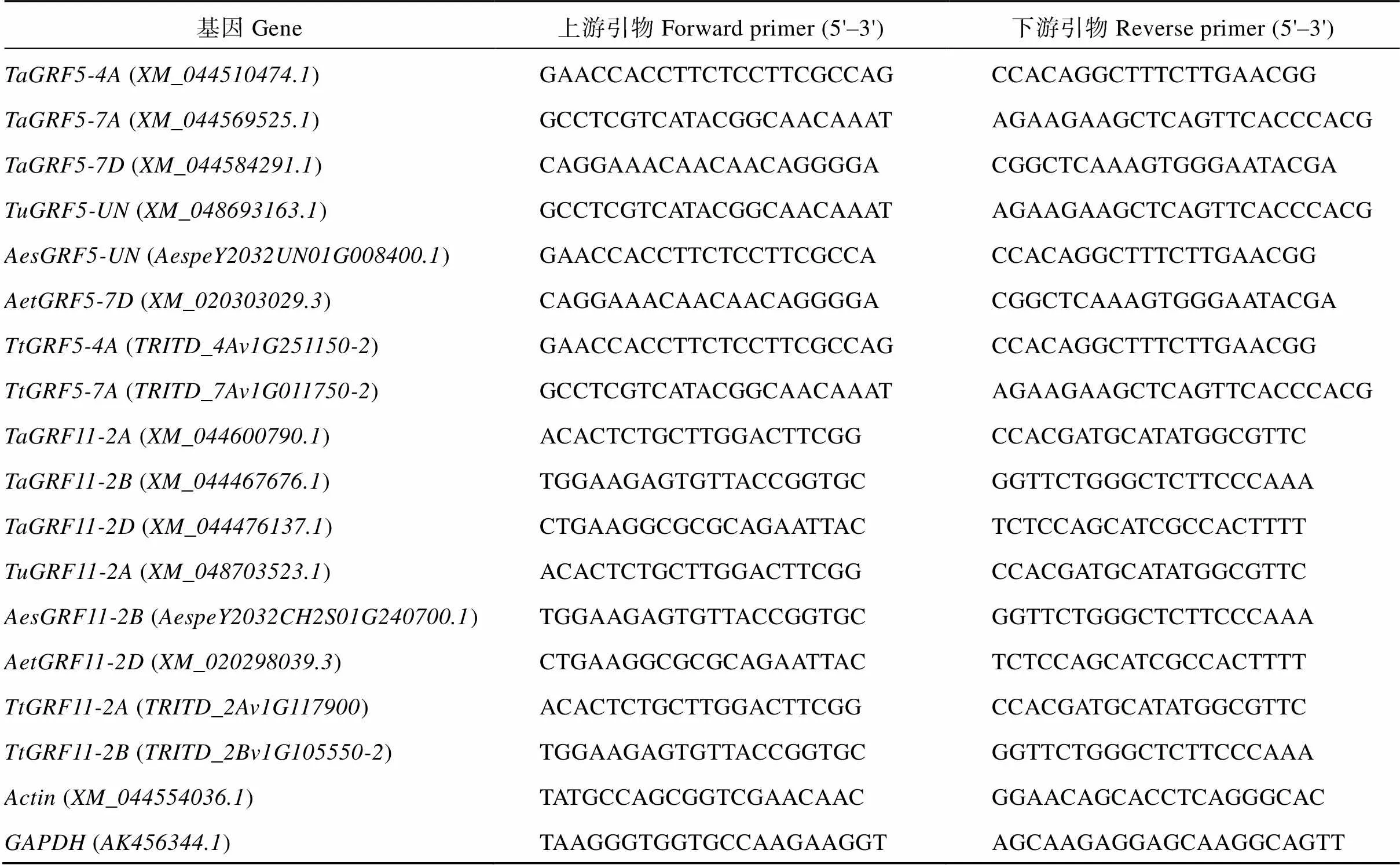
表1 本试验所用引物
2.2 小麦及其祖先物种GRF转录因子家族蛋白二级结构、三级结构和亚细胞定位分析
对5个物种GRF蛋白的二级结构进行预测发现(附表2), 128个家族成员的二级结构中均含有α-螺旋、β-转角、延伸链和无规则卷曲。其中α-螺旋和无规则卷曲是GRF家族成员二级结构的主要构成元件, 其利于蛋白质特殊构象的形成。利用SWISS- MODEL对5个物种GRF5蛋白的三级结构进行预测,发现结果与二级结构相符, 如图1所示。同一类GRF在不同物种中具有相似的三级结构, 例如GRF5, 栽培二粒小麦GRF5蛋白三级结构与其祖先物种拟斯卑尔脱山羊草和乌拉尔图小麦的三级结构存在部分相同, 另外在同号染色体上普通小麦与其祖先物种的GRF5蛋白三级结构相似, 这表明栽培二粒小麦和普通小麦蛋白的三级结构可能由其祖先物种进化而来。亚细胞定位预测表明(附表1), 5个物种的128个成员中有12个定位于叶绿体, 9个定位于线粒体, 2个定位于细胞液, 其他均定位于细胞核中。其中在定位于叶绿体或线粒体的21个成员中, 普通小麦有10个成员, 而其他每个物种只有2~4个成员, 这表明他们可能与叶绿体和线粒体的功能有关, 并且随着进化过程, 有更多的基因参与调控叶绿体或线粒体的功能。
2.3 小麦及其祖先物种GRF转录因子家族系统进化分析
为分析GRF家族成员在不同物种间的进化关系,利用MEGA 11软件和MrBayes v3.1采用邻接法和贝叶斯方法对5个物种的128个GRF家族成员构建系统发育进化树(图2)。基于基因的结构功能与系统发育的拓扑结构分析, 将GRF家族成员进一步划分为5组(I~V), 每组成员分布不均匀, 其成员数量分别为34、27、21、21和25个。除粗山羊草中超过三分之一的GRF家族成员和拟斯卑尔脱山羊草中的1个家族成员分布在第I组外, 其他物种均匀分布在I~V组。利用贝叶斯方法建立的系统发育树中, 树的分支长度表示进化距离, 从水平方向上分支的长度来看, 随时间变化, 分支越长物种中的基因变化就越大。从图中可以看出, III~V组的进化距离较长, 这些家族成员的进化时间较早。I和II组中存在15个家族成员进化较晚, 进化速率较慢。另外, 第III组的全部成员均位于6号和7号染色体上, 第V组的成员均位于2号和6号染色体上, 5个物种的GRF家族成员均没有在3号染色体上分布, 表明分布在同一组的基因可能存在进化关系, 在进化树II组中GRF3亚家族的进化距离存在比较远的情况, 这可能是成员在进化的过程中发生了较大的遗传变异,从而出现了2个分支; IV组中的GRF6亚家族与GRF3亚家族相同, 也出现了进化距离较大的2个分支; 其他的亚家族成员距离都比较近, 遗传变异较小。同时通过分组发现, 相同的亚家族成员在同一分支上, 几个亚家族又在一组里, 例如II组中包含GRF3、GRF4和GRF5亚家族, 这3个亚家族成员的进化距离较近, 而与其他亚家族距离较远。-()、-()和-() 3个成员之间属于直系同源关系, 这些亚家族的成员随着进化分支进入到不同的物种, 并保留了原有的序列信息。
2.4 小麦及其祖先物种GRF转录因子家族的染色体定位、基因结构和保守结构域分析
通过对5个物种基因的染色体定位发现, 5个物种在2号、4号和6号染色体上定位的基因较多, 分别含有28、24和35个成员, 在5号染色体上定位到的基因较少, 仅含有7个成员, 其中包含粗山羊草的3个家族成员和普通小麦的4个家族成员, 另外在3号染色体上没有定位到基因(图3)。66.4%基因的位置在进化的过程中较为保守,分别被定位在2号、4号和6号染色体上, 而在3号染色体上没有被定位到基因, 表明基因在进化过程中出现分布不均的情况, 并且随着小麦的进化被遗传了下来。同时在小麦的3个亚基因组(A、B和D)中发现, 在被鉴定到的亚家族中, A基因组有18个家族成员, 包含所有的GRF亚家族成员, B基因组有15个成员, 未鉴定到GRF5, D基因组有20个成员, 未鉴定到GRF9; 另外, GRF1、GRF4和GRF10亚家族成员只在6号的3个染色体组(6A、6B和6D)上各鉴定到1个, GRF11和GRF12只在2号的3个染色体组(2A、2B和2D)上各鉴定到1个。

图2 小麦及其祖先物种GRF家族成员的系统进化树
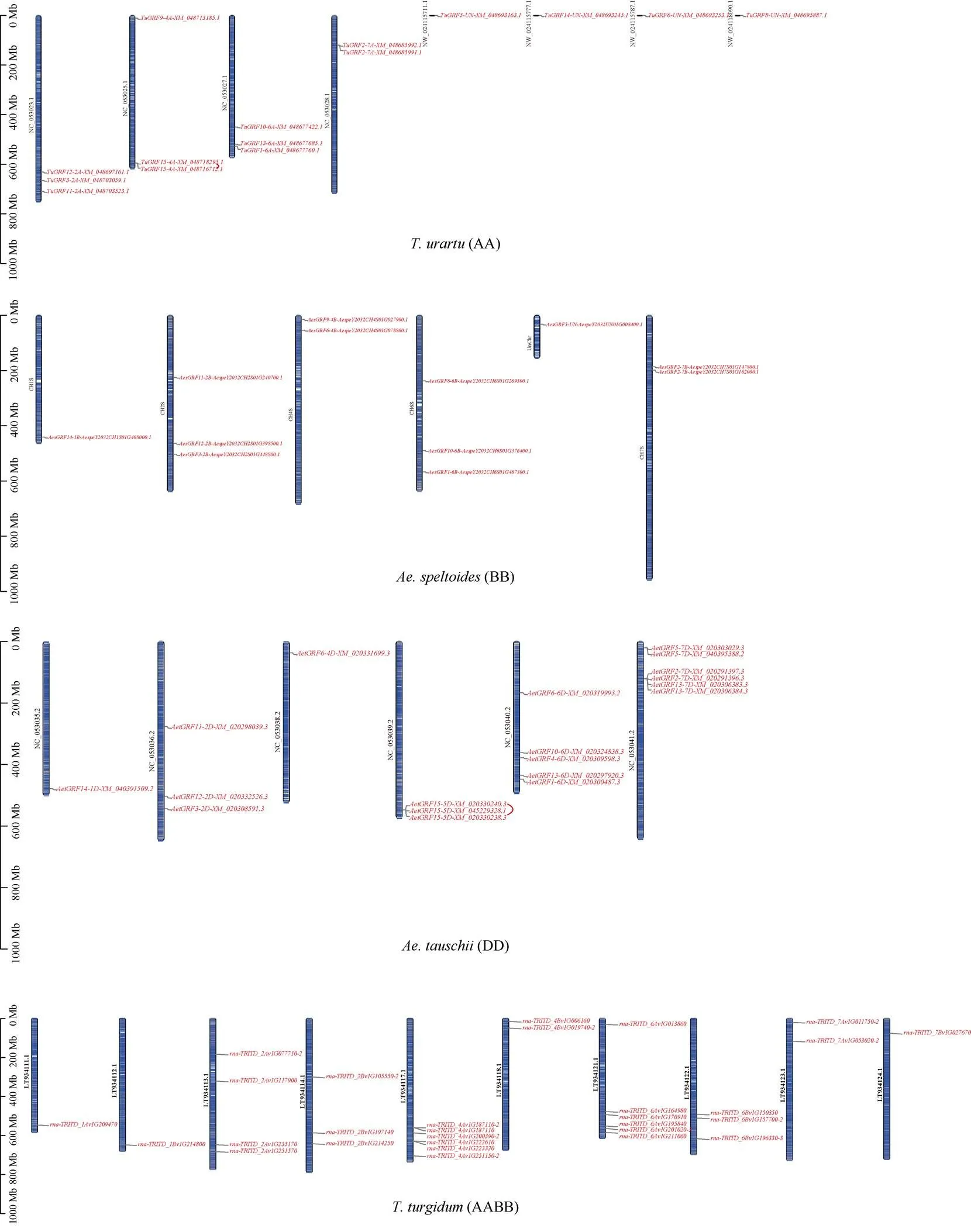
(图3)
利用MEME进行Motif预测(图4), 共鉴定出5个GRF蛋白基序(Motif1~Motif5), 长度分别在15~41个氨基酸之间。其中Motif1、2和3几乎在第II~V组的每个蛋白中均被鉴定到, 分别鉴定到88、92和75个蛋白, 而Motif4在第II、III组大多数蛋白中被鉴定到, 分别鉴定到22个和20个蛋白, Motif5在第V组的25个蛋白中被鉴定到, 另外第I组全部34个蛋白均鉴定到Motif1, 25个蛋白鉴定到Motif3, 仅有4个蛋白鉴定到Motif4。由此发现各个亚家族都有其独特的蛋白基序, 说明基因在进化过程中存在内部分化。
依据基因组注释文件, 获得了5个物种基因的外显子和内含子结构。由图4可知, 所有基因均具有完整的CDS, 并且所有成员均包含不同数量的外显子和内含子, 但在数量和位置上存在差异, 例如普通小麦基因()包含5个外显子, 而栽培二粒小麦基因()只有1个外显子, 表明不同成员之间可能存在功能的差异。另外不同基因存在保守的外显子和内含子结构, 表明他们之间可能具有较近的进化关系。只有93个基因含有上下游调控区 (UTR), 基因中UTR的缺失可能会影响基因的调控功能。而且在系统发育树上距离较近的成员间基因结构存在一定的保守性, 具有相同的外显子和内含子。
2.5 小麦及其祖先物种GRF转录因子家族启动子顺式作用元件预测
基因的表达调控与其启动子顺式作用元件关系密切, 对5个物种基因的启动子区段顺式作用元件进行预测发现(图5), 其上游2 kb区域的启动子顺式作用元件主要包括光响应元件Box 4、AE-box、ACE和Sp1, 逆境响应元件如参与低温反应的LTR和参与干旱反应的MBS, 激素响应元件如赤霉素反应元件P-box和GARE-motif、脱落酸反应元件ABRE和茉莉酸反应元件CGTCA-motif, 以及其他与生长调控相关的元件如厌氧诱导必需元件ARE、生物及非生物胁迫响应元件as-1等。在5个物种的全部128个家族成员中,()含有最少的启动子元件结合位点, 仅含1个(Sp1), 而()含有最多的启动子元件结合位点, 含有24个。基因有较多的关于赤霉素、生长素以及参与干旱胁迫的反应元件结合位点, 并且相同基因类别具有极其相似的元件结合位点, 这说明在不同物种中同类别基因可能参与调控相同的逆境反应, 但又存在一些差别, 表明在进化过程中基因的调控模式被遗传了下来,基因的功能较为保守, 但由于时间的推移, 在进化过程中可能有环境等因素的差异, 导致其调控模式并不完全相同, 例如在GRF2亚家族中, 只有拟斯卑尔脱山羊草中存在AE-box启动子顺式作用元件, 这可能由于在进化中该元件并未遗传下来, 而ACE、ARE、as-1和MBS元件在5个物种中都存在, 证明GRF2家族成员的这些功能在进化过程中得到了很好的保留。

图4 小麦及其祖先物种GRF基因结构和保守结构域分析

图5 小麦及其祖先物种GRF基因启动子顺式作用元件
2.6 小麦及其祖先物种GRF转录因子家族共线性分析
为明确基因在不同物种间的扩展规律, 通过MCScanX对基因家族进行共线性分析(图6), 结果显示了5个物种中分别存在618、4449、1668、24,996、76,238个共线性区域(包含UN染色体), 这些区域是通过片段复制而来, 同时也表明在进化过程中所出现的多倍体化的特点, 5个物种中分别有2、3、3、20和65个基因定位在共线性区域内, 这些片段复制是在进化过程中基因家族扩张的主要途径。图中最外一圈表示这5个物种中的基因密度普遍较大, 而图中的最内圈表示染色体未知碱基的分布, 在乌拉尔图小麦、拟斯卑尔脱山羊草和粗山羊草的染色体上存在较多的未知碱基, 这与中间环带中线的波动相印证。并且同一GRF家族的不同亚基因组成员之间都存在着染色体共线性, 且不同亚基因组的基因在进化树上距离较近, 并聚类在同一分支中, 它们来源于同一物种中的染色体片段复制, 属于旁系同源基因对, 例如GRF12亚家族在不同亚基因组的成员()、()和()之间都存在着共线性, 且分布在系统发育树的同一分支上。
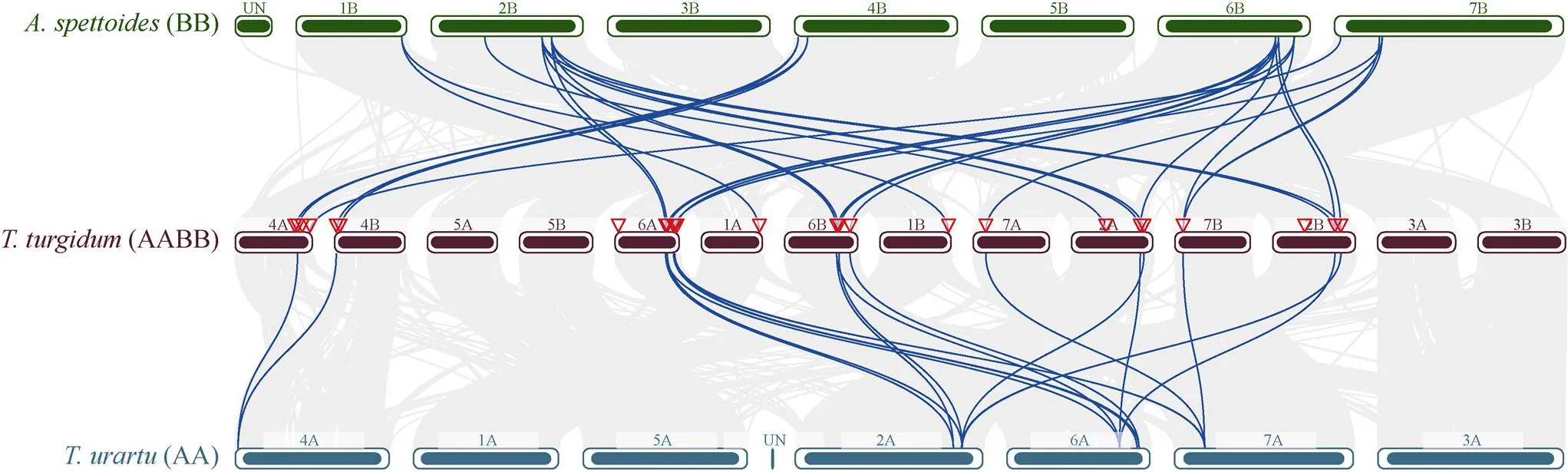
随物种进化方向, 分别对乌拉尔图小麦、拟斯卑尔脱山羊草与栽培二粒小麦以及栽培二粒小麦、粗山羊草与普通小麦进行基因组共线性分析(图7), 并且对共线性的基因进行选择压力分析(附表3)。发现分别有18个和29个基因与和具有共线性, 分别有36个和37个基因与和具有共线性。不同物种间的共线性表明这些成员在物种间的进化过程中得到了保留与复制。同时这些基因的片段复制使得普通小麦中具有了更丰富的家族成员, 也使得小麦中GRF家族的功能更加丰富, 有利于植物自身的生长。另外发现有1个基因对a/s比率大于1, 认为基因在进化过程中受到正选择效应, 可加速基因进化; 其他基因的a/s比率均小于1, 认为基因经过了纯化选择, 而且受到正向选择的成员其蛋白质可能已经在进化中发生了改变。
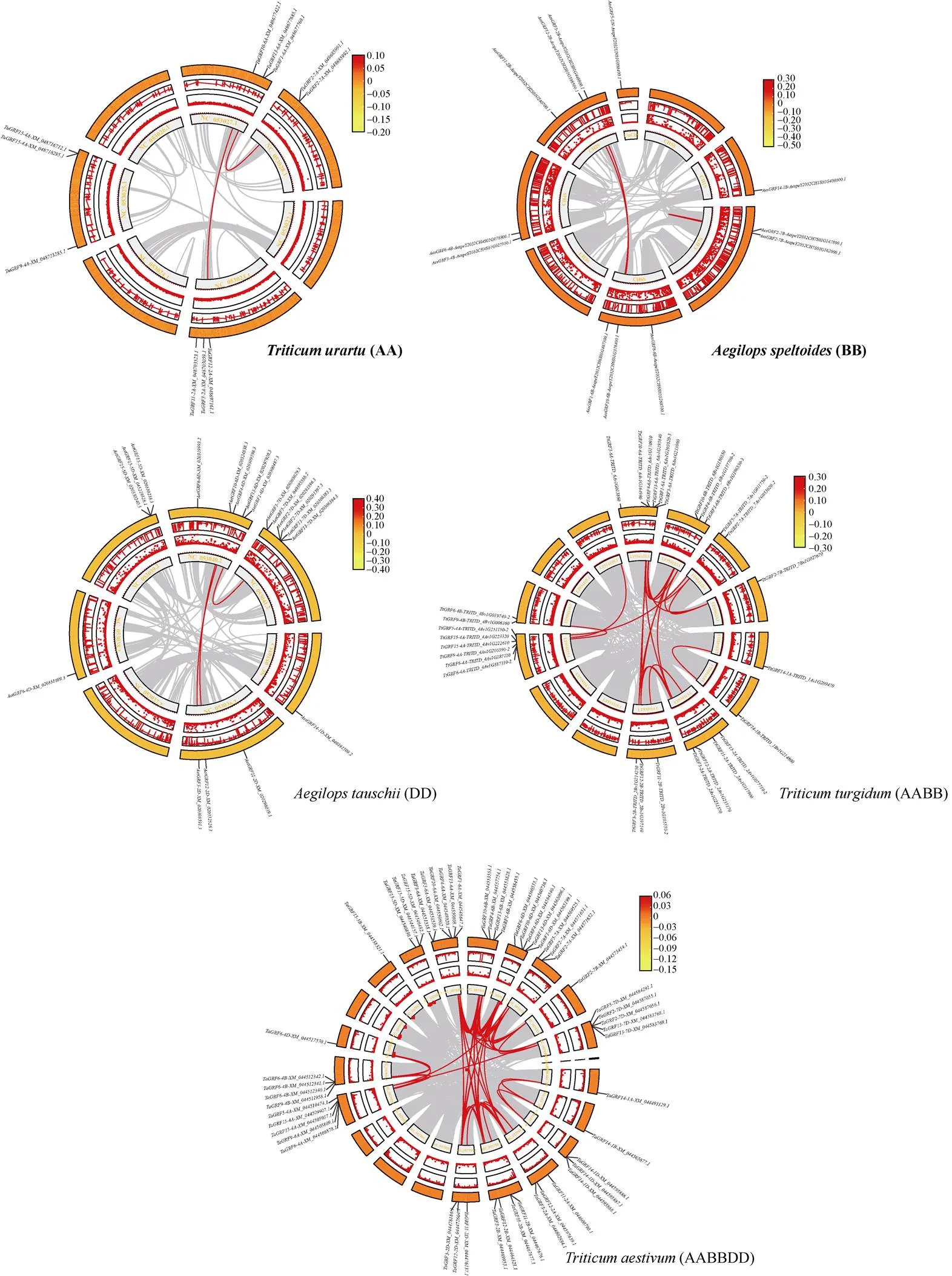
图6 小麦及其祖先物种GRF基因的染色体位置及共线性关系
所有共线性区域和基因由灰线连接, 红线表示基因的片段重复基因对。
All syntenic blocks and genes are linked by the grey lines, and red lines represent the fragment-repeat gene pair of thegenes.
2.7 小麦及其祖先物种GRF转录因子家族表达模式分析
为研究基因在5个物种中对不同外源激素和非生物胁迫的响应, 采用RT-qPCR的方法分析了16个基因在转录水平的表达模式。由图9可知, 在4种激素处理下基因表达量在不同物种间呈现出不同的变化规律。在外源IAA处理下, 基因-、和表达量显著上调, 其中这3个基因表达量分别较CK上调0.7、1.5和6.9倍, 乌拉尔图小麦、普通小麦和栽培二粒小麦-较CK显著下调。在外源ABA处理下, 普通小麦、和以及栽培二粒小麦、和表达量显著上调, 较CK分别上调13.2、5.7和7.5倍以及14.5、29.0和1.7倍, 发现普通小麦和栽培二粒小麦基因对于ABA激素的响应较为强烈。而、、、和这5个基因表达量则显著下调。根据启动子顺式作用元件分析, 在上调和下调的基因中均发现有关于ABA诱导的ABRE作用元件, 猜测这可能与基因的功能不同有关。在外源GA处理下,中除、和表达量略有降低外, 其他基因均呈不同程度的上调表达, 其中上调倍数最大的前3个基因分别是、和, 分别上调15.7、5.6和4.1倍, 而中除、和外, 其他基因均呈不同程度的下调表达, 其中下调倍数最大的前3个基因分别是、和,分别下调1.0、1.0和0.8倍。根据启动子顺式作用元件分析, 上调的基因中有P-box和GARE两个GA响应元件, 而下调基因中没有发现。在外源ZT处理下,、、、、和表达量均呈显著上调变化, 其中上调最为明显, 其表达量较CK上调8.3倍(图8)。
在逆境胁迫条件下(图9), 从整体上看16个基因对于干旱胁迫均有不同的表达趋势, 其中栽培二粒小麦和上调最为显著, 其表达量较CK上调7.0倍和1.2倍, 发现其中都包含有MBS启动子顺式作用元件, 这可能是导致表达量上调的主要因素。在盐胁迫条件下, 基因、、、和呈现出上调的表达趋势, 其余均有不同程度的下调表达趋势, 其中普通小麦下调最为显著, 其表达量较CK下调0.7倍。在高温胁迫下, 除、、和的表达量略有上调外, 其余均有不同程度的下调, 其中下调最为显著, 其表达量较CK下调0.9倍。在低温胁迫下, 整体上16个基因呈现出上调的表达规律, 其中上调倍数最大的前3个基因分别是、和, 分别上调10.9、4.6和3.6倍, 下调倍数最大的前3个基因分别是、和, 分别下调0.9、0.5和0.4倍。在这些基因中都含有LTR顺式作用元件, 因此表达量下调不显著可能与其有关。这些基因在不同的物种中表达趋势存在一定的区别, 这可能是与其在不同物种中表达模式不同有关。
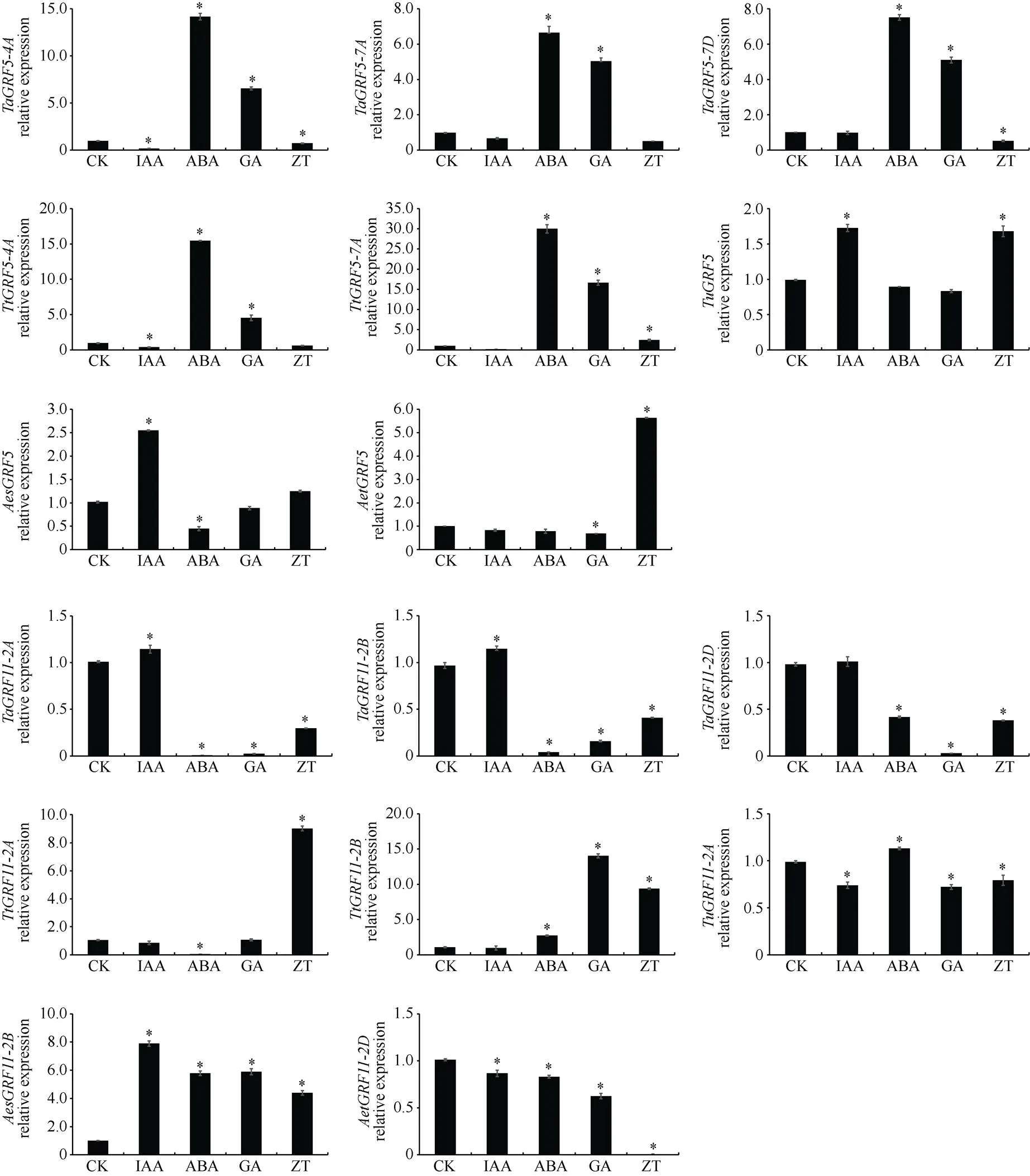
图8 16个GRF基因对外源激素响应的表达模式分析
*表示处理间存在显著差异(< 0.05)。
* indicates significant differences between the stress conditions and the control condition at< 0.05.
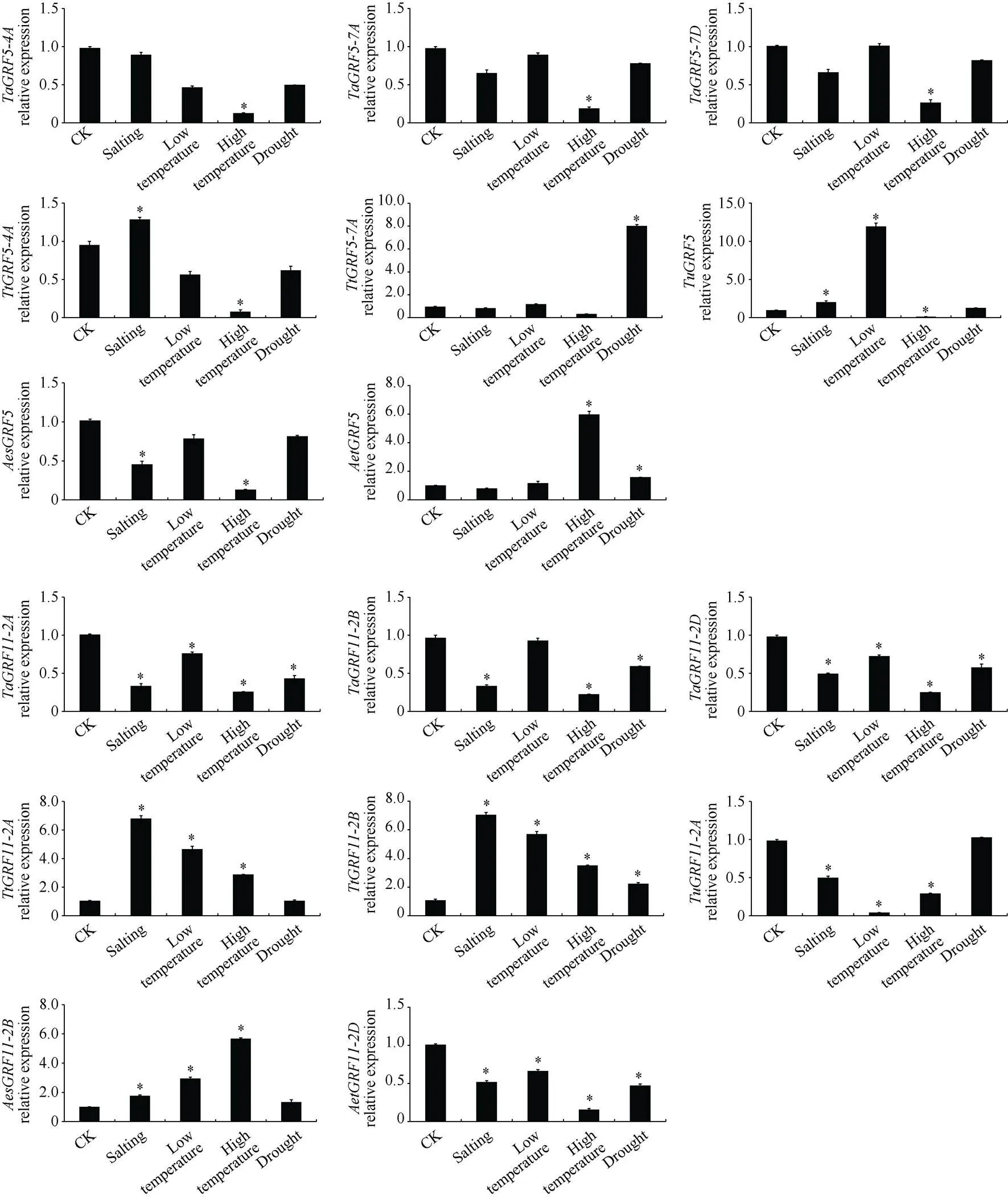
图9 16个GRF基因对环境胁迫响应的表达模式分析
*表示处理间存在显著差异(< 0.05)。
* indicates significant differences between the stress conditions and the control condition at< 0.05.
3 讨论
GRF转录因子广泛参与了植物器官发育、逆境响应、花期和产量等多个生物学过程。目前, GRF转录因子家族已经在拟南芥[7]、水稻[9]、番茄[36]、大豆[37]、谷子[38]等作物中开展了大量的研究。作为一种植物特有的转录因子, GRF在植物生长发育中起着重要调控作用。研究表明GRF可以通过维持或者促进细胞分裂能力来促进植物器官的生长[39]。到目前为止, 在低等植物苔藓中仅鉴定到GRF家族成员2个, 在高等植物的单子叶物种中, 鉴定到GRF家族成员的数量为8 (大麦)~19 (香蕉)个; 且通常表现为双子叶植物GRF家族成员的数目大于单子叶植物,而多倍体也通常大于二倍体[40]。在本研究中, 通过搜索本地基因库数据, 在小麦及其祖先物种中共鉴定出GRF家族成员128个, 其中分别在乌拉尔图小麦、拟斯卑尔脱山羊草、粗山羊草、栽培二粒小麦和普通小麦中鉴定出15、12、19、29和53个基因, 并通过研究发现GRF家族成员的数量在进化过程中随着多倍化出现了扩增的趋势。四倍体栽培二粒小麦GRF家族成员的数量与其祖先物种二倍体乌拉尔图小麦和拟斯卑尔脱山羊草的数量之和大致相同, 六倍体小麦家族成员数量比其祖先物种栽培二粒小麦和粗山羊草的数量之和多5个, 推测这种GRF成员数量的变化可能与增强生物体对复杂环境的适应性有关。基因数量的变化往往和基因进化相关联, 而基因结构的异同也与其进化密不可分。
根据系统发育树的拓扑结构, GRF家族成员被分为5组(I~V), 5个分组分别有34、27、21、21和25个成员, 通过比较不同物种在系统发育树中的位置, 发现具有高度同源性和相似结构的基因家族成员通常聚在一组, 这表明在同一分支上的家族成员可能具有相似的基因功能。另外发现GRF家族成员大多数分布在2号、6号和7号染色体上, 其余染色体上分布较少, 说明GRF在进化过程中出现了分布不均匀的情况, 并且被遗传了下来。此外, 基于MEME分析, 各个组间的蛋白基序差异较大, 而同一分组不同分支的基因具有相似的蛋白基序, 并且几乎所有基因都鉴定到了Motif1。说明GRF在进化过程中存在内部分化, 基因之间可能存在功能的差异。这与在藜麦中GRF家族成员鉴定到的结果相似[39]。相同功能的基因往往存在相同的结构, 这也可能是进化与遗传的结果, 而进化与遗传也与他们之间的共线性关系联系密切。
通过物种内的共线性分析发现, 在小麦及其祖先物种中都存在片段复制, 但是串联复制较少, 推测这5个物种主要是通过片段复制来扩展家族成员的。同时, 乌拉尔图小麦、拟斯卑尔脱山羊草和粗山羊草中上述两种复制事件都相对较少, 这与其基因组大小也有密切的联系。栽培二粒小麦和普通小麦中存在较多的重复事件, 也是这两个物种在进化中GRF家族成员扩张的主要原因。小麦在进化的过程中有很多的祖先基因被保留了下来, 通过种间的同源共线性基因分析发现, 乌拉尔图小麦和拟斯卑尔脱山羊草中的通过片段复制以加倍的方式遗传到了栽培二粒小麦中, 从而造成了栽培二粒小麦中GRF家族成员的扩增, 并且栽培二粒小麦和粗山羊草中的也通过片段复制以加倍的方式遗传到了普通小麦中, 这可能是小麦在进化过程中发生了基因组多倍化现象[41], 前人研究表明, 普通小麦由3个不同的二倍体物种经过两轮异源多倍化过程形成, 这使普通小麦在其适应性方面得到了极大的改善[42]。同时, 在物种间基因的遗传复制大多是直接在自身染色体组上进行, 也有少部分基因出现了不同染色体亚组间的复制, 这可能也是基因进化的一种表现[43]。另外, 除了()和()基因对的a/s比率大于1, 其他基因对的a/s比率均小于1, 这说明在大多数情况下基因在进化过程中是相对保守的, 进化相对较慢。基于比较基因组分析, 虽然不同物种的基因组数量和染色体大小均不同, 但在数百万年的进化过程中, 相关物种的基因顺序仍然高度保守。通过比较5个物种的基因组序列, 发现在早期进化过程中存在一个或多个大规模基因组重复事件, 这些重复事件以及进化过程中的多倍化事件都将显著影响基因在转录水平的表达特性。
启动子区域的顺式作用元件通过对细胞内外不同信号的响应来调控基因转录, 从而调控植物的生长发育等过程[44]。研究发现, MBS、Sp1和AE-box等顺式作用元件在玉米中参与干旱及低温胁迫的诱导[45], ABRE顺式调控元件在水稻中则受到干旱和脱落酸的诱导[46]。本研究发现, 小麦及其祖先物种GRF家族成员的启动子区域含有多种顺式作用元件,主要包括MBS、ABRE和LTR等响应逆境胁迫的元件以及P-box和TGA-box等参与IAA和GA诱导的元件, 表明它们在逆境和激素信号通路中可能发挥着重要作用。前人研究表明, GRF通过参与GA信号通路调节了水稻茎秆的伸长生长[6], 并在小麦干旱胁迫下表现出强烈的反应[12]。本研究通过RT-qPCR分析发现, 在16个基因中, 有12个基因在高温胁迫诱导下呈现出下调趋势, 这与前人研究结果一致, 推测可能是由于高温对于小麦及其祖先生长的影响较大, 导致在植物中的表达下降; 值得注意的是, 外源GA使普通小麦和栽培二粒小麦的呈显著上调表达趋势, 这与在二穗短柄草[40]和水稻[47]中的结果相似, 表明基因参与植物的生长发育以及对逆境胁迫响应的调控模式是保守的。然而, 有关GRF在小麦及其祖先物种中的功能仍然需要进一步的研究。
4 结论
本研究在小麦及其祖先物种中共鉴定到128个GRF家族成员, 基于该基因的结构功能与系统发育的拓扑结构将其分为5组, 其中较多基因被定位在6号染色体上, 表明6号染色体上的家族成员在进化的过程中位置较为保守。GRF家族成员的内含子数为1~5个且都有完整的基因结构, 另外不同的基因含有不同的启动子, 这与基因功能有主要的关系。在5个物种之间都存在着共线性区域, 但不是都存在着基因的串联复制。5个物种中GRF家族成员的表达特性存在特异性, 但大多数与赤霉素诱导有关, 且对于干旱胁迫的响应较为明显, 而且对于其他的激素和逆境胁迫也有不同程度的响应, GRF在5个物种的进化中较保守, 起到的功能较相似。
附表 请见网络版: 1) 本刊网站http://zwxb. chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodicalzuowxb.aspx。
[1] Feng K, Hou X L, Xing G M, Liu J X, Duan A Q, Xu Z S, Li M Y, Zhuang J, Xiong A S. Advances in AP2/ERF super-family transcription factors in plant., 2020, 40: 750–776.
[2] Claus S, Michael B. The regulation of transcription factor activity in plants., 1998, 3: 378–383.
[3] Karam B S, Rhonda C F, Luis O. Transcription factors in plant defense and stress responses., 2002, 5: 430–436.
[4] Du W X, Yang J F, Li Q, Su Q, Yi D X, Pang Y Z. Genome-wide identification and characterization of growth regulatory factor family genes in., 2022, 23: 6905.
[5] Wang F D, Qiu N W, Ding Q, Li J J, Zhang Y H, Li H Y, Gao J W. Genome-wide identification and analysis of the growth-regulating factor family in Chinese cabbage (L. ssp)., 2014, 15: 807.
[6] van der Knaap, Kim J H, Kende H. A novel gibberellin-induced gene from rice and its potential regulatory role in stem growth., 2000, 122: 695–704.
[7] Kim J H, Choi D, Kende H. Thefamily of putative transcription factors is involved in leaf and cotyledon growth in., 2003, 36: 94–104.
[8] Zhang D F, Li B, Jia G Q, Zhang T F, Dai J R, Li J S, Wang S C. Isolation and characterization of genes encoding GRF transcription factors and GIF transcriptional coactivators in maize (L.)., 2008, 175: 809–817.
[9] Choi D, Kim J H, Kende H. Whole genome analysis of thegene family encoding plant-specific putative transcription activators in rice (L.)., 2004, 45: 897–904.
[10] Huang W D, He Y Q, Yang L, Lu C, Zhu Y X, Sun C, Ma D F, Yin J L. Genome-wide analysis of growth-regulating factors (GRFs) in., 2021, 9: e10701.
[11] Yi W, Luan A P, Liu C Y, Wu J, Zhang W, Zhong Z Q, Wang Z P, Yang M Z, Chen C J, He Y H. Genome-wide identification, phylogeny, and expression analysis of GRF transcription factors in pineapple ()., 2023, 14: 1159223.
[12] Zan T, Zhang L, Xie T T, Li L Q. Genome-Wide identification and analysis of the growth-regulating factor () gene family and GRF-Interacting factor family inL., 2020, 58: 1–20.
[13] Kim J H, Kende H. A transcriptional coactivator,, is involved in regulating leaf growth and morphology in., 2004, 101: 13374–13379.
[14] Kim J H. Biological roles and an evolutionary sketch of the GRF-GIF transcriptional complex in plants., 2019, 52: 227–238.
[15] Liang G, He H, Li Y, Wang F, Yu D Q. Molecular mechanism of microRNA396 mediating pistil development in., 2014, 164: 249–258.
[16] Liu X, Guo L X, Jin L F, Liu Y Z, Liu T, Fan Y H, Peng S A. Identification and transcript profiles of citrus growth-regulating factor genes involved in the regulation of leaf and fruit development., 2016, 43: 1059–1067.
[17] Kuijt S J H, Greco R, Agalou A, Shao J, Hoen C C J, Overnäs E, Osnato M, Curiale S, Meynard D, van Gulik R, de Faria M S, Atallah M, de Kam R J, Lamers G E M, Guiderdoni E, Rossini L, Meijer A H, Ouwerkerk P B F. Interaction between the growth-regulating factor and knotted1-like homeobox families of transcription factors., 2014, 164: 1952–1966.
[18] Li S C, Gao F Y, Xie K L, Zeng X H, Cao Y, Zeng J, He Z S, Ren Y, Li W B, Deng Q M, Wang S Q, Zheng A P, Zhu J, Liu H N, Wang L X, Li P. The OsmiR396c-OsGRF4-OsGIF1 regulatory module determines grain size and yield in rice., 2016, 14: 2134–2146.
[19] Wang P, Xiao Y, Yan M, Yan Y, Lei X J, Di P, Wang Y P. Whole- genome identification and expression profiling of growth- regulating factor () and GRF-interacting factor () gene families in Panax ginseng., 2023, 24: 334.
[20] Zhang S W, Li G G, Wang Y D, Anwar A, He B, Zhang J W, Chen C M, Hao Y W, Chen R Y, Song S W. Genome-wide identification ofgenes in flowering Chinese cabbage and preliminary functional analysis ofin nitrogen metabolism., 2023, 14: 1144748.
[21] Yarra R, Krysan P J. GRF-GIF duo and GRF-GIF-BBM: novel transformation methodologies for enhancing regeneration efficiency of genome-edited recalcitrant crops., 2023, 257: 60.
[22] Liu Y T, Guo P, Wang J, Xu Z Y. Growth-regulating factors: conserved and divergent roles in plant growth and development and potential value for crop improvement., 2022, 113: 1122–1145.
[23] Daniela L, Javier F P. MicroRNA miR396, GRF transcription factors and GIF co-regulators: a conserved plant growth regulatory module with potential for breeding and biotechnology., 2020, 53: 31–42.
[24] Montenegro J D, Golicz A A, Bayer P E, Hurgobin B, Lee H, Chan C K, Visendi P, Lai K, Doležel J, Batley J, Edwards D. The pangenome of hexaploid bread wheat., 2017, 90: 1007–1013.
[25] Etienne P, Pierre S, Jérôme S, Cyrille S, Frédéric C, Philippe L, Abraham K, Monika M, Shahryar K, Wolfgang S, Evans L, Daryl S, Andrzej K, Michael A, Sonia V, Hélène B, Kellye E, Rudi A, Jan S, Hana S, Jaroslav D, Michel B, Catherine F. A physical map of the 1-Gigabase bread wheat chromosome 3B., 2008, 322: 101–104.
[26] Zhang M, Qiu X B. Genetic basis of genome size variation of wheat., 2023, 23: 285–285.
[27] El B M, Murat F, Veyssiere M, Molinier M, Flores R, Burlot L, Alaux M, Quesneville H, Pont C, Salse J. Reconciling the evolutionary origin of bread wheat ()., 2017, 213: 1477–1486.
[28] 魏益民. 中国小麦的起源、传播及进化. 麦类作物学报, 2021, 41: 305–309. Wei Y M. Origin, spread and evolution of wheat in China., 2021, 41: 305–309 (in Chinese with English abstract).
[29] Chen F, Yang Y Z, Luo X F, Zhou W G, Dai Y J, Zheng C, Liu W G, Yang W Y, Shu K. Genome-wide identification of GRF transcription factors in soybean and expression analysis offamily under shade stress., 2019, 19: 1–13.
[30] Chen H L, Ge W N. Identification, molecular characteristics, and evolution ofgene family in foxtail millet (L.)., 2022, 12: 727674.
[31] Tamura K, Stecher G, Kumar S. MEGA11: molecular evolutionary genetics analysis version 11., 2021, 38: 3022–3027.
[32] Chen C J, Chen H, Zhang Y, Thomas H R, Margaret H F, He Y H, Xia R. TBtools: an integrative toolkit developed for interactive analyses of big biological data., 2020, 13: 1194–1202.
[33] Li C, Li Q G, Dunwell J M, Zhang Y M. Divergent evolutionary pattern of starch biosynthetic pathway genes in grasses and dicots., 2012, 29: 3227–3236.
[34] Timothy L B, Charles E. Fitting a Mixture Model by Expectation Maximization to Discover Motifs in Biopolymers. Menlo Park, California: AAAI Press, 1994. pp 28–36.
[35] Wang YP,Tang HB,Debarry JD, Tan X, Li JP, Wang XY, Lee TH, Jin HZ, Marler B, Guo H, Kissinger JC, Paterson AH. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity., 2012, 40: e49.
[36] Khadiza K, Arif H K R, Park J, Ujjal K N, Chang K K, Ki-Byung L, Ill S N, Mi-Young C, Hikmet B. Molecular characterization and expression profiling of tomato GRF transcription factor family genes in response to abiotic stresses and phytohormones., 2017, 18: 1056.
[37] Noon J B, Hewezi T, Baum T J. Homeostasis in the soybean miRNA396-GRF network is essential for productive soybean cyst nematode infections., 2019, 70: 1653–1668.
[38] 张立全, 张浩林, 李丛丛, 姚磊, 魏建华, 张杰伟. 谷子GRF基因家族鉴定与分析. 西南农业学报, 2021, 34: 2340–2347.Zhang L Q, Zhang H L, Li C C, Yao L, Wei J H, Zhang J W. Genome-wide analysis and identification of GRF gene family in foxtail millet ()., 2021, 34: 2340–2347 (in Chinese with English abstract).
[39] 时丕彪, 何冰, 费月跃, 王军, 王伟义, 魏福友, 吕远大, 顾闽峰. 藜麦GRF转录因子家族的鉴定及表达分析. 作物学报, 2019, 45: 1841–1850. Shi P B, He B, Fei Y Y, Wang J, Wang W Y, Wei F Y, Lyu Y D, Gu M F. Identification and expression analysis of GRF transcription factor family of., 2019, 45: 1841–1850 (in Chinese with English abstract).
[40] 马超, 宋鹏, 尚申申, 杨夏夏, 杨金华, 韩群威, 李记民, 冯雅岚. 二穗短柄草GRFs基因家族的鉴定及表达模式分析. 核农学报, 2020, 34: 1152–1162. Ma C, Song P, Shang S S, Yang X X, Yang J H, Han Q W, Li J M, Feng Y L. Whole genome identification and analysis of GRFs gene family in., 2020, 34: 1152–1162 (in Chinese with English abstract).
[41] Jiao Y N, Wickett N J, Ayyampalayam S, Chanderbali A S, Landherr L, Ralph P E, Tomsho L P, Hu Y, Liang H Y, Soltis P S, Soltis D E, Clifton S W, Schlarbaum S E, Schuster S C, Ma H, Leebens-Mack J, de Pamphilis C W. Ancestral polyploidy in seed plants and angiosperms., 2011, 473: 97–100.
[42] 赵旭博, 李爱丽, 毛龙. 植物多倍化过程中小分子RNA调控基因表达机制研究进展. 作物学报, 2013, 39: 1331–1338. Zhao X B, Li A L, Mao L. Progress on gene regulatory mechanisms by small RNAs during plant poly-ploidization., 2013, 39: 1331–1338 (in Chinese with English abstract).
[43] Panchy N, Lehti-Shiu M, Shiu S H. Evolution of gene duplication in plants., 2016, 171: 2294–2316.
[44] Kong F L, Wang J, Cheng L, Liu S Y, Wu J, Peng Z, Lu G. Genome-wide analysis of the mitogen-activated protein kinase gene family in., 2012, 499: 108–120.
[45] Tao Y, Wang F T, Jia D M, Li J T, Zhang Y M, Jia C G, Wang D P, Pan H Y. Cloning and functional analysis of the promoter of a stress-inducible gene () in maize., 2015, 33: 200–208.
[46] Lee S C, Kim S H, Kim S R. Drought induciblepromoter is activated by OsDREB1A and OsDREB1D., 2013, 56: 115–121.
[47] Lee S J, Lee B H, Jung J H, Park S K, Song J T, Kim J H. Growth- regulating factor and GRF-interacting factor specify meristematic cells of gynoecia and anthers., 2018, 176: 717–729.
Whole genome identification and analysis of GRFs transcription factor family in wheat and its ancestral species
WANG Tian-Ning1, FENG Ya-Lan2, JU Ji-Hao1, WU Yi1, ZHANG Jun1, and MA Chao1,*
1Henan University of Science and Technology, Agronomy College, Luoyang 471000, Henan, China;2Wuchang University of Technology, College of Life Science, Wuhan 430223, Hubei, China
Growth-regulating factors (GRFs) play important roles in plant growth, stress response, and hormone signal transduction. Systematic analysis of the distribution, structure, evolution, and expression characteristics of the GRF transcription factor family members in the genome of wheat and its ancestral species is of great significance for in-depth research on the biological functions of GRF family and the evolution of wheat. In this study, bioinformatics methods were used to identify the whole genome of GRF members from five species (,,,, and), and their physical and chemical properties, phylogenetic relationships, gene structure, promoter-regulatory element, and expression characteristics were also analyzed. The results showed that there were 15, 12, 19, 29, and 53 GRF members in,,,, and, respectively. Through interspecific colinearity analysis, we found that 18 and 29 members ofwere colinear withand, and 36 and 37 members ofwere colinear withand, respectively. The prediction of promoter-regulatory element found thatgene had basic transcription elements and some binding elements with growth, development, and stress response. RT-qPCR analysis revealed that mostgenes up-regulated under exogenous IAA, GA, and drought stress, but down-regulated under high temperature stress, indicating that members of the GRF family exerted a crucial influence in hormone response and stress. Phylogenetic analysis evidenced that there was a conserved and complex evolutionary relationship between the GRF members of wheat and its ancestral species. The above results provide a theoretical basis for the evolution and functional research of the GRF transcription factor family.
wheat; ancestral species; GRF; bioinformatics; phylogenetic analysis
10.3724/SP.J.1006.2024.31046
本研究由国家自然科学基金项目(32372227), 河南省自然科学基金项目(222300420430)和河南省高等学校青年骨干教师培养计划 (2021GGJS050)资助。
This study was supported by the National Natural Science Foundation of China (32372227), the National Natural Science Foundation of Henan Province (222300420430), and the Training Program for University Young Key Teachers in Henan Province (2021GGJS050).
马超, E-mail: machao840508@163.com
E-mail: haust6626wtn@163.com
2023-08-04;
2023-10-23;
2023-11-10.
URL: https://link.cnki.net/urlid/11.1809.S.20231108.1632.004
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
——以多重共线性内容为例

