微管蛋白α-1B在泛癌中的表达及临床意义
刘 艺,周晔禄,贺文芳,卢瑗瑗,赵晓迪,吴开春
(国家消化系统疾病临床医学研究中心,消化系肿瘤整合防治全国重点实验室,空军军医大学西京消化病医院,陕西 西安 710032)
癌症是世界范围内重要的公共卫生问题,每年全世界约有1 900万新发病例和1 000万死亡病例,给个人、家庭、社区和卫生系统带来了巨大的负担[1]。单一癌症种类的研究限制了我们对基因多面性和潜在机制的全局视野,描述基因在不同肿瘤类型的差异和相似性对识别肿瘤发生的基本动态以及为诊断、预后和治疗提供信息至关重要。泛癌分析是一种跨多种癌症类型的分子畸变分析,可将来自不同谱系癌细胞的特征进行整理总结,以发现失调的关键生物学过程[2],泛癌基因探索已成为揭示癌症基因机制的一种重要手段。
微管蛋白α-1B(tubulin alpha-1B,TUBA1B)基因编码微管蛋白α-1B链,可水解GTP使微管生长[3]。TUBA1B的c端尾部有7个谷氨酸残基,能够发生谷氨酰化、甘氨酸化和去酪氨酸化[4-5]。通过此c端,TUBA1B可与微管结合蛋白相互作用,包括细胞骨架相互作用因子和细胞骨架调节因子。目前已有多项研究表明TUBA1B在多种肿瘤中表达升高,通过影响细胞极性、迁移、骨架形成等参与疾病发展,如鼻咽癌[6]、乳腺癌[7]、肝细胞癌[8]、精神分裂症[9]等。此外,有证据表明TUBA1B可通过影响免疫细胞的生物学过程参与肿瘤发生发展,如参与结直肠癌患者CD8+T细胞耗竭[10];影响肝细胞癌患者NK细胞浸润[11]。
然而,目前TUBA1B在肿瘤发展的机制未得到明确阐述,且缺少其在泛癌中的研究,因此,我们利用癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库和GEO数据库对TUBA1B进行系统分析,在33种肿瘤中通过表达水平、疾病分期、临床预后和基因突变特征等方面评估TUBA1B与肿瘤恶性表型的相关性。随后,我们对TUBA1B相关免疫细胞浸润水平、免疫相关基因共表达水平进行数据挖掘。最后进行TUBA1B的富集分析,包括相互作用蛋白预测、通路富集分析,通过生物信息学的方法探讨TUBA1B参与肿瘤发生发展的可能机制,为基础研究提供线索。
1 资料与方法
1.1 资料
在TCGA数据库中提取33种癌症样本和相应正常样本的mRNA表达谱及相关临床数据,共涉及11 315份样本。在组织基因谱(Genotype-Tissue Expression,GTEx)中下载31个不同组织的正常基因表达数据。将基因表达量进行log2转化后,对31个正常组织和33个肿瘤组织中的TUBA1B表达水平进行评估并将33个癌症样本与配对标准样本进行比较。
1.2 方法
1.2.1 基因和蛋白表达分析 肿瘤免疫评测网站(Tumor Immune Estimation Resource,TIMER)可分析不同肿瘤的基因表达、免疫浸润、临床预后[12]。我们在TIMER2.0网站(http://timer.cistrome.org/)中检索TUBA1B,分析TCGA内33种癌症类型中TUBA1B在肿瘤和癌旁中的表达。基因表达分析(Gene Expression Profiling Interactive Analysis,GEPIA2,http://gepia 2.cancer-pku.cn/)可进行基因相关性、临床生存和自定义分析[13]。我们利用GEPIA2分析了TUBA1B在TCGA肿瘤中的表达,并将匹配的TCGA癌旁和GTEx正常组织数据作为对照。阿拉巴马大学伯明翰分校网站(University of Alabama at Birmingham Cancer Data Analysis Portal,UALCAN,http://ualcan.path.uab.edu)是可利用来自31种肿瘤的转录组和临床资源进行交互式分析的在线网站[14]。在此,分析了TUBA1B在肿瘤和正常样本中的相对表达,以及TUBA1B表达高低与肿瘤分期的相关性。使用蛋白质组学分析网站(Clinical Proteomic Tumor Analysis Consortium,CPTAC,http://ualcan.path.uab.edu/analysis-prot.html)分析TUBA1B在肿瘤中蛋白水平的表达情况。
1.2.2 生存预后分析 使用GEPIA2工具进行患者的生存预后分析。在网站中检索TUBA1B基因。高表达和低表达队列按高(50%)和低(50%)临界值划分。此外,使用Kaplan-Meier工具网站的“泛癌RNA-seq”模块,高低表达组界限按“最佳cut-off值”分界,评估21种癌症总体生存期与TUBA1B表达高低的相关性[15]。
1.2.3 基因突变分析 cBioPortal网站(https://www.cbioportal.org)可用于分析肿瘤多组学数据并做可视化分析[16-17]。本文利用此网站进行了TUBA1B的基因突变分析,包括突变频率、突变类型、拷贝数改变、突变与生存预后相关性的分析。
1.2.4 免疫浸润水平分析 使用TIMER2.0在线分析平台对多种肿瘤免疫浸润水平评估[12]。在免疫模块中检索TUBA1B基因,选择免疫浸润类型,在此分析了重要的免疫浸润细胞和免疫相关基因在多种肿瘤中的相关性。
1.2.5 基因富集分析 通过互作蛋白检索网站(https://string-db.org)筛选了TUBA1B的结合蛋白,并进行蛋白-蛋白相互作用网络的富集分析[18]。使用TIMER2.0分析TUBA1B与不同癌症类型中TUBA1B结合的12个相互作用因子的相关性。利用GEPIA2筛选出在多种肿瘤中与TUBA1B相似的100个基因,并将这100个基因与STRING网站筛选出的12个基因取交集,得出2个共同基因。利用ShinyGO图形化基因富集工具分析100个TUBA1B相似基因的富集通路[19],随后使用微生信在线网站(https://www.bioinformatics.com.cn)中的GO、通路气泡图将富集结果可视化。
1.2.6 统计学分析 非参数Wilcoxon秩和检验应用于组间差异;相关分析使用Spearman相关检验;Kaplan-Meier曲线应用于生存预后分析,组间生存率采用Cox检验,检验水准为α=0.05,P<0.05表示差异有统计学意义。
2 结果
2.1 TUBA1B在泛癌中的表达差异分析
进行TUBA1B的泛癌分析包括癌与癌旁表达差异分析、肿瘤TNM分期差异分析、预后差异分析、免疫浸润分析、基因突变特征分析和富集分析,以探索TUBA1B参与多种肿瘤发生发展的可能机制。本研究使用的数据库概览如图1所示。
图1 数据库概览
为了研究TUBA1B在多种癌与癌旁的表达差异,我们分析了TUBA1B在mRNA水平上的表达差异(图2A)。结果表明在膀胱尿路上皮癌、宫颈鳞状上皮细胞癌、结肠癌等16种肿瘤中TUBA1B表达显著高于癌旁组织(P<0.05)。
此外,鉴于TIMER2.0数据库个别肿瘤缺少癌旁数据,我们又利用GTEx数据集作为对照组进行分析,结果表明在弥漫大B细胞淋巴瘤等6种肿瘤中,TUBA1B的mRNA表达高于癌旁(P<0.05,图2B)。
进一步针对TUBA1B蛋白表达差异展开研究。结果显示TUBA1B蛋白表达在胰腺癌、肝细胞癌和肾细胞癌中显著升高(P<0.05,图2C)。
2.2 TUBA1B在泛癌临床分期间的表达差异分析
鉴于TUBA1B在多种肿瘤中表达升高,我们进一步探索了TUBA1B与多种肿瘤病理分期之间的相关性。TUBA1B在多种肿瘤的临床早期即有表达升高,包括头颈鳞状细胞癌、胃腺癌、膀胱尿路上皮癌、子宫内膜癌、肾脏乳头状细胞癌、侵袭性乳腺癌、肺腺癌、胆管癌、结肠癌、食管癌、宫颈鳞状上皮细胞癌、肝细胞癌、肺鳞状细胞癌、甲状腺癌(P<0.05,图3A)。而在另一些肿瘤包括肾上腺皮质癌等3种肾脏肿瘤中,TUBA1B的表达随疾病进展呈升高趋势(P<0.05,图3B)。
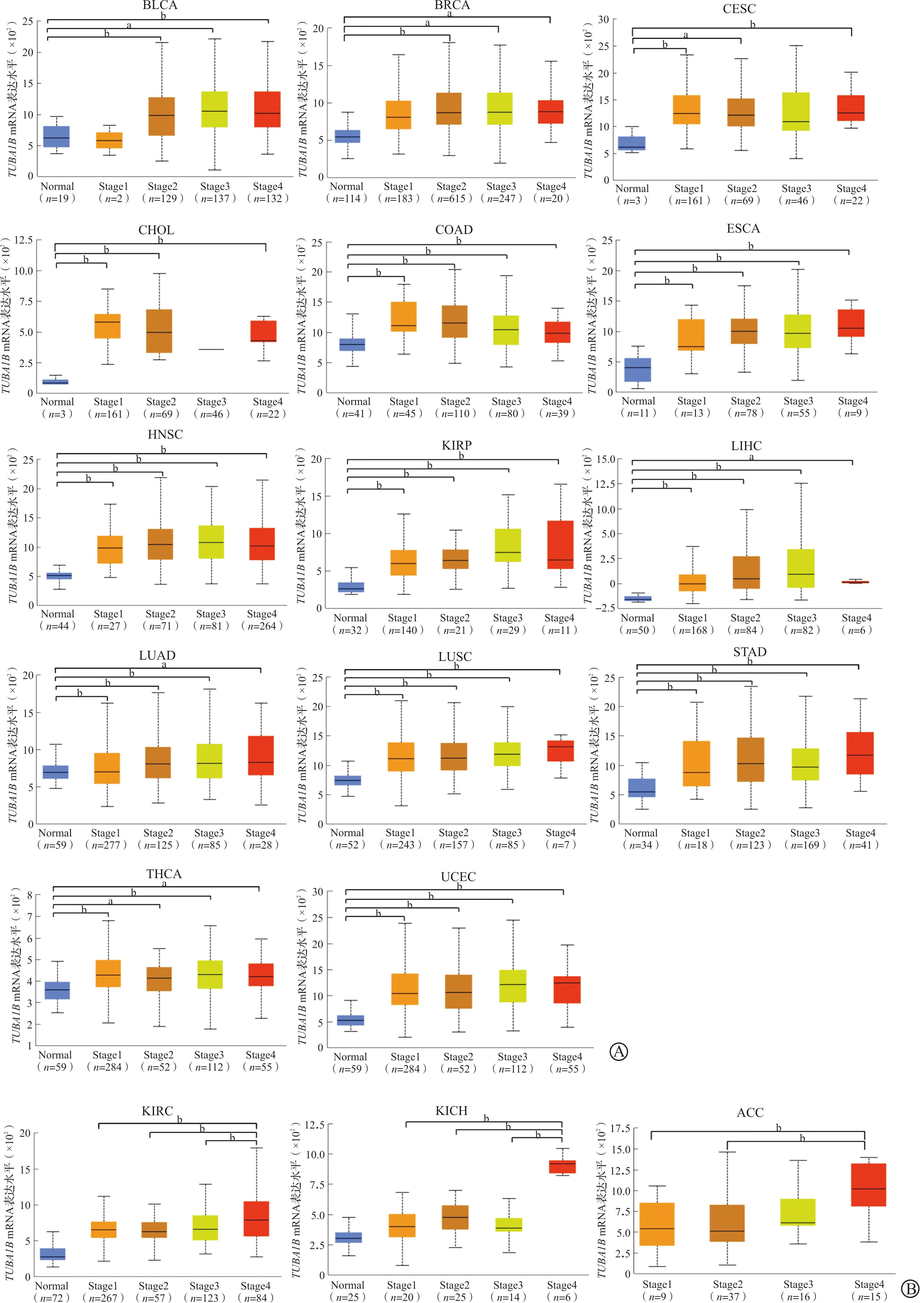
A:UALCAN数据库分析TUBA1B与肿瘤临床早期的相关性;B:UALCAN数据库分析TUBA1B与肿瘤临床晚期的相关性。Normal:癌旁组织;Stage:临床分期;BLCA:膀胱尿路上皮癌;BRCA:乳腺浸润癌;CESC:宫颈鳞状上皮癌;CHOL:胆管癌;COAD:结肠癌;ESCA:食管癌;GBM:多形成性胶质细胞瘤;HNSC:头颈癌;KIRP:肾乳头状细胞癌;LIHC:肝癌;LUAD:肺腺癌;LUSC:肺鳞状细胞癌;STAD:胃癌;THCA:甲状腺癌;UCEC:子宫内膜样癌;ACC:肾细胞癌;KICH:肾嫌色细胞癌;KIRC:肾透明细胞癌;TUBA1B:微管蛋白α-1B;UALCAN:阿拉巴马大学伯明翰分校网站。aP<0.05,bP<0.01。
2.3 TUBA1B表达与泛癌的生存预后分析
分析TUBA1B表达与肿瘤患者生存预后的关系。结果显示在肾上腺皮质癌、肾嫌色细胞癌、脑低级别胶质瘤、间皮瘤中TUBA1B高表达与较差的总生存期有关(P<0.05,图4A)。此外,更换预后数据库进行分析,结果显示,高表达TUBA1B组在膀胱尿路上皮癌、肾透明细胞癌、侵袭性乳腺癌、肾乳头状细胞癌、宫颈鳞状上皮细胞癌、肝细胞癌、肉瘤、肺腺癌、胰腺癌中预示差的总生存期(P<0.05,图4B)。
2.4 TUBA1B在泛癌中的突变特征
为了全面展示TUBA1B在肿瘤发生发展中的生物学功能,我们分析TUBA1B在泛癌中的基因突变状态。结果如图5A所示,突变类型被分为:碱基突变、结构变异、扩增、深度缺失4种,其中TUBA1B在成熟B细胞肿瘤中突变频率最高,且最高的突变种类为扩增突变,突变频率高达4.17%。此外,在肾上腺皮质瘤和精原细胞瘤中同样观察到扩增突变为主要突变类型,频率超过2%。相关性分析表明TUBA1B的扩增突变与其mRNA高表达显著相关(图5B)。此外,碱基突变是子宫内膜癌、结直肠癌、膀胱癌、头颈部肿瘤的主要突变类型,其中子宫内膜癌和结直肠癌中的占比超过1%。膀胱癌中TUBA1B突变患者总体生存期和无进展生存期均较差(P<0.05,图5C)。

A:TUBA1B在泛癌中的突变频率;B:TUBA1B表达水平与基因突变类型间的相关性;C:TUBA1B突变与临床预后的相关性。TUBA1B:微管蛋白α-1B。
2.5 TUBA1B与免疫浸润的相关性分析
分析TUBA1B与多种免疫细胞浸润的相关性,TUBA1B与多种肿瘤的肿瘤相关成纤维细胞、髓源性抑制细胞等免疫细胞呈正相关关系,与造血干细胞表达呈负相关(图6A)。同时,将TUBA1B与免疫相关基因的表达进行相关性分析,重点关注趋化因子、趋化因子受体、免疫激活和免疫抑制相关基因。TUBA1B与多种肿瘤的趋化因子(CCL3、CCL4),趋化因子受体(CCR3、CCR4)、免疫检查点(CTLA-4)呈正相关,但在胸腺瘤中,TUBA1B表达与免疫细胞浸润及免疫分子呈负相关(图6B~C)。

A:TUBA1B与肿瘤内免疫细胞浸润能力相关性;B~C:TUBA1B与免疫相关基因相关性。ACC:肾上腺皮质癌;BLCA:膀胱尿路上皮癌;BRCA:乳腺浸润癌;CESC:宫颈鳞状上皮癌;CHOL:胆管癌;COAD:结肠癌;DLBC:弥漫性大B细胞淋巴瘤;ESCA:食管癌;GBM:多形成性胶质细胞瘤;HNSC:头颈癌;KICH:肾嫌色细胞癌;KIRC:肾透明细胞癌;KIRP:肾乳头状细胞癌;LGG:脑低级别胶质瘤;LIHC:肝癌;LUAD:肺腺癌;LUSC:肺鳞状细胞癌;MESO:间皮瘤;OV:卵巢癌;PAAD:胰腺癌;PCPG:嗜铬细胞瘤;PRAD:前列腺癌;READ:直肠癌;SARC:肉瘤;SKCM:黑色素瘤;STAD:胃癌;TGCT:睾丸癌;THYM:胸腺瘤;UCEC:子宫内膜样癌;UCS:子宫癌肉瘤;UVM:眼部黑色素瘤。TUBA1B:微管蛋白α-1B。aP<0.05,bP<0.01。
2.6 TUBA1B相关基因的富集分析
为了进一步探索TUBA1B参与肿瘤发生发展中的作用机制,我们分析了与TUBA1B相互作用的结合蛋白,得出与TUBA1B相互作用的12个蛋白(图7A)。对挖掘出的12个互作蛋白与TUBA1B进行相关性分析,发现驱动蛋白家族2C(kinesin family member 2C,KIF2C)、丝/苏氨酸蛋白激酶、微管蛋白α-1A链、微管蛋白β(β-tubulin,TUBB)、微管蛋白β-2A链、微管蛋白-酪氨酸羧肽酶1、微管蛋白酪氨酸羧肽酶2、卵黄羧肽酶与TUBA1B在多种肿瘤中关系密切(图7B)。随后对TUBA1B相似基因进行挖掘,将前100个与其相似的基因与其相互作用蛋白进行交集分析,得到TUBB和KIF2C两个分子(图7C),二者与TUBA1B在膀胱尿路上皮癌(KIF2C,r=0.75,P<0.01;TUBB,r=0.69,P<0.01)、侵袭性乳腺癌(KIF2C,r=0.74,P<0.001;TUBB,r=0.60,P<0.01)、结肠癌(KIF2C,r=0.53,P<0.01;TUBB,r=0.56,P<0.01)、食管癌(KIF2C,r=0.72,P<0.01;TUBB,r=0.68,P<0.01)、头颈鳞状细胞癌(KIF2C,r=0.69,P<0.01;TUBB,r=0.6,P<0.01)中均有很强的正相关性(图7D)。

A:TUBA1B相互作用蛋白网络;B:TUBA1B与相互作用基因的相关性分析;C:TUBA1B相互作用基因和100个相似基因的交联分析;D:TUBA1B与TUBB、KIF2C的相关性分析;E~F:针对TUBA1B相似基因的KEGG、GO富集分析。ACC:肾上腺皮质癌;BLCA:膀胱尿路上皮癌;BRCA:乳腺浸润癌;CESC:宫颈鳞状上皮癌;CHOL:胆管癌;COAD:结肠癌;DLBC:弥漫性大B细胞淋巴瘤;ESCA:食管癌;GBM:多形成性胶质细胞瘤;HNSC:头颈癌;KICH:肾嫌色细胞癌;KIRC:肾透明细胞癌;KIRP:肾乳头状细胞癌;LAML:急性髓系白血病;LGG:脑低级别胶质瘤;LIHC:肝癌;LUAD:肺腺癌;LUSC:肺鳞状细胞癌;MESO:间皮瘤;OV:卵巢癌;PAAD:胰腺癌;PRAD:前列腺癌;RCC:肾细胞癌;READ:直肠癌;SARC:肉瘤;SKCM:黑色素瘤;STAD:胃癌;TGCT:睾丸癌;THYM:胸腺瘤;UCEC:子宫内膜样癌;UCS:子宫癌肉瘤;UVM:眼部黑色素瘤;TUBA1B:微管蛋白α-1B;TUBB:微管蛋白β;KIF2C:微管解聚激酶。
随后对得出的100个TUBA1B相似基因进行富集分析,KEGG通路分析显示TUBA1B可能通过有丝分裂、微管细胞骨架、细胞周期等参与肿瘤发生发展(图7E)。GO富集分析则显示TUBA1B与DNA复制、错配修复、细胞周期、P53信号通路相关(图7F)。
3 讨论
本文利用来自TCGA数据库的33种肿瘤数据,通过基因表达差异、分期差异、预后差异、基因突变特征、免疫浸润和基因富集分析,探索适用于广谱肿瘤诊断和疗效预测的生物标志物。通过单一基因的泛癌分析,我们发现TUBA1B在多种肿瘤中表达显著上调,并与临床病理分期和预后有着明显的相关性,因此进一步探讨了其作为肿瘤标志物的临床价值和致肿瘤发生发展的可能机制。
TUBA1B是微管蛋白家族中的一员,通过与微管蛋白β构成二聚体参与微管形成,发挥细胞运动、分裂、信号传导、物质运输等功能。已有多篇文章报道TUBA1B与抗肿瘤药物作用相关,如针对尼替丁氯抗肝细胞癌的研究显示,尼替丁氯治疗前后组差异表达的circRNA可通过miRNA海绵作用竞争TUBA1B转录水平的表达,影响肝癌治疗[8];在尤文肉瘤治疗中,TUBA1B基因突变可导致小分子抑制剂产生耐药性。此外,TUBA1B在肝癌中可作为肿瘤相关骨髓细胞标记基因,预测患者预后和指示免疫浸润水平[20]。虽然目前已有多篇文章提示TUBA1B密切联系肿瘤,但在肿瘤起源和进展中的机制未阐明,且缺少泛癌水平的研究分析。
在本研究所涉及的33种肿瘤数据中,TUBA1B在22种肿瘤组织的表达均显著高于正常组织。遗憾的是,由于数据库中正常样本数量过少,TUBA1B在多形性成胶质细胞瘤、肺腺癌、直肠腺癌表达差异分析中的数据无统计学意义。由于数据库中肾上腺皮质癌、乳腺浸润癌等缺乏正常样本,TUBA1B无法在其中分析表达差异。随着肿瘤正常组织基因表达数据的积累,TUBA1B在泛癌水平的表达分析值得进一步探索。
随后我们对TUBA1B进行了肿瘤分期差异分析,以探讨其作为癌症早期筛查和疾病进展监测的潜力。结果提示14种肿瘤存在早期TUBA1B表达升高,提示TUBA1B具有作为早癌筛查指标的意义。此外,TUBA1B在3种肾癌晚期表达显著升高,提示TUBA1B可作为这3种肾脏肿瘤进展的检测标志物。生存预后分析结果表明TUBA1B高表达与多种肿瘤预后较差相关,综上反映TUBA1B可作为良好的肿瘤临床标志物。但预后分析中值得注意的是,GEPIA2与KM plotter预后分析结果存在较大差异,我们通过查阅数据库原始文献发现二者在cut-off值的选取模式是不同的。GEPIA2中cut-off值选用的是中位数,KM plotter中的cut-off值根据网站自身算法计算出差异最显著的cut-off值,两者选择的cut-off值未统一,导致高低表达TUBA1B的分群标准不一致,因此我们认为两个数据库在预后分析时所得出的结果差异较大可能与cut-off值的设定有关,此外数据来源不同可能也是两个数据库对预后分析差异的原因。
通过分析TUBA1B基因表达与肿瘤免疫细胞浸润水平的关系,我们发现TUBA1B表达与多种免疫细胞浸润呈现正相关,且既往研究表明TUBA1B的表达与NK细胞浸润相关[11]、可作为肿瘤相关骨髓细胞标记基因。此外,TUBA1B与免疫相关基因存在显著正相关性,其中与细胞因子、免疫检查点关系密切,提示TUBA1B可能成为免疫检查点抑制剂或细胞因子疗效的预测指标。
蛋白互作网络显示TUBB和KIF2C在多数癌症中与TUBA1B较强的正相关性,TUBB基因编码TUBB蛋白,与TUBA1B编码的微管蛋白α组装构成微管。研究表明TUBB与肿瘤发生发展关系密切,表达增加可导致肿瘤细胞对微管靶向药物的耐受性增加,且其高表达与肺腺癌的低生存率相关[21];针对髓母细胞瘤的研究发现,TUBB可作为靶点阻断肿瘤生长[22]。KIF2C编码有丝分裂着丝粒相关运动蛋白,与KIF18B构成复合物,在有丝分裂中发挥微管解聚活性[23]。KIF2C在多种肿瘤中表达升高,近期有关肝细胞癌的研究显示KIF2C可沟通Wnt/β-catenin与mTORC1信号通路,给两种明星通路交联提供分子基础,强调了KIF2C作为肝细胞癌治疗靶点的潜力[24]。除此之外,KIF2C还可通过调节DNA修复、微管稳定性、免疫细胞浸润[25]等参与肿瘤的发生发展[26]。综合以上文献回顾和本文蛋白互作结果,TUBA1B蛋白互作网络是癌症相关的主要网络之一,与肿瘤的耐药、转移、免疫、治疗靶点等有着密切的联系,进一步探究其在肿瘤中的功能可为癌症治疗提供新的线索。
总之,本研究表明TUBA1B可能成为多种肿瘤的独立预后因素,且其表达在不同分期的肿瘤中存在明显差异,提示TUBA1B可成为肿瘤早期筛查和晚期进展监测的重要标志物。此外,肿瘤免疫细胞浸润水平及免疫相关基因分析显示TUBA1B表达与肿瘤免疫多呈正相关,提示TUBA1B有望成为免疫治疗疗效的预测标志物或免疫治疗潜在靶点。
——水芹主要害虫识别与为害症状

