基于自监督深度学习的抗癌药物敏感性评估方法研究
柴 华,辜晓纯,苏咏纯,邓伟振,林俊淇
(佛山科学技术学院 数学与大数据学院,广东 佛山 528000)
1 相关工作
癌症是一种复杂的疾病,涉及基因和环境之间的一系列相互作用。大量的临床试验和科学研究发现,对于不同的癌症患者,相同药物导致的治疗结果不尽相同。因此,根据患者的差异性,判断癌症治疗药物的有效性,对实现癌症的精准治疗至关重要。随着计算机科学和生物医学研究的迅速发展,利用人工智能算法预测药物有效性因为其成本低、效率高等优势,对于筛选具有良好药效和安全性的药物候选物,缩短药物研发周期,降低药物研发成本等方面具有重要意义。
目前,大量的传统机器学习方法已经被用于这一领域。例如,Huang 等[1]使用基于正则化的逻辑回归模型预测抗癌药物的有效性,Dong 等[2]结合患者的基因表达,使用支持向量机预测药物敏感性,Riddick等[3]使用随机森林寻找对乳腺癌有效的潜在治疗药物。尽管这些方法在很多方面已经取得了一定的成果,但是高维的患者数据特征限制了此类方法的预测性能。深度学习在处理高维非线性数据方面展现了巨大优势。Theodore 使用深度神经网络评估不同药物的有效性和以及不同药物和患者生存之间的关系[4]。在此基础上,为了更好地学习不同数据之间的分布信息,Ladislav 使用变分自编码器构造癌症患者基因的低维表示,并将其输入传统机器学习分类器,用于预测药物的敏感性[5]。在Wang 等[6]的工作中,基于患者基因通路构建的图卷积神经网络被用来更好地提取患者基因数据中的隐藏信息。而在最近的研究中,JIA 等[7]提出使用生成式自编码器的药物有效性预测框架,通过对抗生成式策略自主学习其特定的数据空间分布特性,从而更好地区分患者间的差异性。尽管这些方法和传统的机器学习方法相比,取得了更加准确的药物敏感性预测表现,然而,有限的癌细胞小样本数据限制了此类方法性能的进一步提升。为了解决深度学习中的小样本学习困境,算法设计者提出了迁移学习[8],在模型训练中通过利用其他相似数据引入更多的信息。然而,迁移学习对数据的同质性有很强的假设,不同数据集的批次效应限制了模型的性能。
针对以上问题,本文设计了一种基于深度学习的抗癌药物敏感性预测框架(SSLGP),通过结合自监督学习减少高维基因数据中的冗余信息和噪声,从而获得低维数据的稳健表示,并将所获得的低维数据用于训练XGBoost 预测模型。与迁移学习不同的是,在最近的研究中,基于自监督学习的方法被设计用于应对小样本学习的挑战,而无需考虑不同数据集的同质性[9]。在我们的预测框架中,自监督学习策略通过让模型学习样本之间的相似性以及差异性来提取患者的基因特征。该框架在8 种药物数据集中进行了测试,结果证明获得的结果比以往常用的基线方法的AUC 平均高出>6.5%,实验证明了方法的先进性。
2 方法介绍
2.1 药物敏感性数据
本研究使用了8 个收集自癌症药物敏感性基因组学数据库(genomics of drug sensitivity in cancer,GDSC,https://www.cancerrxgene.org)的药物数据,包括药物敏感性数据以及癌细胞的mRNA 表达数据(Erlotinib 429 例,Irinotecan 453 例,Lapatinib 434 例,Nilotinib 434 例,Paclitaxel 434 例,PLX4720 452 例,Sorafenib 434 例,Topotecan 411 例)。对于mRNA 特征数据,首先删除缺失超过20%的特征及样本,之后使用中位数填补其缺失值。经log2 变换后的细胞系特征数据,其所有mRNA 表达数据均标准化为均值为零,标准差为1。
2.2 方法流程
方法流程如图1 所示。

图1 方法流程图
本研究设计了一种基于对比学习的癌症药物预测框架,以基因表达数据作为输入变量,半最大抑制浓度IC50 作为输出响应。本框架包含3 个步骤:1)采用SSLGP 自监督学习框架实现基因表达数据的低维特征提取;2)将学习到的低维特征表示与对应的代表药物敏感性的IC50 值合并为用于模型训练的数据集;3)构建XGBoost 分类预测模型。
2.3 基于自监督学习的深度学习框架
深度学习框架的关键思想是利用自监督学习有效的细胞表示,假设 x=(x1,xg)表示一个多组学特征列表,其中,g 表示特征数量。在自监督学习的数据增强阶段,样本数量n 将增加到2n。在每个批次中,将同一样本产生的两个相似点设置为正对,而将其他点视为负对。在自监督学习模块中,为了学习正负对的区别,给出的损失函数为
在队列中,xk代表第k 个变形的样本嵌入,xi和xj指的是来自同一个原始样本的一对正样本,温度系数τ 用于调节模型对负样本的区分能力。
SSLGP 自监督学习框架学习了一个局部平滑的非线性映射函数fθ,并使用了两个编码器:键编码器(fk)和查询编码器(fq)。键编码器的进化速度较慢,而查询编码器的进化速度较快。键编码器的参数由θk表示,查询编码器的参数由θq表示。使用常规的反向传播算法来更新查询编码器的参数。对于键编码器,使用动量方法来更新参数,以确保更新的方向保持一致。每次更新键编码器时,使用较小的步长,并通过线性组合之前的键编码器参数和新计算得到的查询编码器参数来保留先前步骤中的信息,这样做有助于保持模型更新的稳定性和一致性。θk和θq的关系式为
这里m∈[0,1)是动量系数。较大的m 使键编码器更新缓慢,而较小的m 则迫使键编码器变得非常像查询编码器,动量更新使得编码器网络进化顺利。模型训练完成后,查询编码器网络fq作为最终的生产网络,输出降维后的基因表达特征。
编码器神经网络采用两个全连接层构成,其中包括查询编码器和键编码器,两者共享相同的架构。第1 层包含1 024 个节点,而第2 层则包含128 个节点。线性变换后的非线性激活函数采用了ReLU 函数,定义为ReLU(x)=max(0,x)。在训练过程中,我们采用了Adam 优化器,并设置学习率为1,同时应用了余弦学习时间表。对配对神经网络进行了200 个epoch 的训练。在目标函数中,我们将温度τ 设为0.2,并设置动量系数m 为0.999。超参数的确定通过网格搜索和交叉验证的方式进行。
2.4 基于XGBoost 构建药物敏感性预测模型
通过深度学习框架重构细胞系的低维特征后,将其用于XGBoost 模型训练敏感性预测模型。XGBoost 算法是一种可扩展的用于树提升集成学习算法,它将多棵决策树的预测相结合,最后得出最终分数,即
其中,k 是树的数量,fk是函数空间F 的一个函数,F 是所有可能的分类回归树的集合。目标函数为
对每步训练目标函数二阶泰勒展开,即
记
可得
则目标函数最优解为
这是衡量树结构好坏的标准,值越小代表树结构越好。通常不可能枚举所有可能的树结构,而是使用从单叶开始并迭代地向树添加分支的贪婪算法。假设一个叶子分裂为两个叶子,则它的得分增加为
如果增益小于γ,则不再将此叶子分裂。在本研究中,从[2,8]中选取深度,从9 个值(0.01 和0.05*[1,8])中选取学习率。通过10 倍交叉验证(CV)最小化均方误差来优化这些参数。所有其他参数设置为“XGboost”包中的默认值。
2.5 性能评估
为了综合评估本框架的预测性能,基于相同的数据集,将本研究中的框架与其他常用的基于机器学习的癌症药物敏感性预测算法,包括逻辑回归(Logisitc)、随机森林(RF)、支持向量机(SVM)、DNN、Dr.VAE、GADRP 进行比较。通过比较不同方法在5 折交叉验证中的药物敏感性预测指标,包括曲线下剂量-反应面积(AUC)、准确率(ACC)、F1-score 等,来比较各模型的性能,以此评估本方法是否先进和稳健。
AUC 用于衡量二分类模型的性能,指正样品的预测值大于负样品的概率,也表示ROC 曲线下方的面积。ROC 曲线是以真阳性率(TPR)为纵轴、假阳性率(FPR)为横轴所绘制的曲线,因此AUC 的取值范围在0 到1 之间,越接近1 表示模型的性能越好,计算公式为
ACC 指分类模型的预测准确率,通过计算在总体样本中被正确预测的样本比例来衡量模型预测的准确程度。ACC 值越高,说明分类模型的性能越好,计算公式为
其中,TP 指模型预测为正例且实际也是正例,TN 指模型预测为负例且实际也是负例,FP 指模型预测为正例而实际是负例,FN 指模型预测为负例而实际是正例。
F1-score 是模型精确率(precision)和召回率(recall)的调和平均值,反映了分类模型对于正负样本的分类能力。F1-score 值越大,说明模型的精确率和召回率越趋于平衡,计算公式为
3 结果分析
3.1 SSLGP 评估方法的准确性
图2~4 分别给出了SSLGP 在5 倍交叉验证获得的预测准确度评估指数ACC、AUC 以及F1 得分,其在预测8 类药物数据库平均值分别为0.635、0.670 和0.647。图2 展示了8 种抗癌药物敏感性的ACC指数,其值范围为0.500~0.700,其中在Sorafenib 中最高,Paclitaxel 最低。

图2 SSLGP 在不同数据集上的ACC 得分
图3 展示了8 种抗癌药物敏感性的AUC 指数,这8 种药物的AUC 得分集中在0.600~0.700,在AUC 指标评估上表现相似,并且Erlotinib,Nilotinib 和Sorafenib 中的AUC 得分普遍高于0.650,在Erlotinib 中AUC 得分最高达到0.731。
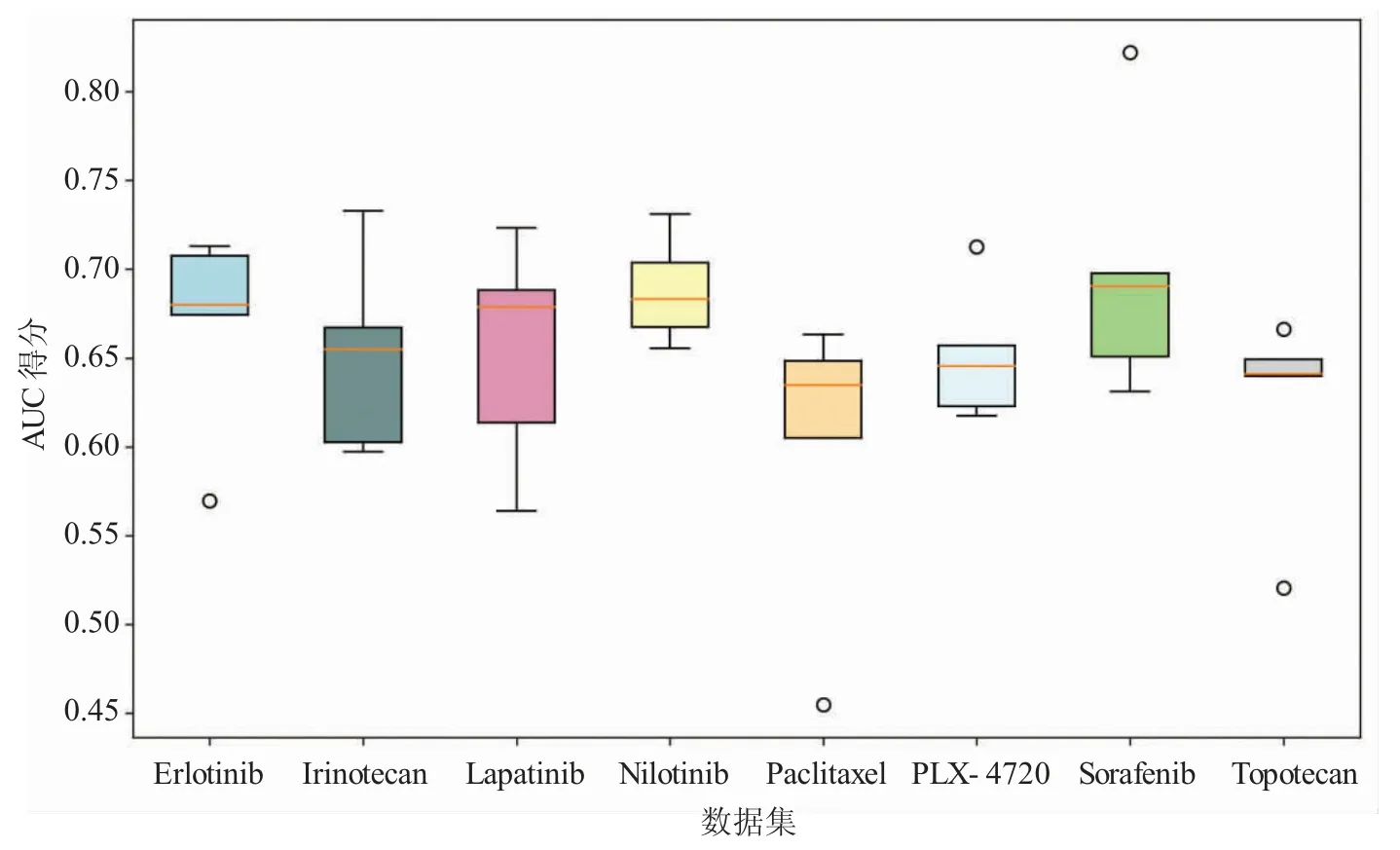
图3 SSLGP 在不同数据集上的AUC 得分
图4 展示了8 种抗癌药物敏感性的F1 得分,这些药物的F1 得分的中位数基本上分布在0.600 左右,其中在Sorafenib 中最高,在Paclitaxel 中最低,并且在Irinotecan、Paclitaxel 等数据集中有超出一半的F1 值高于中位数。结果表明,SSLGP 具有较好的准确性和稳健性。

图4 SSLGP 在不同数据集上的F1 得分
3.2 方法比较
表1 给出了不同算法在8 个抗癌药物数据集中5 折交叉验证得到的平均AUC 值,AUC 是ROC 曲线下的面积,AUC 值越大,说明该模型分类能力越好。如表1 所示,SSLGP 取得AUC 在0.597(Irinotecan)和0.731(Nilotinib)之间,平均值为0.670。与其他方法相比,SSLGP 平均提高了5.18%的AUC 指数值。在其他方法中,Logistic 的AUC 指数值最低,平均为0.606,其他两种传统方法RF 和SVM的AUC 指数值平均为0.616 和0.624,均低于现有的深度学习方法。在三种用于比较的深度学习方法中,Dr.VAE 优于DNN,但低于GADRP。这三种深度学习方法均低于我们提出的SSLGP。实验证明了本文方法的准确性以及稳健性。

表1 在8 个数据集上应用不同方法下的AUC 得分
4 结论
在以往的研究中,用于预测抗癌药物敏感性方法的性能受到了样本量的限制。为了解决这一问题,本文设计了一个基于自监督学习策略的深度学习的框架,通过构建正负样本对扩增数据,从而更好地提取高维小样本生物数据中的有效信息。与以往方法相比,SSLGP 方法在预后预测中表现出更好的性能,平均AUC 指数优于基线方法5.18%。
尽管该方法已经在预测药物敏感性方面取得了一定的成果,但仍然存在许多问题需要进一步探讨和解决。首先,现有研究已经发现药物的有效性和药物结构密切相关,而本研究没有用到其相关的信息。如果能引入此类的信息,可以帮助模型实现跨药物的敏感性预测。其次,本研究仅仅利用了相关细胞系的mRNA 表达数据,提供的有效信息优先。据过往研究,利用不同类型的多组学数据(如DNA 甲基化,拷贝数变异等)可以构建多元化的预测模型,较好地弥补其他高通量数据的不足,对于提升模型的预测精度具有一定的潜在作用。在未来的工作中,将考虑结合细胞系的不同组学特征,并引入药物相关的化合物结构信息,并对深度神经网络进行改良,从而进一步优化方法模型。

