利用荧光SSR 标记鉴定茶树自然杂交后代遗传背景
杨 军,游小妹,陈常颂
(福建省农业科学院 茶叶研究所/国家茶树改良中心福建分中心/农业部福建茶树及乌龙茶加工科学观测实验站,福州 350000)
茶树[Camelliasinensis(L.) O.Kuntze]属山茶科(Theaceae)山茶属(CamelliaL.),起源于中国西南地区,至今已有3 000多年的栽培历史,并先后传入日本、印度、斯里兰卡等50多个国家,目前已成为全世界三大饮料作物之一[1]。茶树是多年生、常绿木本、异花授粉植物,在遗传组成上高度异质杂合,在品种选育初期通过早期鉴定和准确选择目标性状提高育种效率尤为重要[2-3]。分子标记技术是茶树种质资源鉴定的重要手段之一[4-5]。余继忠等[6]比较‘福鼎大白茶’、云南大叶茶杂交后代遗传多样性与亲缘关系,显示以云南大叶茶为母本比以福鼎大白茶为母本遗传多样性更高;姜晓辉等[7]鉴定结果显示,5个云南白莺山茶树种质资源亚群间存在基因交流;陈立杰等[8]分析贵阳花溪古茶树资源,结果表明贵阳花溪古茶树资源由乔木型向灌木型进化;安红卫等[9]对贵州茶树种质资源进行鉴定,贵州茶树种质资源间亲缘关系、遗传距离与取样的海拔高度与密切相关;毛娟等[10]认为野生大理茶居群大雪山与栽培大理茶居群香竹箐和白莺山遗传距离相对较大,大理茶栽培居群和阿萨姆茶通过渐渗杂交,可能造成大理茶栽培居群的遗传背景较复杂;张明泽等[11]分析黔南野生茶树种质资源来源地间遗传距离与地理距离不存在显著相关性。以分子标记技术对多个茶树自然杂交后代进行亲缘关系、遗传多样性、亲本模拟分析的相关研究较少,利用分子标记辅助多个茶树自然杂交后代进行鉴定,对于明确茶树自然杂交后代遗传组成上的差异,加快茶树品种选育进程,具有重大的意义。
亲本模拟分析是了解自然杂交后代遗传背景的重要手段之一,主要用于分析亲本的遗传方式(花粉转播距离、自交不亲性)。常用的亲本模拟分析软件有PAPA、CERVUS、FAMOZ、和COLONY,其中李博等[12]对比PAPA、CERVUS、COLONY 亲本模拟鉴定的准确率,认为CERVUS 亲本模拟分析的准确率最高。近些年,在不同的植物与动物研究中都有大量利用CERVUS 软件亲本模拟分析,分别确定了马尾松25粒种子[13]、油松126粒种子[14]、桉树7个半同胞子代[15]、油橄榄29个子代[16]、圆口铜鱼297尾子一代[17]、许氏平鲉18个子代[18]、鳙42尾子代[19]、长鳍吻鮈96个子代[20]的亲本。茶树上利用CERVUS软件对茶树自然杂交后代进行模拟分析,并将其结果与遗传距离、遗传多样性分析相结合,研究茶树自然杂交后代遗传背景的报道较少。试验以福建省主要栽培品种为参照,对‘丹桂’、‘白鸡冠’、‘黄观音’自然杂交后代进行亲本模拟、亲缘关系、遗传多样性、基因流等研究,旨在对茶树自然杂交后代进行初步鉴定,确定自然杂交后代遗传背景,为茶树育种、茶树自然杂交后代筛选提供初步依据。
1 材料和方法
1.1 试验材料
供试材料包括福建主要栽培品种与茶树自然杂交后代种质资源一共116个,其中‘丹桂’(母本)自然杂交后代58个,‘白鸡冠’(母本)自然杂交后代15个,‘黄观音’(母本)自然杂交后代9个,福建省主要栽培品种34个(表1)。2021年10月在福建省农业科学院茶叶研究所采集116个材料的1芽2叶嫩叶后用液氮保存带回实验室,保存在-70 ℃冰箱中备用。

表1 116份供试材料的名称与来源Table 1 Name and origin of 116 materials used in this study
1.2 样品DNA提取与SSR扩增
1.2.1基因组DNA提取与检测
采用金基强等[21]的方法,利用CTAB法提取茶树嫩叶的总DNA。用1.0%的琼脂糖凝胶电泳进行茶树基因组分子量大小检测,用756-MC 型紫外分光光度计测定茶树基因组DNA 的纯度(OD260/280比值)。
1.2.2引物合成
参照文献[22]引物序列(上海Sangon 公司合成),引物信息见表2。

表2 24对 SSR 引物及其序列Table 2 Nucleotide sequences of the 24 primer pairs
1.2.3PCR扩增和产物鉴定
PCR 反应体系为:ddH2O 18.8 μL,10×Buffer 2.5 μL(Mg2+),dNTP(10 mmol/L)0.5 μL,上、下游引物(10 μmol/L)各0.5 μL,Taq 聚合酶 0.2 μL(0.5 U/μL),模板DNA 1.0 μL。PCR 反应于美国ABI-9600型扩增仪上进行,热循环程序为:94 ℃预变性4 min,使模板DNA 充分变性,然后进入下列温度循环:94 ℃变性45 s,不同温度条件退火60 s,72 ℃延伸75 s,重复35 个热循环;72 ℃延伸10 min,最后4 ℃保存。引物统一在反向引物(R)的5′段标记荧光(FAM/TAMRA),由上海百力格生物科技有限公司合成。扩增产物0.5 μL,GeneScanTM500 ROXTM0.5 μL,HiDi 9.0 μL,混匀后使用ABI公司3730XL进行毛细管电泳。
1.2.4数据处理
采用ABI公司的GeneMapper4.0软件,选择GeneScanTM500 ROXTM作为分子量标准,对每1个扩增出的条带记录大小。运用PopGen3.2软件计算等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He),Shannon's信息指数(I)和Nei's基因多样性指数(H);群体间遗传分化系数(Fst)、基因流(Nm),遗传相似度(GI)、遗传距离(GD)。使用PIC-CALC软件计算各引物的多态性信息含量指数(PIC)。运用NTSYSpc2.1计算材料间遗传距离,利用MEGA7.0进行UPGMA 聚类分析,并采用网站https://itol.embl.de/进行图片优化。
1.2.5亲本模拟分析
利用CERVUS 3.0软件对子代模拟亲本鉴定。对‘丹桂’、‘白鸡冠’和‘黄观音’自然杂交后代,34个候选亲本进行亲子鉴定分析,分别运行Simulation 程序与Parentage Analysis程序中的paternity子程序,模拟10 000次亲子鉴定,候选亲本检测率为80%,位点检测率为90%,基因型判别错误率为0.01,计算候选父本基因型似然对数比(LOD)值和95%置信水平,根据LOD 值大小判定亲子关系。
2 结果与分析
2.1 引物的多态性信息量
从表3看出,24对SSR 引物在供试样品中共获得157 个等位基因,如图1 为毛细管电泳对引物TM352扩增片段进行分析的部分结果,其中最多的为16个,最少为3个,平均为6.542;有效等位基因数(Ne)的变化范围是1.235~6.166,平均为2.959。本试验中PIC值变化范围在0.226~0.819,平均大于0.5(PIC值平均值=0.542),因此,24对引物整体多态性较高。Shannon's多样性指数(I)变化范围在0.398~2.073,平均值为1.182,说明供试样品具有较高的遗传多样性。

图1 引物TM352的部分毛细管电泳图谱Fig.1 Partial capillary electrophoresis patterns of TM352

表3 24对引物多样性信息Table 3 Diversity information of 24 SSR primers
2.2 茶树自然杂交后代亲缘关系与亲子模拟分析
从遗传距离聚类(图2)看出,供试材料主要分布在4个群体。群体1主要为‘丹桂’及其自然杂交后代;群体2主要为‘丹桂’、‘黄观音’自然杂交后代与福建省乌龙茶品种;群体3主要为‘白鸡冠’及其自然杂交后代;群体4主要为福建省绿茶品种,‘福鼎大毫茶’、‘歌乐茶’与‘福鼎大白茶’及其杂交后代,其中‘福鼎大毫茶’、‘歌乐茶’与‘福鼎大白茶’均为福鼎市当地品种;从4个群体茶树品种分布看出,茶树品种自然杂交后代大多与其母本亲缘关系较近,遗传基础受母本遗传信息影响较大。群体1中主要为‘丹桂’及其自然杂交后代,按亲缘关系分为3个亚群。将遗传距离聚类与亲子分析结果(表4)相结合可以看出,亚群a由‘丹桂’及其29个自然杂交后代与BJG5组成,亲子模拟母本(‘丹桂’)与真实母本(‘丹桂’)相符合为26个,准确率为89.6%,不符合的DG26、DG34模拟父本为‘肉桂’(‘肉桂’为‘丹桂’的母本),亲子模拟受祖母‘肉桂’遗传基础的影响;BJG5 真实母本(‘白鸡冠’)与模拟母本(‘白鸡冠’)相符合,模拟父本为‘丹桂’,BJG5 与‘丹桂’聚类在一起,说明BJG5遗传基础更偏向父本‘丹桂’;29个‘丹桂’自然杂交后代模拟父本中21个为绿茶品种,8个为乌龙茶,推测亚群a整体遗传基础与同母本‘丹桂’的自然杂交后代具有更多绿茶属性。
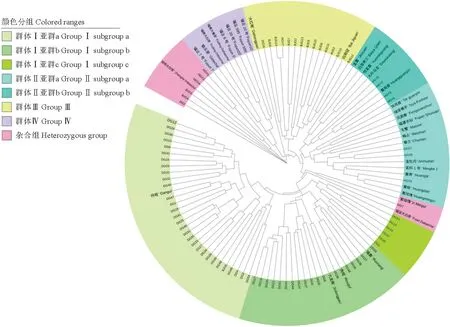
图2 遗传距离聚类图Fig.2 Cluster diagram of genetic distance

表4 82个茶树品种亲子分析结果Table 4 Parentage analysis result of the 82 tea varieties
亚群b由18个‘丹桂’自然杂交后代组成,亲子模拟母本(‘丹桂’)与真实母本(‘丹桂’)相符合为14个,准确率为77.8%;18个‘丹桂’自然杂交后代模拟父本中17个为乌龙茶品种,1个为绿茶品种,推测亚群b整体遗传基础与同母本‘丹桂’的自然杂交后代具有更多乌龙茶属性。亚群c由6个‘丹桂’自然杂交后代组成,亲子模拟母本(‘丹桂’)与真实母本(‘丹桂’)相符合为6个,准确率为100%,6个‘丹桂’自然杂交后代4个为乌龙茶品种,2个为绿茶品种。
群体2由5个‘黄观音’、2个‘丹桂’自然杂交后代与17个乌龙茶品种组成,按亲缘关系分为2个亚群。5个‘黄观音’自然杂交后代亲子模拟母本(‘黄观音’)与真实母本(‘黄观音’)相符合为4个,准确率为80.0%,4个与母本‘黄观音’亲缘关系较近,HGY8与‘黄旦’(‘黄观音’的父本)亲缘关系较近。DG29、DG52与乌龙茶品种‘春兰’亲缘关系较近,未与母本‘丹桂’聚类在一起,可能遗传基础受父本影响较大。
群体3由12个‘白鸡冠’自然杂交后代,2个乌龙茶品种,DG16、HGY7组成。12个‘白鸡冠’自然杂交后代亲子模拟母本(‘白鸡冠’)与真实母本(‘白鸡冠’)相符合为12个,准确率为100%,10个模拟父本为乌龙茶品种,且母本‘白鸡冠’聚类在一起,说明‘白鸡冠’自然杂交后代多具有乌龙茶品种特征,遗传基础主要来自于母本‘白鸡冠’。DG16、HGY7模拟父本均为‘白鸡冠’,与‘白鸡冠’亲缘关系较近,说明DG16、HGY7受模拟父本遗传基础影响较大。
2.3 茶树自然杂交后代遗传多样性分析
Hardy-Weinberg遗传偏离指数D与Shannon's遗传多样性指数是衡量群体结构的重要指标之一。从表5看出,按材料来源分析,‘丹桂’、‘白鸡冠’、‘黄观音’自然杂交后代遗传偏离指数D值均大于0,均表现为杂合子过剩现象,属于亲本单一,基本遗传信息主要来自与其自然杂交后代母本,‘丹桂’、‘白鸡冠’、‘黄观音’自然杂交后代间Shannon's遗传多样性指数相当,遗传多样性均低于福建参照品种群体;福建参照品种遗传偏离指数D值小于0,存在杂合子缺失现象,材料遗传来源复杂,Shannon's遗传多样性指数最较高,遗传多样性高。按遗传距离聚类群体分析,8个群体(包含亚群与杂合组)遗传偏离指数D值均大于0,说明8个群体遗传信息较为一致;其中群体3与杂合组Shannon's遗传多样性指数较高,群体3主要为‘白鸡冠’自然杂交后代,推测‘白鸡冠’父本的遗传多样性强于‘丹桂’、‘黄观音’的父本;杂合组的遗传来源丰富,遗传杂合度高。116个材料整体遗传偏离指数D值小于0,Shannon's遗传多样性指数为1.181,说明整体遗传多样性丰富。

表5 4个群体遗传多样性对比Table 5 Contrasting the genetic diversity of 4 groups
2.4 遗传距离与遗传相似度分析
从表6遗传距离看出,‘丹桂’自然杂交后代与福建主要栽培品种遗传距离数值最小,亲缘关系最近;3个自然杂交后代群体中,‘丹桂’与‘白鸡冠’的遗传距离最小,亲本模拟分析结果显示DG16的模拟父本为‘白鸡冠’,BJG1、BJG9的模拟父本为‘丹桂’,‘丹桂’与‘白鸡冠’自然杂交后代亲缘关系较近,受互为其模拟父本的影响。

表6 4个群体间遗传距离和遗传相似度Table 6 Genetic distance and genetic identity among 4 groups
群体2(亚群a)、群体2(亚群b)内主要材料为福建省乌龙茶品种,群体4主要材料为福建省绿茶品种;‘丹桂’自然杂交后代分布在群体1(亚群a、b、c)内,群体1(亚群b)与群体2(亚群a)、群体2(亚群b)的遗传相似度分别为0.899、0.822,高于群体1(亚群a)、群体1(亚群c),群体1(亚群b)优先进行乌龙茶品种筛选;群体1(亚群a)与群体4的遗传相似度为0.805,群体1(亚群a)优先进行绿茶品种筛选。
群体3主要为‘白鸡冠’自然杂交后代,除杂合组外,与其遗传相似度最高的为群体1(亚群b),亲缘关系较近(表7)。

表7 8个群体间遗传距离和遗传相似度Table 7 Genetic distance and genetic identity among 8 groups
2.5 遗传分化与基因流分析
参考Wright等[23]对遗传分化系数Fst值大小对种群的分化程度高低进行评估,‘黄观音’自然杂交后代与‘丹桂’、‘白鸡冠’自然杂交后代的Fst值分别为0.065、0.054,达到中度分化,其他组合间几乎无分化(表8)。从表9中可以看出,群体间Fst值范围0.043~0.144之间,群体1(亚群b)与群体2(亚群a)小于0.05,两群体几乎无分化外,其他群体间均达到了中度分化,因此,按遗传距离分类群体能使群体分化更为显著,群体代表的遗传信息特征更为明显。群体间的基因流大小是衡量群体间遗传差异的重要指标,Nm>4,说明群体间基因流充分;Nm<1,说明群体间可能发生遗传漂变,基因交流较少[24]。本试验中‘丹桂’、‘白鸡冠’、‘黄观音’自然杂交后代与福建主要栽培品种的基因流分别为8.331、5.806、6.706,说明‘丹桂’自然杂交后代与福建主要栽培品种基因交流最为充分,‘白鸡冠’自然杂交后代与福建主要栽培品种基因交流为最低。按遗传距离分类8个群体,群体间Nm值为1.476~5.480,群体1(亚群c)与群体4 间基因交流值为1.476,基因流最少,按遗传距离分类群体能使群体基因交流降低。

表8 4个群体间遗传分化系数和基因流Table 8 Genetic differentiation index and gene flow among 4 groups

表9 8个群体间遗传分化系数和基因流Table 9 Genetic differentiation index and gene flow among 8 groups
2.6 分子方差分析
从表10中可以看出,供试材料取样来源与遗传距离聚类分类的群体内遗传变异分别为88.52%、79.53%,群体内遗传变异主要来自于材料间个体差异。遗传距离聚类分类与材料取样来源分类相比,群体内个体间遗传变异变小,群体间遗传变异增加。说明遗传距离聚类分类比自然杂交后代群体来源分类,在一定程度降低自然杂交后代未知父本带来遗传的影响,能够更清晰、准确掌握自然杂交后代的遗传背景。
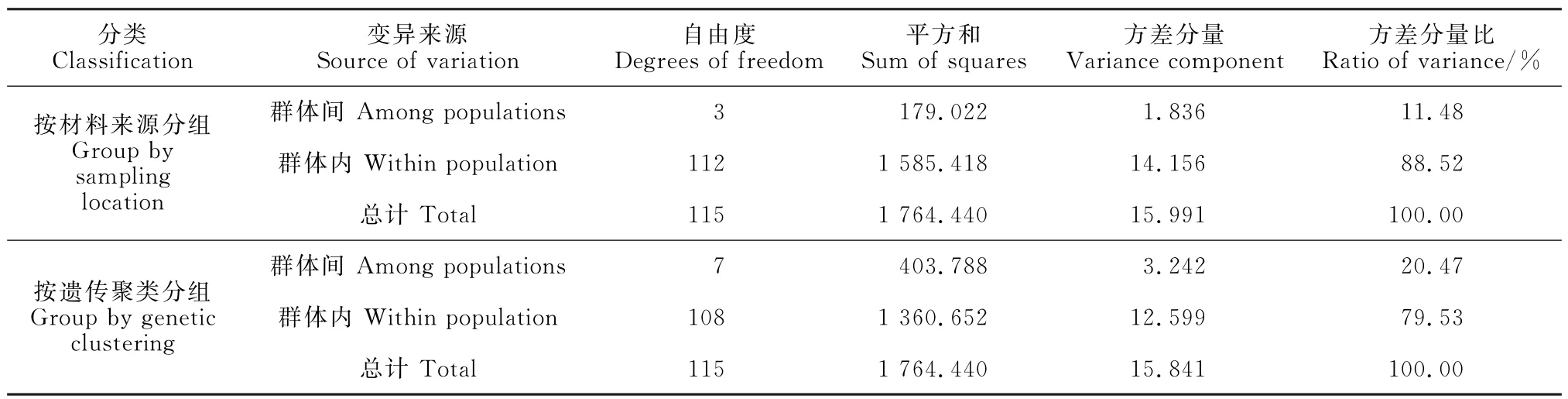
表10 群体内和群体间AMOVA分析结果Table 10 AMOVA results for within and among population variations
3 讨论
3.1 茶树自然杂交后代遗传背景分析
亲缘关系分析是鉴定种质资源遗传背景的重要手段之一。郭俊等[25]对32份油梨资源进行亲缘关系分析,亲缘关系与地理分布有一定的关系;王鑫等[26]明确了17个类群的野生铁线莲亲缘关系,部分铁线莲形态上十分相似,亲缘关系较远,尤其是槭叶铁线莲与其他16种亲缘关系较远;王留彬等[27]鉴定表明广西南渡镇茶树群体与六堡镇茶树群体、栽培种间亲缘关系较远,材料间亲缘关系远近与栽培种、野生种关系密切。宋艳画等[28]分析没有血缘记录的黑山羊亲缘关系,依据利用CERVUS 软件以父权分为11个群体,得出整个群体的亲缘关系,为群体的选育选配提供参考。试验按照材料间遗传距离进行分类,显示多数自然杂交后代都与其母本亲缘关系较近。‘丹桂’自然杂交后代多数与‘丹桂’聚类在群体1,‘丹桂’的自然杂交后代亲缘关系都较为接近;群体1中的3个亚群,按模拟父本(父权)分类群体1亚群a的模拟父本主要为绿茶品种,可以优先进行早生、高香绿茶鉴定筛选;群体1亚群b的模拟父本主要为乌龙茶品种,可以优先进行高香乌龙茶筛选。说明同一母本遗传背景情况下,亲缘关系分析与按模拟亲本(父权)分群结果具有相关性,且亲本模拟分析比亲缘关系分析能更深刻了解供试材料的遗传背景,有助于利于分子标记进行辅助育种选择。
3.2 茶树自然杂交后代遗传多样性分析
Shannon's多样性指数是衡量植物遗传多样性重要指标之一,在茶树种质资源鉴定上进行大量研究。杨阳等[29]利用EST-SSR 标记研究黄金茶群体遗传多样性,结果显示湖南黄金茶38 个材料的Shannon's多样性指数为0.550;王松琳等[30]采用SSR 标记对16个白化和黄化茶树品种遗传多样性分析,Shannon's信息指数0.790;杨军等[31]对‘金牡丹’、‘悦茗香’、‘紫玫瑰’自然杂交后代进行遗传多样性分析,结果显示Shannon's 信息指数分别0.899、1.041、1.010。彭靖茹等[32]分析14 份广西德保县和隆林县野生古茶树遗传多样性,茶树种质资源Shannon's多样性指数0.970;周萌等[33]对122个云南野生茶树种质资源进行遗传多样性分析,Shannon's多样性指数为0.880;姜晓辉等[34]分析广东曲江罗坑茶与丰顺马图茶的Shannon's多样性指数分别为1.217与1.228;本试验中,‘丹桂’、‘黄观音’、‘白鸡冠’3个群体的Shannon's多样性指数分别为1.029、1.035、1.044,与前人研究相比,高于黄金茶群体、浙江白化和黄化茶、云南野生茶树,与‘金牡丹’、‘悦茗香’、‘紫玫瑰’自然杂交后代相当,低于广东曲江罗坑茶与丰顺马图茶,初步说明‘丹桂’、‘黄观音’和‘白鸡冠’3个群体遗传多样性丰富。

