基于邻近切片注意力融合的直肠癌分割网络
兰冬雷,王晓东,姚宇,王辛,周继陶
基于邻近切片注意力融合的直肠癌分割网络
兰冬雷1,2*,王晓东1,2,姚宇1,2,王辛3,周继陶3
(1.中国科学院 成都计算机应用研究所,成都 610213; 2.中国科学院大学,北京 100049; 3.四川大学华西医院 腹部肿瘤科,成都 610041)(∗通信作者电子邮箱461779062@qq.com)
针对直肠癌目标靶区在磁共振成像(MRI)图像的大小、形状、纹理和边界清晰程度不同等问题,为了克服患者之间的个体差异性并提高分割精度,提出一种基于邻近切片注意力融合的直肠癌分割网络(ASAF-Net)。首先,使用高分辨率网络(HRNet)作为主干网络,并在特征提取过程始终保持高分辨率特征表示,以减少语义信息和空间位置信息的损失;其次,通过邻近切片注意力融合(ASAF)模块融合并增强相邻切片之间的多尺度上下文语义信息,使网络能够学习相邻切片之间的空间特征;最后,在解码网络使用全卷积网络(FCN)和空洞空间金字塔池化(ASPP)分割头协同训练,并通过添加相邻切片间的一致性约束作为辅助损失缓解训练过程中出现的相邻切片差异过大的问题。实验结果表明,与HRNet相比,ASAF-Net在平均交并比(IoU)、平均Dice相似系数(DSC)指标上分别提升了1.68和1.26个百分点,平均95%豪斯多夫距离(HD)降低了0.91 mm。同时,ASAF-Net在直肠癌MRI图像多目标靶区的内部填充和边界预测方面均能实现更好的分割效果,有助于提升医生在临床辅助诊断中的效率。
直肠癌;图像分割;注意力机制;特征融合;深度学习
0 引言
世界卫生组织国际癌症研究机构(International Agency for Research on Cancer, IARC)发布的2020全球癌症统计数据显示,结直肠癌的发病率位居全球第三,它的发病率和死亡率均保持上升趋势[1]。2020年我国结直肠癌的发病率和死亡率分别位居我国恶性肿瘤的第3位和第5位[2]。结直肠癌已经成为常见的严重危害国人身体健康的恶性肿瘤之一,因此,结直肠癌的筛查和治疗是一个迫切的需求。
针对肿瘤距肛门小于等于10 cm的直肠癌患者,医疗工作者一般参考肿瘤大小、癌细胞转移到直肠系膜内/外淋巴结的情况和直肠系膜是否被侵犯等信息对患者进行TNM(Tumor Node Metastasis)分期诊断[3]。根据直肠癌的诊断标准,医疗工作者首先从医学影像上确定肿瘤的位置,其次通过判断肿瘤对直肠壁固有肌层和直肠系膜筋膜的浸润程度诊断直肠癌分期;因此,准确分割肿瘤和直肠系膜等目标区域对指导直肠癌TNM分期和制定个体化治疗方案至关重要。
磁共振成像(Magnetic Resonance Imaging, MRI)对人体软组织具备成像清晰的特点,是膀胱、直肠和子宫等人体腹部器官较优的检查方案,且选择不同的成像参数能提供丰富的信息。基于MRI的辅助诊断分析方法具备无创、非侵入性、获取成本相对较低和对人体无辐射损伤等优点,对疾病的诊断、分析和预后等方面具有重要的研究价值[4]。直肠癌的人工分割需要多序列图像和临床信息准确定位肿瘤,目前临床上大多采用纯手工或半工的方式勾画,不仅复杂、耗时,且病灶区域的勾画受医生专业水平影响较大,分割标注获取成本高。
基于深度学习的自动分割技术大幅减少了医疗工作者的工作量,并在一定程度上减小因个人偏见和临床经验造成的分割误差;然而,现有方法集中于肺部分割[5-6]、肝脏分割[7]、大脑分割[8]和视网膜血管分割[9]等,直肠癌的自动定位和分割方面研究较少。相较于其他类型的癌症,由于直肠是一个中空的内脏,所以直肠壁和肿瘤周围的背景在不同的切片上变化较大,肿瘤和直肠壁之间的对比度低,难以准确定位和分割。目前的研究集中在单目标分割任务上,如肿瘤分割[10]和淋巴结分割[11]等,单目标分割难以满足直肠癌TNM分期诊断需求。
针对目前直肠癌分割问题,本文设计了一种基于邻近切片注意力融合的直肠癌分割网络(Adjacent Slice Attention Fusion Network for rectal cancer segmentation, ASAF-Net),以实现直肠癌MRI中肿瘤、直肠系膜和外淋巴结区的多目标分割。ASAF-Net在编码网络和解码网络之间引入一个邻近切片注意力融合(Adjacent Slice Attention Fusion, ASAF)模块,利用注意力机制融合和增强相邻切片之间的上下文信息,将三维(Three-Dimensional, 3D)体积信息融合到二维(Two-Dimensional, 2D)卷积神经网络;在解码网络中引入相邻切片间的一致性约束作为辅助损失,缓解训练过程中出现的相邻切片差异过大的问题。实验结果表明,ASAF-Net与其他主流的分割网络相比具有较稳健的性能提升。
1 相关工作
1.1 深度学习分割方法
语义分割是医学图像分析中的一项重要技术手段。随着数据量的增加和算力的提升,深度学习依靠强大的特征提取能力,在图像处理(分割、分类、检测等)任务中取得了突破性的成果,并逐渐开始在图像处理领域占据主导地位。Long等[12]提出了全卷积网络(Fully Convolutional Network, FCN),FCN将经典的卷积神经网络中的全连接层全部替换为卷积层,利用反卷积和跳跃连接对特征图进行上采样恢复至与输入图像相同的尺寸,帮助网络从抽象的特征中挖掘每个像素所属的类别,将图像级别的分类进一步延伸至像素级别的分类。Ronneberger等[13]提出的U-Net与FCN都是编码器-解码器(Encoder-Decoder)结构,U-Net采用更巧妙的解码器设计和跳跃连接的叠操作(Concatenation),更好地融合多尺度特征信息。包含压缩路径和扩展路径的对称U形结构网络由此成为了医学图像分割的经典方法。DeepLab网络[14]利用空洞卷积在不增加参数量的同时增大了感受野,并且在解码器中设计了空洞空间金字塔池化(Atrous Spatial Pyramid Pooling, ASPP)模块。ASPP是一种特殊的金字塔池化模块[15],通过并行设计多种空洞率的空洞卷积特征提取路径,再将它们进行融合,以挖掘多感受野特征信息,增强网络识别同一目标不同尺度的能力。Wang等[16]从维持高分辨率特征的角度出发,提出了高分辨率网络(High Resolution Network, HRNet),极大缓解了上下采样过程中导致的大量的有效信息丢失问题。HRNet在整个编码-解码过程中维持高分辨率特征,并通过设置并行的多分辨率分支不断地进行多分辨率分支之间的信息交互,减少语义信息和位置信息的损耗,使网络更适合位置敏感的语义分割任务。
1.2 注意力机制
注意力机制不存在严格的数学定义,因此有大量变种和不同的实现方式。本质上,注意力机制的核心是让网络专注于“重要”的信息,忽略“相对不重要”的信息,提升网络的表示能力。注意力机制在自然语言处理中主要用于解决机器翻译中的长序列问题[17]。Mnih等[18]提出的RAM(Recurrent Attention Model)在循环神经网络中引入注意力机制分类图像,首次将注意力机制应用于计算机视觉领域。Hu等[19]提出的SENet(Squeeze-and-Excitation Network)通过建模特征通道,强化或弱化相关特征以提升网络的准确率。Woo等[20]和Wang等[21]分别提出了CBAM(Convolutional Block Attention Module)和ECA(Efficient Channel Attention module),隐式和自适应地预测潜在的关键特征的代表性模块。自注意力由Vaswani等[22]提出,Wang等[23]将自注意力引入计算机视觉领域,提出的非局部网络可以捕捉两个位置之间的远距离依赖关系,在视频分类和目标检测任务上取得了成功。基于注意力机制的方法高效、简单、通用,在多数任务场景下都能显著地提高网络的速度、结果质量,增强泛化能力。本文利用注意力设计多尺度特征融合模块,提升多尺度之间的融合效率,改善分割结果。
1.3 直肠癌分割
目前的直肠癌分割研究大多集中在单目标分割任务上。Trebeschi等[24]利用卷积神经网络自动定位和分割140名局部晚期直肠癌患者的MRI数据,在独立的验证数据集上单目标Dice相似系数(Dice Similarity Coefficient, DSC)为68%;Rao等[25]利用通道拼接和空洞卷积改进U-Net,在DSC指标上提高了2.7个百分点;Meng等[26]基于592例数据集,提出了带边界约束的掩码分割网络(Mask Segmentation with Boundary Constraints Network,MSBC-Net),MSBC-Net的参数量是U-Net的4倍,平均DSC为80.1%,对于直肠系膜包裹的直肠壁的分割DSC为75.6%。直肠癌的单目标分割结果对临床诊断的辅助效果有限,由于不同目标之间存在较大差异性,多目标分割通常面临各目标分割精度方差较大、目标误分割等问题,因此直肠癌的多目标分割是一项具有挑战性的任务。本文从邻近切片的多尺度特征融合的角度出发,通过注意力机制挖掘空间上下信息并融合到多尺度特征中,改善网络的多目标分割性能。
2 ASAF‑Net
2.1 网络整体架构设计
ASAF-Net采用编码器-解码器结构,由主干网络(backbone)、颈部网络(neck)和解码网络(decoder)这3个组件构成。主干网络采用卷积网络堆叠,用于特征提取;颈部网络对相邻切片的深层高级语义特征进行注意力融合和增强,学习相邻切片之间的一致性上下文信息;解码网络用于处理颈部网络输出的特征图,对图像的每个像素进行分类,得到最终的分割结果。ASAF-Net的整体架构如图1所示。
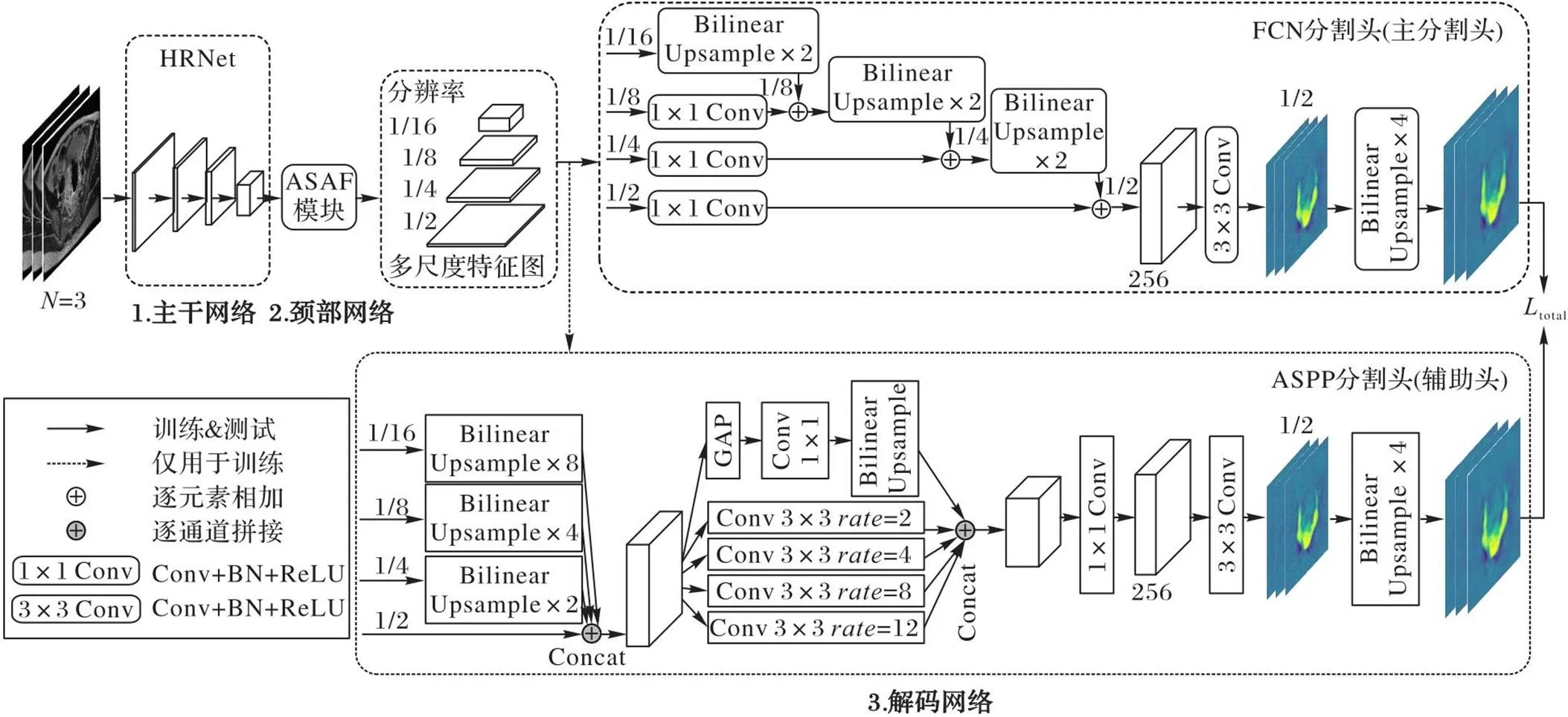
图1 ASAF-Net整体架构
2.2 主干网络
以前的一些先进方法如SegNet[27]、DeconvNet[28]、U-Net[13]和SimpleBaseline[29]等都采用高分辨率恢复过程(如图2(a)所示),先降低分辨率,再从低分辨率中提高表示的分辨率。语义分割任务依赖高分辨率的特征表示,而高分辨恢复过程不可避免地会造成上下文信息的丢失,不利于位置敏感的语义分割任务。相较于常用的医学图像分割网络U-Net,本文采用HRNet作为特征提取的主干网络,参数量和计算量更少。HRNet在整个特征提取过程始终保持高分辨率的特征表示,输出的高分辨率特征保持了强语义信息,并且学到的语义表征在空间上也更精确[16]。高分辨率保持过程如图2(b)所示。
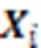
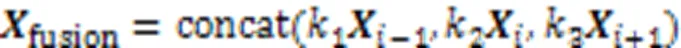
其中:concat()表示以通道维度进行拼接;、和表示权重超参数,通过几组不同经验值的实验对比,分别设置为0.8、1.0和0.8。以相同模式读取N张相邻图像作为网络的输入,图1以N=3为例,主干网络进行特征提取后,输入颈部网络进行进一步特征融合操作。
2.3 颈部网络
颈部网络利用注意力机制融合和增强相邻切片之间多尺度上下文信息。图3展示了同一病人的3张连续MRI和它们对应的多目标标注结果。
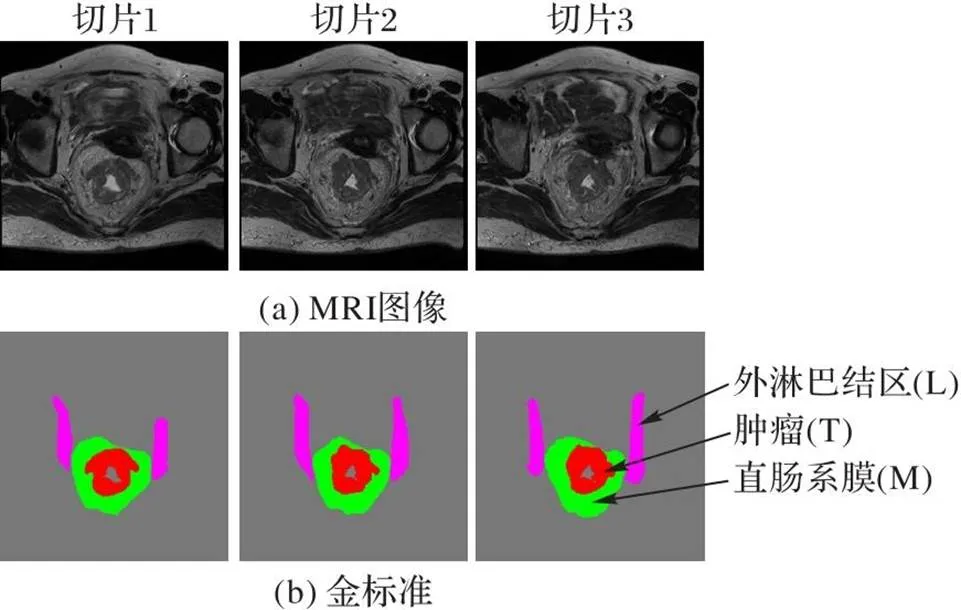
图3 同一病人的3张邻近MRI切片及其对应分割标注
由图3可见,同一病例的相邻切片之间的目标区域在空间位置、形态、大小和对比度等方面呈现一定的相似性。挖掘和利用相邻切片之间的上下文信息融合至二维分割网络是一种利用先验信息提升分割精度的方法,这种先验信息融合操作可以简单分为两类:一类是先利用主干网络提取特征,再进行融合,如Zhang等[30]将相邻3张切片提取的特征利用空间注意力融合改善分割结果;另一类在主干网络提取特征的同时进行融合,如Lv等[31]利用残差Inception模块替换U-Net的基本模块,将相邻切片上下文信息融合作用于整个特征提取阶段。本文采用先提取特征后融合的方式,不同于文献[30],本文选用通道注意力进行融合,在融合时有效利用了多尺度特征。
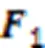
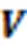
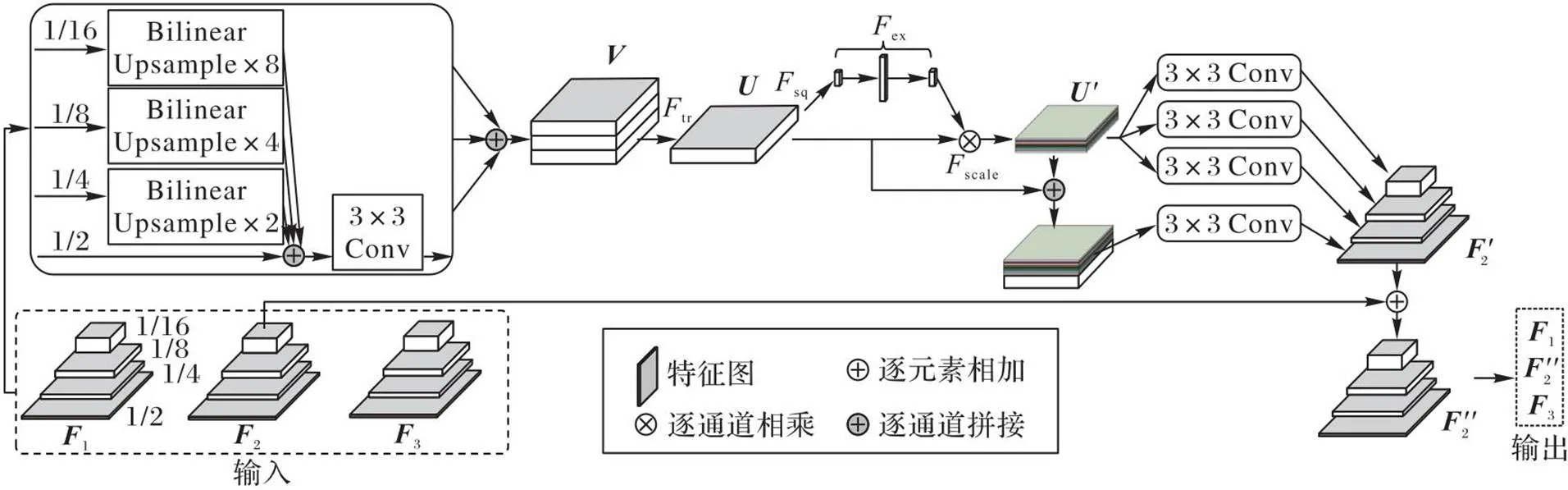
图4 ASAF模块
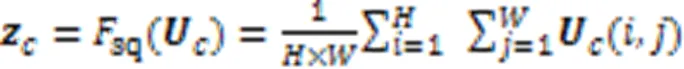
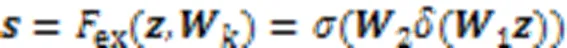
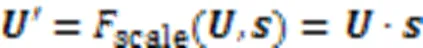
2.4 解码网络
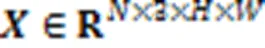
2.5 损失函数
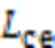
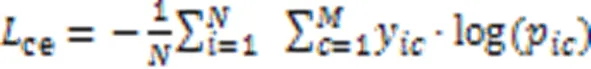
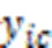
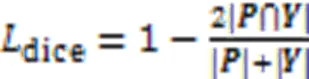
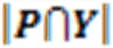
训练时,网络的损失为主分割头FCN的损失与辅助分割头的ASPP的损失加权和。如式(7)所示:
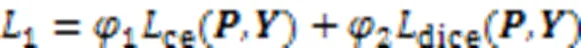
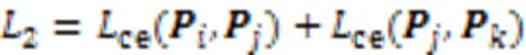
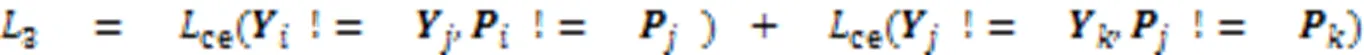
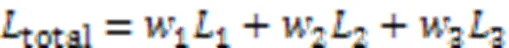
3 实验与结果分析
3.1 实验环境与数据集
本文的实验环境如下:Windows 10操作系统,CPU 13th Gen Intel Core i7-13700K,GPU NVIDIA GeForce RTX 4090,显存大小24 GB。深度学习框架为PyTorch 1.9.0,Python 3.8.8。
实验数据为四川大学华西医院腹部肿瘤科2015至2020年间收治的127例确诊Ⅱ/Ⅲ期直肠癌患者MRI,其中每名患者的MRI中含有17~55张切片。每例直肠癌患者都是由一名具有5年诊断经验的低年资放疗医师勾画,并由另一名具有10年诊断经验的高年资放疗医师校正。
127例病例转换为2D切片后共有2 917张,在划分数据集时,以病人为单位进行划分,即不能出现单个病人的一部分2D切片出现在训练集,而另一部分2D切片又出现在验证集中的情况。将127例原始数据划分为5组(5-Flod),每个子集分别做一次验证集,其余的4组子集作为训练集,将训练后得到的5个最优网络的平均结果表示网络的最终性能。五折交叉验证有效利用了有限的数据,更好地验证网络的泛化性能。
3.2 实验参数设置
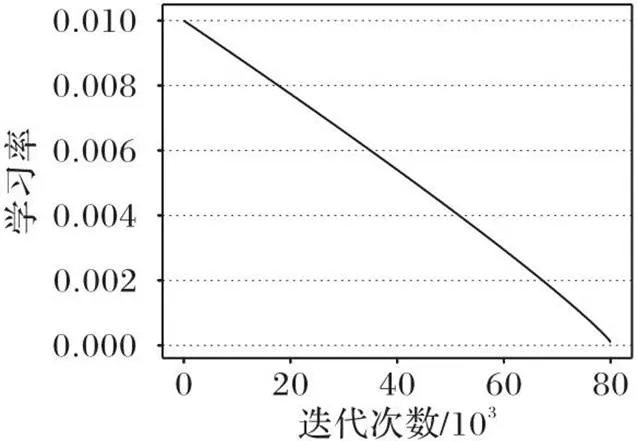
图5 学习率随迭代次数增加的衰减
3.3 评价指标
为了定量评估不同网络的分割性能,本文采用交并比(Intersection over Union, IoU)、DSC和95%豪斯多夫距离(Hausdorff Distance, HD)评价指标,其中IoU也被称为Jaccard相似系数(Jaccard Similarity Coefficient, JSC)。IoU、DSC和95%HD指标旨在将预测分割图与标注图从内部填充区域和边界区域两个角度结合进行综合评估,更有利于语义分割任务,以获取更精确的分割性能。
3.4 结果与分析
为了评估本文网络的有效性,将本文网络与FCN、U-Net、DeepLabV3和HRNetV2对同一分割数据集进行分割对比。分割结果如表1、2所示。由表1可得,本文网络的IoU、DSC和95%HD均值高于其他网络,IoU、DSC和95%HD均值分别取得63.07%、75.96%和25.46 mm。表2则分别列出了每一目标的分割精度,结果显示,HRNetV2在直肠系膜的分割上取得最优,肿瘤和外淋巴结区的表现均不如本文网络。特别地,本文方法在外淋巴结区的分割上,相较于HRNetV2,IoU和DSC分别提升了4.65和3.38个百分点,95%HD降低了3.49 mm。实验结果表明,本文的ASAF模块融合了邻近切片上下文信息,能够带来较为显著的性能提升。
图6为不同分割网络的可视化对比,第一组和第二组为相邻3张切片结果,第三组为独立的两张不相邻切片。对于第一组中相邻切片的不同网络的预测结果,FCN和DeepLabV3只在第一张和第三张切片上分割了肿瘤,从结果上几乎无法看出相邻切片之间存在相关性;U-Net、DeepLabV3和HRNetV2对肿瘤靶区的分割结果都较差,并且还错误地分割了外淋巴结区,特别地,HRNetV2在直肠系膜的分割上的轮廓最接近金标准;本文网络在肿瘤分割上取得了更好的结果,在轮廓上也更接近金标准。对于第二组相邻3张切片结果图,FCN的表现最差,没有正确分割外淋巴结区;U-Net成功预测了所有目标,但肿瘤轮廓与金标准差别较大,且所预测的直肠系膜边界轮廓尖锐,不平滑;DeepLabV3和HRNetV2在外淋巴结区和直肠系膜的预测上,边界轮廓得到较大改善,但对肿瘤的分割预测依然较差。对于第三组中的第1张切片,HRNetV2对直肠系膜的预测结果最接近金标准,本文网络次之;对于第三组中的第2张切片,FCN、U-Net和DeepLabV3对肿瘤的预测与金标准差异较大,HRNetV2则没能分割肿瘤。图6表明了本文网络在直肠癌的多目标分割任务上有着更稳健的分割结果,取得了多目标分割较权衡的结果。
表1不同方法的分割结果对比
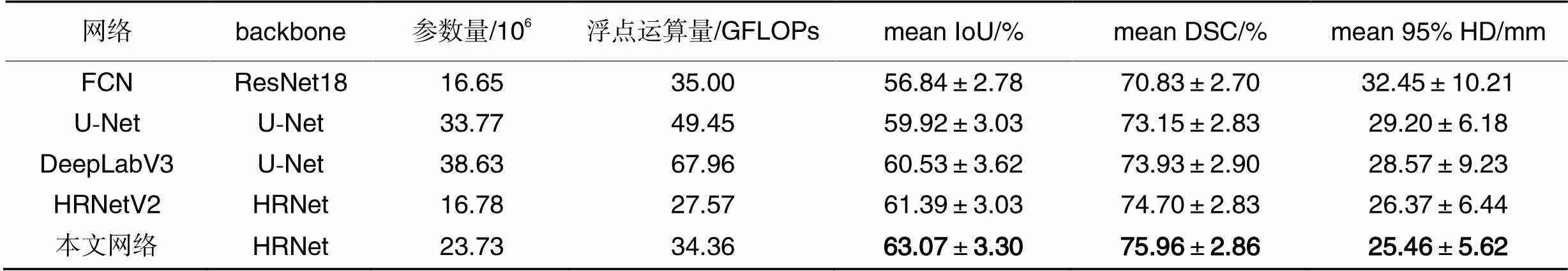
Tab.1 Comparison of segmentation results of different networks
表2不同方法的多目标分割结果对比

Tab.2 Comparison of multi-object segmentation results of different networks
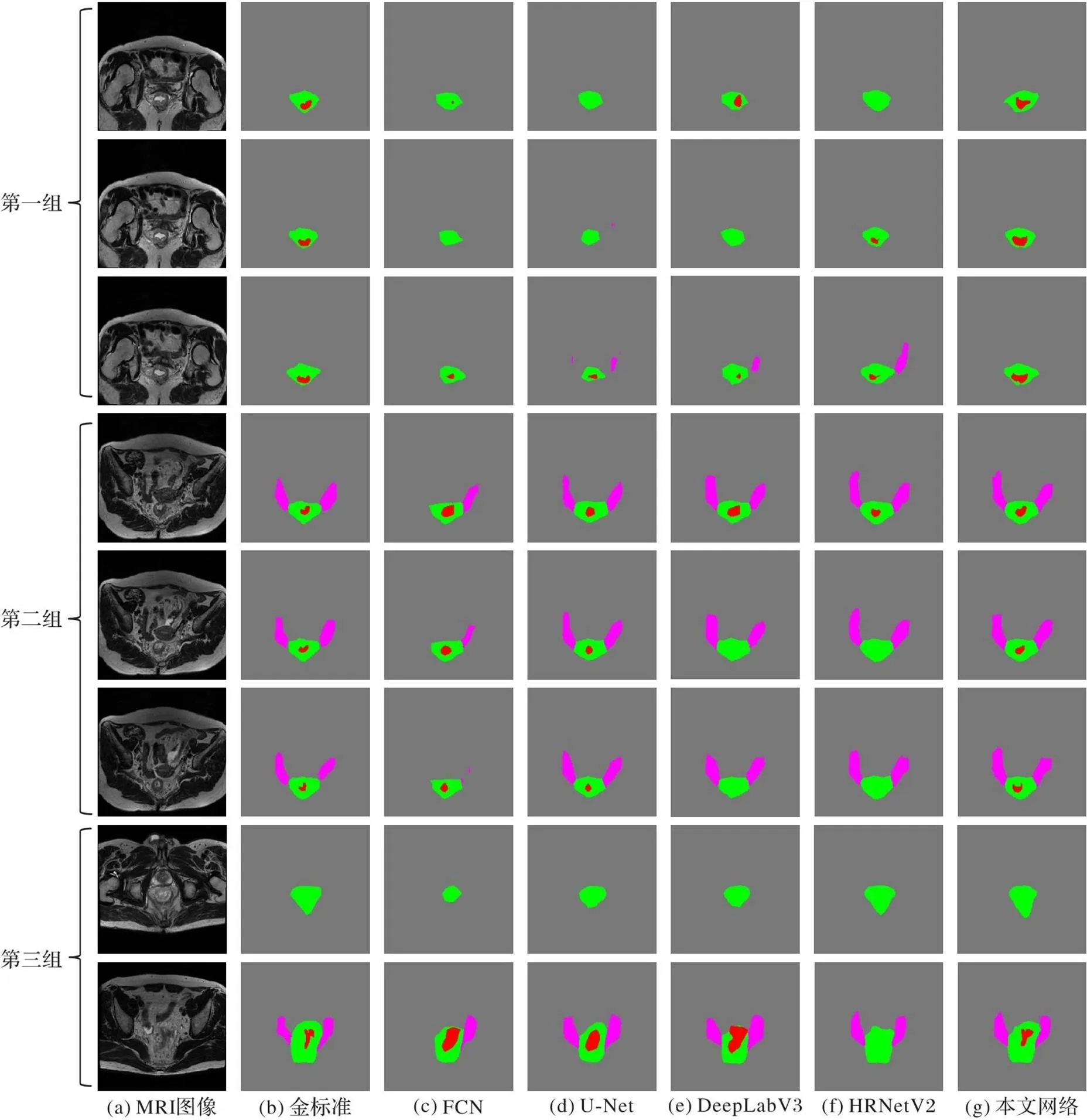
图6 不同网络分割结果的可视化对比
不同分割方法的参数量、计算量见表1。本文网络在HRNetV2基础上,增加了ASPP辅助分割头、ASAF模块和相邻切片一致性约束,参数量和计算量分别增加了6.95×106和6.67 GFLOPs,多目标分割的平均IoU和平均DSC指标分别提高了1.68和1.26个百分点,平均95%HD降低了0.91 mm。
3.5 消融实验
为了验证本文网络的ASAF模块的有效性,进行了消融实验,结果如表3和图7所示。
图7中,方法①作为基线方法(Baseline),它在HRNetV2的基础上增加ASPP辅助分割头。相较于HRNetV2(表1),基线方法的平均IoU提升从61.39%提升到了61.83%,平均DSC从74.70%提升到了74.89%。方法②在方法①的基础上加入了ASAF模块后,平均IoU、平均DSC和平均95%HD指标均优于基线方法,平均IoU和平均DSC分别提升了1.11和0.58个百分点,平均95%HD降低了0.55 mm。方法③引入了约束相邻两张切片之间的一致性损失,缓解优化过程中出现的相邻切片差异大的问题,实验结果表明,一致性损失约束能够给分割结果带来一定的性能提升,平均IoU和平均DSC分别提升了0.28和0.16个百分点,平均95%HD降低了0.17 mm。一致性损失带来的性能提升较为有限,且在实验时需要不断地调参寻找最优的损失权重,泛化能力差,推广到其他分割任务难度和代价高。方法④同时加入ASAF模块和一致性损失约束,取得了最佳分割结果,相较于方法①,平均IoU和平均DSC分别提升了1.24和1.07个百分点,平均95%HD降低了0.96 mm。表3中的实验结果说明本文网络对于改善分割结果是有利的。
图7为消融实验中不同网络对3张直肠癌MRI分割结果的可视化对比。从第1张切片不同网络的分割结果可以看出,图7(f)加上ASAF模块后,肿瘤分割得到了明显改善(箭头所指目标),并且ASAF模块和一致性损失都提升了外淋巴结区的分割精度,外淋巴结区更接近金标准(圆圈圈出的目标)。第2张切片分割结果的外淋巴结区也同样得到了改善。在第3张切片不同网络的结果中,方法④通过结合ASAF模块和一致性损失,成功分割了肿瘤目标(箭头所指)。
最后,为了验证ASAF模块的泛化能力,本文将它与不同的主干网络进行消融实验,实验结果如表4所示。方法⑤和⑦分别为ResNet和U-Net分割网络,方法⑥⑧则分别在⑤⑦基础上加上ASAF模块。表4实验结果表明,ASAF模块作用于其他基干网络也能带来一定的性能提升。

表3 网络结构消融实验结果对比

图7 消融实验分割结果对比
表4ASAF模块泛化能力的消融实验结果
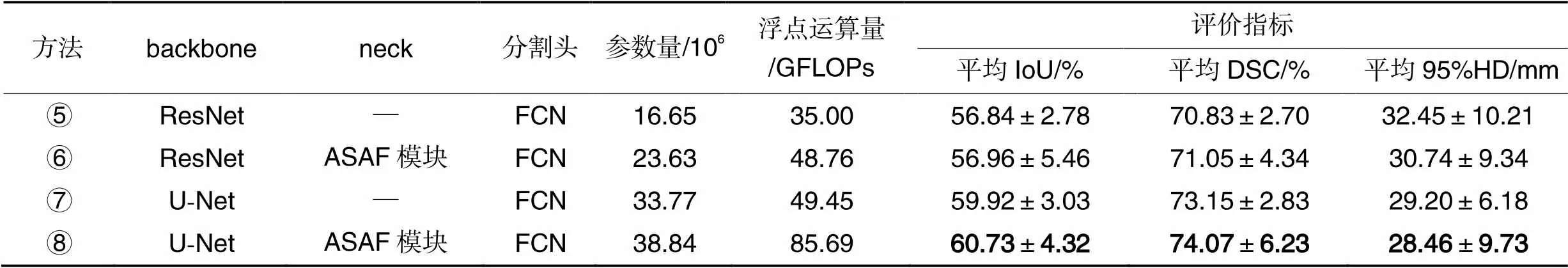
Tab.4 Ablation experimental results of ASAF module generalization ability
4 结语
本文提出一种基于邻近切片注意力融合的直肠癌分割网络(ASAF-Net),用于直肠癌MRI的多目标语义分割。该网络以HRNet作为特征提取的主干网络,解码网络中使用FCN和ASPP分割头协同训练,并在编码网络和解码网络之间引入ASAF模块。该模块输入当前切片与它前、后切片的多尺度特征,利用注意力机制融合和增强相邻切片特征之间上下文信息,使得网络能够学习相邻切片之间的空间信息。最后,在解码网络中加入相邻切片间的一致性损失约束作为辅助,缓解训练过程中出现的相邻切片差异大的问题。通过在127例直肠癌患者的MRI多目标分割数据集上进行评估,实验结果表明,本文提出的ASAF-Net与其他主流的分割网络相比有着较为稳健的性能提升,实验评估指标IoU取得63.07%,DSC取得75.96%,95%HD取得25.46 mm,实验结果均优于其他主流的分割网络。
本文实验所采用的直肠癌MRI数据获取成本较高,数据量仍不足,同时图像的语义信息较简单且目标靶区较固定,使用架构更复杂的网络反而使得分割精度降低。如何基于有限的数据样本量,设计一个架构简洁高效的直肠癌多目标分割网络,减小各目标之间的分割精度方差是下一步的研究重点。
[1] SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2021, 71(3): 209-249.
[2] WANG F H, ZHANG X T, LI Y F, et al. The Chinese Society of Clinical Oncology (CSCO): clinical guidelines for the diagnosis and treatment of gastric cancer[J]. Cancer Communications, 2021, 41(8): 747-795.
[3] 冯超,卢方明,李晓军,等. 探讨高分辨MRI对直肠癌术前T、N分期及环周切缘评估的准确性[J]. 世界最新医学信息文摘, 2019, 19(68):189-190.(FENG C, LU F M, LI X J, et al. Exploring the accuracy of high-resolution MRI for preoperative T and N staging and peri-annular margin assessment of rectal cancer[J]. World Latest Medicine Information, 2019, 19(68): 189-190.)
[4] 熊赤,牛朝诗. 多模态磁共振成像在脑胶质瘤鉴别诊断的应用[J]. 立体定向和功能性神经外科杂志, 2013, 26(1):49-54.(XIONG C, NIU C S. Application of multimodal magnetic resonance imaging in the differential diagnosis of glioma[J]. Chinese Journal of Stereotactic and Functional Neurosurgery, 2013, 26(1): 49-54.)
[5] WANG S, ZHOU M, LIU Z, et al. Central focused convolutional neural networks: developing a data-driven model for lung nodule segmentation [J]. Medical Image Analysis, 2017, 40: 172-183.
[6] SONG J, YANG C, FAN L, et al. Lung lesion extraction using a toboggan based growing automatic segmentation approach [J]. IEEE Transactions on Medical Imaging, 2016, 35(1): 337-353.
[7] BEN-COHEN A, DIAMANT I, KLANG E, et al. Fully convolutional network for liver segmentation and lesions detection[C]// Proceedings of the 2016 International Workshop on Deep Learning in Medical Image Analysis/ International Workshop on Large-Scale Annotation of Biomedical Data and Expert Label Synthesis, LNCS 10008. Cham: Springer, 2016:77-85.
[8] DE BRÉBISSON A, MONTANA G. Deep neural networks for anatomical brain segmentation [C]// Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition Workshops. Piscataway: IEEE, 2015: 20-28.
[9] ZHUANG J. LadderNet: multi-path networks based on U-Net for medical image segmentation[EB/OL]. (2019-08-28)[2022-12-25].https://arxiv.org/pdf/1810.07810.pdf.
[10] ZHU H T, ZHANG X Y, SHI Y J, et al. Automatic segmentation of rectal tumor on diffusion-weighted images by deep learning with U-Net [J]. Journal of Applied Clinical Medical Physics, 2021, 22(9): 324-331.
[11] ZHAO X, XIE P, WANG M, et al. Deep learning-based fully automated detection and segmentation of lymph nodes on multiparametric-mri for rectal cancer: a multicentre study[J]. eBioMedicine, 2020, 56: No.102780.
[12] LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2015, 39(4): 640-651.
[13] RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation [C]// Proceedings of the 2015 Medical Image Computing and Computer-Assisted Intervention, LNCS 9351. Cham: Springer, 2015: 234-241.
[14] CHEN L C, PAPANDREOU G, KOKKINOS I, et al. DeepLab: semantic image segmentation with deep convolutional nets, atrous convolution, and fully connected CRFs [J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 40(4):834-848.
[15] HE K, ZHANG X Y, REN S Q, et al. Spatial pyramid pooling in deep convolutional networks for visual recognition[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2015,37(9): 1904-1916.
[16] WANG J, SUN K, CHENG T, et al. Deep high-resolution representation learning for visual recognition[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2021, 43(10): 3349-3364.
[17] BAHDANAU D, CHO K, BENGIO Y. Neural machine translation by jointly learning to align and translate [EB/OL]. (2016-05-19)[2022-12-28].https://arxiv.org/pdf/1409.0473.pdf.
[18] MNIH V, HEESS N, GRAVES A, et al. Recurrent models of visual attention[C]// Proceedings of the 27th International Conference on Neural Information Processing Systems — Volume 2. Red Hook, NY: Curran Associates Inc., 2014: 2204-2212.
[19] HU J, SHEN L, SUN G, et al. Squeeze-and-excitation networks[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(8): 2011-2023.
[20] WOO S, PARK J, LEE J Y, et al. CBAM: convolutional block attention module[C]// Proceedings of the 2018 European Conference on Computer Vision, LNCS 11211. Cham: Springer, 2018: 3-19.
[21] WANG Q, WU B, ZHU P, et al. ECA-Net: efficient channel attention for deep convolutional neural networks [C]// Proceedings of the 2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Piscataway: IEEE, 2020: 2235-2239.
[22] VASWANI A, SHAZEER N, PARMAR N, et al. Attention is all you need [C]// Proceedings of the 31st International and Workshop on Neural Information Processing Systems. Red Hook, NY: Curran Associates Inc., 2017: 6000-6010.
[23] WANG X, GIRSHICK R, GUPTA A, et al. Non-local neural networks [C]// Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Piscataway: IEEE, 2018:7794-7803.
[24] TREBESCHI S, VAN GRIETHUYSEN J J M, LAMBREGTS D M J, et al. Deep learning for fully-automated localization and segmentation of rectal cancer on multiparametric MR[J]. Scientific Reports, 2017, 7: No.5301.
[25] RAO Y, ZHENG W, ZENG S, et al. Using channel concatenation and lightweight atrous convolution in U-Net for accurate rectal cancer segmentation[C]// Proceedings of the 4th International Conference on Pattern Recognition and Artificial Intelligence. Piscataway: IEEE, 2021: 247-254.
[26] MENG P, SUN C, LI Y, et al. MSBC-Net: automatic rectal cancer segmentation from MR scans [EB/OL]. [2023-02-20].https://www.semanticscholar.org/reader/5deddb5627e2a4805d72836b4039254c4ee94ebf.
[27] BADRINARAYANAN V, KENDALL A, CIPOLLA R. SegNet: a deep convolutional encoder-decoder architecture for image segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(12): 2481-2495.
[28] NOH H, HONG S, HAN B. Learning deconvolution network for semantic segmentation[C]// Proceedings of the 2015 IEEE International Conference on Computer Vision. Piscataway: IEEE, 2015:1520-1528.
[29] YUAN J, LIU Y, SHEN C, et al. A simple baseline for semi-supervised semantic segmentation with strong data augmentation[C]// Proceedings of the 2021 IEEE/CVF International Conference on Computer Vision. Piscataway: IEEE, 2021: 8209-8218.
[30] ZHANG Y, YUAN l, WANG Y, et al. SAU-Net: efficient 3D spine MRI segmentation using inter-slice attention[C]// Proceedings of the 3rd Conference on Medical Imaging with Deep Learning. New York: JMLR.org, 2020:903-913.
[31] LV P, WANG J, WANG H. 2.5D lightweight RIU-Net for automatic liver and tumor segmentation from CT [J]. Biomedical Signal Processing and Control, 2022, 75: No.103567.
Rectal cancer segmentation network based on adjacent slice attention fusion
LAN Donglei1,2*, WANG Xiaodong1,2, YAO Yu1,2, WANG Xin3, ZHOU Jitao3
(1,,610213,;2,100049,;3,,,610041,)
Aiming at the problem that the target regions of rectal cancer show different sizes, shapes, textures, and boundary clarity on Magnetic Resonance Imaging (MRI) images, to overcome the individual variability among patients and improve the segmentation accuracy, an Adjacent Slice Attention Fusion Network for rectal cancer segmentation (ASAF-Net) was proposed. Firstly, using High Resolution Network (HRNet) as the backbone network, the high-resolution feature representation was maintained during the feature extraction process, thereby reducing the loss of semantic information and spatial location information. Secondly, the multi-scale contextual semantic information between adjacent slices was fused and enhanced by the Adjacent Slice Attention Fusion (ASAF) module, so that the network was able to learn the spatial features between adjacent slices. Finally, in the decoder, the co-training of Fully Convolutional Network (FCN) and Atrous Spatial Pyramid Pooling (ASPP) segmentation heads was carried out, and the large differences between adjacent slices during training was reduced by adding consistency constraints between adjacent slices as an auxiliary loss. Experimental results show that compared with HRNet, ASAF-Net improves the mean Intersection over Union (IoU) and mean Dice Similarity Coefficient (DSC) by 1.68 and 1.26 percentage points, respectively, and reduces the 95% mean Hausdorff Distance (HD) by 0.91 mm. At the same time, ASAF-Net can achieve better segmentation results in both internal filling and edge prediction of multi-objective target regions in rectal cancer MRI image, and helps to improve physician efficiency in clinical auxiliary diagnosis.
rectal cancer; image segmentation; attention mechanism; feature fusion; deep learning
This work is partially supported by National Natural Science Foundation of China (82073338), Key Research and Development Project of Sichuan Provincial Science and Technology Program (2022YFS0217).
LAN Donglei, born in 1998, M. S. candidate. His research interests include deep learning, medical image processing.
WANG Xiaodong, born in 1973, research fellow. His research interests include network engineering.
YAO Yu, born in 1980, Ph. D., research fellow. His research interests include machine learning, pattern recognition.
WANG Xin, born in 1977, Ph. D., professor. Her research interests include rectal tumor diagnosis and treatment.
ZHOU Jitao, born in 1985, Ph. D., chief physician. Her research interests include rectal tumor diagnosis and treatment.
TP391.41
A
1001-9081(2023)12-3918-09
10.11772/j.issn.1001-9081.2023010045
2023⁃01⁃17;
2023⁃03⁃15;
2023⁃03⁃16。
国家自然科学基金资助项目(82073338);四川省科技计划项目重点研发项目(2022YFS0217)。
兰冬雷(1998—),男,广西河池人,硕士研究生,主要研究方向:深度学习、医学图像处理;王晓东(1973—),男,四川乐山人,研究员,主要研究方向:网络工程;姚宇(1980—),男,四川宜宾人,研究员,博士,主要研究方向:机器学习、模式识别;王辛(1977—),女,四川成都人,教授,博士,主要研究方向:直肠肿瘤的诊断与治疗;周继陶(1985—),女,四川成都人,主治医师,博士,主要研究方向:直肠肿瘤的诊断与治疗。

