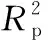基于高光谱成像技术的斜生四链藻(T. obliquus)碳水化合物和蛋白质判别研究
楚秉泉, 李成峰, 丁 黎, 郭正彦, 王世宇, 孙伟杰, 金唯一, 何 勇
1. 浙江科技学院生物与化学工程学院, 浙江 杭州 310023 2. 浙江大学生物系统工程与食品科学学院, 浙江 杭州 310058 3. 杭州方回春堂集团有限公司, 浙江 杭州 311500
引 言
微藻是一种水生光养型单细胞微生物, 具有易于培养、 生长周期短、 单位面积产量大和不占用农业用地等特点, 在添加剂、 生物固碳、 可再生生物能源等多领域具有广泛应用。 较高的培养成本始终是限制微藻商业化发展的主要瓶颈之一[1]。 在养殖过程中诸多因素如光照、 温度、 培养基、 磁场等都会影响微藻生长及胞内物质合成。 因此, 开发快速、 简单、 有效的无损检测方法, 实现微藻生长过程代谢信息的实时获取, 并据此及时调整培养条件, 对保障微藻高效优质生产、 降低养殖成本至关重要。 传统检测微藻生长和代谢产物(如碳水化合物、 蛋白质等)的方法虽具有很高的灵敏度和特异性, 但高昂的设备及专业操作人员不仅增加了微藻养殖成本, 同时也费时费力且无法实现大规模实时无损检测。
在微藻商业化养殖过程中, 应用智慧农业, 可以带来新的技术力量和科学的管理方法, 实现智能化养殖, 精准化管理, 提高微藻产量, 降低培养成本。 智慧农业是指利用物联网技术, 采集农业数据, 并通过云计算、 人工智能、 大数据等技术传输分析并建立决策模型, 对农业生产各个环节进行智能化管理的新兴技术手段。 在智慧农业中, 如何方便、 快速、 准确地获取作物信息, 是其关键问题之一[2]。 高光谱成像(hyperspectral imaging, HSI)技术作为一种综合技术, 集精密光学机械、 信号探测、 信息处理和计算机技术等于一体, 能同时表征像元光谱信息和空间物理特性, 目前已有许多研究致力于将其引入农业生产, 助力智慧农业。 例如, 农作物生化特性检测、 营养状况监测以及物种分类判别等。 HSI具有快速、 高效、 无损、 低价等特点, 近些年也有相关研究将其应用在微藻生长监测中。 例如, Xu等[3]采用透射光谱对棕囊藻(phaeocystis)生长阶段进行监测; Lian等[4]基于高光谱荧光成像技术分析蓝藻色素的分布和浓度; Lorenzo等[5]利用高光谱成像技术检测集胞藻(Synechocystissp.)中聚羟基丁酸酯(polyhydroxybutyrates)的累积情况。 前期文献检索发现, 将HSI应用于微藻生长监测方面研究多集中在油脂及其特性、 色素等方面, 对于同作为微藻胞内重要营养成分的碳水化合物、 蛋白质等的HSI研究则尚未见报到, 以往研究中所涉及的预处理方法、 特征选择方法和建模算法相对较单一, 模型优化有待进一步完善[6]。
斜生四链藻(Tetradesmusobliquus)是一种在淡水中生长、 适合大规模养殖的微藻, 富含油脂、 碳水化合物等营养成分, 具有很大的商业应用潜力。 碳水化合物、 蛋白质和油脂是微藻细胞内碳存在的主要形式, 各自合成代谢存在相互影响、 共同竞争碳源底物现象。 即通过理解碳水化合物、 蛋白质和油脂在微藻中的代谢分配规律及机制, 进而采取合理手段进行干预诱导, 是提高微藻生产效率、 降低养殖成本的重要途径。 要实现以上目标, 首先需要实现斜生四链藻培养过程中碳水化合物、 蛋白质和油脂的快速、 无损和实时监测。 前期利用可见/近红外(visible/near infrared, VIS/NIR)HSI对斜生四链藻油脂及脂肪酸不饱和度进行了建模分析及可视化研究, 取得了较好的效果[7]。 鉴于碳水化合物和蛋白质较油脂具有完全不同的特性和化学基团, 国内外未有相关报道, 故十分有必要对这两种成分进行HSI分析研究。
本研究以斜生四链藻为对象, 采用VIS/NIR HSI技术结合12种光谱预处理方式、 3种特征选择算法和4种建模方法, 对HSI应用于斜生四链藻生长代谢信息实时无损获取的可行性进行了探究, 并对藻液中生物量、 碳水化合物和蛋白质的空间分布和丰度进行可视化展示。 以期为斜生四链藻规模化培养过程的优化控制提供实时、 快速检测方法。
1 实验部分
1.1 藻种与培养条件
斜生四链藻(T.obliquus)购置于中国科学院淡水藻种库(FACHB), 采用BG11培养基(配方由FACHB提供)置于条件为温度25 ℃、 湿度60%、 光强(100±10) μmol·m-2·s-1、 光照时间12 h·d-1的人工智能气候箱中培养, 培养期间每天早、 中、 晚各摇微藻1次。 稳定培养5 d后进行实验。
由于前期研究发现30 mT的磁场结合100 μmol·m-2·s-1环绕式LED绿光对斜生四链藻生长具有显著促进作用[8], 故本实验采用此条件培养微藻, 重复25组。 其他条件与稳定培养期相同。
1.2 光谱数据获取
准确量取45 mL混匀的藻液, 倒于直径为90 mm的培养皿中, 立即采用ImSpector V10E可见光(VIS)高光谱成像(HSI)系统(Spectral Imaging Ltd., Specim, Finland, 380~1 030 nm, 512 wavebands)进行扫描获得VIS光谱数据。 考虑到NIR光谱需要更高的反射率, 故将藻液浓缩10倍后于35 mm培养皿, 采用ImSpector N17ENIR HSI(Spectral Imaging Ltd., Specim, Finland, 870~1 730 nm, 256 wavebands)获得样本NIR数据[7]。 每隔1 d(即培养的第1、 3、 5、 7、 9、 11、 13和15天)进行取样HSI扫描, 即一共采集25组×8次=200个样本数据。
图1为斜生四链藻VIS(a)和NIR(b)扫描图像及感兴趣区域(region of interest, ROI)选取示意图(红色框)。 采用ENVI软件(ITT Visual Information Solutions, United States)截取可见光谱40 000个像素点作为ROI并得到ROI的平均光谱; 截取近红外光谱400个像素点作为ROI并得到ROI的平均光谱。
图2(a, b)展示了斜生四链藻在生长周期内不同时间获得的200个样本的VIS(a)和NIR(b)平均光谱。 由于光谱首尾存在噪声(图中阴影部分), 因此截取VIS区392~1 023 nm 共500个波段和NIRS区931.40~1 645.82 nm共213个波段, 作为光谱建模波段。 VIS/NIRS光谱主要反映含氢基团(如C-H、 O-H、 N-H等)振动的倍频与合频吸收, 可通过光谱数据处理及建模分析获取样本中丰富的结构和组成信息。 例如, 属于色素吸收波段的430和680 nm以及属于水分子吸收波段的980 nm处有明显的反射波谷[5]; 1 159 nm 附近的反射波谷则与O-H、 C-H有关[9]; 位于1 490~1 600 nm归属于N-H吸收谱带[10], 也出现反射率降低。
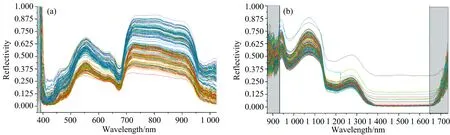
图2 斜生四链藻藻液的VIS(a)与NIRS(b)均值光谱图Fig.2 VIS (a) and NIRS (b) average spectral data of T. obliquus liquid
1.3 理化测定与分析
样本的光谱数据采集后, 立刻采用相应方法对样本中生物量、 碳水化合物和蛋白质含量进行分析, 具体方法参考文献[8]。
1.4 数据校正
由于数据采集过程中设备存在一定的暗电流且不同波段下光源的分布不均匀会产生较大的噪声, 因此需要对高光谱图像进行黑白校正, 校正公式如式(1)
(1)
式(1)中:R0为原始高光谱图像,Rd为反射率0%的黑板高光谱图像,Rw为反射率100%的白板高光谱图像,R为校正后的高光图图像。
1.5 光谱预处理
光谱采集过程中难免会受到环境、 设备等各种条件的影响, 使得光谱出现基线漂移、 噪声、 散射等问题。 因此需要对光谱数据进行预处理, 从中选择合适的预处理方法提高模型的预测能力。
本研究采用12种较为常用的预处理方法[10], 包括原始光谱(raw)、 标准化(autosacling)、 标准正态化(standard normal variate transform, SNV)、 均值中心化(mean centering, MC)、 最大最小归一化(min-max)、 矢量归一化(vector normalization, VN)、 多元散射校正(multiplicative scatter correction, MSC)、 S-G(Savitzky-Golay smoothing)变换、 滑动平均滤波(moving average filter, MAF)、 小波变换(wavelet transform, WT)、 一阶差分(first difference, FD)和二阶差分(second difference, SD)。
1.6 特征波长选取
特征波长的选取可以大大降低样本的维度, 简化模型; 同时可以剔除不相关或冗余信息, 提升模型预测能力和稳健性。 本研究采用竞争自适应重加权采样算法(competitive adaptive reweighted sampling, CARS)、 区间随机蛙跳算法(interval Random Frog, iRF)和模拟退火算法(simulated annealing, SA)进行特征波长选取。
1.7 校正建模
采用了多元线性回归(multiple linear regression, MLR)、 偏最小二乘(partial least squares, PLS)、 支持向量机回归(support vector regression, SVR)以及随机森林回归(random forest regression, RFR), 用于构建光谱信息与目标组分之间的分析模型。
1.8 模型评估

2 结果与讨论
2.1 生物量的高光谱分析
2.1.1 高光谱采集及预处理
将每次采集的25份斜生四链藻(T.obliquus)藻液样本的高光谱数据按18∶7分为训练集和预测集, 即200个光谱数据中随机选择144个作为训练集, 56个作为预测集。 同时, 采用偏最小二乘(PLS)进行全波段建模, 评估不同预处理方法的优劣, 最终选定较为良好的预处理方法。 表1为200个藻液样本生物量范围, 表2为不同光谱预处理方法处理后的建模效果。

表1 斜生四链藻藻液样本中生物量范围(mg·L-1)Table 1 Biomass range of T. obliquus liquid samples

2.1.2 光谱特征选择

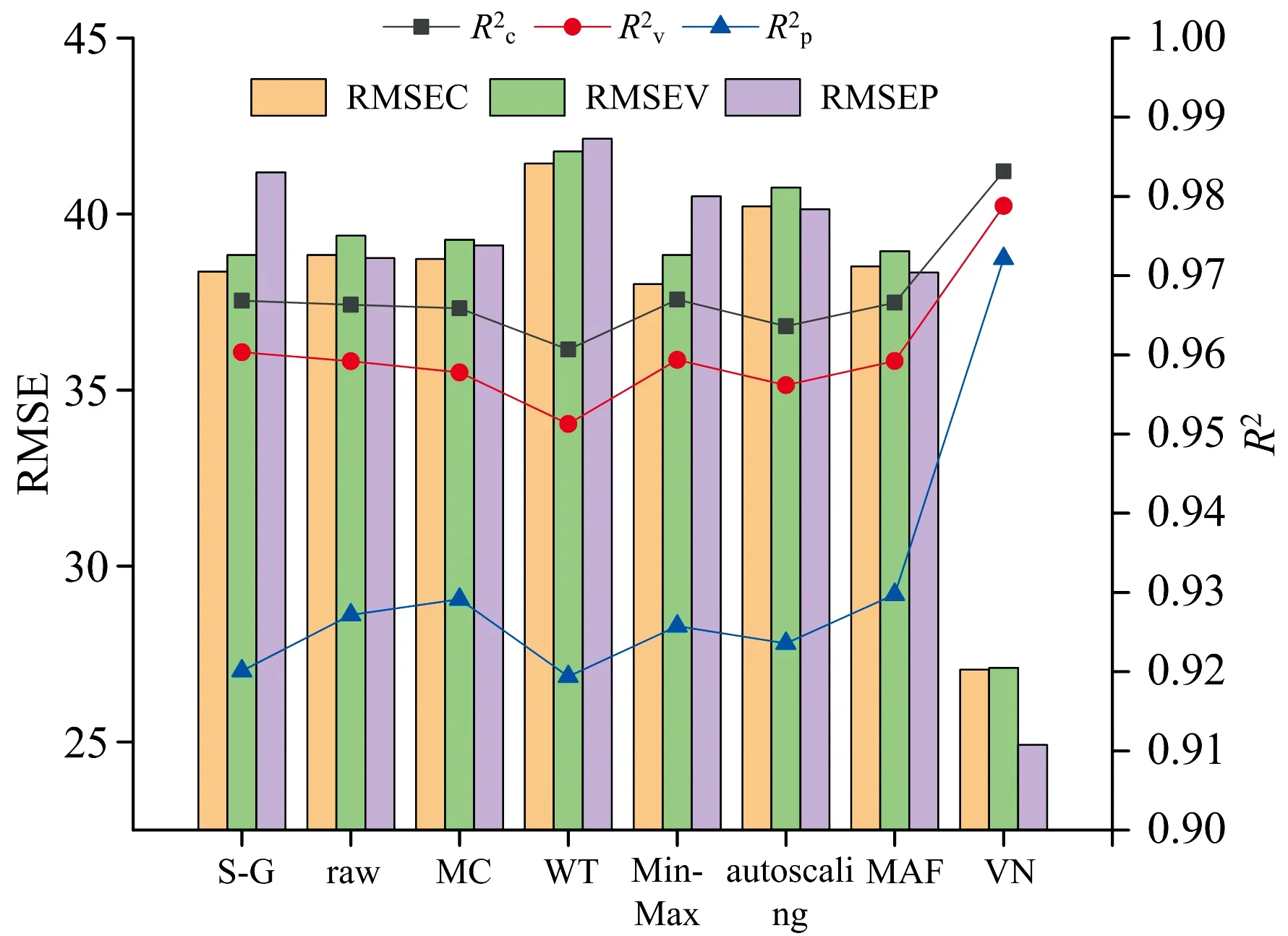
图3 不同预处理方式下藻液中生物量CARS特征选择建模结果评估Fig.3 Evaluation of microalgal liquid biomass model by CARS feature selection under different preprocessing methods
2.1.3 光谱建模
根据特征波段分析结果, 最终选择VN预处理光谱数据, 然后采用CARS选择特征波段建立CARS-PLS、 CARS-MLR模型和CARS-SVM模型。 其结果见表3。
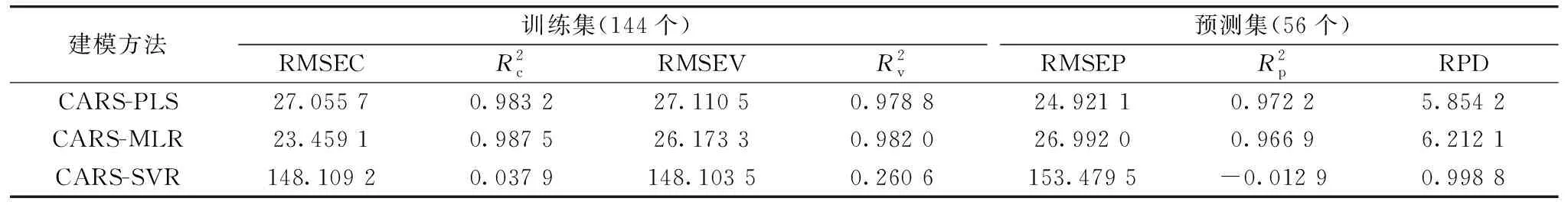
表3 经不同算法处理的藻液中生物量建模评估结果Table 3 Evaluation results of biomass model by different modeling algorithms
由表3可知, CARS-PLS和CARS-MLR预测精度, 泛化能力都很好, 较全波段建模, 整体模型预测能力有较大提升, 而CARS-SVR模型效果较差。 综合考量, 本实验最终选择VN光谱预处理方式结合CARS-MLR进行建模。 其模型公式如下
y=-41.096 6-21 806.072 34X452.35 nm+
16 779.819 6X487.46 nm+126 600.494 1X536.53 nm-
89 997.815 2X537.76 nm-165 549.437X593.75 nm+
83 791.885 7X610.06 nm-399 601.426 1X654.22 nm+
380 768.452 4X655.49 nm+214 234.526 9X714.04 nm-
188 444.272 9X715.32 nm-33 342.143 5X940.81 nm
(1)
式(1)中,Xi为波段第i个波段经过VN处理后的光谱值。 所选波段中452.35、 487.46、 536.53、 537.76和593.75 nm波段与色素有关; 610.06、 654.22、 655.49、 714.04和715.32 nm波段属于C-H基团振动谱带[7]; 940 nm属于水的吸收范围[9]; 生物量特征波段选择与前人研究一致[12]。 图4为光谱数据经VN预处理后基于CARS-MLR模型的斜生四链藻藻液生物量的预测结果。

图4 基于CARS-MLR模型的斜生四链藻藻液生物量的预测结果Fig.4 Prediction result of biomass of T. obliquus liquid based on CARS-MLR model
2.1.4 光谱成像
高光谱图像不仅能够对生物量浓度做出准确预测, 还可将模型应用于图像的每个像素点。 通过对每个像素点的生物量浓度进行预测, 从而得到生物量在高光谱图像中的分布图。 图5(a-h)为CARS-MLR模型预测斜生四链藻藻液样本中生物量的高光谱图像反演热力图。 可以看出, 从培养第9天起[图5(a-e)], 斜生四链藻生长速率显著增加, 分析认为培养前期(D1-D9)微藻以适应环境为主(调整期), 生物量增长较为滞缓, 而从D9起进入生长指数期。

图5 磁场和绿光培养下斜生四链藻藻液中生物量分布的化学成像图Fig.5 Chemical imaging of biomass distribution in liquid suspense of T. obliquus under MF and green LED exposure
2.2 碳水化合物的高光谱分析
2.2.1 光谱预处理
按同“2.1.1”的方式将藻液样本分为训练集和预测集。 斜生四链藻藻液中碳水化合物浓度范围如表4所示。

表4 斜生四链藻藻液样本中碳水化合物含量范围(mg·L-1)Table 4 Carbohydrate range of T. obliquus liquid samples (mg·L-1)
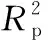

表5 不同光谱预处理方法对藻液中碳水化合物含量建模结果评估Table 5 Evaluation of carbohydrate model using different spectral preprocessing methods
2.2.2 光谱特征选择
在光谱预处理的基础之上, 选择raw光谱、 autoscaling、 MC、 S-G变换、 MAF, WT这6种效果相对好的预处理方式结合区间随机蛙跳(iRF)的方法进行光谱特征选择, 利用PLS算法建模, 其评估结果见图6。
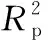
2.2.3 光谱建模

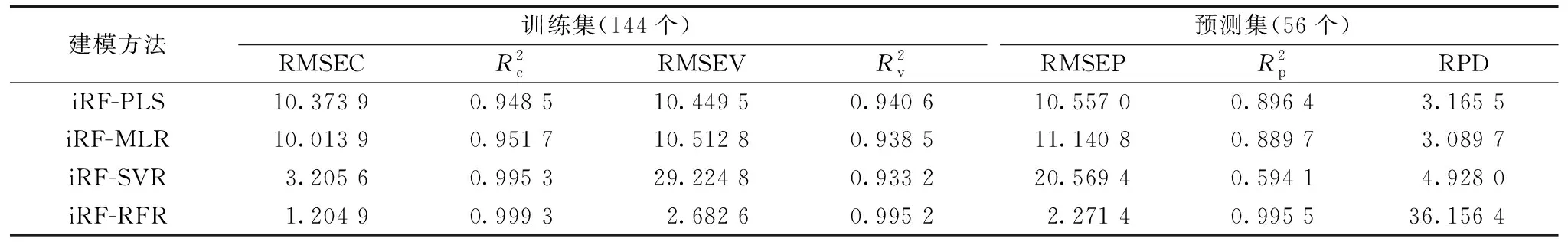
表6 经不同算法处理的藻液中碳水化合物含量建模评估结果Table 6 Evaluation results of carbohydrate model of different modeling algorithms

图7 基于iRF-RFR模型的斜生四链藻藻液中碳水化合物含量的预测结果Fig.7 Prediction result of carbohydrate of T. obliquus liquid based on iRF-RFR model
2.2.4 光谱成像
采用iRF-RFR模型, 对斜生四链藻选定的藻液ROI区域的每个像素点进行反演, 得到每个像素点碳水化合物的浓度值, 形成其在藻液ROI区域的空间分布图。 图8为采用30 mT的磁场结合100 μmol·m-2·s-1环绕式LED绿光连续培养15 d的斜生四链藻藻液中碳水化合物的丰度变化。 可以看出, 随着培养时间延长, 藻液中碳水化合物浓度逐渐增大, 尤其是从培养第11天起, 碳水化合物的累积效率明显增加。 但相对于生物量的增长有一定的滞后性, 可能的解释是随着微藻密度的增加, 藻液透光性明显降低, 反过来抑制了微藻的光合作用, 从而导致光合作用的主要产物--碳水化合物合成的减缓[15]。

图8 磁场和绿光培养下斜生四链藻藻液中碳水化合物分布的化学成像图Fig.8 Chemical imaging of carbohydrate distribution in liquid suspense of T. obliquus under MF and green LED exposure
2.3 蛋白质的高光谱分析
2.3.1 光谱预处理
按同“2.1.1”的方式将200个斜生四链藻藻液光谱数据分为训练集和预测集。 藻液中蛋白质的浓度范围见表7。 对比分析12种光谱数据预处理方法的效果, 表明raw光谱、 autoscaling、 MC、 S-G变换、 MAF和WT效果相对较好(表8)。

表7 斜生四链藻藻液样本中蛋白质含量范围(mg·L-1)Table 7 Protein range of T. obliquus liquid samples

表8 不同光谱预处理方法对藻液中蛋白质含量建模结果评估Table 8 Evaluation of protein model using different spectral preprocessing methods
2.3.2 光谱特征选择
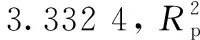

图9 不同预处理方式下藻液中蛋白质含量SA特征选择建模结果评估Fig.9 Evaluation of microalgal liquid protein model by SA feature selection under different pretreatment methods
2.3.3 光谱建模
在前期SA-MLR的基础上, 对选择的特征波段采用PLS、 SVR以及随机森林回归(RFR)算法进行建模, 并比较这几种方法的建模效果。 结果如表9所示, 总体以SA-RFR建模效果最佳。 图10为光谱数据经WT预处理后基于SA-RFR模型的斜生四链藻藻液中蛋白质含量的预测结果。
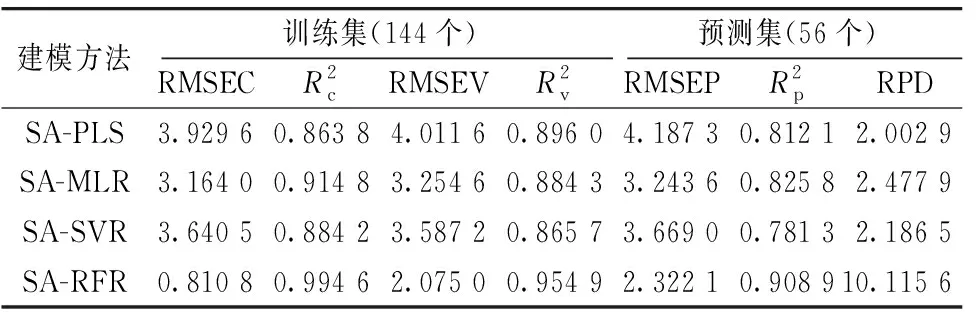
表9 经不同算法处理的藻液中蛋白质含量建模评估结果Table 9 Evaluation results of protein model by different modeling algorithms
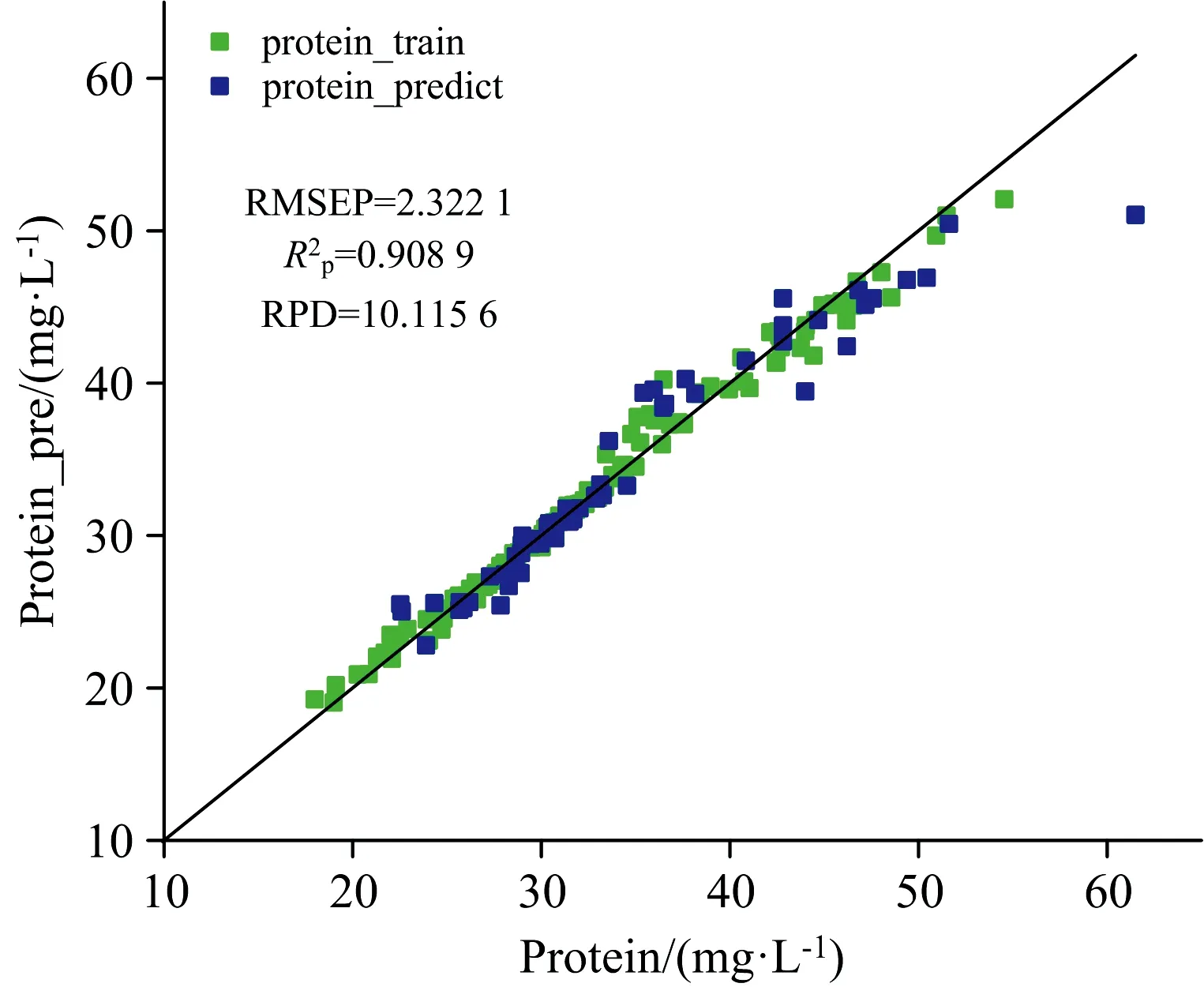
图10 基于SA-RFR模型的斜生四链藻藻液中蛋白质含量的预测结果Fig.10 Prediction result of protein of T. obliquus liquid based on SA-RFR model
2.3.4 光谱成像
采用前期优化筛选的SA-RFR模型对斜生四链藻藻液中的ROI区域的每个像素点进行反演, 得到每个像素点的蛋白质浓度值, 进而描绘出蛋白质在ROI区域上的丰度分布图(图11)。 从图可以看出, 培养前7天, 藻液中蛋白质含量增加速率较缓慢, 但从培养D9起, 蛋白质累积效率显著增高, 与生物量的变化较为同步, 可能是本研究采用的培养条件对斜生四链藻中蛋白质含量占比影响较小[8]。
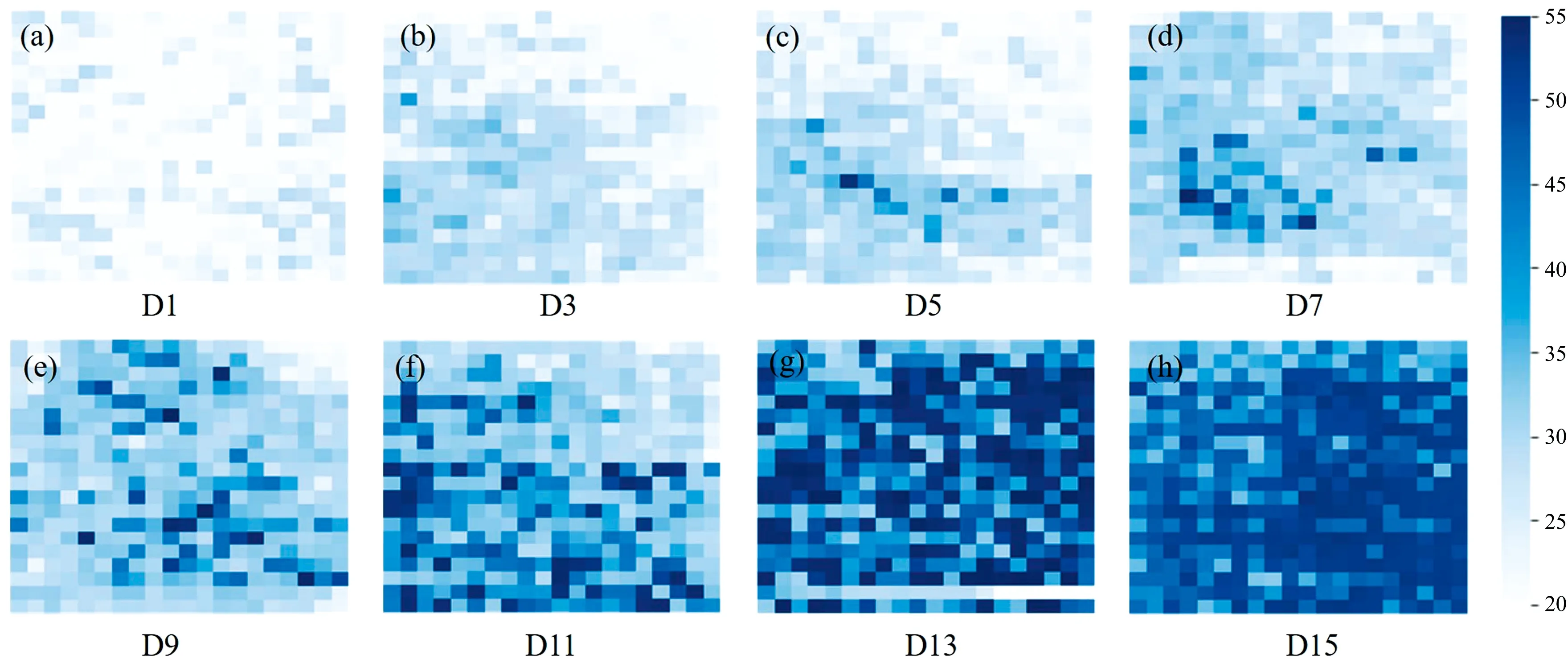
图11 磁场和绿光培养下斜生四链藻藻液中蛋白质分布的化学成像图Fig.11 Chemical imaging of protein distribution in liquid suspense of T. obliquus under MF and green LED exposure
3 结 论