阿尔茨海默症的多模态分类方法
李伟汉,侯北平,胡飞阳,朱必宏
浙江科技学院自动化与电气工程学院,浙江杭州310023
阿尔茨海默病[1](Alzheimer’s disease,AD)是一种起病隐匿的进行性发展神经系统退行性疾病,临床表现为记忆力衰退、失语、抑郁、执行功能障碍以及人格和行为改变等症状。目前该病的发病机制尚未确定,暂时无法治愈,只能通过药物延缓疾病进展,延长生存期。中国约有1 400 万阿尔茨海默患者,预计到2050 年将突破3 000 万[2],AD 的诊断和治疗已成为当今社会的主要医疗卫生问题之一。该病可划分为AD 前阶段和AD 阶段,AD 前阶段医学认定为轻度认知障碍[3](mild cognitive impairment,MCI),这一阶段介于正常衰老和AD 之间,是认知障碍症阶段,患者轻度认知功能减退,但日常能力没有受到明显影响,MCI 早期阶段在临床上认定为早期轻度认知障碍(early mild cognitive impairment,EMCI),这一阶段的诊出对病人的及时治疗、延缓疾病进展、改善症状具有重要意义。
随着深度学习领域的不断发展,越来越多的学者利用医学影像通过深度学习的方法对AD 进行分级诊断。主要方法有两种:一种是基于单模态医学影像的分类方法,利用核磁共振成像[4](magnetic resonance imaging,MRI)对AD 进行分类识别,例如文献[5] 提出的基于视觉几何组(visual geometry group,VGG)神经网络的弱监督细粒度分类方法,文献[6] 提出的基于Alexnet 模型的AD 分类方法;另一种是基于多模态医学影像的分类方法,利用MRI图像和正电子发射计算机断层成像[7](positron emission computed tomography,PET)加权融合作为多模态输入,如文献[8] 提出的一种基于卷积循环神经网络的阿尔茨海默症早期诊断方法,这两种方法均存在不足,主要体现在以下两个方面:
1)脑图像切片选取问题:MRI 图像和PET 图像均为三维影像,按照剖切面[9]的不同可分为多张横断面、冠状面、矢状面切片。AD、EMCI 和NC 大脑萎缩程度只在部分脑区内有所差别,上述分类方法所用的图像是随机取大脑横断面单张切片或连续多张切片作为神经网络输入,随机单切片法所取切片难以完整表征三类人群脑结构差异,无法充分发挥神经网络模型性能。连续切片法虽然保证了少部分输入图像能够表征三者脑区域差异性,但引入了大量的无关图像降低了神经网络的分类精度,增加了神经网络计算量。
2)生物标志物选取问题:在AD 的多模态分类识别中,仅利用MRI 图像和PET 图像作为两个不同的输入模态,未加入生物标志物模态。MRI 图像和PET 图像均反映大脑萎缩程度,作为两个多模态输入图像对模型性能提升有限,且AD 的早期阶段EMCI 与NC 脑结构差异并不明显,其病理变化首先体现在生物标志物含量的改变。生物标志物作为临床上诊断AD 和EMCI 的首要参考因素,其含量的异常变化具有重大的分类意义,应考虑作为多模态输入,探究其对AD 分类准确率的贡献程度。
以上现状说明,选取更精确的脑图像切片,采用更科学的多模态输入,对于EMCI 的早期发现及AD 精确分级诊断具有重大意义。本文以分析AD、EMCI、NC 脑差异区域为着力点,确定最大差异脑区域位置并在此区域提取三类样本的脑图像切片,对同一名样本对象筛选并引入其生物标志物与脑图像切片一一对应作为多模态输入,采用基于ResNet 的特征融合方法提取脑图像切片深度特征,同时利用多层感知机对生物标志物进行扩维,构建多模态分类模型,经多次实验对比验证,该模型具有优秀的分类能力。
1 基本原理
1.1 数据说明
本文所使用的图像数据、生物标志物均来自ADNI (Alzheimer’s disease neuroimaging initiative)数据库。ADNI[10]数据库由美国国立卫生研究院衰老研究所(National Institute on aging,NIA)、生物医学成像与生物工程研究所(National Institute of biomedical imaging and bioengineering,NIBIB)等组织联合建立。数据库主要由临床数据、遗传数据、和影像数据组成,用于早期检测、研究、跟踪阿尔茨海默病。临床数据为每个受试者的临床信息,包括招募,人口统计学,身体检查和认知评估数据;遗传数据为受试者的基因数据;影像数据为原始、预处理和后处理的图像文件包括MRI、功能性磁共振成像(functional magnetic resonance imaging,FMRI)、弥散张量成像(diffusion tensor imaging,DTI)、PET 图像。
1.2 脑图像配准与分割
本文通过对不同人群进行脑区域差异性分析[11],来获取最大差异脑区域,并确定特征图的选取位置,以选取优质脑图切片提升识别准确率。在进行脑区差异性分析时要对脑图像进行配准处理[12]。不同患者大脑的大小和形状是不一样的,而且在不同次的核磁扫描中患者躺平的姿势也不同,这就会造成脑图像在相同空间坐标下对应的不同大脑的解剖位置也不一致,因此不同组别样本的脑组织图像相减得到的并不是对应组织结构的变化,而这样的脑区差异是没有意义的。脑图像空间坐标配准,能够在保证脑组织特异性的前提下,将不同大脑图像对应到公共脑模板上矫正大脑形状和解剖位置,让不同的受试者具有可比性。图像配准方法大致可分为基于标签的配准方法和基于强度的配准方法[13]:基于标签的配准方法识别源图像和参考图像中的同源特征,并找到最佳叠加变换,标签可以是点、线或曲面,由于标签通常需要人工参与,因此存在主观和耗时的问题;基于强度的配准方法则是寻找特定的变换来优化源图像和模板图像中的一些体素相似性度量,将模板图像g看作待配准图像f根据变换向量α扭曲后,同时加上高斯随机噪声的图像。配准原则是找到最优变换向量α,确定位移场来对图像进行矫正。本文采用基于强度的方法进行脑图像配准,模板图像第i个体素的体素强度为gi,其满足均值为fi(α),标准差为σ的正态分布,调节变换向量α使得损失函数F(α) 最小,公式为
式中:I为图像体素点数目;H(α) 为包含能量密度的杂项,在确定最优变换向量α后,原图像像素点xi形变后位置yi(xi,α) 就可以用一系列的基函数的线性组合表示,则有
式中:Φm(xi) 为xi的余弦变换基函数,公式为
式中:M为基函数的项数,变换向量α维度也为M。实际上因为大脑图像并不存在一一精准对应的关系,所以不需要过高的精准度,M可取有限项。
在脑图像配准后要对其进行组织分割处理[14],可将脑组织分割为灰质、白质和脑脊液,以进行下一步处理,分割方法基于不同脑组织的独立高斯模型组成的高斯混合模型和朴素贝叶斯原理。已知该模型由K个单独的脑组织高斯混合模型组成,其中第k个高斯的均值为µk,方差为,混合比例为γk=1,γk>0)。一幅图像强度为yi的体素点,若已知其属于第k个高斯分布,那么它出现的概率P(yi|ci=k,µk,σk) 可表示为
已知任意体素其属于第k个脑组织高斯分布的概率为P(ci=k)=γk,利用贝叶斯方法可以求得体素i属于第k个脑组织高斯分布概率为
求出体素i属于第k个脑组织高斯分布概率后,可根据概率最大原则对所有体素进行分类。
1.3 脑区域差异性分析实验
根据上文提出的脑组织配准、分割原理就可以结合基本统计方法和图像处理方法对不同人群脑组织进行脑区域差异性分析。在ADNI 数据库中选取具有MRI 脑图像的AD、EMCI、NC 各27 例样本,受试者信息如表1 所示。
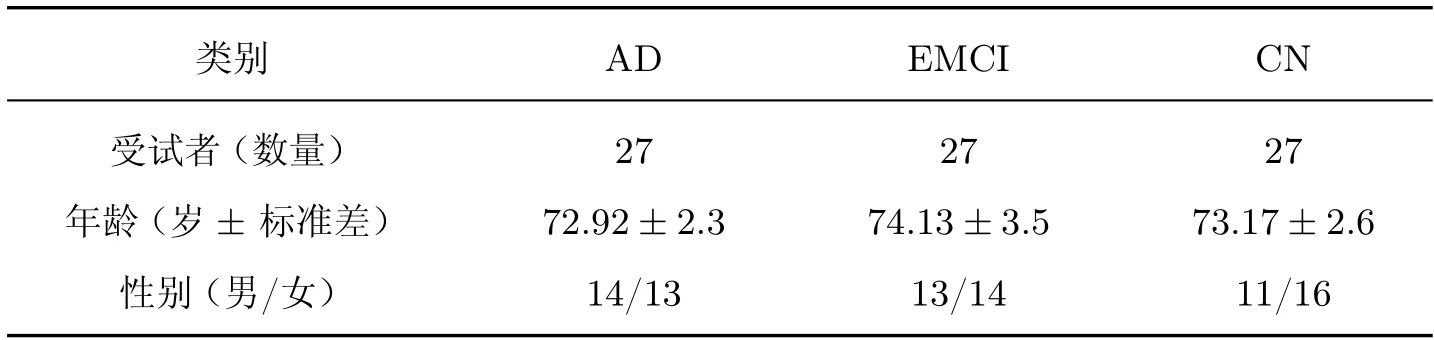
表1 SPM 实验受试者信息Table 1 SPM subjects’ information
将3 组样本的MRI 原生图像利用VBM 软件进行去骨骼、配准和脑组织分割得到灰质和白质,结果如图1 所示,实验流程如图2 所示。随后选择灰质图像进行脑区差异性分析(事实上大脑的灰质和白质在脑空间上是互补的,选用白质也将获得同样的分析结果)。首先将灰质图像进行图像平滑便于统计与分析,然后进行组内相关性分析,去除组内相关性最大和最小的样本,增加实验结果的鲁棒性。此时3 组样本中每组各有25 幅三维灰质图像,将3 组样本进行两两分组,分别为AD&NC、AD&EMCI、EMCI&NC,对应3 次灰质组间差异性实验。以AD&NC 的差异性实验分析为例,将两类样本的三维灰质图像利用SPM 进行双样本t检验(本实验显著性水平α=0.005),其原理是,设AD 患者脑灰质的体素点集合为SAD,NC患者脑灰质的体素点集合为SNC,样本A集合为AD 患者脑灰质的体素点集合SAD的子集合,样本B集合为NC 患者脑灰质的体素点集合SNC的子集合。由上文可知,AD 和NC 样本均经过公共脑图像模板配准,所以SAD与SNC同一空间区域子集合(A,B) 具有相同的组织对应关系,对样本A集合和样本B集合进行双样本t 检验,可得出SAD与SNC同一空间区域脑组织是否有显著性差异。
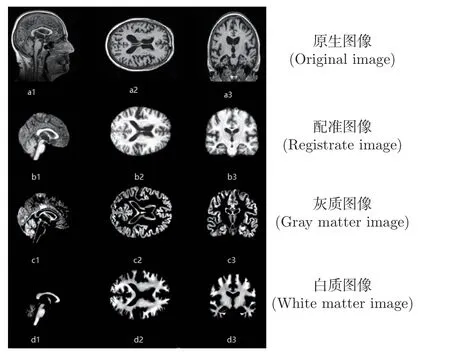
图1 原生脑图和组织图Figure 1 Original brain image and tissue image
图2 实验流程图Figure 2 Flow chart of experimental
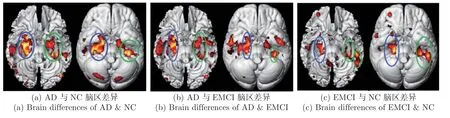
图3 脑区差异对比图Figure 3 Comparison chart of brain differences
1.4 脑图像选取方法
在经过差异性分析后AD、EMCI、NC 脑区差异对比图如图3 所示,同一颜色圆圈所示区域为大脑同一区域(横断面视角)不同层次的观测结果。由3 幅图得知3 组受试者脑结构差异主要存在于左右脑的海马体和颞叶区域。其中右脑海马体和颞叶区域(蓝色圆圈标注区域)较为明显,能很好地涵盖3 类样本。
依照实验结果所示的最大差异脑区域,取右脑海马体-颞叶区域切片作为模型的输入图像,大脑的医学影像视角描述共有3 种,分别为矢状面视角、横断面视角以及冠状面视角(分别对应图1 中的a1、a2、a3),不同视角易于呈现不同的脑组织特征。海马体平行于大脑矢状面斜向下生长,根据矢状面视角取脑切片可以完整地反映出患者大脑海马体-颞叶区域的萎缩程度。
图4 为脑切片选取位置示意图,图中两条红线间区域可做脑切片选取区域,对照图3可知,图4 中绿线所示位置横穿海马体颞叶区域,可以体现出3 类样本间的脑区域差异性。ADNI 数据库中每幅三维MRI 图像都由121 幅二维切片构成,图4 绿线所示位置约在第37∼40 幅矢状面切片处,本文对所有样本的MRI 原生图像进行去骨骼和矫正处理,同时取其第38 幅图像作为神经网络的输入图像。所选脑切片图像如图5 所示。观察图5 可知,AD 海马体区域(红色标注区域)萎缩严重呈现大块黑色空洞,前额叶区域黑色褶皱较多与NC 相关区域呈现出显著差异,EMCI 患者脑萎缩程度不明显,在结构上接近NC,但生物标志物的加入能够提高分类精度。
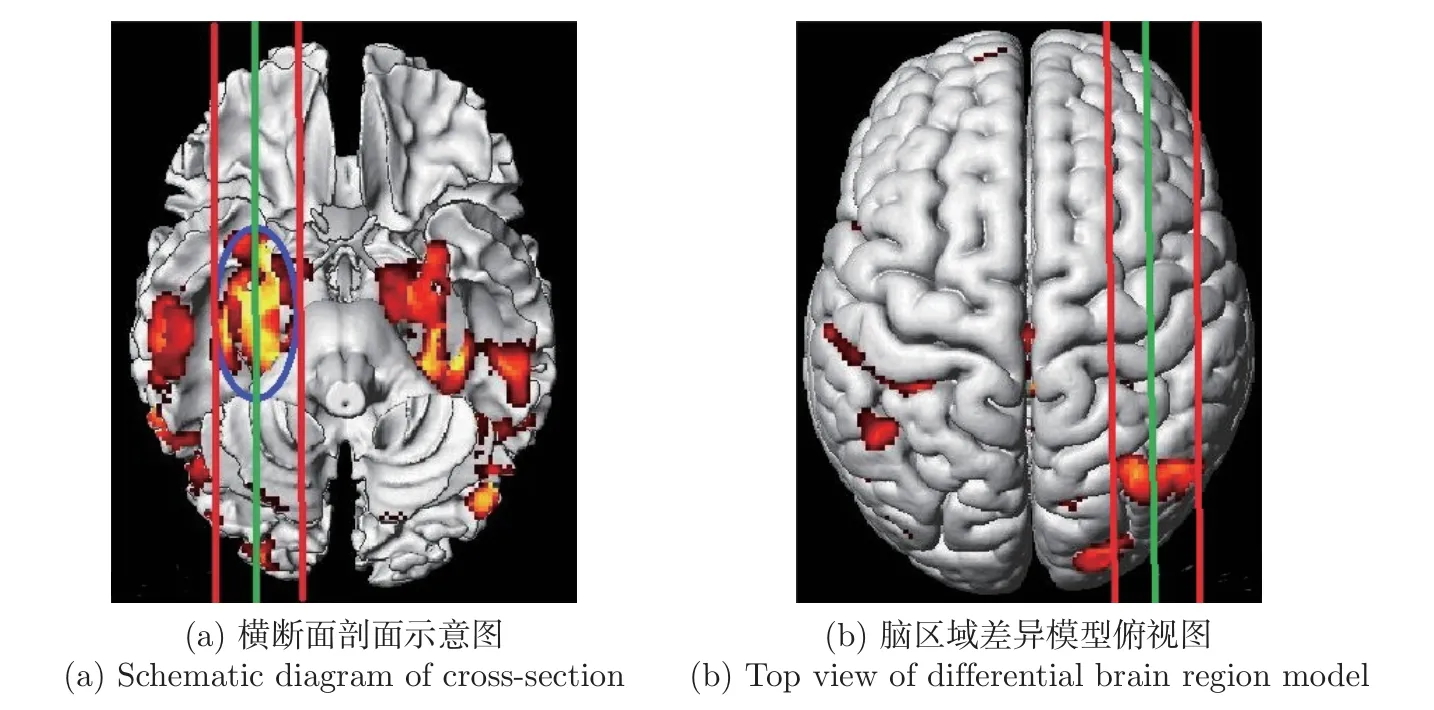
图4 脑切片选取位置示意图Figure 4 Schematic diagram of selected locations of brain slice
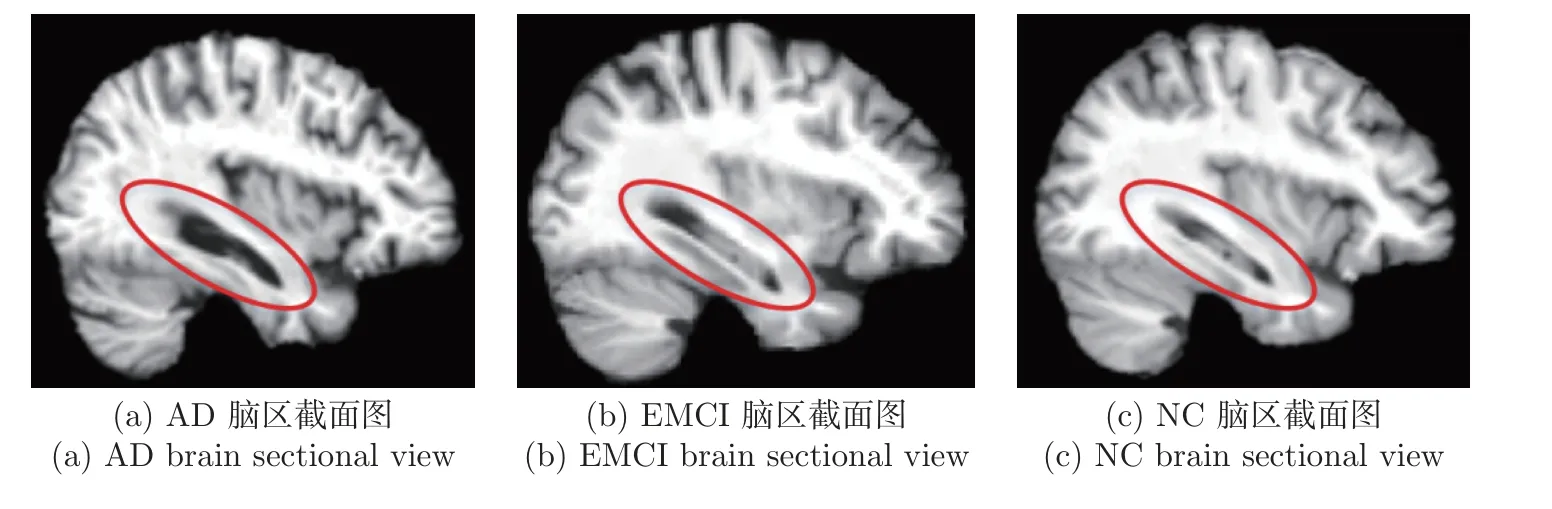
图5 脑区截面图Figure 5 Brain sectional view
1.5 生物标志物分析
AD 患者大脑结构变化为脑萎缩,而生标志物的变化体现在神经元细胞周围β-淀粉样(Amyloid-β,Aβ)蛋白聚集、细胞内微观相关蛋白(microtubule-associated protein tau,tau)、过度磷酸化形成磷化tau 蛋白(phosphorylated tau protein,p-tau)[15]。临床标志物的变化指标是AD 早期诊断、监测疾病的重要依据,寻找到合适的生物标志物对AD 的诊断和分类至关重要。本文从ADNI 数据库中选取50 名受试者和50 名AD 患者的生物标志物,其中包括tau 蛋白、p-tau 蛋白、Aβ蛋白、载脂蛋白E 型基因(Apoli-poproteinE,ApoE)[16-17]等,受试者信息统计表如表2 所示。

表2 差异性实验受试者信息统计表Table 2 Statistical table of difference experiment subjects’ information
对3 组样本的生物标志物进行差异性检验(Z检验)来辨别在AD、EMCI 和NC 组别中相应生物标志物含量的变化是否有显著性差异。对各组样本的生物标志物按照式(10) 求均值,按照式(11) 求标准差,¯x为组内生物标志物含量样本均值,xi为组内第i个样本生物标志物含量,n为样本数量,n取50。
将式(10) 和(11) 代入式(12) 中得出Z值,为组间样本均值,为组间样本方差。
根据Z值表可判定AD、EMCI 与NC 组的tau 蛋白、p-tau 蛋白、Aβ蛋白、ApoE 基因有显著性差异,作为本实验多模态输入。
2 实验设计
2.1 ResNet 网络调整
ResNet[18](residual neural network)神经网络的主要思想是,在网络中增加直连通道以允许保留之前网络层的一定比例输出,构建出一个如图6 所示的残差学习模块。残差模块的引入使ResNet 具有良好的特征提取能力,同时减小了网络的计算量,提高了网络的实际运行速度。本文选择ResNet18 网络为本模型特征提取网络。实验所用图像为121×145 的灰度图像,对ResNet18 网络进行调整使得输入为(121,145,1)大小的图像,并去掉最后一层分类层,图像特征提取网络结构如图7 所示,网络参数如表3 所示,其中s为步长,p为填充。
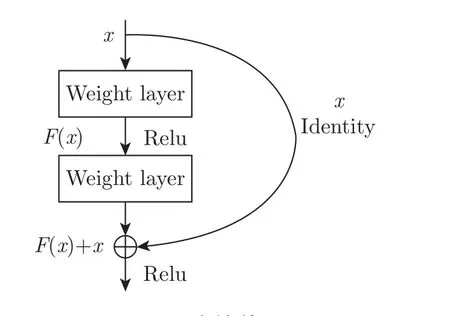
图6 残差单元Figure 6 Residual unit
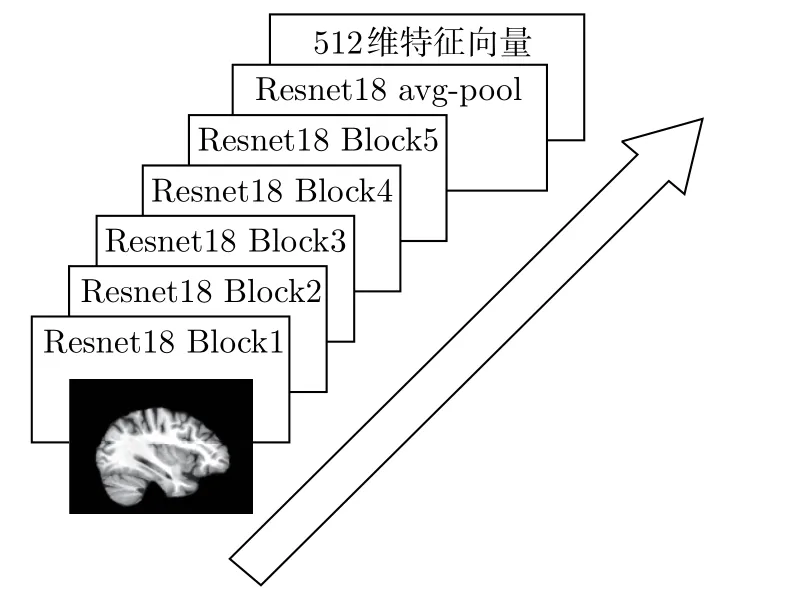
图7 特征提取结构Figure 7 Feature extraction structure
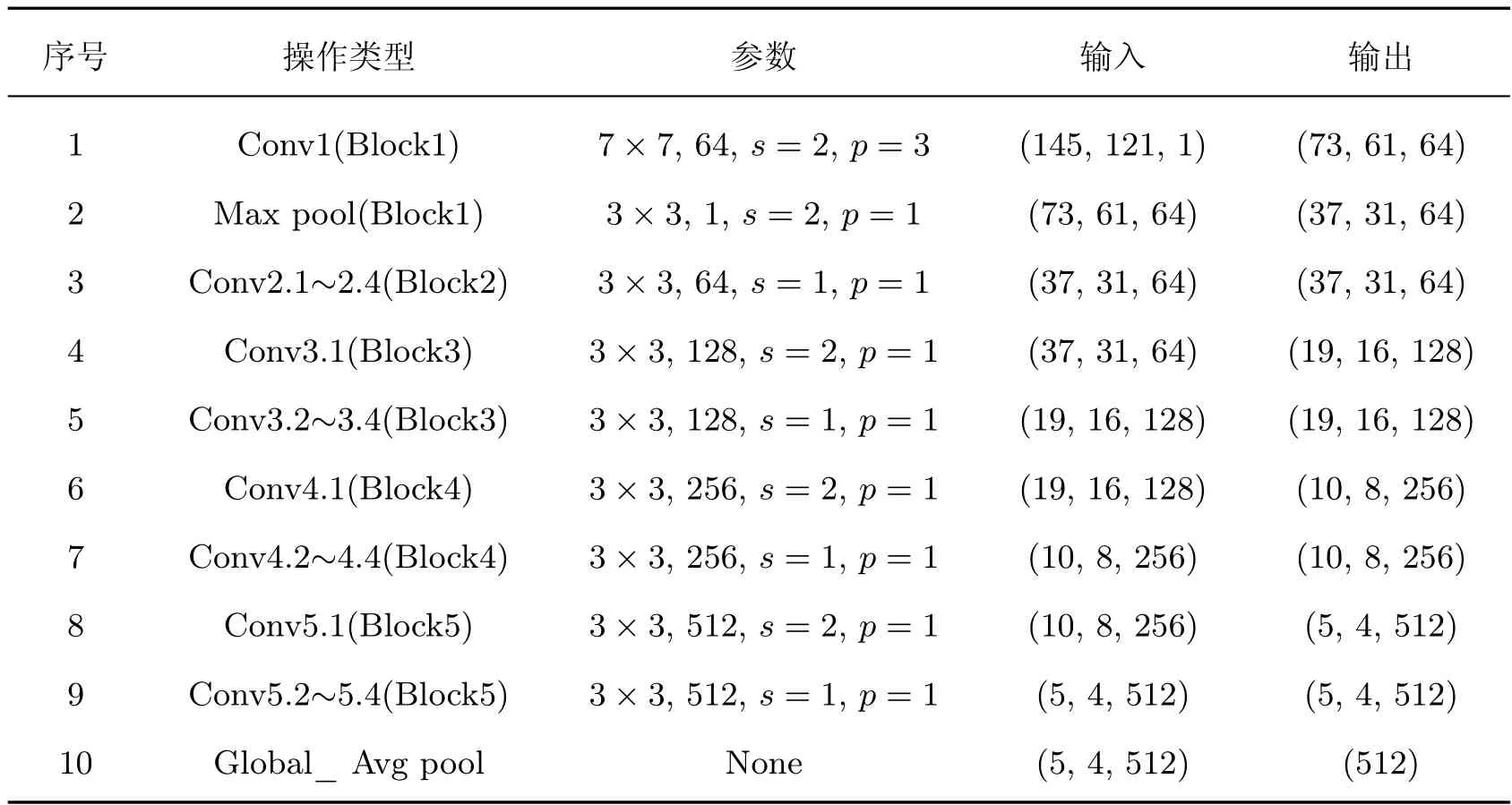
表3 ResNet18 网络参数Table 3 ResNet18 network parameters
2.2 联合网络
本实验利用ResNet18 神经网络模型作为图像特征提取模块,提取脑图像特征并降维至512 维特征向量,利用4 层感知机将生物标志物扩展为512 维特征向量;对图像特征和生物标志物特征进行特征加权拼接至1 024 维特征向量,图像特征向量加权系数为w1,生物特征向量加权系数为(1-w1);最后利用多层感知机将1 024 维的向量降维至512 维并利用Softmax层对融合后的特征向量进行分类,网络流程图如图8 所示,多层感知机网络参数如表4 所示。
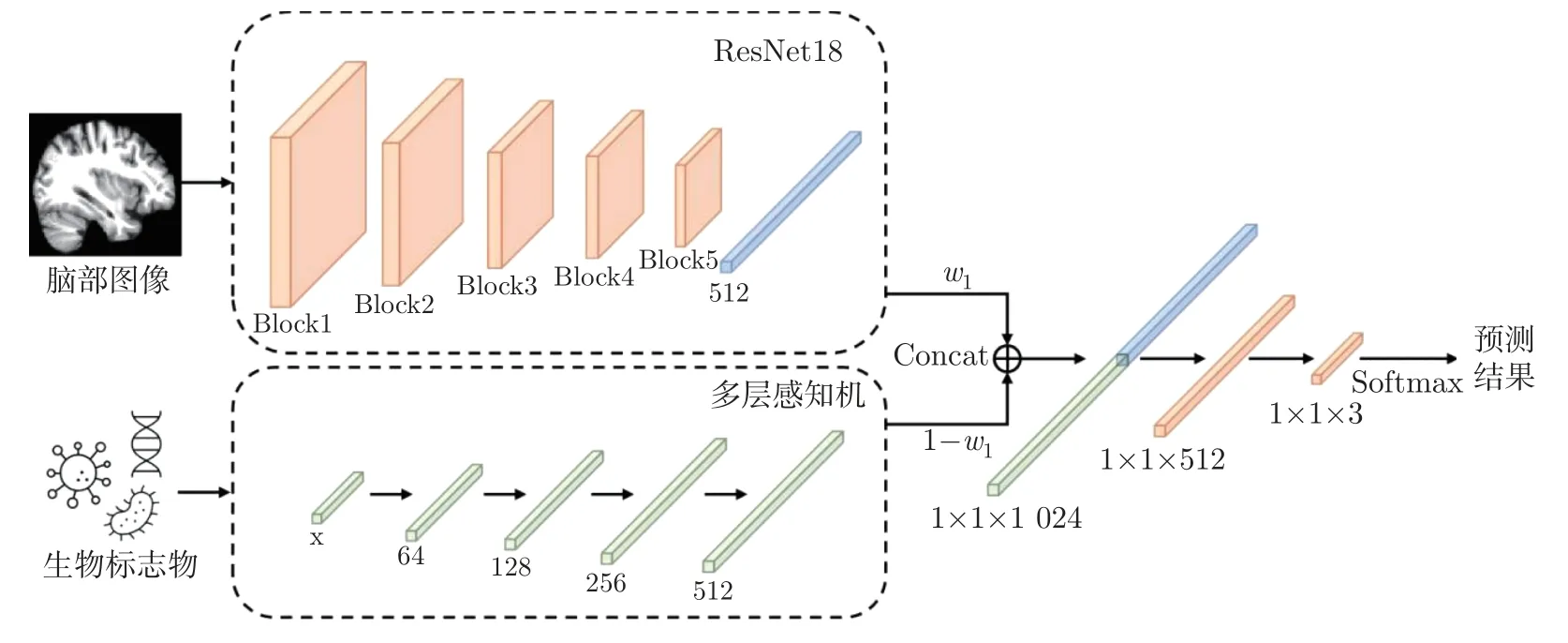
图8 分类网络模型流程图Figure 8 Classification network model flow chart
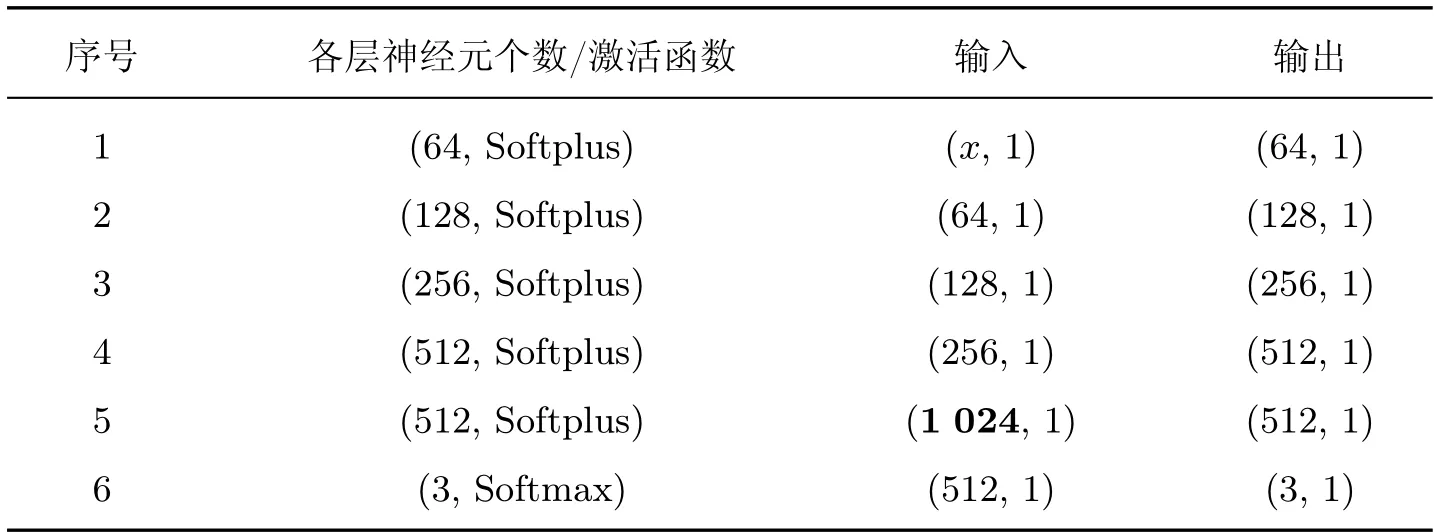
表4 多层感知机网络参数Table 4 MLP network parameters
3 实 验
3.1 分类实验数据集说明
本文分类实验数据来自于ADNI 数据库,其中AD 样本、NC 样本来源于ADNI1 阶段,EMCI 样本来自于ADNIGO 阶段。ADNI1 阶段有1 673 例AD 样本、4 518 例NC 样本,同时包含MRI(T1) 图像和生物标志物信息的AD 样本共有344 例,由233 名患者提供,包含MRI(T1) 图像和生物标志物信息的NC 样本共有651 例,由285 志愿者提供。ADNIGO 阶段有2 798 例EMCI 样本,包含MRI(T1) 图像和生物标志物信息的EMCI 样本共有455 例,由281 名患者提供,同一名患者可能提供多例样本。
分类实验选用的数据包含年龄在57∼89 岁的AD、NC、EMCI 样本各300 例,训练集AD、NC、EMCI 各200 例,测试集AD、NC、EMCI 各100 例。数据库中AD 样本共有344例,由233 名患者提供,因此分类实验中选用的300 例实验样本会包含纵向数据,为保证实验的严谨性与科学性,分类实验测试集选用的100 例数据由100 名患者提供,均不包含纵向数据,训练集选用的200 例数据由133 名患者提供,每名患者最多提供2 例样本,且提供样本的时间跨度至少为1 年,在扩充数据集的同时,保证了实验的严谨性,EMCI、NC 数据集划分规则与AD 数据集划分规则一致。
每例样本的核磁共振图像均经过矫正、配准、分割处理,依据脑区差异性实验选取右脑海马体-颞叶区域的矢状面第38 图像作为模型的输入图像。模型以样本单张脑图像切片预测样本患病的可能性,选取样本信息如表5 所示。
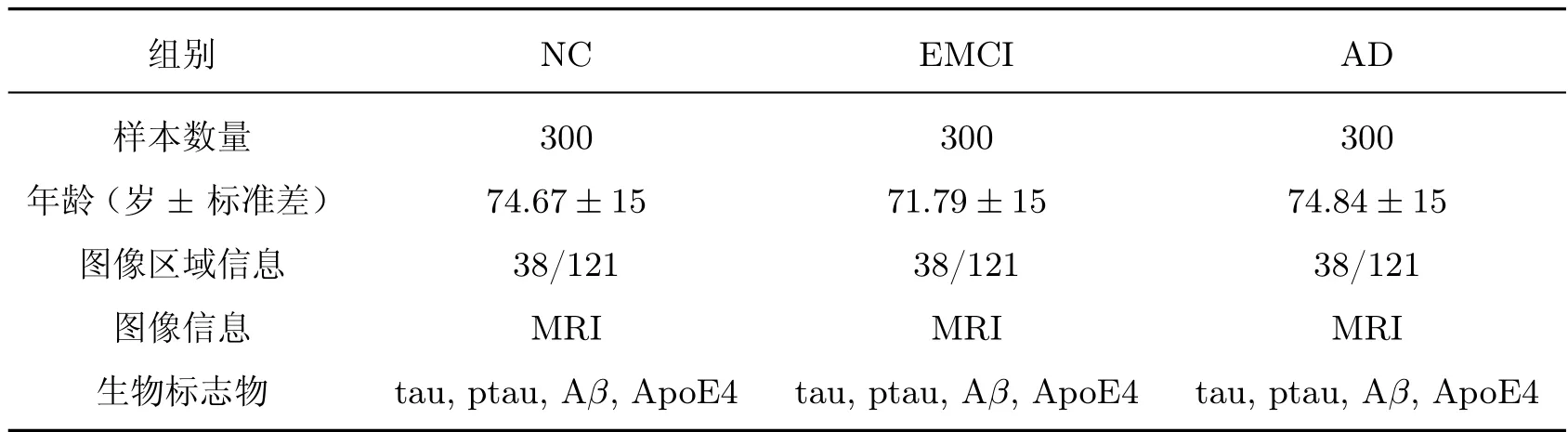
表5 分类实验受试者信息统计表Table 5 Statistical table of classification experiment subjects’ information
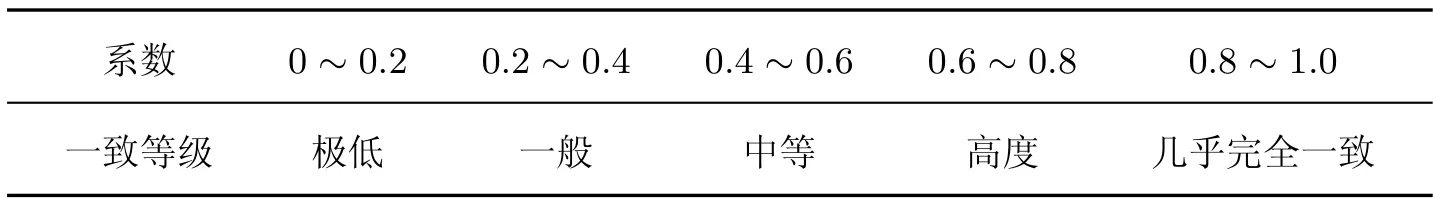
表6 Kappa 系数表Table 6 Kappa Coefficient table
3.2 实验环境和实验方法
网络模型训练使用Keras 深度学习框架,GPU 型号为NVIDIA 3090Ti,显存24 G,CPU为XEON 2680V4,操作系统为Ubantu-18.04。网络为多输入单输出模型,同时输入样本的脑图像信息和临床生物标志物信息,输出分类结果,批次大小设置为20,使用Adam[19]优化器,初始学习率为0.000 1,w1初始权值为1,程序每完全执行一次w1的权值减少0.05 直至w1值为0,记录实验结果并选取最优模型。为与传统分类方法对比,本文部分实验选取目前脑图分类选取较多的横断面区域与本文所选海马体相关区域特征脑图作对比实验。为探究多模态模型对分类准确率的提升效果,本文生物标志物模态由1 种逐步增加至4 种并且尝试不同的组合的分类效果。
3.3 评价指标
为评估模型的分类性能,本实验采用准确率(Accuracy,A)、精确率(Precision,P)、召回率(Recall,R)作为二分类模型的评价指标,采用Kappa (K) 系数[20]作为三分类评价指标。准确率即对于给定的数据,分类正确的样本数占总样本数的比例;精确率是指在预测为正类的样本中真正类所占的比例;召回率是指在所有正类中被预测为正类的比例。Kappa 系数是用在统计学中评估一致性的一种方法,取值范围在一般应用中是[0,1],系数越高,则代表模型分类的准确度越高,相关公式和图表如下。
式中:TP 表示把正类预测为正类;FP 表示把负类预测为正类;TN 表示把负类预测为负类;FN 表示把正类预测为负类。
式中:po为总体分类精度;pe为所有类别分别对应的“实际与预测数量的乘积”之总和,除以“样本总数的平方”。
3.4 实验结果
为探究特征区域选取的有效性,本文利用ResNet18 网络对目前脑图分类选取较多的横断面图像和本文选取的特征区域图像进行AD、CN 和EMCI 的分类,分类效果表7 所示。
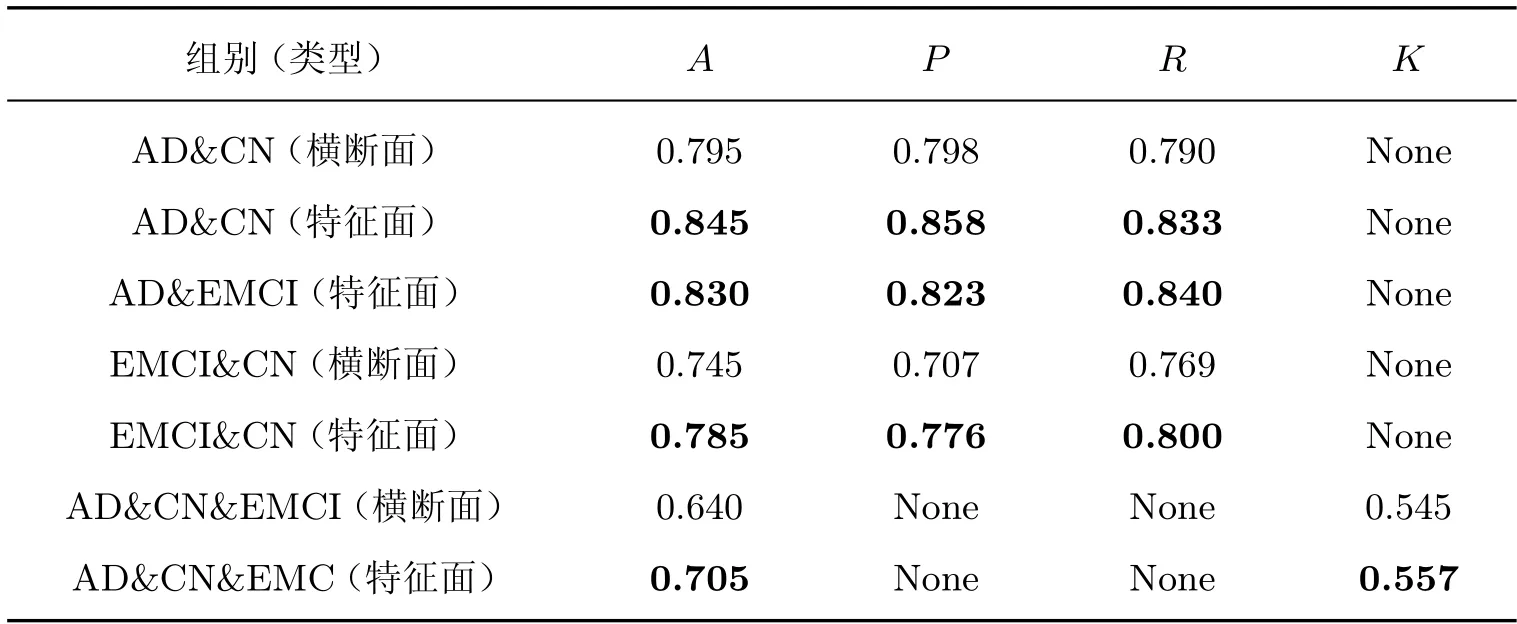
表7 横断面和轴状面分类效果对比Table 7 Comparison of classification results between cross-sectional and axial plane
由表7 可知,在利用特征面做分类脑图之后,因提取到了更为显著的脑图像特征,分类效果显著提升。
为探究加入临床标志物后模型分类效果,本实验在特征面脑图像的基础上逐步增加生物标志物模态的种类,利用图8 的联合分类网络对AD、CN、EMCI 进行分类识别,分类效果如表8 所示。
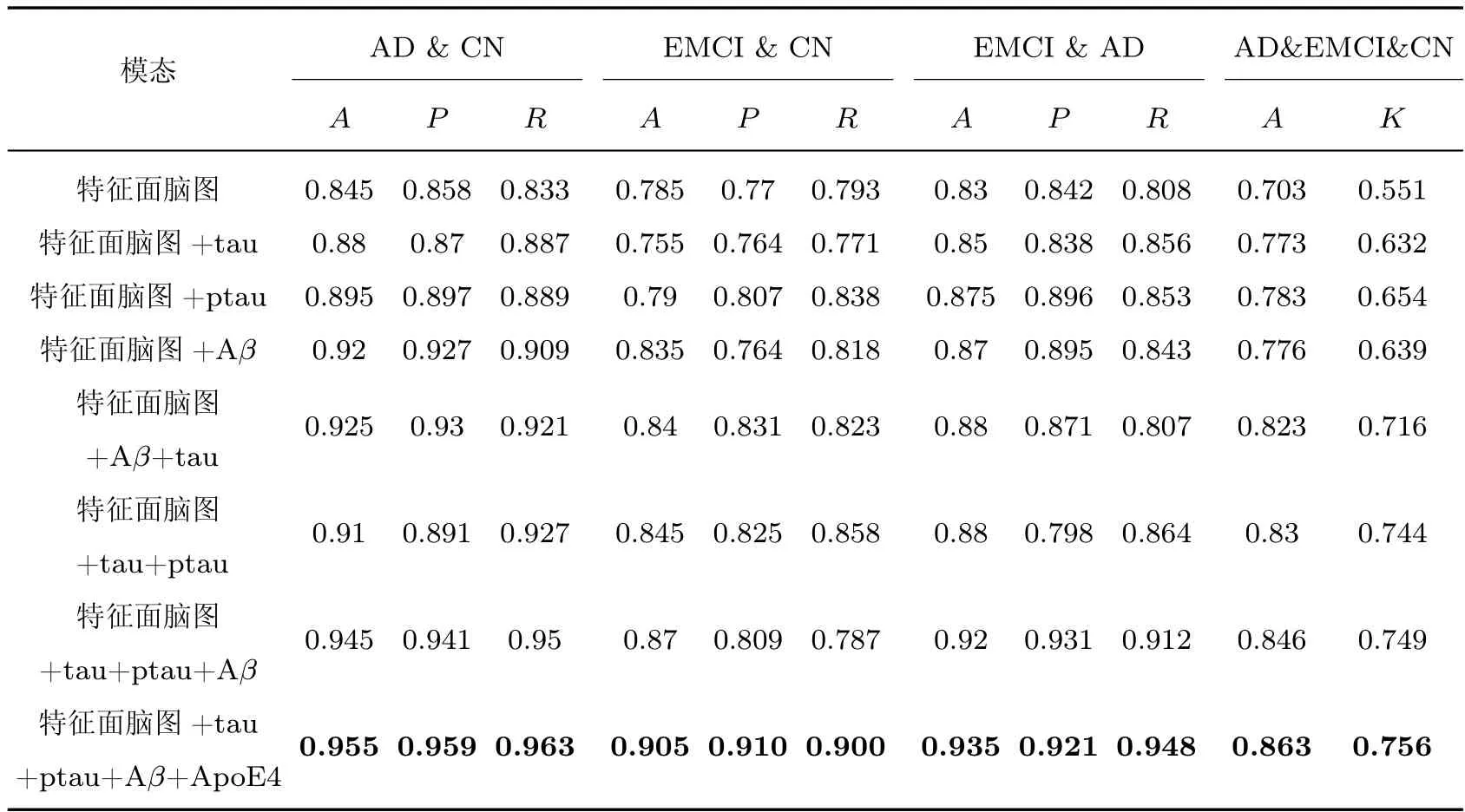
表8 不同模态组别分类效果Table 8 Classification results of different modal groups
上述结果显示在逐步增加生物标志物的种类时,模型的分类性能提高,这表明生物标志物对AD 分类具有提升效果。
为探究生物标志物的特征表达能力,利用图8 的联合分类网络对AD、CN、EMCI 样本的生物标志物进行分类识别,将图像特征的权重调节为0,特征融合方式由拼接改为加和,分类准确率如表9 所示。
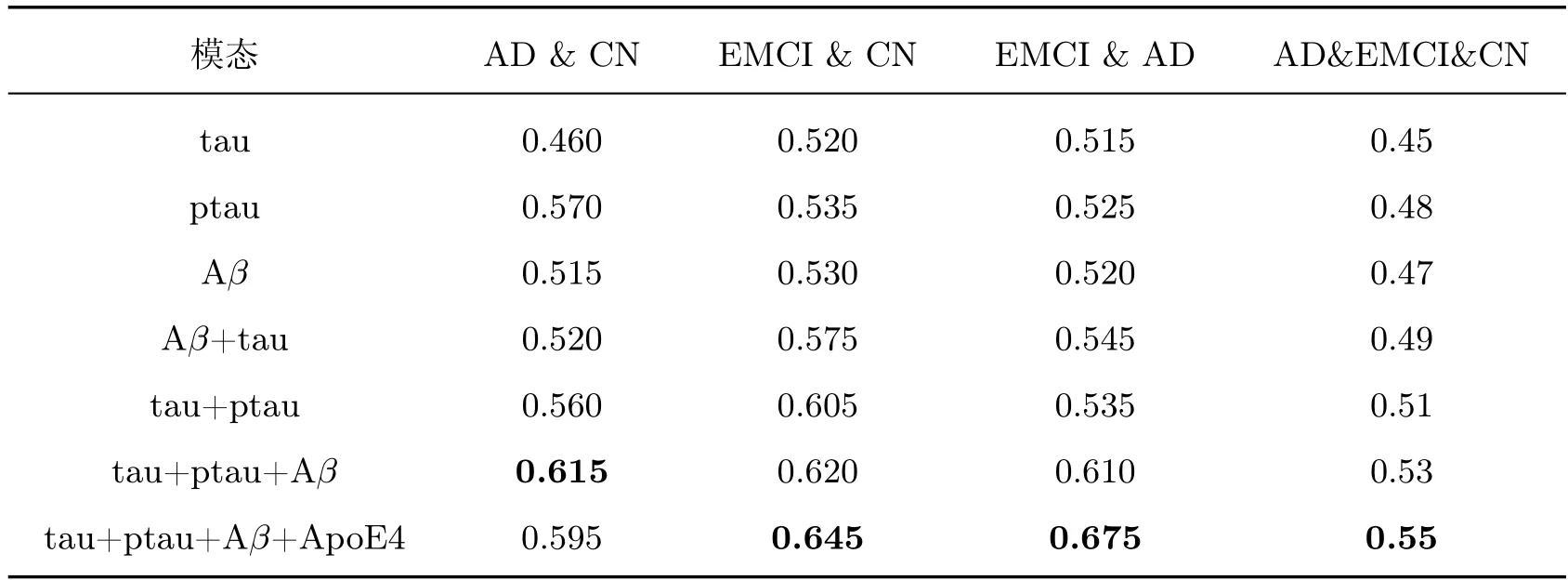
表9 不同生物标志物分类效果Table 9 Classification results of different biomarker groups
上述分类实验显示仅利用生物标志物对三类样本分类识别,模型的分类效果一般,而脑切片影像和生物标志物结合的分类方式会取得良好的分类效果。
最后,将本文方法与文献[5-6,8] 分类方法相比较,由于不同方法衡量指标有差别,这里仅选取3 种方法都选用的准确率衡量指标作为不同方法结果对比指标,比较结果如表10所示。
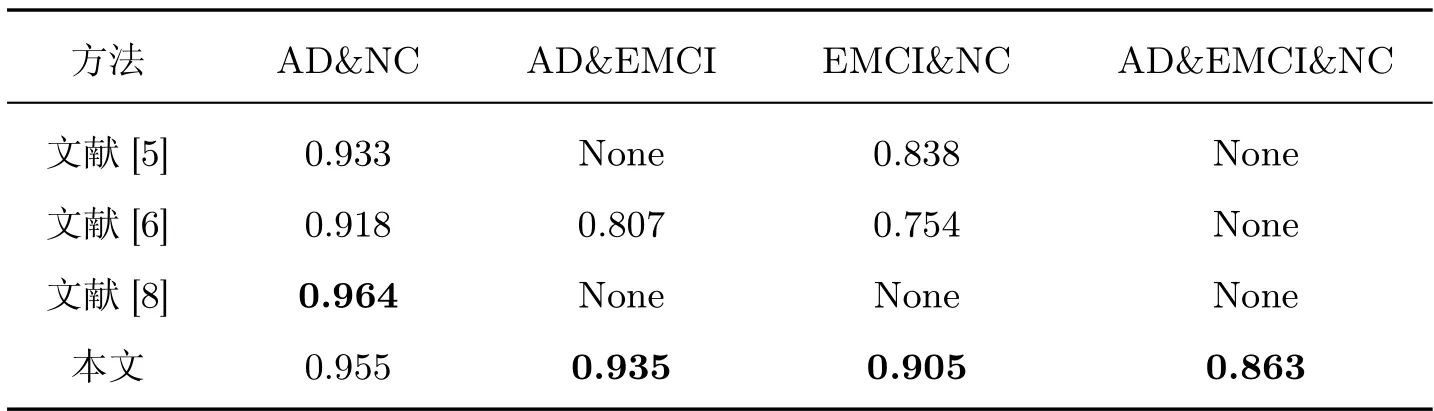
表10 不同方法分类结果Table 10 Classification results of different methods
比较3 种分类方法的分类效果可知,本文通过选取特征区域脑切片结合生物标志物的多模态分类方法,在各组别分类中均优于文献[5] 所用基于VGG 神经网络的弱监督细粒度分类方法和文献[6] 所用基于Alexnet 模型的AD 分类方法。文献[8] 提出的基于卷积循环神经网络的阿尔茨海默症早期诊断方法仅对AD 和NC 组别进行了分类,每组仅有100 个样本,而本文方法每组别各300 个样本,均包含MRI 图像信息和生物标志物信息,在结果上更具有说服力。同时本文分类组别更完善,所用神经网络架构也更为轻量级,同时适用于任意组别分类,具有广泛的应用前景。
4 结语
本文提出了一种选取大脑萎缩区域脑切片并结合生物标志物的多模态疾病分类方法。该方法通过将不同组别MRI 脑图像进行组间差异性分析,获得最大脑萎缩区域。依照最大脑萎缩区域所示位置对所有组别进行特征图选取,将选取的脑切片和生物标志物分别利用ResNet18 网络和多层感知机进行特征的提取和加权融合,最后通过Softmax 层进行分类识别。
实验表明,选取大脑萎缩区域脑切片并结合生物标志物的多模态疾病分类方法能够有效提高网络模型的分类效果,可以广泛应用于智慧医疗领域。此外,该模型可推广到癫痫、小儿麻痹症等脑病的分析与诊断,具有广泛的推广价值和适用性。

