青海草原毛虫化学感受蛋白基因的鉴定与组织表达分析
南彦斌, 唐德靖, 杨永超, 孔一森, 周渊涛
(青海大学农牧学院, 青海 西宁 810016)
化学感受蛋白(Chemosensory proteins,CSPs)是类似六棱柱的小球状蛋白质,以其在信息素感知方面复杂的功能而闻名[1]。它有四个保守的半胱氨酸位点,构成C1-C2和C3-C4两个二硫键,具有6个α螺旋[2]。在CSPs的研究进程中,其命名发生数次变动,起初由于其在触角中优先表达因此为其命名为嗅觉特异-D蛋白质(Olfactory-specific-D proteins,OS-D)或信息素结合蛋白A-10 (Pheromone-binding proteins A-10)[3],后又在一些昆虫中的感觉器官中(如下唇须和足等)发现该家族基因也可以表达,故又称为感受器官蛋白(Sensory appendage protein,SAP)。事实上,它们属于同一类基因,通常作为气味分子和化学信号刺激物的载体蛋白,在感受器树突膜上的受体位点与受体蛋白结合。当前,这类蛋白被称为化学感受蛋白(CSP)[4]。CSPs可感知宿主气味和性信息素[5],且其功能也可能与肢体修复、昼夜节律周期调节、生长发育、免疫反应、摄食行为和杀虫剂抗性有关[6]。昆虫触角和下颚须等化学感受部位可以合成化学感受蛋白,合成的CSPs被分泌到淋巴液中,其携带的信息素或挥发物等疏水性物质进入化学感受器的神经元中,引起下游行为反应的发生,因此,化学感受蛋白在昆虫感知外部环境的过程中CSPs起到重要作用[7]。CSPs在昆虫中具有高度的保守性,它们的主要功能是与气味分子结合,然后将这些分子运输到受体[8]。近年来,随着转录组测序技术的发展,各种昆虫基因组序列被检测,大量的昆虫CSPs基因被鉴定出来。CSPs的数量因不同的昆虫物种而异。例如,在家蚕Bombyxmori中发现20个CSP,在东亚飞蝗Locustamigratoria中发现70个CSP,在意大利蜜蜂Apismellifera中仅发现6个CSP[9]。目前大多数CSPs的功能是未知的。因此,CSPs的嗅觉和非嗅觉功能需要进一步研究[10]。随着生物技术的快速发展,利用实时荧光定量PCR技术成为研究基因表达与定位的重要手段。
青海草原毛虫(Gynaephoraqinghaiensis)俗称红头黑毛虫,属于鳞翅目(Lepidoptera)毒蛾科(Lymantriidae)草原毛虫属(Gynaephora)[11]。迄今为止,全球共有15个物种被发现,其中8个是青藏高原特有种[12,13]。青海草原毛虫幼虫具有7个龄期,雌雄成虫具有显著的不同,其雌成虫翅、触角、胸足皆退化[14]。在青海地区重点分布在门源、海晏、天俊、泽库、玛多、甘德、祁连、杂多和治多等地方[15]。此外,其幼虫背部有毒腺,可导致家畜和野生动物的口腔溃疡和断舌病,使它们无法进食,最终导致死亡[16,17]。近年来,青海草原毛虫在青藏高原爆发成灾的报道很多,反映出其已能够很好的适应青藏高原缺氧、严寒、强紫外辐射的极端恶劣的环境[18]。青海草原毛虫适应环境能力和繁殖能力强,防治困难,当前青海草原毛虫主要采取化学防治,长期的化学防治必然导致抗药性和农药残留的问题[19]。昆虫的CSPs具有作为害虫防控新靶标的潜力,为害虫的绿色防治提供了方向[4]。当前,已经获得多种鳞翅目昆虫的CSPs基因[20],但是在青海草原毛虫嗅觉方面的研究较少,且未见关于该虫CSPs基因的研究报道。因此,本研究基于青海草原毛虫幼虫及雌雄成虫转录组拟筛选鉴定青海草原毛虫CSPs并进行生物信息学分析,通过RT-qPCR技术分析青海草原毛虫CSPs基因在不同组织和发育阶段中的表达量,以期为深入研究青海草原毛虫化学感受机制奠定基础。
1 材料与方法
1.1 供试昆虫
供试青海草原毛虫幼虫及成虫来源于2020年5—8月采集自青海省海北州(36°59′06.6″ N,100°52′19.1″ E,海拔3 095.1 m)的野外种群。在人工气候箱中将采集的部分青海草原毛虫成虫雌雄配对,置于塑料盒(d=10 cm)中进行短期饲养,用牧草提供营养且作为产卵场所收集卵期样品。饲养条件:温度(26±1)℃,光周期16L∶8D,相对湿度60%~80%。对采集的青海草原毛虫幼虫根据头壳宽度挑选出7龄幼虫,进行短期饲养直至化蛹以收集蛹期样品。收集青海草原毛虫雄成虫的触角、头(去除触角)、胸、腹、翅和足,获得雄成虫共6种部位的样品。青海草原毛虫雌成虫触角、翅和足发生退化,收集到头(去除触角)、胸、腹共3种部位的样品,另外收集了青海草原毛虫卵、2至7龄的幼虫和蛹,每种样品取3个生物学重复。每个重复收集数量根据组织或个体大小从3~30不等,用液氮速冻分装储存在-80℃冰箱中备用[21]。
1.2 总RNA提取及cDNA合成
通过Trizol法提取青海草原毛虫各个样品的总RNA。用1%琼脂糖凝胶电泳分析样品RNA完整性及是否存在DNA污染。同时,利用超微量分光光度计检测RNA纯度,当OD260nm/OD280nm的范围为1.8~2.2,表明提取的RNA合格。之后对检测纯度合格的RNA样品利用反转录试剂盒TRUEscript 1 st Strand cDNA Synthesis Kit(艾德莱,北京)的说明进行cDNA第一链的合成并置于—20℃冰箱中保存备用。
1.3 引物设计与合成
根据青海草原毛虫转录组数据信息确定并获得青海草原毛虫CSPs基因序列,利用Oligo7软件设计引物,内参基因(EF1-α-F)引物由兰州大学昆虫实验室筛选获取[18],引物委托北京睿博兴科生物技术有限公司合成。
1.4 青海草原毛虫CSPs基因序列获取
基于前期通过第二代高通量测序技术对青海草原毛虫4龄幼虫、雌雄成虫进行转录组测序的结果[15],以“CSP”和“chemosensory proteins”为关键词在各个库中进行查找,为确定候选基因将所有化学感受蛋白基因序列在NCBI网站的数据库中进行BLASTn比对。
利用ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)预测出ORF完整的核苷酸和氨基酸序列,之后在NCBI中对预测出的ORF氨基酸序列进行BLASTp验证,设定e值小于10-5,相似性大于40%,得到推测的CSPs蛋白序列。
1.5 青海草原毛虫CSPs基因的生物信息学分析
利用ExPASy-ProtParam tool(http://web.expasy.org/protparam/)软件对化学感受蛋白氨基酸序列组成、相对分子量、等电点、正负电荷残基数、不稳定系数、脂肪系数和总平均疏水性进行计算[22]。采用Signalp4.1Server(http://cbs.dtu.dk/services/Signalp/)预测其信号肽。
利用在线预测网站Clustal Omega (http:∥www.ebi.ac.uk/Tools/msa/clustalo/)对筛选获得的青海草原毛虫CSPs进行同源性比对[23]。
采用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.plpage=npsa%20_sopma.html)进行蛋白质二级结构预测;SWISS MODEL(https://www. swissmodel.expasy.org/)进行蛋白质的三级结构预测[24]。
采用DNAMAN6.0软件进行氨基酸多序列比对。使用MEGA7.0[25]软件中的邻位相邻算法同其他昆虫构建系统进化树。设置参数为重复频次1 000,替换方法Jones-Taylor-Thornton(JTT)model,空缺数据的处理95%部分删除,其他参数默认。
1.6 青海草原毛虫CSPs基因的qPCR检测
以1.2节合成的cDNA样品为模板,每个样品含3次生物学重复,每次生物学重复进行3次技术重复,通过熔解曲线评估引物的特异性。扩增体系为20 μL:SYBR Green Real-time PCR Master Mix 10 μL,正反向引物(10 μmol·μL-1)各0.8 μL,cDNA 1 μL(100 ng),ddH2O 7.4 μL。在ABI 7 500型实时荧光定量PCR仪中进行qRT-PCR反应,运行条件:95℃ 1 min;95℃ 15 s,60℃ 15 s,循环40次;72℃ 45 s。在成虫的各组织中以每个CSP在雌性头部的表达量为基准,在幼虫中以7龄幼虫为阳性对照。
1.7 数据分析
青海草原毛虫CSPs基因在不同组织及不同龄期中的相对表达量利用2-ΔΔCt值法进行计算,其中,ΔCt=Ct目的基因-Ct内参基因;ΔΔCt=ΔCt样品-ΔCt对照[26]。通过SPSS 26.0软件进行统计学分析,对CSPs基因在不同组织、不同发育阶段的表达量差异进行单因素方差分析(ANOVA),同时利用Duncan氏新复极差法进行多重比较;最后使用Graphpad prism绘制基因的组织表达谱。
2 结果与分析
2.1 青海草原毛虫CSPs的生物信息学分析
在青海草原毛虫的基因组数据库中筛选出11个CSPs(表2),经过ORFfinder与NCBI BLASTp验证后,其核苷酸长度大小介于222~381 bp之间。GqinCSPs的氨基酸大小差异很大,最短的有74个氨基酸,最长的有127个氨基酸,平均长度为112个氨基酸。采用软件TransDecoder预测出该11个GqinCSPs的编码区序列都是完整的。采用在线网站SignaIP4.1预测其信号肽,结果表明GqinCSP5和GqinCSP8两个基因没有信号肽。将GqinCSPs与其他昆虫序列相似性进行比较,GqinCSP6基因与菊黄花粉蝶Zerenecesonia基因序列(Gen Bank登录号为XP_038221853)相似度最高,达到84.11%。其次,GqinCSP2基因与苜蓿盲蝽Adelphocorislineolatus基因序列(Gen Bank登录号为AXS78221)的相似度达到了83.33%。GqinCSPs基因与其他昆虫物种相似性高于60%的有8个,占总数的72.73%,表明GqinCSPs与这些鳞翅目昆虫序列可能同源。

表2 青海草原毛虫CSPs生物信息学分析Table 2 Bioinformatics analysis of CSPs in G. qinghaiensis
利用在线预测网站Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clustalo/)对筛出的11条GqinCSPs进行比对(表3),其中相似性最高的是GqinCSP4与GqinCSP9,为56.76%,相似性最低的是GqinCSP5与GqinCSP8,为8.05%,GqinCSPs序列之间的相似性反映出它们之间的同源性,由此可以得出GqinCSP1与GqinCSP2和GqinCSP4,GqinCSP2和GqinCSP9以及GqinCSP4和GqinCSP9之间的同源性都比较高。
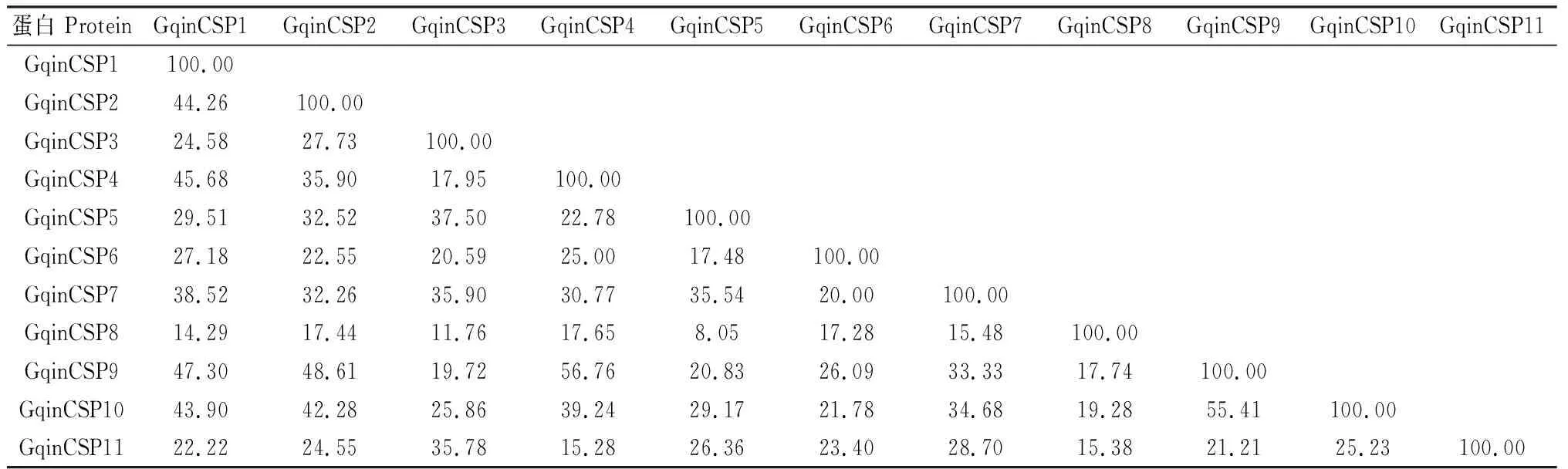
表3 青海草原毛虫化学感受蛋白CSPs氨基酸序列之间的相似性Table 3 The similarity of amino acid sequences of chemosensory proteins CSPs from G. qinghaiensis 单位:%
2.2 青海草原毛虫CSPs理化性质分析
利用ExPASy-ProtParam tool(http://web.expasy.org/protparam/)软件对青海草原毛虫化学感受蛋白理化性质进行预测(表4),青海草原毛虫11个CSPs的氨基酸长度区间是74~127aa,相对分子量在8~15 kD之间。其理论等电点大多数位于6附近,如GqinCSP3-5、GqinCSP7、GqinCSP9-11,表明其属于偏酸性蛋白。GqinCSP1,GqinCSP2,GqinCSP6,GqinCSP8蛋白理论等电点位于8附近,表明其属于偏碱性蛋白。CSP带正电荷和负电荷的残基数目基本相同。GqinCSP1-3,GqinCSP6-11这7个化学感受蛋白的不稳定系数高于40,表明这些蛋白的性质不稳定。从脂肪系数可以看出11个CSPs均为热稳定性蛋白。除GqinCSP1,GqinCSP2,GqinCSP8,GqinCSP10四个蛋白外,其余青海草原毛虫CSPs的总平均疏水性都介于-0.5~0.5之间。因此,判定剩余蛋白为两性蛋白质(+值为疏水性,-值为亲水性,-0.5~0.5之间为两性蛋白质)。

表4 青海草原毛虫化学感受蛋白理化性质Table 4 The physicochemical properties of chemosensory proteins CSPs from G. qinghaiensis
2.3 青海草原毛虫CSPs的二级结构预测分析
利用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.plpage=npsa%20_sopma.html)在线网站预测GqinCSPs二级结构(图1),发现α螺旋是青海草原毛虫化学感受蛋白的二级结构主要组成部分,除GqinCSP8和GqinCSP9外,其他GqinCSPs的α螺旋含量都比较高,其中GqinCSP3 α螺旋含量最高。其次是无规则卷曲。β折叠和延伸链的氨基酸残基较少。由此, 我们推测青海草原毛虫CSPs的二级结构主要由α螺旋和无规则卷曲形成并维持稳定。
2.4 青海草原毛虫CSPs的三级结构的预测
采用SWISS MODEL(https://www.swissmodel.expasy.org/)网站同源建模得到青海草原毛虫CSPs蛋白的三级结构模型,结果发现,GqinCSP1-3,GqinCSP5-7,GqinCSP10和GqinCSP11,都由6个α螺旋组成,各个螺旋之间都由半胱氨酸形成二硫键相互缔合,二硫键对三级结构起到支撑作用,其三维结构形成一个亲水的表面和一个疏水的空腔,由此形成一个闭合的可以容纳小分子的疏水性结合腔(图2)。GqinCSP4,GqinCSP8和GqinCSP9建模后,与其他的蛋白三级结构差异较大,α螺旋结构较少。
2.5 青海草原毛虫CSPs基因的多序列比对
利用DNAMAN6.0软件对GqinCSPs进行多序列比对,通过比对得到10个GqinCSPs的半胱氨酸模式识别序列为C1-X6-C2-X11-19-C3-X2-C4 (C为半胱氨酸,X为任意氨基酸)(图3),这和已经发现的鳞翅目昆虫CSPs家族无差异。但是,图中反映出GqinCSP8序列不属于保守半胱氨酸序列,原因可能是在青海草原毛虫的进化过程中某些基因片段自然消除。

图3 青海草原毛虫11个化学感受蛋白CSP1~CSP11的氨基酸序列比对Fig.3 Sequence alignment of amino acids of 11 chemosensory proteins CSP1-CSP11 in G. qinghaiensis注:图中黑色C1-4代表保守半胱氨酸模式序列Note:The black C1-4 represents the conserved cysteine pattern sequence in the figure
2.6 青海草原毛虫CSPs基因的系统进化分析
为明确青海草原毛虫CSP与其他近缘种的进化关系,采用NJ法构建青海草原毛虫化学感受蛋白与其他昆虫的CSPs系统发育树,参与建树的包括青海草原毛虫的11个CSP氨基酸序列和其他5种昆虫的68个CSP氨基酸序列。结果显示青海草原毛虫11个CSP在整个进化系统中分布比较分散,但所有的GqinCSPs与鳞翅目昆虫CSPs聚到一起(图4)。其中GqinCSP1和HarmCSP19、GqinCSP2和HarmCSP20、GqinCSP5和PxutCSP12、GqinCSP8和EhipCSP5、GqinCSP10和PxutCSP8及GqinCSP11和PxutCSP11b聚到同一个小分支中。因此,青海草原毛虫CSPs与棉铃虫和柑橘凤蝶亲缘关系最近。
2.7 青海草原毛虫CSP基因的组织表达谱
为明确青海草原毛虫功能,以雄虫头部作为阳性对照,通过RT-qPCR技术测定11个CSP基因在青海草原毛虫不同部位的相对表达量差异情况,结果表明,11个GqinCSPs基因在雌雄成虫头部和雄虫触角中均有表达,且体现不同的表达水平,其中:GqinCSP2在雄虫头部的表达量显著高于雌虫头部(P<0.05),其余CSP基因在雌雄成虫头部的表达无显著差异,而GqinCSP1,GqinCSP2,GqinCSP4,GqinCSP5,GqinCSP10在雌性成虫头部的相对表达量显著高于其他组织(P<0.05);GqinCSP3,GqinCSP7,GqinCSP10,GqinCSP11在雄成虫触角的相对表达量显著高于其他组织(P<0.05)。此外,GqinCSP1,GqinCSP3,GqinCSP4,GqinCSP5,GqinCSP9,GqinCSP10在雌雄成虫胸部不表达或微量表达;GqinCSP1,GqinCSP2,GqinCSP8,GqinCSP10在雌雄成虫腹部的表达存在显著差异,且呈现雄性偏好表达模式,而GqinCSP3则呈现雌性偏好表达模式(P<0.05);GqinCSP2,GqinCSP10在雌雄成虫胸翅部不表达或微量表达;GqinCSP4,GqinCSP5,GqinCSP6,GqinCSP8,GqinCSP9在雄性成虫足中的相对表达量显著高于其他组织(P<0.05)(图5)。

图5 青海草原毛虫雌雄成虫化学感受蛋白基因组织表达谱分析Fig.5 The tissue expression profile of chemoreceptor protein genes in male and female adults of G. qinghaiensis注:雌虫;雄虫;头(去除触角);胸;腹;触角;翅;足。EF-1-α作为内参基因进行校正各组织的表达量,目的基因在雄性头部中表达量作为阳性对照。误差线表示3次独立试验的标准误,不同的大小写字母表示不同的雌雄组织之间的显著性差异(Duncan:P<0.05)。单星号和双星号表示目的基因在同一组织的相对表达量雌雄间差异显著(t检验,*P<0.05;**P<0.01)Note:Head (with antennae removed) (Hd),Thorax (Th),Abdomen (Ab),Antennae (An),Wings (Wg),Legs (Lg). EF-1- α was used as a reference gene to correct the expression level in each tissue,and the expression level of the target gene in the male head served as a positive control. Error lines represent the standard error of 3 independent trials. Different uppercase letters indicate a significant difference between different tissues of male and female (Duncan:P<0.05). A single asterisk or double asterisk indicate a significant or very significant difference of the relative expression of the target gene in the same tissue (t-test,*P<0.05;**P<0.01)
2.8 青海草原毛虫CSP基因不同发育阶段表达谱
不同发育阶段,青海草原毛虫CSP基因表达水平显著不同。以7龄幼虫作为阳性对照,测定11个CSP基因在青海草原毛虫不同发育阶段的相对表达量差异情况。结果表明,11个青海草原毛虫CSP基因在2龄与7龄阶段均有表达,且体现不同的表达水平,GqinCSP1,GqinCSP3,GqinCSP5,GqinCSP8,GqinCSP9在2龄阶段的相对表达量显著高于其他阶段(P<0.05)。此外,GqinCSP11在卵中的相对表达量明显高于其他发育阶段(P<0.05);GqinCSP7在3龄阶段的表达量极高,是4龄幼虫表达量的140倍;GqinCSP9和GqinCSP4分别在三龄和四龄幼虫中不表达(图6)。
3 讨论
本研究从青海草原毛虫的基因组数组库中,共鉴定得到11个青海草原毛虫CSPs基因,其数目少于菜粉蝶Pierisrapae(38个CSPs)[27],悬铃木方翅网蝽Corythuchaciliata(26个CSP)[28],蚜虫Acyrthosiphonpisum(13个CSPs)但数目多于意大利蜜蜂Apismellifera(6个CSPs)[29],甘蓝蚜Lipaphiserysimi(8个CSPs)[30],点蜂缘蝽Riptortuspedestris(4个CSP)基因[31]。青海草原毛虫CSPs属于分泌型疏水性蛋白,热稳定性较高。除GqinCSP5和GqinCSP8外都有信号肽。全长的CSPs序列,无信号肽的原因推测是不具有信号肽剪切位点造成的[24]。二级结构预测显示,GqinCSPs主要由α螺旋和无规则卷曲组成。三级结构预测显示,8个GqinCSPs三级结构具有6个α螺旋,表明青海草原毛虫CSPs种内结构相似度高,三维结构相似的化学感受蛋白可能在昆虫寻找寄主、识别定位中具有相似的功能[32]。多序列比对的结果表明GqinCSP8蛋白序列不属于保守半胱氨酸序列,推测其原因可能有两点:一是青海草原毛虫在进化时自然丢失[33]。二是在高通量转录过程中造成基因片段的缺失。GqinCSPs系统发育分析表明,大部分GqinCSPs与柑橘凤蝶Papilioxuthus和棉铃虫Heliothisarmigera聚到同一个小分枝上。只有少数几个GqinCSPs没有聚在同一个小分枝上,但聚集在同一个大进化枝上,表明这些物种的CSP基因可能拥有共同的祖先,但为了适应不同的环境,后来逐渐分化成不同的基因型。
基因功能研究的重要参考手段是表达谱分析[34]。本研究利用用qRT-PCR技术对青海草原毛虫不同发育阶段和成虫不同组织中的表达情况进行了分析,GqinCSPs基因在雌雄成虫头部和雄虫触角中均有表达,且体现不同的表达水平。这与张志春等[35]研究的小菜蛾CSP不仅在化学器官中表达,而且在其它非感受器官也有表达的结果相同,表明GqinCSPs可能参与昆虫化学感受以外的其它生理过程。GqinCSP3在雌虫的腹部高表达,推测其原因:GqinCSP3对雌虫激素分泌、卵巢发育及产卵等众多生理过程起着重要作用[36]。GqinCSP2在雄虫头部的表达量显著高于雌虫头部,这表明该基因可能与性别特异性行为有关[37]。本研究获得的GqinCSP3,GqinCSP7,GqinCSP10,GqinCSP11在雄成虫触角中表达量较高,推测其在雄虫寻找和定位寄主植物的过程中发挥重要作用[38]。GqinCSP4,GqinCSP5,GqinCSP6,GqinCSP8,GqinCSP9在雄足中的表达量最高,推测这五个基因可能与昆虫的运动行为相关,有助于其生长发育并扩大分布范围[39],同时也反应了青海草原毛虫对外环境的一种适应机制。
不同发育阶段的青海草原毛虫表达谱显示,GqinCSP1,GqinCSP3,GqinCSP5,GqinCSP8,GqinCSP9在2龄阶段的相对表达量显著高于其他阶段,推测其原因是这些基因在幼虫信息素运输中发挥着关键作用[40]。GqinCSP11在卵中的相对表达量明显高于其他发育阶段,这与谭瑶等[41]的研究结果,GdHsp70在卵期高表达相同,推测其可能参与胚胎的正常发育[42]。因此,GqinCSPs在不同组织与发育阶段的表达谱非常复杂,表明它们在青海草原毛虫的生长发育和化学感受过程中起着至关重要的作用。
4 结论
本研究基于青海草原毛虫转录组测序数据,利用生物信息学方法首次鉴定出青海草原毛虫的11个CSP基因,之后对其表达谱进行分析,发现11个青海草原毛虫CSP基因在雌雄成虫头部、雄虫触角以及2龄至7龄阶段中均有表达,且体现不同的表达水平,为进一步研究青海草原毛虫嗅觉识别机制提供了理论基础。

