镜鲤体质量、体长、体高和体厚的QTL 定位分析
张晓峰,栾培贤,户国,李超,鲁翠云,曹顶臣
(中国水产科学研究院黑龙江水产研究所,淡水鱼类育种国家地方联合工程实验室,黑龙江 哈尔滨 150070)
水产品是人类的重要营养来源,特别是在食物缺乏的国家和地区[1,2]。持续改良和提高水产品的产量性状,满足人们不断增长的对水产品的需求,一直是水产育种者面临的长期挑战之一[3,4]。作为全球最重要的养殖鱼类之一,鲤(Cyprinus carpio)在全球100 多个国家养殖,全球年产量超过400 万公吨(http://www.fao.org/fishery/statistics/global-aquaculture-production/query/en)。鉴于鲤对水产养殖业的重要性,在过去几十年中,开发了各种基因组资源和遗传工具,以促进鲤遗传改良和育种计划[5-10]。鲤育种方法已经从传统的选择发展到现代生物技术,如标记辅助选择(MAS)和分子育种等[11-13]。
鱼类多数经济性状都为数量性状,如生长、抗病性、性别和肌肉品质等,由多个基因控制,称为数量性状座位(quantitative trait locus,QTL)[14-16]。这些性状的表型变异被认为是由多个数量性状位点(QTL)等位基因分离产生的数量遗传变异引起。通过传统常规选育费时费力,分子标记辅助选择(marker-assisted selection,MAS)可以利用分子标记直接选择基因型,使得优势基因型快速富集,提高目标性状的改良效率。MAS 的实施需要通过QTL定位或关联分析获得与感兴趣的性状紧密连锁的DNA 标记[17,18]。QTL 定位的目的是了解决定性状的基因的数量和作用,并协助育种选择,以加速重要性状的遗传改良[19]。
生长性状作为鲤的重要经济性状之一,揭示其性状复杂的分子机制并挖掘影响该性状的重要功能基因,可以为鲤生长性状分子标记辅助选择育种奠定基础。近年来随着鲤基因组资源的快速开发利用,鲤重要性状相关QTL 定位分析已开展了很多研究。例如生长[20-25]、抗性[5,26]、肌肉品质[16,27]和饲料转化率[28,29]等,大大促进了鲤分子标记辅助选择和分子育种进程。尤其是,不同品种和家系鲤生长相关QTL 定位分析取得了很多研究结果。虽然关于镜鲤生长相关性状的QTL 已有多篇报道,但由于作图群体的样本量和使用的标记数量相对较少,并不是所有性状相关的DNA 变异完全被捕获。因此,亟需构建高密度相对较高的遗传连锁图谱,以利于QTL 精细定位和候选基因的筛选。
本研究拟对镜鲤F1家系进行SLAF-seq(specific-locus amplified fragment sequencing)测序,利用SNP 标记构建鲤高密度遗传连锁图谱。利用该高密度图谱对镜鲤主要生长性状进行QTL 精准定位,挖掘与这些性状相关的分子标记及候选基因,为鲤MAS 育种提供依据。
1 材料与方法
1.1 材料
本实验用镜鲤采自黑龙江水产研究所呼兰试验站。F0(祖父母本)为早期由国家原种和良种审定委员会鉴定为良种的德国镜鲤选育系的后代,经微卫星分子标记检测亲本间的遗传距离,选取遗传距离大的一对亲本构建F1家系,池塘养殖6 个月后,随机选取200 尾子代个体,测量表型性状。
1.2 方法
表型性状的测量:根据中华人民共和国国家标准《养殖鱼类种质检验》(GB/T 18654.3-2008),测量了研究群体的体质量(BW)、体长(SL)、体高(H)和体厚(TH)4 个性状,获取试验群体生长性状表型值。利用SPASS 19.0 对性状进行描述性统计分析和正态分布检验。
基因型分析:表型值测量结束将实验鱼麻醉,尾静脉抽血0.5 mL。采用QIAampDNA Blood Midi Kit 血液提取试剂盒,参照标准流程提取血液样本的DNA。用1.5%琼脂糖凝胶电泳检测基因组DNA的完整性,利用NanoDrop-1000 超微量分光光度计检测基因组DNA 浓度,检测合格的DNA 样本置于-20 ℃冰箱保存备用。委托百迈克生物有限公司,利用Illumina HiseqTM 平台进行SLAF-seq 和SNP 标记的鉴定分型。
遗传连锁图谱的构建和QTL 定位:选择符合孟德尔遗传的多态性SNP 标记用于进一步的连锁分析。连锁分析采用双伪测交策略(CP)。根据分离模式将SNP 分为三类:ABxAA 或ABxBB(仅在母本中1∶1 分离)、AAxAB 或BBxAB(仅在父本中1∶1 分离)和ABxAB(双亲中1∶2∶1 分离)。利用Join-Map 4.0 软件构建镜鲤高密度遗传图谱[30]。分析模型为CP(cross pollinators),作图函数为Kosambi 函数。以极大似然法构建遗传连锁图谱,LOD 值设置为6.0;采用Kosambi 函数计算标记间的遗传距离(cM)。图谱绘制软件为MapChart 2.2[32]。
用MapQTL5.0[31]软件包中区间作图(interval mapping,IM)对四个生长性状进行QTL 定位分析,取LOD 阈值为2.5。通过逐步回归(stepwise regression)估计表型方差(PVE)。QTL 最近的SNP 标记附近的生长基因做为候选基因,注释基因的功能。
2 结果与分析
2.1 表型性状的统计分析
利用SPSS19.0 对生长性状数据进行描述性统计,计算了性状的平均值、偏度、峰度、最小值、最大值和P 值(表1)。表1 表明,体质量测量值介于88.90~273.30 g,平均(162.84±42.71)g;体长测量值介于11.73~17.90 cm,平均为(15.91±1.32)cm;体高测量值介于5.58~8.32 cm,平均为(6.83±0.66)cm;体厚测量值介于2.94~4.59 cm,均值为(3.51±0.33)cm。根据Shapiro-Wilk 正态分布检验的显著性阈值,四个性状P 值均大于0.05,符合正态分布(图1)。

图1 体质量、体长、体高和体厚性状的正态分布Fig.1 The normal distribution of body weight(BW)(a),body length(SL)(b),body depth(H)(c)and body thickness(TH)(d)
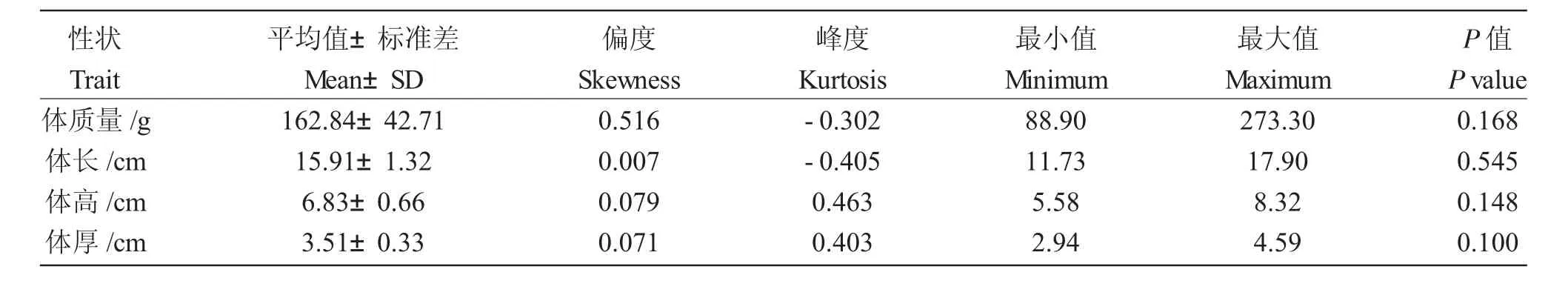
表1 4 种性状表型数据统计及正态分布检验Tab.1 Descriptive statistics of phenotypic data of growth-related traits.
2.2 遗传连锁图谱的构建
SNPs 标记基因分型数据由百迈克生物有限公司自主开发的软件进行处理分析,剔除低质量的基因分型和不符合孟德尔标记后,剩余4 026 个均匀分布鲤基因组的高质量的多态性SNPs 标记用于镜鲤遗传连锁构建图谱。为提高构建图谱的准确性,将个体基因型缺失20%以上的个体剔除,剩余107个子代用于遗传连锁图谱的构建。用JoinMap4.0 软件的“CP”作图模型对这4 026 个分子标记进行连锁分析,共得到50 个连锁群,最终构建的图谱包含3 903 个SNP 标记,图谱总长度为3 923.31 cM,全基因组覆盖度97.26%,每个连锁群有78.06.1 个标记,连锁群长度范围为40.83~120.01 cM,标记间平均距离为0.998 cM。
2.3 生长性状的QTL 定位
采用MapQTL5.0 软件的区间定位作图,取LOD值2.5 做为阈值,共检测到29 个与镜鲤生长性状相关的QTLs,分别定位到11 个连锁群(LG3、LG5、LG6、LG7、LG12、LG16、LG30、LG38、LG44、LG46 和LG47)上,LOD 值介于2.50~4.09 之间,解释表型变异介于9.0%~24.2%之间(表2、图2)。其中,与体质量相关的QTLs 最多,为20 个,其次为体高和体厚性状,均为15 个,体长最少,为11 个。从连锁群分布看,LG5、LG6 和LG38 连锁群最多,均为6 个QTL区间。其次是LG30 和LG12,分别为3 个和2 个,其余6 个连锁群(LG3、LG7、LG16、LG44、LG46 和LG47)均含有1 个QTL 区间。本研究中的29 个生长性状QTLs 中,3 个QTLs(QTL6_5、QTL6_6 和QTL44_1)的单个QTL 解释表型变异率大于20%,为生长性状的主效QTL 区间。解释表型变异最大的为QTL6_5,分别解释体质量和体长性状表型变异的24.2%和24.1%。

图2 QTLs 在联锁群中的分布Fig.2 QTL mapping of growth-related traits distribution in linkage groups
本研究共检测到17 个QTL 区间在不同生长性状间共享,其中与4 个性状都相关的QTL 区间3个,分别为QTL5_3、QTL6_2 和QTL38_4。4 个性状解释表型变异9.1%~13.7%;12.0%~13.3%;和10.6%~16.3%。与3 个性状相关的QTL 区间有9 个,分别为QTL3_1、QTL5_2、QTL5_4、QTL5_5、QTL6_3、QTL6_6、QTL30_1、QTL38_5 和QTL44_1;与两个生长性状相关QTLs 有5 个,分别为QTL5_6、QTL6_5、QTL12_2、QTL38_1 和QTL38_6;其余12 个QTLs(QTL5_1、QTL6_1、QTL6_4、QTL7_1、QTL12_1、QTL16_1、QTL30_2、QTL30_3、QTL38_2、QTL38_3、QTL46_1 和QTL47_1)仅与一个性状相关。
2.4 生长性状的候选基因鉴定
对所有与生长性状相关的29 个QTL 区间与鲤全基因组物理图谱进行了比对分析,在所有QTL 区间上基因进行生长基因筛选(图3),结果在12 个QTL 区间筛选到了生长相关候选基因(表3),分别为:转化生长因子-β 受体(Transforming growth factor beta receptor type I b,tgfbr1b)、生长因子非依赖性新蛋白(Novel protein similar to growth factor independent,gfi)、生长激素调节的TBC 蛋白2(Growth hormone_regulated TBC protein 2,grtp2)、生长激素调节的TBC 蛋白1(Growth Hormone Regulated TBC Protein 1,grtp1)、转化生长因子β 受体3(Transforming growth factor beta receptor 3,tgfb3)、胰岛素样生长因子Ⅱ受体(Insulin-like growth factor II receptor,igf2r)、含转化生长因子β 样结构域蛋白(Transforming growth factor beta like domain containing protein,tgfb)、生长激素受体Ⅱ型(Growth hormone receptor type II,ghr2)、表皮生长因子样蛋白6(Epidermal growth factor like protein 6,egf6)、表皮生长因子样蛋白8(Epidermal growth factor-like protein 8,egf8)、生长因子受体相关结合蛋白2(Growth factor receptor bound protein 2-associated,grb2)和生长激素受体Ⅰ型(Growth hormone receptor type I,ghr1)等,这些候选功能基因为镜鲤生长相关的分子机理研究提供了有价值的信息。
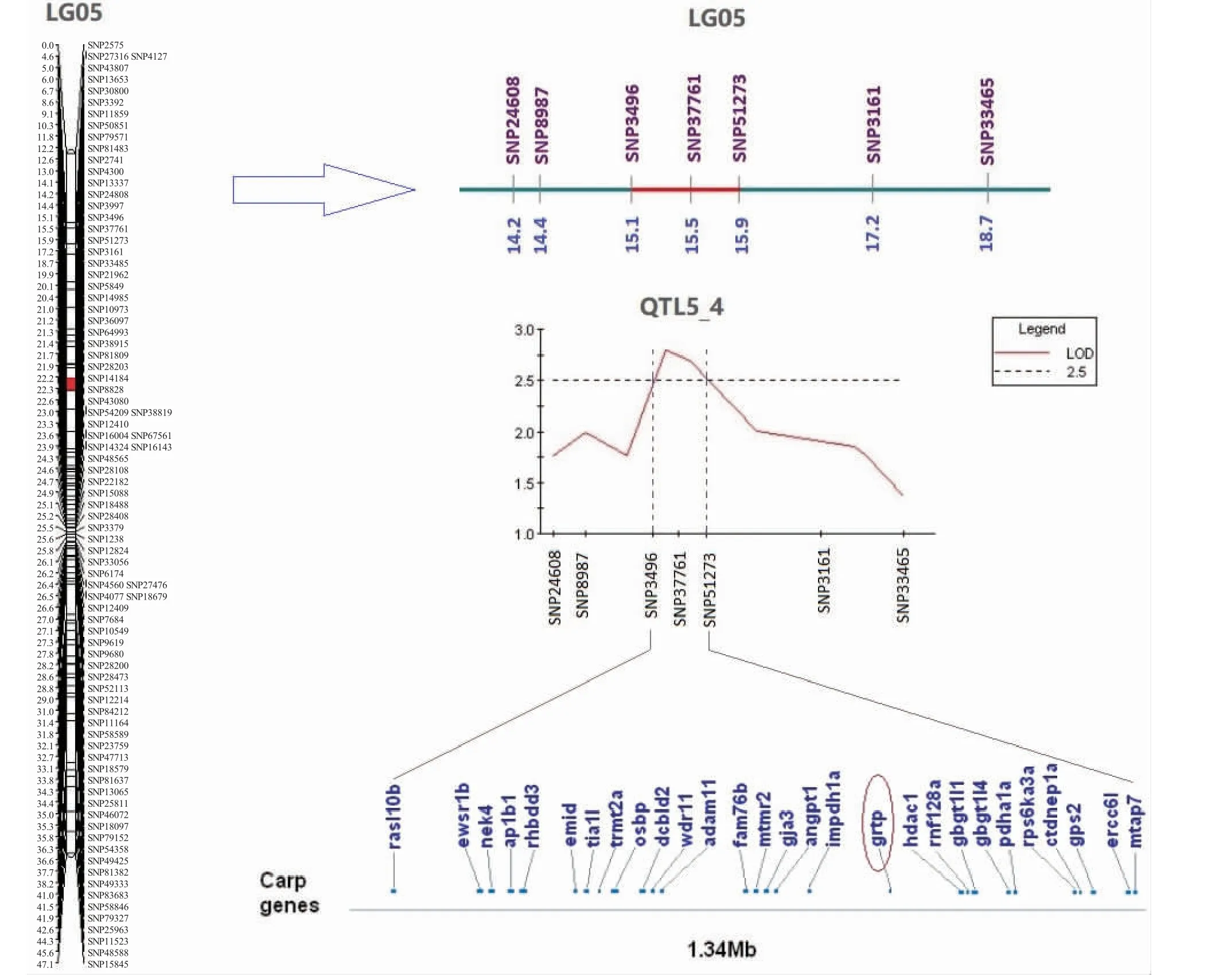
图3 QTL 定位及生长相关的候选基因分析Fig.3 QTL mapping and association analysis of candidate growth-related genes
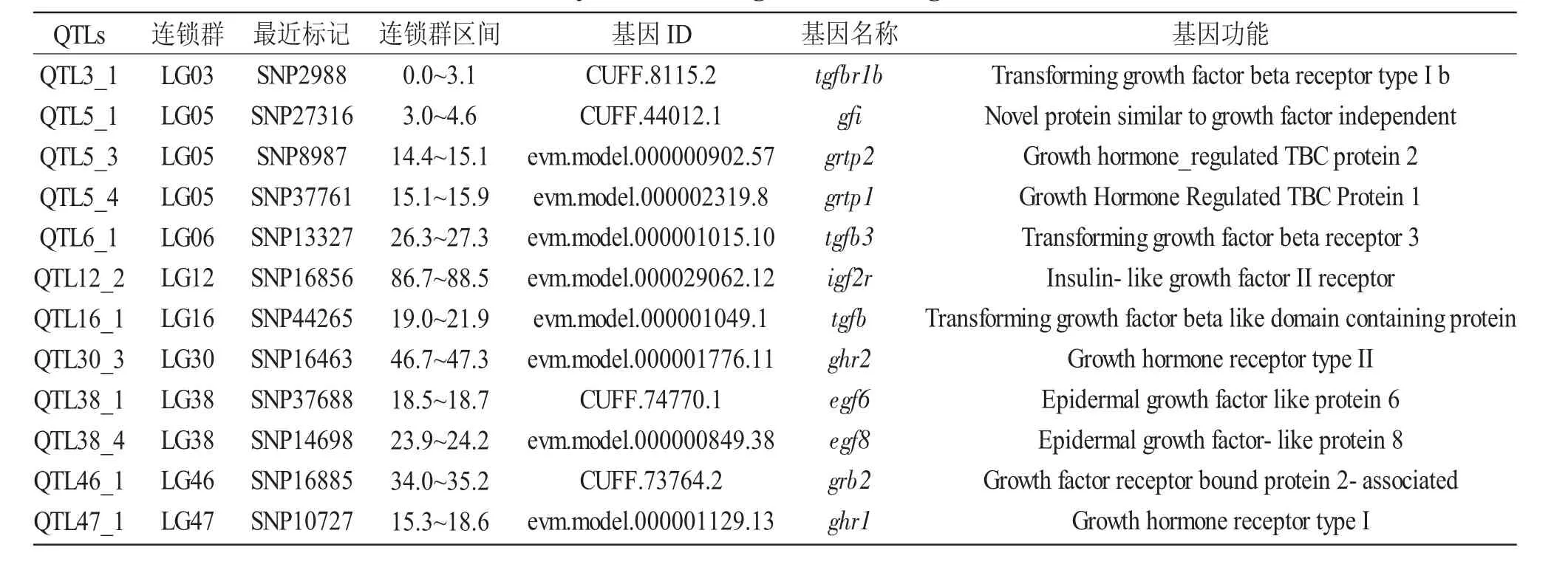
表3 四个生长性状候选基因Tab.3 Summary of candidate genes for four growth-related traits
3 讨论
在过去二十年中,鱼类育种方法已从传统选择发展到现代生物技术,如标记辅助选择(MAS)和分子育种[11]。鱼类最重要的经济性状,大都由多个被称为QTL 的基因控制。这些性状的表型变异是由多个数量性状位点(QTL)等位基因分离产生的数量遗传变异引起[18]。这些QTL 中的大多数对性状影响较小,但多个QTL 位点的累加效应可能对性状产生重大影响。如果识别出与感兴趣的性状相关的基因和遗传标记,遗传变异可作为MAS 分析的工具[33]。
高质量的遗传图谱是进行高效、准确定位QTL分析所必需的至关重要的工具。然而,传统标记数量有限,难以覆盖整个基因组[34]。随着高通量测序成本的降低,利用高通量测序可快速、批量地开发SNP 标记,构建高密度遗传图谱,为后续经济性状QTL 精准定位提供重要工具。SNP 标记是基因组中最丰富的多态性标记,非常适合构建高密度连锁图谱。本研究利用SLAF-seq 高通量测序技术,对镜鲤F1全同胞家系进行高通量测序,构建了镜鲤含有3 903 个SNP 标记的高密度遗传连锁图谱,图谱平均标记间隔0.998 cM,较之前用于镜鲤生长相关性状QTL 定位的图谱的平均图距(5.77~16.8 cM)小很多,标记数量也明显增多[20-23]。
用高密度图谱对生长性状进行QTL 定位,可快速、批量获得生长相关的标记和基因。本研究利用新构建的高密度连锁图谱对6 月龄镜鲤四个生长性状进行QTL 精准定位,共检测到29 个QTLs 与镜鲤幼鱼生长性状显著相关。这些QTLs 多数(18 个)呈簇状分布于第5、6 和38 连锁群上,少数(11 个)位 于LG3、LG7、LG12、LG16、LG30、LG44、LG46 和LG47 上。进一步分析发现,镜鲤幼鱼体质量相关的20 个QTLs 中有17 个均不同程度与其他4 个性状中至少1 个生长性状QTL 位点一致。与本研究中的4 个生长性状均共享的QTLs 有3 个,分别为QTL5_3、QTL6_2 和QTL38_4,说明生长性状之间存在高度的相关性,这与其表型数据相关性结果一致。在获得的29 个鲤长相关的QTLs 中,绝大多数单个QTL 对性状的贡献率在10%以上,3 个QTLs(QTL6_5、QTL6_6 和QTL44_1)的单个QTL 解释表型变异率大于20%,为生长性状的主效QTL 区间。其中,解释表型变异最大的为QTL6_5,分别解释体质量和体长性状表型变异的24.2%和24.1%。
为了获得生长相关的候选基因,本研究比对29个QTL 区间对应的鲤全基因组序列,筛选了12 个生长相关的候选基因。包括转化生长因子-β 受体tgfbr、生长激素调节的TBC 蛋白grtp、胰岛素样生长因子igf2r 和生长激素受体ghr 等。这些基因的功能涉及脂质代谢、碳水化合物代谢、细胞增殖和分化等[35-38],其基因参与鱼体生长的具体生理生化即代谢过程还需进一步研究验证。
本研究采用的6 月龄早期阶段的镜鲤家系检测和估计了QTL 效应,对后期和收获产量存在高度的遗传相关性,幼鱼早期生长速度也会影响最终的收获质量[39,40]。这些研究结果暗示,本研究获得的生长相关的QTLs,可在早期实施对该性状的遗传改良,缩短选育周期。

