耐碳青霉烯类大肠埃希菌耐药基因检测及同源性分析
于江,刘元元,李晓哲,金呈强
耐碳青霉烯类大肠埃希菌耐药基因检测及同源性分析
于江,刘元元,李晓哲,金呈强
济宁医学院附属医院检验科,山东济宁 272000
探讨耐碳青霉烯类大肠埃希菌(carbapenem-resistant,CREco)的耐药基因分布情况,并进行同源性分析,为细菌感染的治疗和预防其传播提供理论依据。自2016年1月至2022年7月济宁医学院附属医院住院患者分离出的大肠埃希菌1311株中筛选出CREco 16株。最低抑菌浓度法进行药敏试验,聚合酶链反应(polymerase chain reaction,PCR)扩增碳青霉烯酶基因及喹诺酮类耐药基因,对阳性基因进行测序比对,用脉冲场凝胶电泳检测菌株间的同源性。16株CREco仅对个别抗生素敏感,对多数抗生素表现为耐药,其中对β-内酰胺类和喹诺酮类耐药率最高。经PCR扩增发现,100%携带碳青霉烯酶基因,包括NDM-1型13株,NDM-5型3株,KPC-2型8株,未检测出OXA、IMP和VIM。喹诺酮类耐药基因中,5株扩增出qnrS基因,14株扩增出aac(6’)-Ib基因。脉冲场凝胶电泳分型共获得12种谱型,C谱型最多,为流行谱型。CREco耐药形势严峻。碳青霉烯酶以NDM-1最常见,其次为KPC-2。喹诺酮类耐药基因aac(6’)-Ib阳性率最高。脉冲场凝胶电泳分型呈现多态性,C谱型在院内存在流行趋势。应加强CREco耐药基因的监测,采取感染控制措施以帮助遏制耐药菌的传播。
大肠埃希菌;碳青霉烯酶;耐药基因型;同源性
大肠埃希菌是社区相关感染的主要病原菌,是引起脑膜炎、尿路感染和败血症等临床感染最常见的细菌之一[1-2]。由于对广谱抗生素的耐药性不断增强,碳青霉烯类抗菌药物被认为是治疗多重耐药肠杆菌科细菌感染的有效选择[3]。然而随着碳青霉烯类抗菌药物使用量的急剧增加,近年来耐碳青霉烯类大肠埃希菌(carbapenem-resistant,CREco)不断被检出,临床治疗面临严峻考验。由于CREco有在医院和社区环境中传播的潜在趋势,所以应严密监测该菌株[4]。本研究旨在调查CREco的耐药情况,并进一步探讨其耐药基因分布和菌株间的同源性,为指导临床抗生素使用和预防医院感染提供理论依据。
1 资料与方法
1.1 菌株来源
收集2016年1月至2022年7月济宁医学院附属医院住院患者检出的非重复大肠埃希菌1311株,其中对碳青霉烯类抗生素耐药的大肠埃希菌16株。
1.2 仪器与试剂
全自动快速微生物质谱检测系统MALDI-TOF MS(法国生物梅里埃公司),VITEK2 compact全自动微生物分析系统(法国生物梅里埃公司),聚合酶链反应(polymerase chain reaction,PCR)扩增仪(美国ABI公司),琼脂糖凝胶电泳仪(北京百晶生物技术有限公司),凝胶成像系统(英国Syngene公司),脉冲场凝胶电泳仪CHEF Mapper(美国Bio-Rad公司),XbaI限制性内切酶(大连Takara公司),DNA marker(大连Takara公司)。
1.3 方法
1.3.1 临床资料收集 回顾CREco患者的临床资料,包括标本来源、科室分布、年龄、性别等。
1.3.2 细菌鉴定及药敏试验 细菌鉴定采用基质辅助激光解吸电离飞行时间质谱MALDI-TOF MS,药敏试验采用AST-GN13药敏板和KB纸片扩散法测定,可疑结果采用Etest试纸条进行确认,结果判读依据美国临床实验室标准化委员会2020版执行。质控菌株为大肠埃希菌ATCC25922。
1.3.3 耐药基因检测 将菌株复苏培养后,溶于装有100μl去离子水的EP管中,100℃煮沸10min,12 000转/min离心10min,取上清液作为DNA模板。PCR反应体系为25μl,包括上下游引物1μl、模板DNA 2μl、Taq Master Mix 12.5μl、去离子水8.5μl。引物序列和扩增程序参照文献[5-8],见表1。扩增完成后,1%琼脂糖凝胶电泳回收。PCR扩增阳性产物送北京华大基因公司进行测序,用NCBI的BLAST软件对基因序列进行比对,获取基因型。
1.3.4 脉冲场凝胶电泳检测菌株间的同源性 首先制备细菌胶块,54℃细胞裂解24h后,XbaI 37℃酶切4h,1% SeaKem Gold Agarose在CHEF Mapper电泳仪上进行电泳。电泳结束后,凝胶放入溴化乙锭溶液(1µg/ml)染色30min,去离子水清洗30min,用紫外成像系统照相并保存图像。
1.3.5 数据处理 采用BLAST、BioNumerics等软件对数据进行处理分析。
2 结果
2.1 临床资料
感染CREco的患者主要来自监护室(43.75%,7/16)、消化道肿瘤科(18.75%,3/16)及外科病房(25.00%,4/16);标本来源于血液(31.25%,5/16)、脓液(18.75%,3/16)、分泌物(12.5%,2/16)、尿液(12.5%,2/16)、痰液(6.25%,1/16)、脑脊液(6.25%,1/16)、腹水(6.25%,1/16)和胆汁(6.25%,1/16),时间分布无明显差别。男女比例为5∶3,年龄最小9个月,最大99岁,平均年龄(54.12±26.35)岁。
2.2 药物敏感性
CREco对常用抗生素的耐药率较高,除对黏菌素、替加环素均敏感外,对其他抗生素均有不同程度耐药,见表2。
2.3 碳青霉烯酶基因检测结果
16株CREco中,均扩增出NDM目的基因片段、经测序分析13株为NDM-1型,3株为NDM-5型。8株PCR扩增出KPC目的基因片段,经测序分析为KPC-2型碳青霉烯酶,见图1、图2。未筛选出VIM、IMP、OXA目的基因片段。
2.4 喹诺酮类耐药基因检测结果
16株CREco中,5株(31.25%)PCR扩增出qnrS基因目标片段,经测序为qnrS喹诺酮类耐药基因,见图3。14株(87.50%)PCR扩增出aac(6’)-Ib基因目标片段,经测序均为质粒介导的喹诺酮类耐药基因aac(6’)-Ib,见图4。
2.5 脉冲场凝胶电泳
对16株CREco进行脉冲场凝胶电泳分析,以85%为界进行分型,可分为12种谱型,其中菌株13、16为A型,菌株15为B型,菌株2、3、5、8为C型,菌株4为D型,菌株1为E型,菌株9为F型,菌株6、7为G型,菌株11为H型,菌株10为I型,菌株14为J型,菌株12为K型,见图5。C谱型菌株有4株,为流行株。
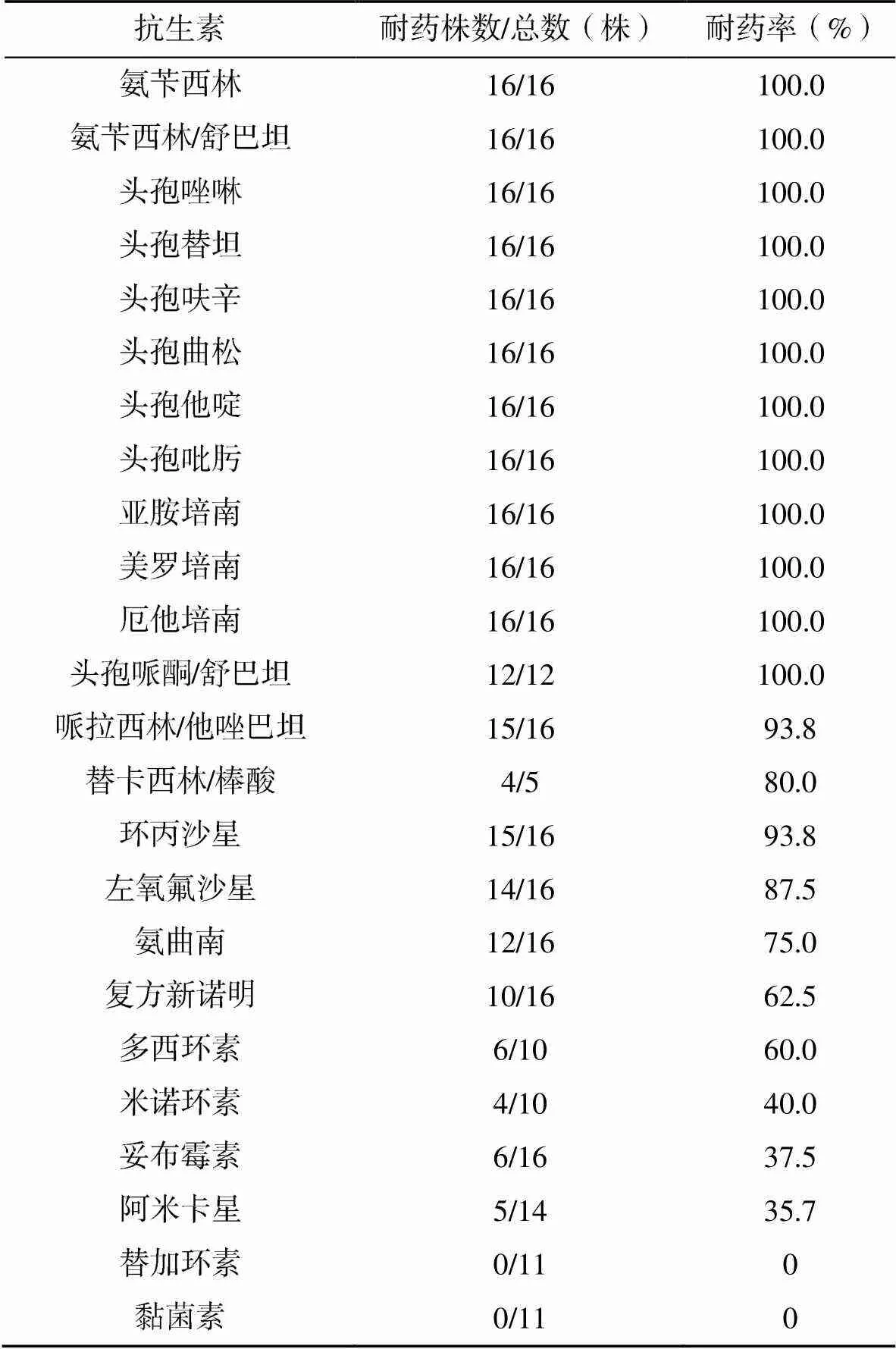
表2 16株CREco对常用抗生素的耐药率

图1 PCR扩增NDM目的基因片段电泳图
注:M为DNA Marker;1~16泳道为1~16号NDM阳性菌株

图2 PCR扩增KPC目的基因片段电泳图
注:M为DNA Marker;1~4,11~13,15泳道为KPC阳性菌株;5~10,14,16泳道为KPC阴性菌株
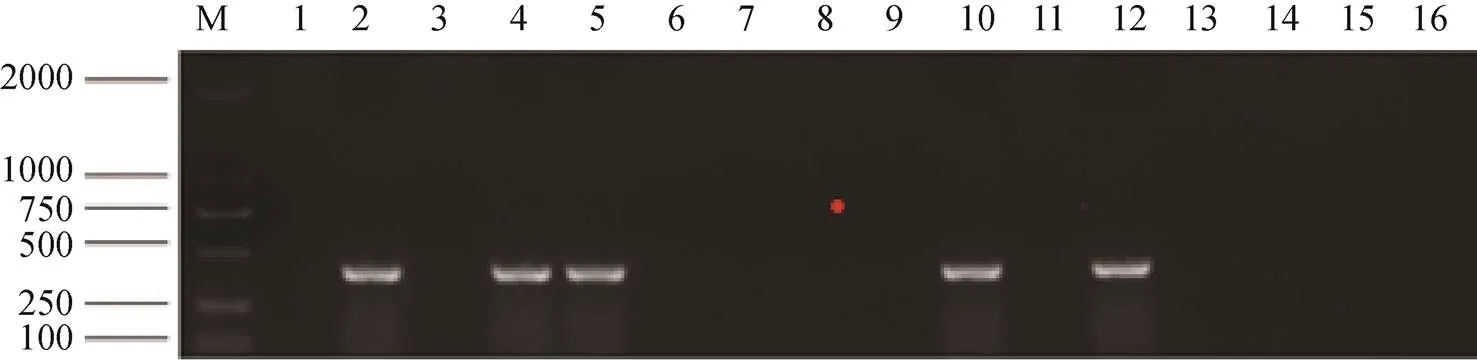
图3 PCR扩增qnrS基因片段电泳图
注:M为DNA Marker;2,4~5,10,12泳道为qnrS阳性菌株;1,3,6~9,11,13~16泳道为qnrS阴性菌株
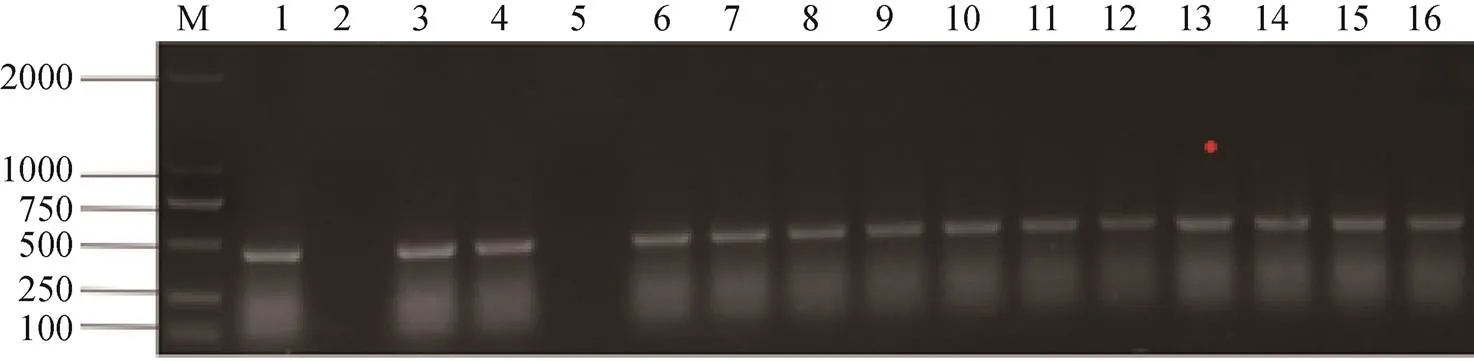
图4 PCR扩增aac(6’)-Ib基因片段电泳图
注:M为DNA Marker;1,3~4,6~16泳道为aac(6’)-Ib阳性菌株;2,5泳道为aac(6’)-Ib阴性菌株

图5 CREco脉冲场凝胶电泳及聚类分析
3 讨论
过去的近20年间,耐碳青霉烯肠杆菌科细菌已成为全球日益严重的公共卫生问题。其感染后治疗选择受限,医疗负担沉重,死亡率较高[9-10]。其中CREco所占比例仅次于肺炎克雷伯菌居第二位。据统计,美国CREco比例由2006—2007年的0.9%上升至2009—2010年的1.9%[11]。欧洲2013—2014年,19%的大肠埃希菌菌株为CREco[12]。在中国,2004—2015年的监测结果显示,10年间CREco的比例保持在0.8%~3.0%[13]。
本研究中共检出非重复大肠埃希菌1311株,其中CREco 16株,占大肠埃希菌总数的1.2%,标本主要来源于血液、脓液、分泌物和尿液,来源科室主要是监护室、消化道肿瘤科及某些外科病房,男性多于女性。国内有报道称河北省2017—2019年CREco占大肠埃希菌的2.65%,尿液中检出比例最高,此外还有痰液、血液、分泌物等,超过50%来自内科病房,男性多于女性[14]。而国外研究指出CREco主要来自重症监护室,女性所占比例反而高于男性[15]。可见CREco感染人群的临床特征并不完全一致,不同地区有各自的特点。
16株CREco除少数对含酶抑制剂的复合物及氨曲南敏感外,对青霉素类、头孢菌素类、碳青霉烯类均表现为耐药,对喹诺酮类耐药率>85%,对部分氨基糖苷类耐药率较低,如阿米卡星耐药率为35.7%,对替加环素、黏菌素均敏感。可见,CREco对大多数抗生素呈现高度耐药性。由于耐药基因通常位于可移动的遗传元件上,能够引起广泛传播[16]。大肠埃希菌对碳青霉烯类抗菌药物耐药主要是由质粒编码的碳青霉烯酶引起的[17]。碳青霉烯酶主要分为三种类型:KPC型、金属-β-内酰胺酶和苯唑西林酶。金属-β-内酰胺酶常见有NDM、IMP、VIM型。本研究检测的16株耐碳青霉烯大肠埃希菌中,NDM基因均阳性,经测序分析发现,13株为NDM-1基因阳性、3株为NDM-5基因阳性。6株菌KPC-2型碳青霉烯酶基因阳性。NDM-1于2009年在一名有印度住院史的游客身上发现,它可与其他耐药基因共存,且可在不同菌种间水平转移,基因的不同变异亚型不断被检出[18]。NDM-5是NDM型碳青霉烯酶的另一种亚型,携带NDM-5与携带NDM-1的大肠埃希菌有2个氨基酸的差别,NDM-5对碳青霉烯类水解活性更强,产NDM-5肠杆菌科细菌在世界各国被发现,检出数量不断增加[18-19]。KPC主要存在于克雷伯菌属,但在其他肠杆菌科和其他革兰阴性菌中也有发现。尽管已有20多种亚型出现,KPC-2和KPC-3仍是最常见的亚型[20]。碳青霉烯酶的检出率因地理区域而异。国内外报道显示,2015—2017年全球36个国家CREco的碳青霉烯酶的总体分布为OXA-181(23%)、NDM-5(20%)、OXA-48(17%)、KPC(15%)、NDM-1(10%)。黎巴嫩CREco检出最多的碳青霉烯酶为OXA-48和OXA-181,泰国CREco主要为NDM和OXA-48,我国24个省市36家医院收集的CREco常见酶为NDM-5、NDM-1和KPC-2,未检测到IMP和OXA-48酶。可见不同国家、不同地区CREco的碳青霉烯酶分布不同,国外OXA酶检出较多,而国内NDM和KPC较多。
质粒介导的喹诺酮类耐药基因主要有aac(6’)-Ib-cr和qnr基因。aac(6’)-Ib-cr是氨基糖苷乙酰转移酶的变异基因,可使环丙沙星及诺氟沙星对细菌的MIC值上升,从而产生喹诺酮类耐药。另一种是qnr基因,携带qnr基因的菌属主要以革兰阴性菌为主,包括大肠埃希菌、肺炎克雷伯菌、沙门菌、变形杆菌等。陈杨等[21]报道2018—2020年间不同国家临床患者标本中分离出的大肠埃希菌qnr耐药基因的分布情况,包括qnrA、qnrB、qnrC、qnrD、qnrS,其中qnrS基因检出率最高。本研究发现,CREco对左氧氟沙星的耐药率为87.5%,对环丙沙星的耐药率为93.75%。耐药基因检测显示,31.25%携带qnrS基因,未检出qnrA和qnrB基因,87.50%携带aac(6’)-Ib-cr基因。说明CREco qnrS或aac(6’)-Ib-cr基因携带率高,两者均可介导低水平喹诺酮耐药,且均位于质粒上,常与β-内酰胺类、多黏菌素等多种耐药基因同时存在。
脉冲场凝胶电泳分析发现16株CREco有12种谱型,其中C谱型4株,其余谱型1~2株,说明出现C谱型克隆株的流行。提醒医院应进一步加强防控措施,持续监测流行情况,以防止CREco的进一步传播。
CREco对大多数β-内酰胺类抗生素耐药,治疗该类细菌感染的药物选择非常有限,喹诺酮类抗生素原本是一个选择,然而本研究发现此类抗生素的耐药情况同样严重,通过检测耐药基因,碳青霉烯类及喹诺酮类耐药与碳青霉烯酶基因和喹诺酮类耐药基因存在相关性,因此对CREco进行耐药监测及流行病学研究十分必要,下一步将继续加大样本量,对其他耐药机制进行研究。
[1] PITOUT J D. Extraintestinal pathogenic Escherichia coli: An update on antimicrobial resistance, laboratory diagnosis and treatment[J]. Expert Rev Anti Infect Ther, 2012, 10(10): 1165–1176.
[2] CANDAN E D, AKSÖZ N. Escherichia coli: Characteristics of carbapenem resistance and virulence factors[J]. Braz Arch Biol Technol, 2017, 60(1): 1–12.
[3] TOMPKINS K, VAN DUIN D. Treatment for carbapenem- resistant Enterobacterales infections: Recent advances and future directions[J]. Eur J Clin Microbiol Infect Dis, 2021, 40(10): 2053–2068.
[4] SCHWABER M J, CARMELI Y. Carbapenem-resistant Enterobacteriaceae: A potential threat[J]. JAMA, 2008, 300(24): 2911–2913.
[5] 杨金辉, 吕媛. 中国肺炎克雷伯菌碳青霉烯类抗生素耐药现状和流行病学分析[J]. 中国临床药理学杂志, 2012, 28(5): 323–326.
[6] 沈继录, 朱德妹, 吴卫红, 等. 革兰阴性杆菌碳青霉烯酶产生与细菌耐药性关系的研究[J]. 中华检验医学杂志, 2008, 31(4): 408–414.
[7] ROBICSEK A, JACOBY G A, HOOPER D C. The worldwide emergence of plasmid-mediated quinolone resistance[J]. Lancet Infect Dis, 2006, 6(10): 629–640.
[8] PARK C H, ROBICSEK A, JACOBY G A, et al. Prevalence in the United States of aac(6’)-Ib-cr encoding a ciprofloxacin-modifying enzyme[J]. Antimicrob Agents Chemother, 2006, 50(11): 3953–3955.
[9] FRIEDMAN N D, CARMELI Y, WALTON A L, et al. Carbapenem-resistant Enterobacteriaceae: A strategic roadmap for infection control[J]. Infect Control Hosp Epidemiol, 2017, 38(5): 580–594.
[10] LEE B Y, BARTSCH S M, WONG K F, et al. The potential trajectory of carbapenem-resistant Enterobacteriaceae, an emerging threat to health-care facilities, and the impact of the centers for disease control and prevention toolkit[J]. Am J Epidemiol, 2016, 183(5): 471–479.
[11] MARTIROSOV D M, LODISE T P. Emerging trends in epidemiology and management of infections caused by carbapenem-resistant Enterobacteriaceae[J]. Diagn Microbiol Infect Dis, 2016, 85(2): 266–275.
[12] GRUNDMANN H, GLASNER C, ALBIGER B, et al. Occurrence of carbapenemase-producing Klebsiella pneumoniae and Escherichia coli in the European survey of carbapenemase-producing Enterobacteriaceae (EuSCAPE): A prospective, multinational study[J]. Lancet Infect Dis, 2017, 17(2): 153–163.
[13] ZHANG R, LIU L, ZHOU H, et al. Nationwide surveillanceof clinical carbapenem-resistant Enterobacteriaceae (CRE) strains in China[J]. EBioMedicine, 2017, 19: 98–106.
[14] ZHANG W, LI Z, WANG N, et al. Clinical distribution characteristics of 1439 carbapenem-resistant Escherichia coli strains in China: Drug resistance, geographical distribution, antibiotic MIC50/90[J]. Infect Drug Resist, 2021, 14: 4717–4725.
[15] GUH A Y, BULENS S N, MU Y, et al. Epidemiology of carbapenem-resistant Enterobacteriaceae in 7 US communities, 2012-2013[J]. JAMA, 2015, 314(14): 1479–1487.
[16] DOLEJSKA M, VILLA L, POIREL L, et al. Complete sequencing of an IncHI1 plasmid encoding the carbapenemase NDM-1, the ArmA 16S RNA methylase and a resistance- nodulation-cell division/multidrug efflux pump[J]. J Antimicrob Chemother, 2013, 68(1): 34–39.
[17] GLASNER C, ALBIGER B, BUIST G, et al. Carbapenemase- producing Enterobacteriaceae in Europe: A survey among national experts from 39 countries, February 2013[J]. Euro Surveill, 2013, 18(28): 20525.
[18] SASSI A, LOUCIF L, GUPTA S K, et al. NDM-5 carbapenemase-encoding gene in multidrug-resistant clinical isolates of Escherichia coli from Algeria[J]. Antimicrob Agents Chemother, 2014, 58(9): 5606–5608.
[19] ZHANG F, XIE L, WANG X, et al. Further spread of bla NDM-5 in Enterobacteriaceae via IncX3 plasmids in Shanghai, China[J]. Front Microbiol, 2016, 7: 424.
[20] MUNOZ-PRICE L S, POIREL L, BONOMO R A, et al. Clinical epidemiology of the global expansion of Klebsiella pneumoniae carbapenemases[J]. Lancet Infect Dis, 2013, 13(9): 785–796.
[21] 陈杨, 刘理慧, 吴翠蓉, 等. 喹诺酮类耐药基因qnr的研究进展[J]. 国外医药(抗生素分册), 2021, 42(4): 193–203.
Analysis of drug resistance genes and homology of carbapenem-resistant
Department of Clinical Laboratory, Affiliated Hospital of Jining Medical University, Jining 272000, Shandong, China
To investigate the distribution of drug resistance genes in carbapenem-resistant(CREco) and analyze homology, so as to provide theoretical basis for the treatment and prevention of bacterial infection.Sixteen CREco strains were selected from 1311 trains offrom inpatients in Affiliated Hospital of Jining Medical University from January 2016 to July 2022. The drug sensitivity test was performed by minimum inhibitory concentration method, the carbapenemase genes and the quinolone resistance genes were amplified by polymerase chain reaction (PCR). The positive genes were sequenced, and the homology between strains was detected by pulsed field gel electrophoresis.The 16 strains were sensitive to only a few antibiotics and resistant to most of them, among them, the resistance rate to β-lactams and quinolones was the highest. 100% carried carbapenemase genes by PCR, including 13 strains of NDM-1, 3 strains of NDM-5 and 8 strains of KPC-2. The genes of OXA, IMP and VIM were not detected. 5 strains amplified the qnrS gene, 14 strains amplified the aac(6’)-Ib gene among the quinolone resistance genes. A total of 12 types were obtained by pulsed field gel electrophoresis, and the C type was the most popular.The situation of drug resistance of CREco is serious. NDM-1 was the most common carbapenemase, followed by KPC-2. The highest positive rate of quinolone resistance gene is the aac(6’)-Ib gene. Pulsed field gel electrophoresis typing showed polymorphism, and the C type was prevalent in hospital. Surveillance of CREco resistance genes should be strengthened and infection control operations should be implemented to help contain the spread of resistant bacteria.
; Carbapenemase; Drug resistance genotype; Homology
R378
A
10.3969/j.issn.1673-9701.2023.25.016
李晓哲,电子信箱:lxz_0057@163.com
(2023–04–01)
(2023–08–25)

