小麦丙氨酸氨基转移酶家族成员鉴定及初步分析
赵 琪,李松硕,王 娇,郭 鹏,吕爱枝,周 硕,焦 博*
(1.河北省农林科学院 生物技术与食品科学研究所/河北省植物转基因中心重点实验室,河北 石家庄 050051;2.河北北方学院 农林科技学院,河北 张家口 075000;3.河北科技大学 食品与生物学院,河北 石家庄 050018)
0 引言
氮是植物中氨基酸、蛋白质、核酸、叶绿素以及激素等生物大分子物质的主要组成成分,是植物生长发育的主要限制因素[1]。施用氮肥是提高农作物产量的有效方法,也是农业生产主要的成本支出。在我国的实际农业生产中,长期存在氮肥施用不合理、农作物氮肥吸收利用率低等主要问题,这不仅导致了土壤酸化、水体富营养化,且可能造成严重的环境污染[2]。此外农作物种植成本的提高也严重制约了我国农业的发展。因此,解析植物对氮的吸收、转运以及代谢过程,培育高氮肥利用率的农作物品种,不仅可以降低氮肥施用量,减少环境污染,还能降低农民的生产成本,有力推动我国农业的可持续发展[3]。
植物的根主要从土壤的硝酸盐(NO3-)、铵(NH4+)等无机氮形态中吸收氮元素,这2种形式的氮源分别通过相应的转运蛋白实现其在细胞和组织器官之间的吸收、转运和移动,其中硝酸盐是植物在有氧条件下从土壤中获取氮源的主要形式[3-4]。硝酸盐通过不同亲和力的硝酸转运系统(NRTs)进入细胞,然后在细胞质中经NADPH-依赖硝酸还原酶(NAR)还原为亚硝酸盐,在叶绿体和前质体中通过铁氧还蛋白与亚硝酸还原酶(NiR)催化转变为铵。土壤中的铵盐也可以通过铵转运蛋白(AMT)进入植物体内,后经谷氨酰胺合成酶/谷氨酰胺-α-酮戊二酸氨基转移酶(GS/GOGAT)催化合成谷氨酰胺或谷氨酸,进一步通过不同的氨基转移酶合成其他氨基酸,最后被植物所利用[5]。
丙氨酸氨基转移酶(AlaAT)又称谷丙转氨酶,是参与植物氮素同化的关键酶类,该酶在辅酶磷酸吡哆醛(PLP)存在时催化丙氨酸和α-酮戊二酸反应形成丙酮酸和谷氨酸的可逆反应。此催化反应中的丙酮酸和α-酮戊二酸也是碳代谢中的重要中间产物,谷氨酸参与了氮素同化以及信号转导过程,丙氨酸是缺氧时氨基酸的主要来源[6],所以丙氨酸氨基转移酶将植物初级碳代谢与氨基酸合成联系在一起,在碳氮代谢中发挥了不可或缺的作用。丙氨酸氨基转移酶在拟南芥[6]、水稻[7]、玉米[8]、大麦[9]、苜蓿[10]、豆类[11]、辣椒[12]、莲花[13]、茶叶[14]、杨树[15]以及小麦[16]等植物中均有一定的研究报道。研究发现,低氧、光照、氮肥施用、高温以及低温[14]均可影响丙氨酸氨基转移酶基因的表达水平,并且在许多组织和器官中均能检测到AlaAT的活性,如拟南芥的叶、根、花[17]以及水稻发育中的种子[7]等。
现有研究报道的拟南芥中有4个AlaAT基因,分别为AtAlaAT1和AtAlaAT2、AtGGAT1和AtGGAT2。GGAT为包含编码过氧化物酶体靶向信号(PTSs)序列的谷氨酸:乙醛酸氨基转移酶[17-18],其能催化谷氨酸和乙醛酸反应生成α-酮戊二酸和甘氨酸,在光呼吸和氨基酸代谢中发挥重要作用。在缺氧条件下,AtAlaAT1和AtAlaAT2均被诱导表达。在拟南芥中突变AtAlaAT1证实,丙氨酸氨基转移酶在低氧胁迫恢复过程中,对于丙氨酸分解代谢至关重要[6]。大豆基因组已鉴定出4个AlaAT基因,GmAlaAT1~GmAlaAT4。在淹水导致的缺氧条件下,GmAlaAT表达增强,并参与到丙氨酸的分解代谢[11]。过表达GmAlaAT1可提高大豆中抗氧化保护酶活性并促进根系发育[19]。同样,在苜蓿中也发现了4个AlaAT基因,它们与幼苗耐缺氧有关[10]。在水稻中鉴定出5个AlaAT基因,其中OsAlaAT1在发育种子中的表达量高于其他4个基因的,并且受缺氧诱导表达最显著。在木本植物杨树中确定了4个丙氨酸氨基转移酶同源基因AlaAT1~AlaAT4,其中AlaAT1和AlaAT2属于GGAT蛋白基因,主要在叶中表达,均能被外施N诱导表达,并且受昼夜变化调节,而AlaAT3和AlaAT4属于AlaAT蛋白基因,在根、茎、叶中均有表达。研究证明AlaAT3能够被谷氨酰胺调控,受外施N的诱导表达,可能在根氮代谢中发挥重要作用[15]。
Miyashita等[6]研究表明AlaAT被证实参与植物中许多生理代谢过程,如氨基酸代谢、光呼吸、糖异生和糖酵解等,其还可作为C和N的反应者和生产者参与C4光合作用以及淀粉的合成[20-21],参与植物应对非生物胁迫以及提高氮素利用率等反应过程。在低N条件下,油菜根特异性启动子btg26启动大麦HvAlaAT能够增加油菜地上部分的生物量和产量[22],水稻启幼子OsAnt1启动大麦HvAlaAT可以提高水稻根和苗的生物量[23]并提高氮素利用率[9]。研究发现,OsAlaAT1可以调节水稻种子发育过程中淀粉的合成,增加籽粒重量。OsAlaAT1基因的缺失会导致籽粒中直链淀粉的含量下降,支链淀粉结构改变,并出现白色胚乳[21,24]。OsAlaAT1的表达还可以调控水稻的产量和品质,提高水稻氮素利用率[25]。
丙氨酸氨基转移酶在植物氮吸收利用过程中发挥重要功能,并影响了农作物的产量和质量。AlaAT基因在拟南芥、水稻、大麦、苜蓿、豆类和杨树等多种植物中均有一定的报道,但在小麦中的研究还不够深入。因此,本研究根据小麦的基因组信息,对丙氨酸氨基转移酶家族成员进行鉴定分析,并进行了生物学特性以及功能分析,以期为提高小麦的氮素利用率及产量奠定理论基础,为培育氮高效小麦品种提供理论支撑。
1 材料和方法
1.1 小麦全基因组丙氨酸氨基转移酶家族成员鉴定
本文结合HMM和BLAST这2种方法筛选小麦丙氨酸氨基转移酶基因。首先从Ensembl(http://plants.ensembl.org/index.html)网站中下载小麦蛋白数据库。根据包含典型氨基转移酶家族结构域的Pfam码(PF00155),通过Pfam数据(http://pfamlegacy.xfam.org/)库查找并下载Stockholm种子文件,并使用HMMER软件在小麦蛋白数据库中筛选小麦丙氨酸氨基转移酶家族成员基因;根据拟南芥和水稻丙氨酸氨基转移酶基因蛋白序列在小麦蛋白数据库进行blast同源蛋白比对,筛选小麦丙氨酸氨基转移酶基因,结合两种方法的结果进行分析,确定目标蛋白;通过NCBI-CDD在线网站(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)筛选剔除缺失或含有不完整氨基转移酶保守结构域的蛋白,最终确定小麦丙氨酸氨基转移酶基因;通过Ensembl数据库(http://plants.ensembl.org/index.html)获得基因序列、CDS序列、蛋白序列、基因染色体位置信息、蛋白分子质量和等电点;通过WoLF PSORT网站(https://wolfpsort.hgc.jp/)进行亚细胞定位预测。
1.2 进化树分析
通过MEGA7对小麦丙氨酸氨基转移酶基因及其在水稻、拟南芥和大麦中的同源基因的氨基酸序列进行进化树分析,经氨基酸序列比对后,再计算确定最佳进化树的算法模型为JTT+G,设定Bootstrap取样值为1000。15个小麦丙氨酸氨基转移酶基因最佳进化树算法模型为LG+G。所有氨基酸序列从Ensembl数据库网站下载。基因ID号如下:5个水稻OsAlaAT1(LOC_Os10g 25130)、OsAlaAT2(LOC_Os03g 08530)、OsAlaAT3(LOC_Os07g42600)、OsAlaAT4(LOC_Os10g25140)、OsAlaAT5(LOC_Os09g26380);4 个拟南芥AtAlaAT1(At1g17290)、AtAlaAT2(At1g72330)、AtGGAT1(At1g233100)、AtGGAT2(At1g70580);2个苜蓿MtmAlaAT(Medtr8g023140);MtcAlaAT(Medtr5g03 3230)。
1.3 蛋白保守结构域分析及基因结构分析
将小麦氨基酸序列通过MEME网站进行保守结构域分析,设置结构域个数设为10。通过GSDS 2.0网站(http://gsds.gao-lab.org/index.php)对小麦成员基因CDS序列与全长基因序列的结构进行分析。
1.4 染色体定位及Ka/Ks计算
根据小麦染色体长度与基因在染色体上的位置信息,利用TBtools软件将15个基因定位至染色体上。
1.5 启动子顺式作用元件预测
通过Ensembl数据库获得基因ATG上游2000 bp核苷酸序列,通过PlantCARE网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行启动子顺式作用元件预测。
1.6 基因表达分析
收集温室培养的中国春小麦灌浆期的根、茎、叶、旗叶和种子。选取均匀饱满无伤的种子,平铺在发芽盘上,避光2 d进行萌发,随后正常光照培养3 d,去掉胚乳,转入96孔板上并使用Hoagland营养液水培3 d,达到2叶1心期进行缺氮处理,处理0、0.5、1.0、1.5、2.0、3.0、6.0、24.0 h后收集根组织。使用Trizol提取不同组织的RNA后,通过Nano-Drop 2000分光光度计测定其浓度。每个样品包含3个生物学重复,每个样品取1 μg RNA通过HiScript RT SuperMix试剂反转录成cDNA。以每个样品的cDNA为模板,使用ChamQ Universal SYBR qPCR Master Mix通过ABI 7500 Real-Time PCR定量PCR仪进行基因表达分析,引物见表1,采用ΔΔCT法计算基因的相对表达量。使用TaActin作为内参基因。每个样品进行3次技术复制。

表1 引物序列
2 结果与分析
2.1 小麦丙氨酸氨基转移酶基因筛选、鉴定及进化树分析
根据典型氨基转移酶家族结构域的Pfam码(PF00155),通过HMM和blast结合,筛选出小麦中的丙氨酸氨基转移酶家族成员,并使用SMART和CDD网站进行蛋白保守结构域检测,除去缺失和不完整保守结构域的基因,鉴定出73个小麦丙氨酸氨基转移酶基因序列(图1)。对73个小麦丙氨酸氨基转移酶基因的进化关系进行分析,通过与其在拟南芥、水稻和苜蓿中的同源基因进行进化树聚类分析,发现只有15个基因与拟南芥、水稻和苜蓿中的同源基因聚类到同一分支上,且这一分支可分为2个亚族,其中12个基因与拟南芥中AtAlaAT和所有的水稻OsAlaAT亲缘关系较近,聚类到第1组。小麦中有3个基因与水稻中OsAlaAT1、OsAlaAT3、OsAlaAT4和OsAlaAT5基因同源,其余3个基因与拟南芥中AtGGAT1/2和苜蓿中的MtcAlaAT的亲缘关系较近,聚类到第2组。

图1 小麦丙氨酸氨基转移酶基因及其同源基因进化树分析
2.2 小麦丙氨酸氨基转移酶家族成员信息分析
对小麦中15个丙氨酸氨基转移酶基因进行分析,根据其在染色体上的位置以及水稻丙氨酸氨基转移酶基因,将小麦的丙氨酸氨基转移酶基因分别命名为TaAlaAT1-A/B/D,TaAlaAT3-A/B/D,TaAlaAT4-A/B/D,TaAlaAT5-A/B/D和TaGGAT1-A/B/D。根据基因序列以及氨基酸序列信息分析这15个基因的化学特性,结果显示15个丙氨酸氨基转移酶基因的等电点在5.6613~6.9316之间,蛋白质分子量介于43.7~65.9 kDa之间,转录本长度介于1209~2434 bp之间。亚细胞定位预测显示TaAlaAT1-A/B定位于细胞质中,TaAlaAT1-D定位于叶绿体中,TaAlaAT3-A/B/D均定位于线粒体,TaAlaAT4-A/B/D均定位于叶绿体中,TaAlaAT5-A/B/D定位于细胞质中,而TaGGAT1-A/B定位于过氧化物酶体,TaGGAT1-D定位于细胞质中(表2)。不同的亚细胞定位结果表明,15个丙氨酸氨基转移酶基因在小麦生长发育过程中发挥了不同的生物学功能。

表2 小麦丙氨酸氨基转移酶家族成员信息分析
2.3 小麦丙氨酸氨基转移酶基因染色体定位分析
普通小麦为异源六倍体(AABBDD),包含3个染色体组(ABD),每个染色体组含有7条染色体,总共21条染色体。基因染色体定位信息结果显示,15个基因主要分布在1、2和5号染色群组上,定位信息(图2)分布如下:Chro1A(TaAlaAT1-A);Chro1B(TaAlaAT1-B);Chro1D(TaAlaAT1-D);Chro2A(TaAlaAT3-A、TaGGAT1-A);Chro2B(TaAlaAT3-B、TaGGAT1-B);Chro2D(TaAlaAT3-D、TaGGAT1-D);Chro5A(TaAlaAT4-A、TaAlaAT5-A);Chro5B(TaAlaAT4-B、TaAlaAT5-B);Chro5D(TaAlaAT4-D、TaAlaAT5-D)。

图2 TaAlaAT基因的染色体分布情况
2.4 小麦丙氨酸氨基转移酶基因序列及蛋白保守结构域分析
根据AlaAT家族成员氨基酸序列信息,通过MEME网站进行蛋白保守结构域预测。设定10个保守结构域进行分析,结果显示在10个TaAlaAT氨基酸序列中,除TaAlaAT1-D外均含有10个完整的保守结构域;TaAlaAT1-D缺少N端的Motif9和Motif6保守结构域;TaGGAT1-A/B均含有8个完整的保守结构域,缺少N端的Motif9和Motif7保守结构域;TaGGAT1-D只含有7个完整的保守结构域,N端缺少Motif9和Motif7,C端缺少Motif8保守结构域。基因序列结构分析显示,所有基因均含有多个外显子和内含子,其中外显子数目分别为10、12、13、15和18个,绝大部分为15个,外显子数目最少的为TaAlaAT1-D,数目最多的为TaAlaAT4-B(图3)。
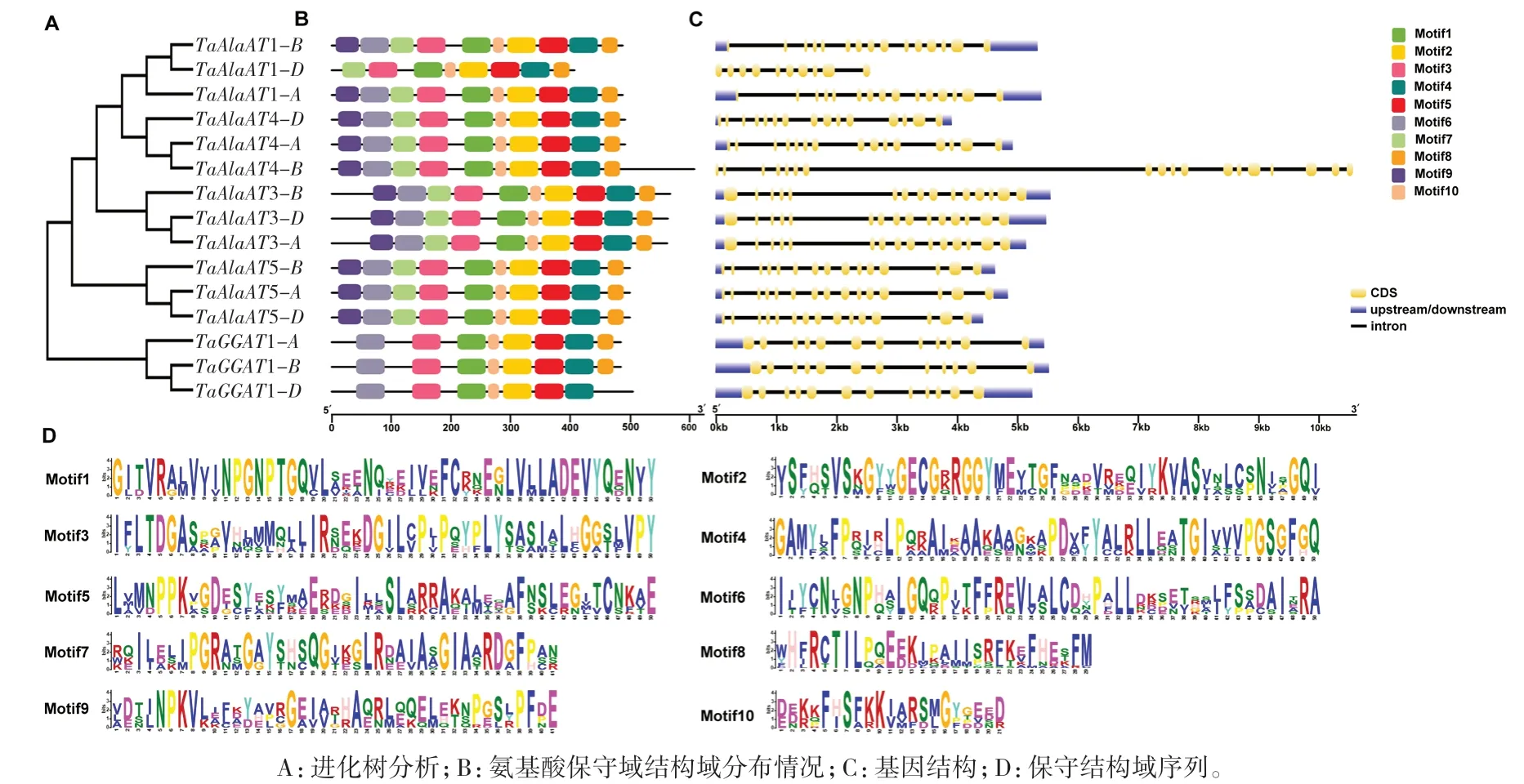
图3 15个TaAlaAT基因氨基酸及基因结构分析
2.5 基因对Ka/Ks分析
通过进化树分析发现,15个小麦丙氨酸氨基转移酶基因可进化为5支,每支有3个同源基因,基因序列高度同源。为了研究每个分支中一组基因在进化过程中是否受到选择压力的影响,对每对基因Ka/Ks值进行分析。由表3可知,5组基因对的Ka/Ks值均小于1,这说明基因在进化过程中受纯化选择作用。

表3 基因对Ka/Ks分析
2.6 小麦丙氨酸氨基转移酶基因启动子顺式作用元件分析
启动子中的顺式作用元件决定了基因的表达特异性。通过对基因启动子的顺式作用元件进行预测,可以为研究AlaAT家族成员的功能提供更多的参考依据。由图4可知,每个基因启动子上均含有多种类型的反应元件,如激素响应元件、胁迫响应元件以及与发育相关的顺式作用元件;激素响应元件包括脱落酸响应元件(ABRE)、生长素响应元件(AuxRR-core/TGA-element)、赤霉素响应元件(P-box/GARE-motif/TATC-box)、茉莉酸响应元件(TGACG-motif/CGTCA-motif)和水杨酸响应性(TCA-Element);胁迫反应相关元件包括缺氧诱导反应元件(ARE)、防御和胁迫响应元件(TC-rich repeats)、低温反应元件(LTR)、干旱诱导响应元件(MYB结合位点,MBS)、光响应相关元件(TCTmotif/TCCC-motif/GATA-motif/L-box/GA-motif/I-box/LAMP-element/chs- cma1a/Sp1/GT1-motif/MRE/Box4/G-Box)和创伤反应元件(WUN-motif);在启动子上还检测到一些与植物生物合成和发育相关的顺式作用元件,如黄酮类生物合成基因调控(MYB结合位点,MBSI)、种子特异性调控(RYelement)、玉米蛋白代谢调控(O2-site)、细胞周期调控(MSA-like)和胚乳表达调控元件(GCN4-motif)、调控分生组织表达(CAT-box)和昼夜节律调控(circadian)。综上,小麦丙氨酸氨基转移酶可能参与小麦的生长发育、激素响应以及胁迫响应等过程。
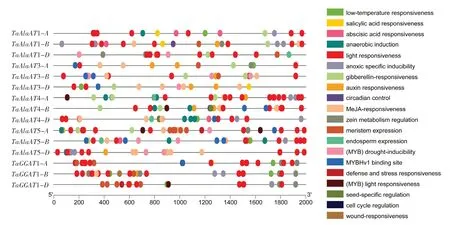
图4 启动子顺式作用元件预测
2.7 丙氨酸氨基转移酶基因在小麦不同组织中的表达模式分析
为了研究丙氨酸氨基转移酶基因在小麦不同组织中的表达模式,本研究以小麦灌浆期根、茎、叶、旗叶以及种子为材料,提取RNA并分析基因表达量。由于同源基因序列相似度较高,分别设计了5对引物,每对引物可同时检测包含了ABD染色体组上的3个同源基因(表1)。由图5可知,所有基因在根、茎、叶、旗叶以及种子中均有表达,且在叶、旗叶中的表达量均较高。

图5 基因的组织表达量分析
2.8 小麦丙氨酸氨基转移酶基因缺氮处理的表达水平分析
丙氨酸氨基转移酶是氮代谢通路中的重要酶类,本研究对小麦丙氨酸氨基转移酶在不同时长缺氮处理的表达模式进行分析。由图6可知,丙氨酸氨基转移酶基因在缺氮处理后表达模式均有变化,其中TaAlaAT1-A/B/D在缺氮处理后相对表达水平上调,在处理0.5 h时的表达量最高,随后下降,在处理24.0 h后,相对表达水平恢复到处理前水平;TaAlaAT3-A/B/D基因在缺氮处理1.0 h后相对表达水平上调,处理2.0 h的表达量达到最高水平,在3.0、6.0、24.0 h时的表达量均高于处理前水平;TaAlaAT4-A/B/D基因对缺氮处理没有显著性的响应;TaAlaAT5-A/B/D基因在缺氮处理后表达量显著下调;TaGGAT1-A/B/D基因在缺氮处理1.0、2.0、3.0、6.0、24.0 h的表达量均上调,在处理2.0 h时表达量达到最高值。综上,丙氨酸氨基转移酶能够参与小麦响应氮胁迫的调控过程。
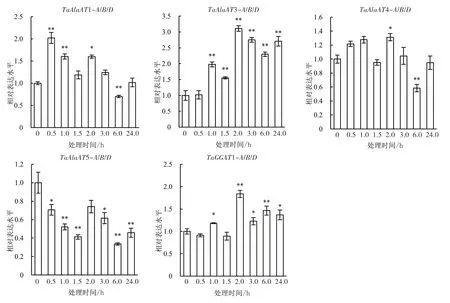
图6 基因在缺氮胁迫不同时间的表达量分析
3 结论与讨论
氮是植物生长所必需的基本元素,对植物施用氮肥可以促进植物的生长发育,提高植物的生物量和产量。小麦(Triticum aestivumL.)是世界“三大谷物”之一,为人类和牲畜提供蛋白质、淀粉和能量。本文结合HMM、BLAST这2种方法在小麦中筛选鉴定出73个丙氨酸氨基转移酶基因,通过进化树分析发现:只有15个基因与拟南芥、水稻、苜蓿中的AlaAT基因亲缘关系比较近(图1),在5个水稻丙氨酸氨基转移酶基因中,除了OsAlaAT2基因,其余4个OsAlaAT基因在小麦中均有3个同源基因,并且3个同源基因分布于ABD不同染色体组上(图2)。目前,有关水稻中GGAT基因的研究还鲜有报道,而在拟南芥中有2个AtGGAT1/2,大麦中有1个MtcAlaAT,在小麦中有3个TaGGAT分别分布于ABD不同染色体组上。研究报道拟南芥中AlaAT1定位于细胞质中,AlaAT2预测定位于线粒体中,GGAT定位于过氧化物酶体中[10,18],水稻OsAlaAT1定位于细胞质中[21]。亚细胞定位预测结果显示,小麦中与水稻OsAlaAT1亲缘关系较近的TaAlaAT1-A/B定位于细胞质中,TaAlaAT1-D定位于叶绿体中,说明OsAlaAT1和TaAlaAT1-A/B可能在细胞质中发挥相同的生物学功能;与拟南芥AlaAT2亲缘关系较近的TaAlaAT3-A/B/D也定位于线粒体;与GGAT亲缘关系较近的TaGGAT1-A/B也定位于过氧化物酶体中,TaGGAT1-D则定位于细胞质中,这是由于TaGGAT1-A/B氨基酸序列C端含有过氧化物酶体靶向信号-PTS1的3个保守多肽(SRL)[17],而TaGGAT1-D氨基酸序列C端则不含有(表2)。蛋白保守结构域分析结果显示:所有基因均含有典型的依赖于磷酸吡哆醛的天冬氨酸转氨酶超家族AAT-like保守结构域,磷酸吡哆醛通过与赖氨酸残基形成希夫碱中间化合物。TaGGAT1与TaAlaAT氨基酸序列保守结构域对比发现,TaGGAT1氨基酸序列N端缺少Motif9和Motif7保守结构域(图3),说明TaGGAT和TaAlaAT功能上的差异可能是由于这2个结构域的缺失导致。
有研究报道,Kendziorek等[16]在小麦中分离出4个丙氨酸氨基转移酶基因,并分别命名为AlaAT1-2和GGAT1-2,定量结果显示只有AlaAT1在缺氧胁迫下转录水平升高,其余3个基因在缺氧胁迫后表达没有显著性差异。这4个基因在缺氮后表达量均下调,其中AlaAT1-2基因表达下调显著,而GGAT1基因下调不显著,GGAT2基因在缺氮处理24.0 h后才开始下调表达。AlaAT基因不仅受缺氧调控,还受光照影响。小麦中AlaAT和GGAT的活性在黄化幼苗中均能检测到,并且其活性在放入有光环境后增强。根据序列比对确认AlaAT1在本文中命名为TaGGAT1-A/B/D,AlaAT2在文中为TaGGAT5-A/B/D,GGAT1在文中为TaAlaAT3-A/B/D,GGAT2在文中为TaGGAT1-A/B/D。本文定量结果显示TaAlaAT1-A/B/D、TaAlaAT3-A/B/D和TaGGAT1-A/B/D基因在缺氮处理后均被诱导上调表达,其中TaAlaAT1-A/B/D基因在处理0.5 h时表达水平上调,随后下降,在处理24.0 h后恢复到处理前水平;TaAlaAT3-A/B/D基因缺氮处理1.0 h后表达水平上调,处理2.0 h时达到最高水平,随后在处理24.0 h内的表达量均高于处理前水平;TaGGAT1-A/B/D基因在缺氮处理1.0 h后表达水平上调,处理2.0 h时达到最高表达水平;而TaAlaAT5-A/B/D在基因缺氮处理后表达水平下调。上述基因可能在不同时间参与小麦响应氮胁迫调控过程。TaAlaAT4-A/B/D基因在缺氮处理后表达量与处理前相比没有显著性的差异,说明该基因可能未参与小麦氮代谢的调控过程。本文定量结果与前人报道上存在的差异可能是由于小麦品种差异造成。小麦中AlaAT基因被证实在小麦根、茎、叶、雄蕊和雌蕊中均有表达,但在叶片中的表达量最高,该基因参与小麦响应镉胁迫调控过程,经镉胁迫处理后,该基因的表达量先上升后下降[26]。AlaAT基因即本文中的TaGGAT1-B基因,本文组织表达分析显示15个丙氨酸氨基转移酶基因在根、茎、叶、种子和旗叶中均有表达,且在叶、旗叶中的表达量均较高。

