菱叶绣线菊彩叶园艺品种‘粉霜’和‘黄金喷泉’的叶绿体基因组研究
张巧玲, 陈琳, 张淑燕, 颜海飞
菱叶绣线菊彩叶园艺品种‘粉霜’和‘黄金喷泉’的叶绿体基因组研究
张巧玲1, 陈琳1, 张淑燕2, 颜海飞3*
(1. 杭州西溪国家湿地公园生态文化研究中心,杭州 310013;2. 华南农业大学生命科学学院,广州 510642;3. 中国科学院华南植物园,广州 510650)
基于菱叶绣线菊(×)培育出了很多的彩叶园艺品种,其中‘粉霜’彩叶绣线菊(‘Pink Ice’)和‘黄金喷泉’菱叶绣线菊(‘Gold Fountain’)是两个性状优良的品种,这两个彩叶品种的形成机制尚缺乏深入研究。该研究基于二代测序的浅层测序技术,对‘粉霜’和‘黄金喷泉’的叶绿体基因组进行组装、注释、绘制其叶绿体基因组图谱;结合网上已有的绣线菊属植物的叶绿体全基因组开展比较基因组学研究。结果表明,两个品种的叶绿体基因组均为典型的四分体结构,即含有1个LSC、1个SSC及2个IR;‘粉霜’和‘黄金喷泉’的叶绿体基因组大小分别为155 953和155 941 bp,各含有130个基因,包括85个蛋白编码基因,37个转运RNA基因和8个核糖体RNA基因;分别含有67、69个简单重复序列,其中,单核苷酸重复序列最多。筛选出这两个叶绿体基因组内7个高变异区域,分别为、、、、、、。菱叶绣线菊、‘粉霜’和‘黄金喷泉’虽有非常近缘的关系,但并未聚成单系。该研究首次获得了两种绣线菊属彩叶品种的叶绿体基因组,为进一步理解绣线菊属及其彩叶园艺品种的亲缘关系提供了大量有用信息,并为今后该属更多园艺资源的发掘奠定了基础。
绣线菊属;园艺品种;叶绿体基因组;系统发育分析
叶绿体是植物细胞中一种半自主复制的细胞器,是植物进行光合作用的场所,能将光能转化为化学能进行储存,因此,叶绿体对植物乃至人类的生存至关重要。目前,多数观点认为叶绿体的起源来源于真核细胞早期捕获蓝藻或光合细菌共生形成(内共生假说[1])。叶绿体基因组是叶绿体具有的独立遗传物质,最早完成叶绿体基因组测序的物种为烟草()[2]与地钱()[3]。在早期,叶绿体基因组序列需首先提取叶绿体DNA,并随机打断或酶切后才能通过克隆和桑格测序技术获得,获取过程费时费力[4],随后,通过设计大量保守引物及长PCR方法[5]使叶绿体基因组学研究进入了快车道。
随着高通量测序技术的快速发展,其测序数据通量和准确率都得到了极大提升,而且测序费用较为低廉,使该技术已普遍应用于当前的生物学研究中。基因组浅层测序是一种二代全基因组测序方法,可高效便捷地获取基因组中高重复的基因或区域(如重复元件、核DNA的核糖体内转录间隔区等)[5]。叶绿体基因组在光合组织中的含量较高,约为细胞内总DNA含量的10%~20%[6],因而基因组浅层测序技术可获得高丰度的叶绿体基因组信息。此外,不断发展的组装及注释技术[7–9]也使近年来叶绿体基因组数据呈爆发式增长。
植物叶绿体基因组在园艺植物研究方面具有重要价值[6],叶绿体基因组可用于园艺植物的起源、演化历史及遗传多样性方面的研究。叶绿体基因组通常为单亲遗传(且绝大多数为母系遗传),其序列结构及组成相对保守,核苷酸变异虽低于核基因但高于植物线粒体基因。叶绿体基因组的有效群体大小是雌雄异株植物的1/4或雌雄同株植物的1/2,因此叶绿体单倍型的固定时间及溯祖时间都较短,有利于我们发现其遗传变异并追溯其演化历史[6]。因此,叶绿体基因组在植物起源、演化历史、遗传多样性、杂交方向等领域具有重要研究价值。叶绿体基因组有利于园艺植物的分子定向育种和杂交选育研究。叶绿素含量是植物叶片的重要生理指标, 但杂交育种或基因工程会导致核质不亲和或叶绿素合成不正常而形成花叶或白化,增加园艺植物的观赏价值[6,10–11]。叶绿体的合成或质体形成过程中的自发突变是导致一些园艺嵌合体植物[如天竺葵(×)、牵牛花()、紫茉莉()、大丽菊()等]花斑现象形成的原因[12]。最近,有研究比较了20种嵌合体植物的叶组织(绿叶组织和白化组织)的叶绿体基因组,发现了8个叶绿体编码的RNA聚合酶或光系统有关的基因中存在14个与白化现象产生相关的点突变[11]。
蔷薇科(Rosaceae)绣线菊属()是一类具有重要园艺观赏价值的植物类群。根据最新的蔷薇科分类系统,绣线菊属归属于桃亚科(Amygda- loideae)绣线菊族(Spiraeeae)[13–14],为北温带分布的大属,全球包含约90种(http://www.plantsoftheworld online.org),间断分布于欧、亚和北美洲地区。我国是绣线菊属的现代分布和分化中心,多达70种(其中47种为我国特有[15])。因此,我国拥有丰富的绣线菊属的种质资源。绣线菊属植物多为伞形、伞形总状、复伞房或圆锥花序,多数种类具有美丽的花朵、细致的叶片,生性强健,具有耐寒、耐旱、繁殖简便等优点,可应用于花篱、花径、花坛的栽植及园林主景的构造[16],是具有重要经济价值的观赏类园艺植物。然而,我国绣线菊属植物的园林应用仅限于几种常见种类,与我国丰富的种质资源十分不符。基于菱叶绣线菊(×)培育出了多种彩叶园艺品种,其中‘粉霜’彩叶绣线菊(‘Pink Ice’)和‘黄金喷泉’菱叶绣线菊(‘Gold Fountain’)是两个性状优良的菱叶绣线菊品种,表现出较好的园艺价值。
近年来,绣线菊属植物的叶绿体基因组研究得到了一些发展。目前,已公开8种绣线菊属植物的叶绿体基因组数据[14,17–23],绣线菊属叶绿体基因组具有典型的四分体结构,长度为156 226~155 981 kb,编码基因的数量变化较大,菱叶绣线菊有129个编码基因[23],而渐尖叶粉花绣线菊(var.)有125个编码基因[22],这可能是注释不完整导致的,因此需进行更详尽的注释及比较基因组学研究。此外,‘粉霜’和‘黄金喷泉’的叶色形成机制及与绣线菊属其他植物的演化关系尚未开展研究。鉴于叶绿体基因组在叶片色素形成及植物亲缘关系及进化方面的重要价值,本研究基于二代测序的浅层测序技术,对‘粉霜’和‘黄金喷泉’的叶绿体基因组进行组装、注释、绘制其叶绿体基因组图谱;结合网上已有的绣线菊属植物的叶绿体全基因组开展比较基因组学研究,探讨‘粉霜’和‘黄金喷泉’的叶绿体基因组的组成及高变异位点;以及这两个品种与绣线菊属植物的亲缘关系,为深入解析菱叶绣线菊及栽培品种的遗传基础和开发绣线菊属的园艺资源奠定基础。
1 材料和方法
1.1 植物材料和DNA测序
‘粉霜’彩叶绣线菊(凭证标本号:Yan Hai-Fei Y1940)和‘黄金喷泉’菱叶绣线菊(凭证标本号:Yan Hai-Fei Y1948)植株购自浙江桐乡四季花园家庭农场,并栽种于杭州植物园。凭证标本保存在中国科学院华南植物园标本馆。采集新鲜健康叶片保存在透气的纸袋中,并放入带有变色硅胶的封口袋中干燥保藏。在实验室采用CTAB法[24]从干燥叶片中提取总DNA,并在深圳华大基因股份有限公司(武汉)的MGISEQ-2000基因组测序平台上开展基因组浅层测序,获得原始读长数据(约2 Gbp)。
1.2 叶绿体基因组组装及注释
原始读长数据在去除接头和低质量数据后,使用软件GetOrganelle v1.7.3.5[8]进行叶绿体基因组的从头组装。将组装好的序列使用在线注释网站Geseq (https://chlorobox.mpimp-golm.mpg.de/geseq.html[25])进行基因注释,并在Geneious Prime 2019软件[26]中进行适当的手工调整。通过线上工具OGDRAW v1.3.1 (https://chlorobox.mpimp-golm.mpg.de/OGDraw.html)绘制叶绿体基因组序列物理图谱[27]。
1.3 叶绿体基因组的比较分析
将叶绿体基因组导入Geneious Prime 2019软件,比较叶绿体基因组总长度及大单拷贝区(LSC)、小单拷贝区(SSC)和反向重复区(IR)的长度、基因组成、GC含量等叶绿体基因组的基本信息。利用mVISTA在线软件(https://genome.lbl.gov/vista/mvista/submit. shtml)的Shuffle-LAGAN模型检测叶绿体基因组的序列异质性。利用在线软件Reputer (https://bibiserv. cebitec.uni-bielefeld.de/repuer)鉴定叶绿体基因组的重复序列类型(如正向重复、倒置重复、互补重复、回文重复)[28];其参数设置为:最小重复长度设置为8,最小排列值为50。
1.4 叶绿体简单重复序列及核苷酸多态性分析
使用简单重复序列(SSR)识别软件MISA[29]鉴定叶绿体基因组的单核苷酸、二核甘酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸SSR;每种类型最小重复参数设置依次为10、5、4、3、3、3;两个SSR之间的最小距离设置为100 bp;当两个SSR之间的距离小于100 bp时识别为复合微卫星(compound SSR)。核苷酸多态性分析采用DnaSP 6.0软件[30],窗口大小设置为500 bp,步移大小设置为200 bp。
1.5 系统发育分析
从NCBI的GenBank中下载所有绣线菊属物种的叶绿体基因组序列数据,以中华绣线梅() (MT683856)和华北珍珠梅() (NC_062461)为外类群(表1)。本研究提取出所有叶绿体基因组中共有的蛋白质编码区(Protein-coding genes, PCGs),并采用MAFFT v7.308软件[31]比对后形成串联序列矩阵。利用RAxML v8.2.10软件[32]序列矩阵进行最大似然树的构建,核苷酸替代模型设置为GTRGAMMA,执行1 000次重复自举抽样以获得系统树上每个分支的支持率。
2 结果和分析
2.1 叶绿体基因组的结构
‘粉霜’和‘黄金喷泉’的叶绿体基因组长度分别为155 953和155 941 bp,均稍小于菱叶绣线菊(155 957 bp),也小于绝大多数已知绣线菊属植物,除毛枝绣线菊()和蒙古绣线菊()外(表1), ‘粉霜’和‘黄金喷泉’的叶绿体基因组与其他绣线菊属物种的一致,均为典型的四分体结构,即含有1个LSC、1个SSC和2个IR (图1)。‘粉霜’叶绿体基因组的LSC、SSC和IR的长度分别为84 384、18 883和26 342 bp,‘黄金喷泉’分别为84 395、18 860和26 343 bp。‘粉霜’与菱叶绣线菊叶绿体基因组的GC含量均为36.8%,而‘黄金喷泉’为36.7%,说明绣线菊属植物叶绿体基因组的GC含量相近。通过重新注释并仔细检查,除渐尖叶粉花绣线菊缺失基因外,该属叶绿体基因组的基因组成高度保守。
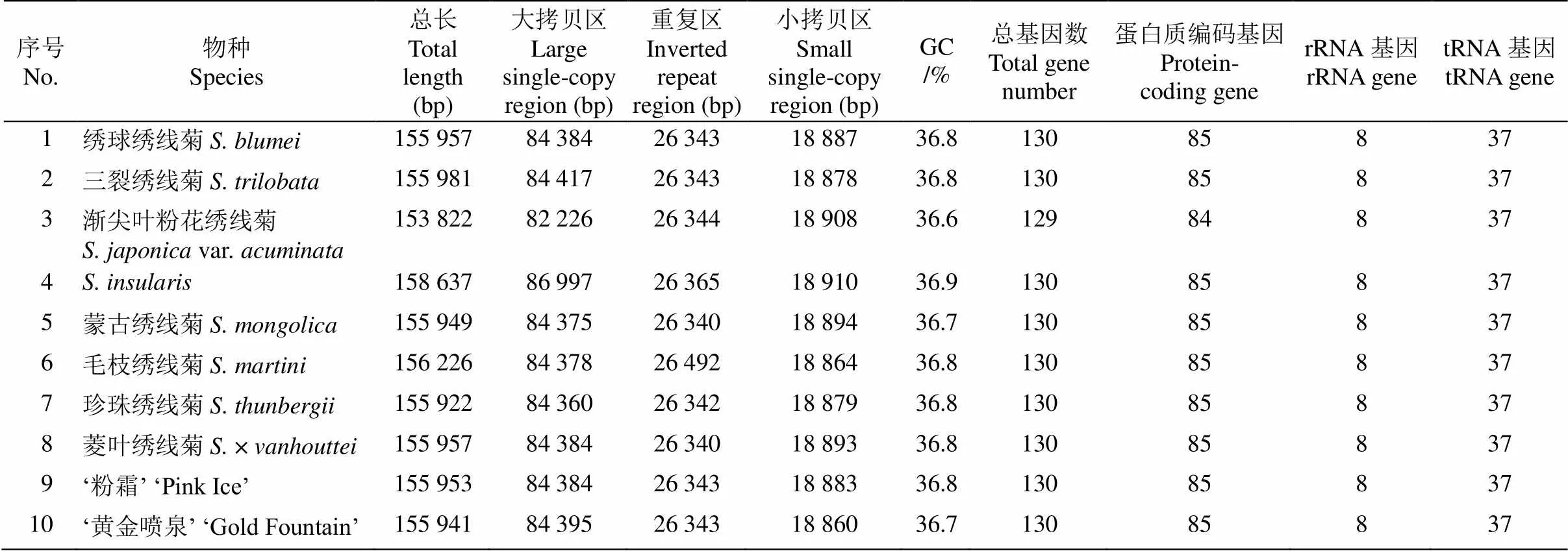
表1 绣线菊属叶绿体基因组的基本特征
2.2 叶绿体基因组的基因组成
基因注释结果表明, ‘粉霜’与‘黄金喷泉’叶绿体基因组各含有130个基因,包括85个蛋白质编码基因,37个转运RNA基因(tRNA gene)和8个核糖体RNA基因(rRNA gene)。这些基因可划分成光合作用相关基因、自身表达相关基因和其他基因3类(表2)。‘粉霜’与‘黄金喷泉’叶绿体基因组中这3类基因的种类和数量均完全一致。光合作用相关的基因均有45个,其中5个基因(F、B、D、A和B)含1个内含子,B分布在IR上,因此具有2个拷贝。自身表达相关基因有61个基因, 其中C1、2、rps12、16、A、G、I、K、L、V均有1个内含子,而P含有2个内含子, 其中, 有15个基因分布于IR上(2237124.551623AIILNRV),因此均有2个拷贝。其他基因中的1和2分布于IR上,均有2个拷贝,3含有2个内含子。

表2 ‘粉霜’和‘黄金喷泉’叶绿体基因组的注释基因及其归类
(2): 含有2个重复单元;*: 含有1个内含子;**: 含有2个内含子。
(2): Two repeat units; *: With one intron; **: With two introns.
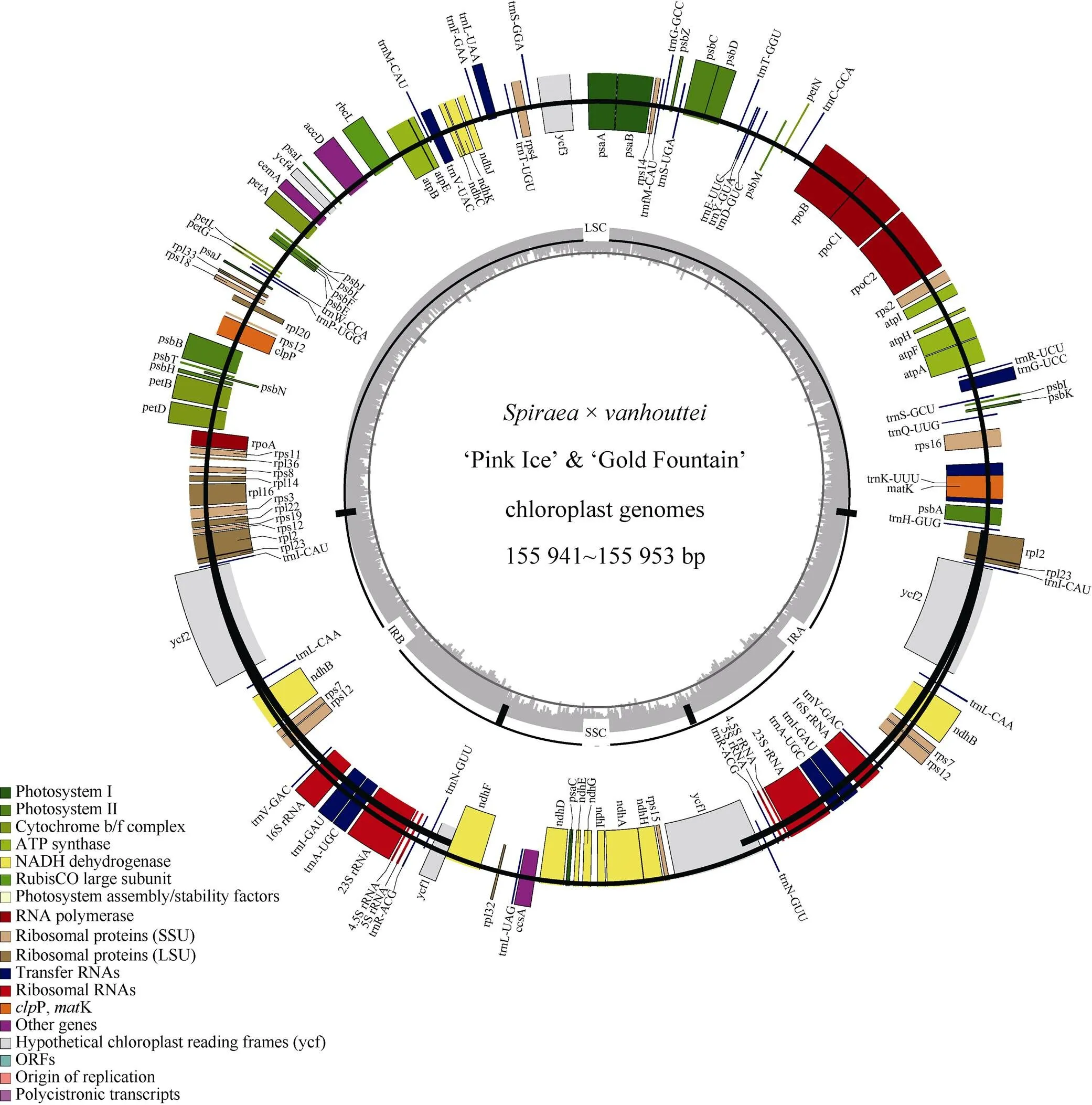
图1 ‘粉霜’和‘黄金喷泉’叶绿体基因组
2.3 重复序列及简单重复序列(SSR)分析
在‘粉霜’叶绿体基因组中共鉴定出49个重复序列,其中正向重复和回文重复各19个,反向重复9个,互补重复2个; 在‘黄金喷泉’中鉴定出49个重复序列,其中正向重复和反向重复均与‘粉霜’一致,但回文重复和互补重复分别为20个和1个; 在菱叶绣线菊中鉴定到49个重复序列,其中2个互补重复、21个回文重复、19个正向重复、7个反向重复(表3)。
‘粉霜’、‘黄金喷泉’和菱叶绣线菊的叶绿体基因组分别含有67、69和65个简单重复序列(表4)。其中,单核苷酸重复序列最多,4碱基重复序列次之,2碱基重复序列均为6个。‘粉霜’叶绿体基因组有1个5碱基重复序列,而其余2种菱叶绣线菊均不存在这类重复。本研究的3种菱叶绣线菊均不存在3碱基重复和6碱基重复,其叶绿体基因组的简单重复特征也基本反映了绣线菊属的情况。
2.4 叶绿体基因组变异分析
以三裂绣线菊()的叶绿体全基因组为参考序列,利用mVISTA在线软件基于Shuffle- LAGAN模型检测绣线菊属叶绿体基因组的序列异质性(图2)。结果表明, 绣线菊属叶绿体基因组较为保守,特别是IR区的变异程度整体较低。变异多发生在大小拷贝区(LSC和SSC区),特别是基因间隔区(Intergenic spacers: IGSs)的变异信号更多。
使用DnaSP6软件的滑动窗口方法筛选‘粉霜’和‘黄金喷泉’叶绿体高变区域(图3),结果存在7个高变异区域,分别为HA、K、RA、TD、C、32和1。
2.5 系统发育分析
对10个有叶绿体基因组数据的绣线菊属物种进行了系统发育分析(图4)。结果表明,除1个节点外,其余分支节点均有很高的支持率(>95%);菱叶绣线菊、‘粉霜’和‘黄金喷泉’虽有非常近缘的关系,但并未聚成单系;菱叶绣线菊与绣球绣线菊()形成姐妹群,该分支再与‘黄金喷泉’组成姐妹关系,但其支持率非常低(37%),其关系并未解决。
3 结论和讨论
当前,欧美国家已在原产于亚洲的绣线菊属物种中进行了大量的新品种培育,基于粉花绣线菊()培育出‘金焰’绣线菊(‘Gold Flame’)、‘金山’绣线菊(‘Gold Mound’)、‘魔毡’绣线菊(‘Walbuma’PBR)等大量园艺品种。菱叶绣线菊是麻叶绣线菊()和三裂绣线菊的人工杂交种,广泛栽培于我国的山东、江苏、广东、广西、四川[33]。基于菱叶绣线菊培育出了很多的彩叶园艺品种,其中‘粉霜’彩叶绣线菊和‘黄金喷泉’菱叶绣线菊是两个性状优良的菱叶绣线菊品种,具有极高的园艺价值。我国是绣线菊属的现代分布和分化中心,种类多达70种(其中47种为我国特有种[15]),拥有丰富的绣线菊植物资源,但其园艺资源亟待开发。绣线菊属叶绿体基因组的研究还未深入开展,据统计,仅有8个绣线菊属物种具有叶绿体基因组数据[14,17–23]。通过绣线菊属叶绿体基因组学研究将有助于厘清该属植物的遗传资源及亲缘关系,有利于进一步发掘其园艺资源。
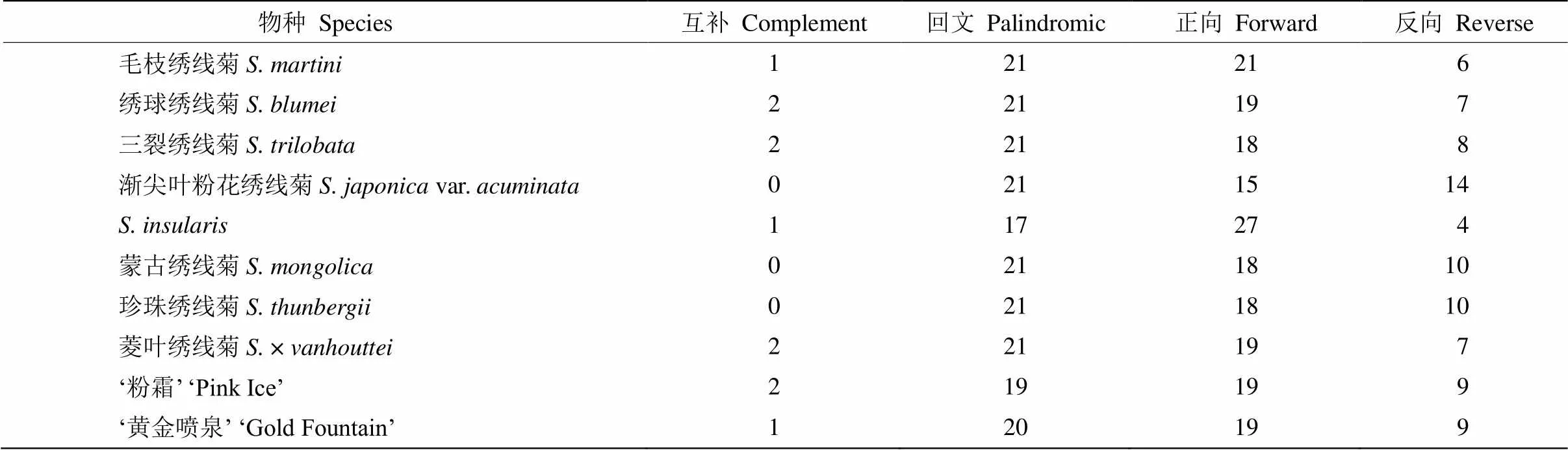
表3 绣线菊属植物叶绿体基因组的重复序列

表4 绣线菊属叶绿体基因组的简单重复序列SSR类型

图2 绣线菊属植物叶绿体基因组的比对分析

图3 ‘粉霜’和‘黄金喷泉’叶绿体基因组核苷酸多态性(Pi)的滑动窗口分析
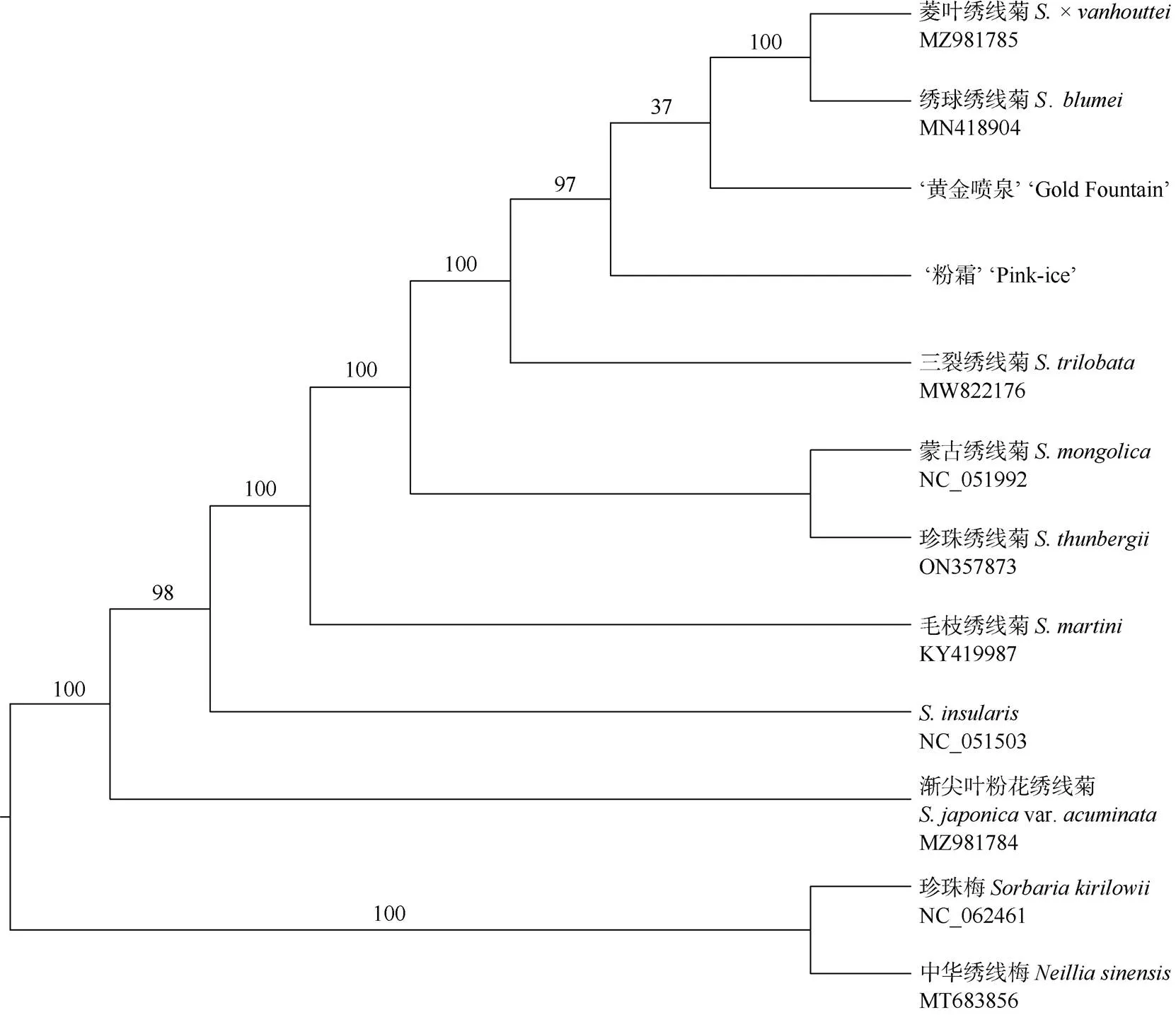
图4 基于叶绿体基因组中编码基因构建的绣线菊属植物最大似然树。分支上的数字为支持率
本研究首次测序组装并分析了绣线菊属中两种重要彩叶绣线菊‘粉霜’和‘黄金喷泉’的叶绿体基因组,发现这两种彩叶绣线菊的叶绿体基因组均为典型的四分体结构,即含有1个LSC、1个SSC和2个IR,并与其他绣线菊属物种的叶绿体基因组结构一致,是植物叶绿体基因组中最常见的结构类型[34]。‘粉霜’和‘黄金喷泉’叶绿体基因组分别为155 953和155 941 bp,是绣线菊属叶绿体基因组中较小的。此外,本研究通过比较绣线菊属叶绿体基因组,发现除渐尖叶粉花绣线菊缺失基因外,该属的叶绿体基因组在基因组成和GC含量均较稳定。一般而言,光合作用相关的基因以往在寄生植物中大量缺失,可能与寄生植物无需进行光合作用有关[35–37],而这类基因的丢失现象近年来也在其他植物中发现,可能是由于其他基因(如核基因)发挥了其功能有关[38]。渐尖叶粉花绣线菊叶绿体基因组丢失基因的原因值得后续深入研究。
叶绿体基因组的编码区序列进化速率慢,因而相对保守,而基因内含子及基因间隔区,以及简单重复序列均有较高的变异速率,叶绿体基因组内不同的区域具有不同变异速率的特点非常适用于不同分类阶元的系统发育研究。例如,保守的编码基因适用于科、目及以上分类阶元的系统发育分析[39],而其非编码区可用于近缘物种或种下的进化研究[40]。叶绿体简单重复序列(SSR)是研究植物系统与进化的重要遗传标记,主要为单核苷酸重复序列[41–42]。在本研究中,‘粉霜’、‘黄金喷泉’及菱叶绣线菊的叶绿体基因组均含有最多的单核苷酸重复序列,与以往的研究结果一致[41]。此外,本研究还发现4碱基重复序列也较多,为后续开展相关研究奠定了基础。除叶绿体简单重复序列外,本研究使用滑动窗口方法筛选出‘粉霜’和‘黄金喷泉’的7个高变异区域,这些高变位点有4个处于编码区,其余位于基因间隔区。在这些高变位点中,HA和1在其他植物中也具有很高的变异,因此曾被推荐为DNA条形码候选位点[43–44]。此外,其他片段也具有很高的变异率(特别是32),说明这些位点在绣线菊属中具有作为DNA条形码位点的潜力,为后续开展相关研究提供了重要研究方向。
本研究对10个有叶绿体基因组数据的绣线菊属物种进行了系统发育分析,结果表明几乎所有的分支节点均有很高的支持率(>95%)。在以往的研究中,菱叶绣线菊与绣球绣线菊、三裂绣线菊等具有非常近缘的亲缘关系,但彼此的关系并未解决[45]。本研究支持以上结论,并进一步厘清了这些种的关系,说明绣线菊属叶绿体基因组的蛋白质编码区已积累了较大变异,有充足的系统发育信号,为今后大规模开展相关研究奠定了基础。菱叶绣线菊是麻叶绣球和三裂绣线菊的人工杂交种,本研究由于缺少麻叶绣线菊的样品,并不能使用叶绿体基因组进行杂交方向的判定,后续通过引入麻叶绣线菊的样品将有助于解决上述问题。本研究还发现,菱叶绣线菊、‘粉霜’和‘黄金喷泉’虽有近缘关系,但并未聚成单系,这说明两个菱叶绣线菊园艺品种可能分别通过杂交培育的,也可能是由于菱叶绣线菊(尤其是叶的颜色)经历了强烈的人工选择,从而导致其叶绿体基因突变很快积累,并通过无性繁殖的方式保持了这种变异。叶绿体基因组研究可为我们进一步理解绣线菊属及其彩叶园艺品种的亲缘关系提供大量有用信息,为今后发掘该属更多有价值的园艺资源奠定了基础。
[1] RAVEN J A, ALLEN J F. Genomics and chloroplast evolution: What did cyanobacteria do for plants? [J]. Genome Biol, 2003, 4(3): 209. doi: 10.1186/gb-2003-4-3-209.
[2] SHINOZAKI K, OHME M, TANAKA M, et al. The complete nucleotide sequence of the tobacco chloroplast genome: Its gene organization and expression [J]. EMBO J, 1986, 5(9): 2043–2049. doi: 10.1002/j.1460-2075.1986.tb04464.x.
[3] OHYAMA K, FUKUZAWA H, KOHCHI T, et al. Chloroplast gene organization deduced from complete sequence of liverwortchloroplast DNA [J]. Nature, 1986, 322(6079): 572–574. doi: 10.1038/322572a0.
[4] FAN L J. Plant Genomics [M]. Beijing: Science Press, 2020: 181–183. [樊龙江. 植物基因组学 [M]. 北京: 科学出版社, 2020: 181–183.]
[5] STRAUB S C K, PARKS M, WEITEMIER K, et al. Navigating the tip of the genomic iceberg: Next-generation sequencing for plant syste- matics [J]. Am J Bot, 2012, 99(2): 349–364. doi: 10.3732/ajb.1100335.
[6] RAUWOLF U, GOLCZYK H, GREINER S, et al. Variable amounts of DNA related to the size of chloroplasts: III. Biochemical determina- tions of DNA amounts per organelle [J]. Mol Genet Genom, 2010, 283(1): 35–47. doi: 10.1007/s00438-009-0491-1.
[7] DIERCKXSENS N, MARDULYN P, SMITS G. NOVOPlasty:assembly of organelle genomes from whole genome data [J]. Nucleic Acids Res, 2017, 45(4): e18. doi: 10.1093/nar/gkw955.
[8] JIN J J, YU W B, YANG J B, et al. GetOrganelle: A fast and versatile toolkit for accurate de novo assembly of organelle genomes [J]. Genome Biol, 2020, 21(1): 241. doi: 10.1186/s13059-020-02154-5.
[9] SHI L C, CHEN H M, JIANG M, et al. CPGAVAS2, an integrated plastome sequence annotator and analyzer [J]. Nucl Acids Res, 2019, 47(W1): W65–W73. doi: 10.1093/nar/gkz345.
[10] GREINER S, RAUWOLF U, MEURER J, et al. The role of plastids in plant speciation [J]. Mol Ecol, 2011, 20(4): 671–691. doi: 10.1111/j. 1365-294X.2010.04984.x.
[11] PARK H S, JEON J H, CHO W, et al. High-throughput discovery of plastid genes causing albino phenotypes in ornamental chimeric plants [J]. Hort Res, 2022, 10(1): uhac246. doi: 10.1093/hr/uhac246.
[12] ZHOU Y, ZHOU H G, ZHANG X L. The study on variegation and chimerism in ornamental plants [J]. J Guangxi Agric Biol Sci, 1999, 18(4): 304–309. [周焱, 周厚高, 张西丽. 观赏植物花叶现象研究现状 [J]. 广西农业生物科学, 1999, 18(4): 304–309.]
[13] POTTER D, ERIKSSON T, EVANS R C, et al. Phylogeny and classification of Rosaceae [J]. Plant Syst Evol, 2007, 266(1): 5–43. doi: 10.1007/s00606-007-0539-9.
[14] ZHANG S D, JIN J J, CHEN S Y, et al. Diversification of Rosaceae since the late Cretaceous based on plastid phylogenomics [J]. New Phytol, 2017, 214(3): 1355–1367. doi: 10.1111/nph.14461.
[15] WU Z Y, Peter H R. Flora of China, Vol. 9 [M]. Beijing: Science Press & St. Louis: Missouri Botanical Garden Press, 2003: 47–73.
[16] CHENG Y H, WU R H. Plant resource and application in garden ofin Heilongjiang Province [J]. For Invest Des, 2008(2): 62–63. [程银虎, 武荣贵. 黑龙江省绣线菊属植物资源及在园林中的应用 [J]. 林业勘查设计, 2008(2): 62–63.]
[17] QIN H, ZHU X X, ZHANG X, et al. Characterization of the complete plastome of(Rosaceae), a perennial shrub [J]. Mitochondrial DNA B, 2022, 7(1): 249–250. doi: 10.1080/23802359. 2021.2018948.
[18] YANG J Y, KANG G H, PAK J H, et al. Characterization and comparison of two complete plastomes of Rosaceae species (var.and) endemic to Ulleung Island, Korea [J]. Int J Mol Sci, 2020, 21(14): 4933. doi: 493310.3390/ ijms21144933.
[19] SHEN W Y, LIN J, LIN H F. The chloroplast genome of(Rosaceae) [J]. Mitochondrial DNA B, 2022, 7(10): 1879– 1881. doi: 10.1080/23802359.2022.2135406.
[20] MA Y J, GUO Y P, ZHU Y, et al. The complete chloroplast genome ofmaxim [J]. Mitochondrial DNA B, 2021, 6(5): 1614–1616. doi: 10.1080/23802359.2021.1926351.
[21] HUO Y, YAN M, ZHAO X Q, et al. The complete chloroplast genome sequence ofG. Don (Rosaceae) [J]. Mitochondrial DNA B, 2019, 4(2): 3671–3672. doi: 10.1080/23802359.2019.1678434.
[22] WANG Q, CHEN M M, HU X F, et al. The complete chloroplast genome sequence ofvar.Franch. (Rosaceae) [J]. Mitochondrial DNA B, 2022, 7(1): 275–276. doi: 10. 1080/23802359.2022.2028590.
[23] CHEN M M, WANG R H, SHA H K, et al. The complete chloroplast genome sequence of×(Briot) Zabel (Rosaceae) [J]. Mitochondrial DNA B, 2022, 7(3): 505–506. doi: 10.1080/23802359. 2022.2052369.
[24] DOYLE J J, DOYLE J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochem Bull, 1987, 19: 11–15.
[25] TILLICH M, LEHWARK P, PELLIZZER T, et al. GeSeq: Versatile and accurate annotation of organelle genomes [J]. Nucleic Acids Res, 2017, 45(W1): W6–W11. doi: 10.1093/nar/gkx391.
[26] KEARSE M, MOIR R, WILSON A, et al. Geneious basic: An inte- grated and extendable desktop software platform for the organization and analysis of sequence data [J]. Bioinformatics, 2012, 28(12): 1647– 1649. doi: 10.1093/bioinformatics/bts199.
[27] GREINER S, LEHWARK P, BOCK R. OrganellarGenomeDRAW (OGDRAW) version 1.3.1: Expanded toolkit for the graphical visuali- zation of organellar genomes [J]. Nucl Acids Res, 2019, 47(W1): W59– W64. doi: 10.1093/nar/gkz238.
[28] KURTZ S, CHOUDHURI J V, OHLEBUSCH E, et al. REPuter: The manifold applications of repeat analysis on a genomic scale [J]. Nucl Acids Res, 2001, 29(22): 4633–4642. doi: 10.1093/nar/29.22.4633.
[29] BEIER S, THIEL T, MÜNCH T, et al. MISA-web: A web server for microsatellite prediction [J]. Bioinformatics, 2017, 33(16): 2583–2585. doi: 10.1093/bioinformatics/btx198.
[30] ROZAS J, FERRER-MATA A, SÁNCHEZ-DELBARRIO J C, et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets [J]. Mol Biol Evol, 2017, 34(12): 3299–3302. doi: 10.1093/molbev/msx 248.
[31] KATOH K, STANDLEY D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability [J]. Mol Biol Evol, 2013, 30(4): 772–780. doi: 10.1093/molbev/mst010.
[32] STAMATAKIS A. RAxML version 8: A tool for phylogenetic analysis and post-analysis of large phylogenies [J]. Bioinformatics, 2014, 30(9): 1312–1313. doi: 10.1093/bioinformatics/btu033.
[33] LU L T. The evolution and distribution of subfam. Spiraeoideae (Rosaceae) of China, with special reference to distribution of the subfamily in the world [J]. Acta Phytotaxon Sin, 1996, 34(4): 361–375. [陆玲娣. 中国蔷薇科绣线菊亚科的演化、分布——兼述世界绣线菊亚科植物的分布 [J]. 植物分类学报, 1996, 34(4): 361–375.
[34] WICKE S, SCHNEEWEISS G M, DEPAMPHILIS C W, et al. The evolution of the plastid chromosome in land plants: Gene content, gene order, gene function [J]. Plant Mol Biol, 2011, 76(3): 273–297. doi: 10.1007/s11103-011-9762-4.
[35] BRAUKMANN T, KUZMINA M, STEFANOVIĆ S. Plastid genome evolution across the genus(Convolvulaceae): Two clades within subgenusexhibit extensive gene loss [J]. J Exp Bot, 2013, 64(4): 977–989. doi: 10.1093/jxb/ers391.
[36] DONAHER N, TANIFUJI G, ONODERA N T, et al. The complete plastid genome sequence of the secondarily nonphotosynthetic alga cryptomonas paramecium: Reduction, compaction, and accelerated evolutionary rate [J]. Genome Biol Evol, 2009, 1: 439–448. doi: 10. 1093/gbe/evp047.
[37] GRAHAM S W, LAM V K Y, MERCKX V S F T. Plastomes on the edge: The evolutionary breakdown of mycoheterotroph plastid genomes [J]. New Phytol, 2017, 214(1): 48–55. doi: 10.1111/nph. 14398.
[38] MOHANTA T K, MISHRA A K, KHAN A, et al. Gene loss and evolution of the plastome [J]. Genes, 2020, 11(10): 1133. doi: 10.3390/ genes11101133.
[39] SOLTIS D E, SMITH S A, CELLINESE N, et al. Angiosperm phylogeny: 17 genes, 640 taxa [J]. Am J Bot, 2011, 98(4): 704–730. doi: 10.3732/ajb.1000404.
[40] YAN H F, ZHANG C Y, WANG F Y, et al. Population expanding with the phalanx model and lineages split by environmental heterogeneity: A case study ofin subtropical China [J]. PLoS ONE, 2012, 7(9): e41315. doi: 10.1371/journal.pone.0041315.
[41] EBERT D, PEAKALL R. Chloroplast simple sequence repeats (cpSSRs): Technical resources and recommendations for expanding cpssr discovery and applications to a wide array of plant species [J]. Mol Ecol Resour, 2009, 9(3): 673–690. doi: 10.1111/j.1755-0998.2008. 02319.x.
[42] YAN H F, PENG C I, HU C M, et al. Phylogeographic structure of(Primulaceae) inferred from chloroplast micro- satellites (cpSSRs) markers [J]. Acta Phytotaxon Sin, 2007, 45(4): 488–496. doi: 10.1360/aps06214.
[43] DONG W P, XU C, LI C H, et al., the most promising plastid DNA barcode of land plants [J]. Sci Rep, 2015, 5: 8348. doi: 10.1038/ srep08348.
[44] KRESS W J, ERICKSON D L. A two-locus global DNA barcode for land plants: The codingL gene complements the non-codingspacer region [J]. PLoS ONE, 2007, 2(6): e508. doi: 10.1371/ journal.pone.0000508.
[45] YU S X, GADAGKAR S R, POTTER D, et al. Phylogeny of(Rosaceae) based on plastid and nuclear molecular data: Implications for morphological character evolution and systematics [J]. Perspect Plant Ecol Evol Syst, 2018, 34: 109–119. doi: 10.1016/j.ppees.2018. 08.003.
Chloroplast Genome Analyses ofבPink Ice’ and ‘Gold Fountain’
ZHANG Qiaoling1, CHEN Lin1, ZHANG Shuyan2, YAN Haifei3*
(1. Hangzhou Xixi National Wetland Park Ecology & Culture Research Center, Hangzhou 310013, China; 2. College of Life Sciences, South China Agricultural University,Guangzhou 510642 China; 3. South China Botanical Garden, Chinese Academy of Sciences,Guangzhou 510650, China)
Several foliage horticultural varieties have been bred based on, including ‘Pink Ice’ and ‘Gold Fountain’, which exhibit excellent horticultural traits. However, the underlying mechanism of these two colorful leaf varieties remain poorly understood. To address this knowledge gap, the complete chloroplast genomes of ‘Pink Ice’ and ‘Gold Fountain’ were assembled by using genome skimming sequencing. Comparative genomic analysis of all available chloroplast genomes of the genusto date was studied, including variation hotspot analysis, simple repetitive sequence analysis, and phylogenetic analysis. The results showed that both varieties had a typical quadripartite structure, consisting of one LSC, one SSC, and two IRs. The chloroplast genomes of ‘Pink Ice’ and ‘Gold Fountain’ were 155 953 and 155 941 bp in size, respectively, and each contains 130 genes, including 85 protein-coding genes, 37 transfer RNA genes and 8 ribosomal RNA genes. The two chloroplast genomes also contained 67 and 69 simple repetitive sequences, respectively, with single nucleotide repeat sequences being the most abundant. There were seven highly variable regions within these two chloroplast genomes, includingHAKRATDC32and1. Finally, phylogenetic analysis revealed that ‘Pink Ice’ and ‘Gold Fountain’ could not be considered a monophyletic group, despite their close relationship. Overall, these would provide valuable insights into the chloroplast genomes of two foliage varieties of, which would aid in the development of more horticultural resources for this genus in the future.
; Horticultural variety; Chloroplast genome;Phylogenetic analysis
10.11926/jtsb.4769
2023-01-04
2023-02-10
杭州西湖风景名胜区(市园文局、市运保委)科技发展计划项目(2018-006)资助
This work was supported by the Project for Science and Technology Development of the West Lake of Hangzhou (Grant No. 2018-006).
张巧玲(1982年生),女,硕士研究生,从事植物应用及湿地生物多样性保护研究。E-mail: 3905397@qq.com
通讯作者 Corresponding author.E-mail: yanhaifei@scbg.ac.cn

