细菌非编码小RNA的生物学特征及作用机制研究进展
王海燕 贺婷停 李昊阳


摘要:细菌非编码小RNA(sRNA)是一类长度为50~500 nt、一般不编码蛋白质的具有调控功能的小RNA,主要通过与靶mRNA碱基配对或与靶蛋白质结合,在转录后水平发挥调控作用.sRNA参与细菌氨基酸代谢和转运、环境胁迫响应以及毒力作用等众多重要生理过程的调控,与传统的转录因子等构成多层级的调控网络,协同作用使细菌对环境变化做出快速且精细的响应.由于sRNA在细菌调控网络中的重要作用,发现新的sRNA并阐明它们的调控功能成为原核生物基因表达调控研究的一个热点.本综述对细菌sRNA的筛选、鉴定、作用机制以及生物学功能等方面取得的进展进行总结,希望给相关领域的研究者提供一个较为全面的视角.
关键词:细菌; 非编码sRNA; 转录后调控
中图分类号:Q939.1 文献标志码: A 文章编号:1001-8395(2023)05-0581-10
细菌常常暴露在恶劣的自然环境中,为了应对外界环境的变化,细菌已进化形成一套精密的应答机制来适应各种环境变化.长期以来,普遍认为细菌基因的表达调控主要存在于转录水平,现在发现细菌中同样存在由非编码RNA介导的转录后调控.细菌非编码RNA是一大类具有调控功能的RNA分子,通过各种机制调控多种生理过程,这些RNA包括核开关(riboswitch)、CRISPR RNA、非编码小RNA(sRNA)等[1],其中sRNA是研究最多的一类转录后调控因子.细菌sRNA通常不进行翻译(但是也有例外[2-3]),主要通过与被调控的靶mRNA进行碱基配对来调控靶标的翻译或稳定性[4],少数sRNA通过与蛋白质结合发挥功能[5].和蛋白類调控因子相比,sRNA分子小,合成及周转所需能量和时间少,sRNA结构可以在不同构象之间快速切换,确保了对环境压力信号的迅速反应.因此,sRNA介导的转录后基因表达调控在细菌中扮演重要角色[6-7].
近年来,高通量测序特别是RNA-seq已经成为发现新sRNA的主流方法,研究者相继从大肠杆菌、沙门氏菌、结核杆菌、霍乱弧菌、枯草芽孢杆菌、地衣芽孢杆菌、金黄色葡萄球菌、李斯特菌等多种细菌中预测到大量sRNA,每种细菌的sRNA数目约在几十到200 个之间[8].然而,对sRNA的实验鉴定及功能研究还比较有限,已有的研究主要集中于大肠杆菌和沙门氏菌等几种革兰氏阴性细菌,对于革兰氏阳性菌,即使在模式菌枯草芽孢杆菌中经过功能鉴定的sRNA也只有十来个.细菌sRNA作为基因表达的转录后调控因子,参与细胞生理的多个层面,包括碳源、氨基酸和金属离子的利用,细胞分裂,DNA水平转移,群体感应,生物膜形成和毒力基因表达等,对细菌应对环境变化产生快速响应至关重要[9-13].本文对细菌sRNA的发现方法、功能机制以及研究策略进行简要总结,以期能对细菌sRNA有一个更加全面和清晰的认识.
1细菌sRNA的分类
细菌sRNA长度多在50~500 nt,绝大部分不编码蛋白质,转录后可形成稳定的二级结构,通过各种作用机制调控细菌的多种生理过程,影响遍及细菌的整个生命周期.目前已鉴定的细菌sRNA,根据其功能及作用方式可分为3大类:具有看家功能的sRNA,与靶蛋白结合的sRNA,与靶mRNA碱基配对结合的sRNA.已发现的细菌sRNA绝大多数属于第三种类型,是sRNA发挥调控功能最普遍的一种方式,即调节mRNA的翻译和稳定性.这类调控RNA根据其编码基因的染色体位置进一步被分为两类:顺式编码(cis-encoded)sRNA和反式编码(trans-encoded)sRNA.顺式编码sRNA(又叫antisense sRNA)由其调控靶基因的反义链转录产生,通过与靶mRNA完全互补配对影响其稳定性.反式编码sRNA通常由染色体的基因间隔区独立转录产生,还有一部分由mRNA的5或3UTR加工产生,这类sRNA通过不完全碱基配对方式结合靶mRNA调控其稳定性或翻译.虽然细菌中sRNA的数量并不多,但同一个sRNA可以调控多个靶基因,同一个靶基因也可以被多个sRNA所调控,使sRNA能够参与对调控网络不同节点、不同严谨性的协同控制.
2细菌sRNA的发现
由于sRNA是一类序列较短、通常不编码蛋白的转录后调控因子,不受无义或移码突变的影响,且很多只在特定环境下(如生长状态、温度、pH、离子浓度等)诱导表达,这给细菌sRNA的发现带来一定的困难.
早期发现的sRNA主要是通过实验分离得到,包括放射性标记法、鸟枪克隆法(RNA组学)以及免疫共沉淀法[14-17].这些方法主要适合高丰度、组成型sRNA的筛选,例如4.5S、tmRNA、RNase P以及Spot 42等[18-20].随着高通量测序技术的发展及相应的生物信息学分析工具不断开发,利用基因组或转录组测序数据,大量sRNA被预测出来[21],成为发现细菌sRNA的主流方法.生物信息学预测软件主要是参考细菌sRNA的序列保守性、二级结构和热动力学稳定性以及转录信号等特征,包括基于比较基因组学开发的QRNA、ERPIN、MSARI、INFERNAL、ISI,基于二级结构与热稳定性开发的RNAZ和CARNAC,以及基于孤儿启动子转录信号特征开发的sRNAscanner、sRNAPredict3/SIPHT等[22].相比于实验方法,生物信息学方法通过对高通量测序数据的分析,可在短期内获得大量sRNA的相关信息,具有成本低、时间快等优点.但该方法的一个缺点是预测具有一定的不可靠性,因此,需结合生物学实验进行验证,方法包括qRT-PCR和Northern杂交等,其中Northern杂交是验证非编码sRNA存在的金标准.
3sRNA靶基因的高通量筛选及实验鉴定
细菌非编码sRNA主要通过碱基互补配对的方式对靶基因mRNA进行调控,如何有效找出靶基因是研究sRNA功能的关键因素.由于sRNA与靶mRNA是不完全碱基配对,配对核心区域一般只有7个碱基左右,因此,靶基因的预测及筛选具有相当大的挑战性.迄今为止,已发展多种方法来筛选sRNA所调控的靶基因,包括生物信息学预测、组学筛选、实验与高通量测序相结合的方法等.
生物信息学软件主要根据RNA序列保守性、sRNA-mRNA碱基配对及结合自由能、RNA二级结构等因素预测sRNA的靶基因,是最简单、快速的靶基因预测方法.迄今为止,有TargetRNA2[23]、CopraRNA/IntaRNA[24]和sTarPicker[25]等软件被开发用于预测sRNA的靶mRNA,不同软件预测方式不同,预测出的靶基因有时差异较大.此外,由于大部分预测软件都是基于大肠杆菌及沙门氏菌中已经鉴定的sRNA及靶基因进行学习的,因此,用于其他非模式菌以及革兰氏阳性菌也可能出现较多假阳性结果,给后续的实验验证造成干扰.
转录组测序(RNA-seq)是目前使用最多的靶基因筛选方法,其一般流程是:1) 在宿主菌中转入一个诱导型sRNA表达质粒,对照组转入不含sRNA的空载体;2) 在细菌生长对数期诱导sRNA表达,短暂诱导后收集菌体提取总RNA;3) 将sRNA诱导表达菌株和对照菌株的RNA进行转录组测序,比较找出诱导后上调或下调的基因,即为候选靶基因.不过,这种方法筛选到的是sRNA诱导表达后的差异基因,但无法区分这些差异基因来自于sRNA的直接调控还是间接调控.一般来说,采用较短的诱导时间(15~30 min)可以尽可能减少间接调控基因的RNA水平变化.此外,RNA-seq只能检测mRNA水平发生变化的靶基因,无法获得翻译水平受到影响的靶基因.因此,该方法仍然具有一定的局限性.
实验与高通量测序相结合筛选靶基因的方法主要分为两大类.一类是基于革兰氏阴性菌中sRNA-mRNA结合通常需要伴侣蛋白(如RNA结合蛋白Hfq和ProQ)辅助、或结合后会招募核酸酶(如RNase E)参与mRNA降解等特性,通过免疫沉淀特定蛋白来捕获sRNA-mRNA-蛋白复合体,再用RNA连接酶将配对的sRNA-mRNA首尾相连,最后对连接RNA进行高通量测序.RIL-seq(RNA interaction by ligation and sequencing)[26]、CLASH(UV-crosslinking, ligation and sequencing of hybrids)等就是基于该原理设计的靶基因筛选策略,通过测序数据分析,理论上可以捕获细胞特定时期天然状态下所有sRNA的体内靶标.另一类是基于革兰氏阳性菌中sRNA-mRNA结合无固定结合蛋白这一特点,主要方法如MAPS(MS2-affinity purification coupled with RNA-Seq)[27].MAPS的原理是在sRNA上融合一段来自于噬菌体的RNA序列MS2,然后利用可结合MS2 RNA的蛋白进行免疫共沉淀捕获sRNA及其结合的靶mRNA或靶蛋白,最后通过RNA-seq或蛋白质谱分析获得靶基因.
这些通过高通量测序筛选获得的靶基因,还需要进行体外、体内相互作用的实验验证,以确认其可靠性.体外相互作用的检测方法主要包括凝胶阻滞实验EMSA、足迹法footprinting[28]以及趾纹法toeprinting[29]等,用于检测RNA-RNA以及RNA-蛋白质的相互作用.体内相互作用实验主要是利用报告基因(GFP或lacZ)来检测sRNA-mRNA的相互作用,是验证sRNA与靶mRNA相互作用最常用的方法.首先利用copraRNA/IntaRNA软件预测二者的相互作用位点,由于大部分sRNA与靶mRNA的结合位于靶mRNA的5UTR或起始密码子附近的区域,因此,可以构建靶基因5UTR及起始密码子附近区域与报告基因编码区融合的表达质粒,sRNA诱导表达单元可以与报告基因构建在同一个载体上,也可以构建在不同的载体上.将含有sRNA以及融合报告基因的质粒转入同一个细胞,通过sRNA诱导前后报告基因表达水平的变化来判断sRNA是否调控该mRNA.体内相互作用实验不仅可以验证sRNA对靶基因的调控,还可以通过碱基突变方式寻找sRNA-mRNA的结合位点,因此成为檢验sRNA-mRNA相互作用最可靠的方法.
4细菌sRNA的作用机制及生物学功能
细菌sRNA的表达通常受不同生长时期或各种胁迫因素的诱导,sRNA水平在某种特定条件下达到最高.当sRNA被诱导产生后,可通过不同方式调控靶基因的表达.通常情况下,少数sRNA直接与蛋白质结合发挥调控功能,大多数sRNA通过碱基配对与靶基因mRNA结合对其进行调控,最新的研究显示,sRNA还可以通过结合其他sRNA发挥调控功能.
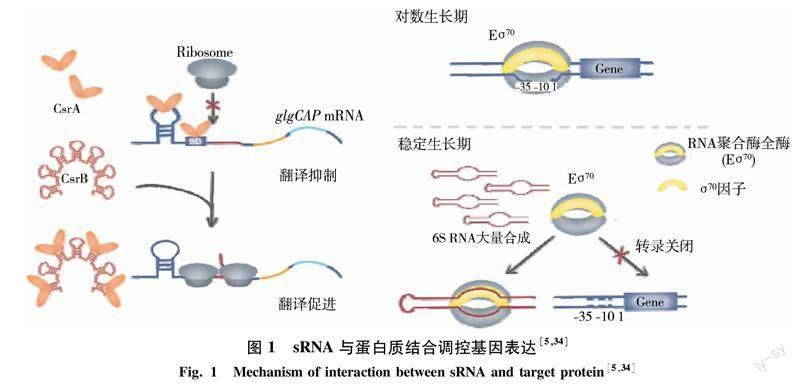
4.1sRNA与蛋白质结合 这类sRNA在体内通过模拟靶蛋白的天然底物结构,与底物竞争性结合靶蛋白从而影响靶蛋白的功能.例如大肠杆菌全局性调控蛋白CsrA是一种RNA结合蛋白,可结合在mRNA分子上进而影响其稳定性或翻译,CsrA的结合严格依赖于RNA上的GGA基序.非编码sRNA CsrB上含有多个GGA基序可同时结合超过18个CsrA,从而与mRNA竞争性结合CsrA,释放CsrA对靶mRNA的调控.例如大肠杆菌glgCAP操纵子参与糖原合成,CsrA与glgC的SD序列结合,抑制glgC mRNA的翻译并加速glgCAP mRNA降解,导致糖原合成减少.当CsrB大量表达时,CsrB螯合CsrA释放glgCAP mRNA的核糖体结合位点,进而促进mRNA翻译,使糖原合成增加[5,30](图1).与CsrB和CsrA类似的sRNA和RNA结合蛋白广泛存在于多种细菌的基因组中,例如荧光假单胞菌有3个CsrB类似sRNA(RsmX/Y/Z)和两个CsrA类似蛋白(RsmA/E),表明这种sRNA与蛋白结合的调控机制在细菌中是常见的[31].此外,6S RNA也是一个研究比较清楚的与蛋白结合的sRNA,其二级结构与基因转录起始区的DNA构象类似,可以竞争结合σ70 RNA聚合酶(Eσ70),从而影响相关基因转录[32-34].大肠杆菌的Eσ70主要负责对数生长期相关基因的转录,当细菌生长进入稳定期,6S RNA大量产生并螯合Eσ70,使依赖于Eσ70的对数期基因转录关闭,而依赖于其他σ因子的稳定期或胁迫基因转录得以开启(图1).6S RNA在原核生物中高度保守,枯草芽孢杆菌有两个6S RNA同源RNA,分别为6S-1和6S-2[35-36],二者均可以与RNA聚合酶结合抑制下游基因的转录.
4.2sRNA与mRNA结合 顺式编码sRNA通常由蛋白基因的反义链转录产生,大多是通过高通量测序被发现的,研究最多的是细菌的毒素-抗毒素系统.细菌毒素蛋白基因反义链编码的抗毒素实质是一个非编码sRNA,抗毒素sRNA与毒素mRNA完全配对,影响mRNA的稳定性或翻译,从而中和毒素[37].例如大肠杆菌I型毒素-抗毒素系统TisAB/IstR-1,在正常生长条件下,抗毒素sRNA IstR-1(70 nt)表达,通过其5端的25个碱基与毒蛋白tisAB mRNA的5UTR互补配对,抑制翻译并招募RNase III加速其降解,使毒蛋白低表达细菌得以正常生长.在SOS应激条件下,IstR-1表达水平下降,tisAB mRNA持续翻译促进毒蛋白积累,导致细菌生长减缓[38-39].枯草芽孢杆菌染色体上的原噬菌体区域编码的I型毒素-抗毒素系统BsrG/SR4也具有类似的调控机制[40].除了毒素-抗毒素系统外,地衣芽孢杆菌的枯草菌素家族蛋白酶基因apr的反义链可以转录产生sRNA AprA,它与蛋白酶基因mRNA相互作用抑制其翻译,从而降低蛋白酶水平[41].
反式编码sRNA在细菌中数量最多,通过部分碱基配对调控靶mRNA的表达,其作用机制主要包括以下4种方式(图2):1)sRNA与mRNA结合后会招募核糖核酸酶对sRNA-mRNA复合体进行降解,促进靶mRNA的降解.例如在α变形杆菌中,细胞生长到稳定期以及处于胁迫条件时,sRNA StsR高表达,然后与细胞分裂/细胞壁基因簇dcw mRNA的5UTR结合,招募内切核酸酶RNase E切割dcw mRNA,进而加速外切核酸酶对mRNA的降解,最终导致细胞分裂速度减慢[42].2)sRNA结合封闭靶mRNA的核酸酶切割位点,增加mRNA的稳定性.如在肠沙门氏菌中,细菌通过改变细胞膜性质来抵抗外界环境的变化,基因cfa1(编码环丙烷脂肪酸合酶)参与细胞膜重塑过程.在正常生长过程中,sRNA RydC可与Hfq共同作用结合在cfa1 mRNA的RNase E识别位点,保护cfa1 mRNA免遭RNase E切割,从而维持细胞在生长过程中细胞膜的特性[43].3)sRNA结合于靶mRNA的核糖体结合位点及附近,阻止核糖体与mRNA结合,从而抑制靶mRNA的翻译.该方式是sRNA最主要的作用方式,也是生物信息学预测sRNA靶基因的一个重要参数.如大肠杆菌的sRNA Spot 42,长度为109 nt,特异性结合在操纵子galETKM mRNA的galK(编码半乳糖激酶)的翻译起始区域,阻断核糖体结合抑制galK翻译,使半乳糖激酶与UDP-半乳糖差向异构酶(由galE编码)的比率发生改变,从而适应不同的生存环境[44].4)sRNA与mRNA的5UTR结合,改变5UTR区的构象使核糖体结合位点得以暴露,促进靶基因的翻译.如在铁离子不足的条件下,大肠杆菌sRNA RyhB高表达,与大肠杆菌素(colicin)受体基因cirA mRNA的5UTR结合暴露核糖体结合位点,促进cirA mRNA的翻译,使细菌对大肠杆菌素更加敏感[45].
4.3双功能sRNA 传统意义上,sRNA为不编码蛋白的非编码RNA,但随着研究的深入,发现个别sRNA也可以作为mRNA编码小肽行使功能.迄今为止,共发现5个双功能sRNA,即大肠杆菌的SgrS[46]、金黄色葡萄球菌的RNA III[47]和Psm-mec[48]、脓性链球菌的Pel RNA[49]以及枯草芽孢杆菌的SR1[50],其中SgrS的特性及功能在大肠杆菌和沙门氏菌中研究得较清楚.细菌利用细胞膜上的PTS系统将葡萄糖运输到胞内,同时磷酸化生成6-磷酸葡萄糖(G6P),当糖的转运和代谢不能协调匹配,就可能导致胞内G6P积累,产生细胞毒性.在G6P胁迫下,转录因子SgrR大量产生并激活sRNA SgrS的转录,SgrS可与PTS系统的葡萄糖渗透酶基因ptsG mRNA的SD序列结合阻止核糖体结合,并招募RNase E加速mRNA降解,从而抑制新的PtsG蛋白合成.此外,sRNA SgrS还编码一个长度仅为43个氨基酸的小肽SgrT,SgrT可与细胞膜上已经存在的PtsG蛋白结合抑制其转运活性[2](图3).SgrS的这两个功能似乎是互斥的,即一个SgrS分子只能以一种方式发挥作用,表现出对G6P胁迫响应的时间顺序:即SgrS首先以sRNA形式与靶mRNA配对快速响应胁迫,而SgrT蛋白要在胁迫后30 min才产生,进一步发挥抑制PtsG蛋白活性的功能.
4.4作为sRNA“海绵”间接调控靶基因的表达 sRNA除了结合靶蛋白和靶mRNA,还可以作为RNA“海绵”,通过结合具有调控功能的sRNA,间接调控靶mRNA的表达.例如沙门氏菌sRNA SroC,由Glu/Asp转运蛋白操纵子gltIJKL的gltI mRNA的3端加工产生,可以竞争结合具有全局性调控功能的sRNA GcvB,形成sRNA-sRNA配对,进而招募RNase E对GcvB进行降解,以缓解GcvB对氨基酸转运及代谢相关基因(也包括gltIJKL mRNA)的抑制作用[51](图4).此外,SroC还可以结合sRNA MgrR,释放其对eptB mRNA的抑制作用,影响细菌对多黏菌素B的敏感性[52].枯草芽孢杆菌中一个独立转录产生的sRNA RosA也可作为RNA海绵,结合至少两种sRNA,即参与氧化胁迫调节的RoxS以及参与铁胁迫调节的FsrA.RosA的转录受到碳代谢关键调控因子CcpA的抑制,在以阿拉伯糖为碳源时,CcpA表达受到抑制,使RosA的转录增加,RosA竞争性结合RoxS,从而缓解RoxS对TCA循环以及细胞呼吸链相关基因的抑制作用,由此将RoxS对氧化胁迫的调控作用与碳代谢又联系起来[53].
4.5sRNA參与多个代谢通路的协同控制 反式编码sRNA通过短的不完全碱基配对与靶mRNA结合,因此,同一个sRNA可以调节相关通路的多个基因,协同控制相应的生理过程.sRNA参与细菌铁稳态调控是研究得比较深入的案例.铁元素是绝大多数生命必需的营养物质,通常以蛋白辅因子形式存在,大肠杆菌一般拥有上百种含铁蛋白,包括代谢酶、电子传递蛋白、感应与调控蛋白等,参与一系列重要的生物过程.但铁过量又会给生命带来严重威胁,因此,细菌必须随时维持细胞内铁元素水平的相对稳定,即铁稳态.转录调控因子Fur是最关键的铁稳态调控因子,此外,还有多个sRNA参与细菌铁稳态的调控,最著名的是sRNA RyhB.研究发现,当胞内游离Fe2+浓度较低时,RyhB参与调控100多个基因的表达以维持胞内铁离子稳态:1) 抑制非必需含Fe2+蛋白的表达,包括约50个Fe-S蛋白基因以及21个Fe2+结合蛋白基因,以使细胞内有限的铁用于必需的生命过程;2) 抑制Fe-S簇合成的“管家”Isc系统,促进胁迫响应的Suf系统,节约铁离子以满足最需要的Fe-S蛋白合成;3) 促进铁摄取相关蛋白CirA以及ShiA的表达,增加细胞外铁离子的输入[54].
另一方面,同一个靶mRNA又可被不同的sRNA所调节,使细胞内不同的靶mRNA通过sRNA连接成复杂的网络以协调细菌的生理和行为.在大肠杆菌和其他肠道细菌中,生物膜形成涉及对鞭毛合成和生物膜基质合成两个过程的反向控制,不同sigma因子、一系列级联调控转录因子以及大量sRNA协同作用共同调控这一过程.其中,开启鞭毛基因表达最上层的转录激活因子FlhDC,负责生长稳定期基因转录的sigma因子σS,以及生物膜基质重要组分菌毛合成的关键调节因子CsgD,这3个基因的mRNA被证明是多个sRNA的作用中心.研究显示,flhDC mRNA可被3种sRNA ArcZ、OmrA/OmrB以及OxyS结合而抑制表达,被McaS结合则促进翻译.σSmRNA可被sRNA DsrA、RprA 和ArcZ正调控,促进菌毛及生物膜形成.csgD mRNA 可被sRNA McaS、RprA、GcvB、RydC、RybB和OmrA/OmrB负调控,抑制菌毛及生物膜形成[55](图5).综合来看,这些sRNA根据其对鞭毛及生物膜合成的调控效应可分為4类:1) 促进生物膜形成、下调鞭毛合成(如ArcZ)[56-57];2) 作用正好相反,即刺激鞭毛合成、干扰生物膜基质合成(如McaS)[58];3) 下调这两种功能(如OmrA/OmrB)[59];4) 在鞭毛和生物膜功能的控制中具有多种更复杂的影响(如RprA)[60].不同sRNA共同作用的最终效应取决于sRNA和mRNA的表达时期、速率与水平、sRNA与mRNA结合力等多种因素[55].
5小结与展望
非编码sRNA在细菌中广泛存在,其调控作用遍及细菌的整个生命过程.在细菌基因表达调控系统中,sRNA与传统的转录因子等构成多层级的调控网络,协同作用使细菌对环境胁迫做出快速且精细的响应.目前,细菌sRNA的研究主要集中在大肠杆菌和沙门氏菌等革兰氏阴性细菌,对革兰氏阳性菌及非模式菌的sRNA研究还比较缺乏.对在实际应用中具有重要价值的非模式菌(如致病菌、工业生产菌等)开展研究,探索sRNA如何调控细菌的致病性、耐药性及胁迫应答,不仅可以揭示更多参与不同生物学功能调控的sRNA,扩展sRNA的多样性,也将为其实际应用提供理论基础.
参考文献
[1] WATERS L S, STORZ G. Regulatory RNAs in bacteria[J]. Cell,2009,136(4):615-628.
[2] RAINA M, KING A, BIANCO C, et al. Dual-function RNAs[J]. Microbiol Spectr,2018,6(5):471-485.
[3] VANDERPOOL C K, BALASUBRAMANIAN D, LLOYD C R. Dual-function RNA regulators in bacteria[J]. Biochimie,2011,93(11):1943-1949.
[4] H?R J, MATERA G, VOGEL J, et al. Trans-acting small RNAs and their effects on gene expression in Escherichia coli and Salmonella enterica[J/OL]. EcoSal Plus,2020,9(1):10.1128/ecosalplus.ESP-0030-2019.
[5] BAKER C S, MOROZOV I, SUZUKI K, et al. CsrA regulates glycogen biosynthesis by preventing translation of glgC in Escherichia coli[J]. Mol Microbiol,2002,44(6):1599-1610.
[6] MARS R A, NICOLAS P, DENHAM E L, et al. Regulatory RNAs in Bacillus subtilis:a gram-positive perspective on bacterial RNA-mediated regulation of gene expression[J]. Microbiol Mol Biol Rev,2016,80(4):1029-1057.
[7] GOTTESMAN S, STORZ G. Bacterial small RNA regulators:versatile roles and rapidly evolving variations[J]. Cold Spring Harb Perspect Biol,2011,3(12):a003798.
[8] H?R J, GORSKI S A, VOGEL J. Bacterial RNA biology on a genome scale[J]. Mol Cell,2018,70(5):785-799.
[9] MORFELDT E, TAYLOR D, GABAIN A V, et al. Activation of alpha-toxin translation in Staphylococcus aureus by the trans-encoded antisense RNA, RNAIII[J]. Embo J,1995,14(18):4569-4577.
[10] ROMBY P, VANDENESCH F, WAGNER E G. The role of RNAs in the regulation of virulence-gene expression[J]. Curr Opin Microbiol,2006,9(2):229-236.
[11] PAPENFORT K, VOGEL J. Regulatory RNA in bacterial pathogens[J]. Cell Host Microbe,2010,8(1):116-127.
[12] DURAND S, GILET L, CONDON C. The essential function of B. subtilis RNase III is to silence foreign toxin genes[J]. PLoS Genet,2012,8(12):e1003181.
[13] HOE C H, RAABE C A, ROZHDESTVENSKY T S, et al. Bacterial sRNAs:regulation in stress[J]. Int J Med Microbiol,2013,303(5):217-229.
[14] H?TTENHOFER A, VOGEL J. Experimental approaches to identify non-coding RNAs[J]. Nucleic Acids Res,2006,34(2):635-646.
[15] LIVNY J, WALDOR M K. Identification of small RNAs in diverse bacterial species[J]. Curr Opin Microbiol,2007,10(2):96-101.
[16] VOGEL J, GERHART E, WAGNER H. Approaches to Identify Novel Non-messenger RNAs in Bacteria and to Investigate Their Biological Functions:RNA Mining[M]//Handbook of RNA Biochemistry. Weinheim:Wiley-VCH Verlag GmbH,2008:595-613.
[17] BILUSIC I, POPITSCH N, RESCHENEDER P, et al. Revisiting the coding potential of the E. coli genome through Hfq co-immunoprecipitation[J]. RNA Biol,2014,11(5):641-654.
[18] GRIFFIN B E. Separation of 32P-labelled ribonucleic acid components. The use of polyethylenimine-cellulose (TLC) as a second dimension in separating oligoribonucleotides of ‘4.5 S and 5 S from E. coli[J]. FEBS Letters,1971,15(3):165-168.
[19] HINDLEY J. Fractionation of 32P-labelled ribonucleic acids on polyacrylamide gels and their characterization by fingerprinting[J]. Journal of Molecular Biology,1967,30(1):125-136.
[20] IKEMURA T, DAHLBERG J E. Small ribonucleic acids of Escherichia coli:I. Characterization by polyacrylamide gel electrophoresis and fingerprint analysis[J]. Journal of Biological Chemistry,1973,248(14):5024-5032.
[21] WASSARMAN K M, REPOILA F, ROSENOW C, et al. Identification of novel small RNAs using comparative genomics and microarrays[J]. Genes Dev,2001,15(13):1637-1651.
[22] SRIDHAR J, GUNASEKARAN P. Computational small RNA prediction in bacteria[J]. Bioinform Biol Insights,2013,7(7):83-95.
[23] KERY M B, FELDMAN M, LIVNY J, et al. TargetRNA2:identifying targets of small regulatory RNAs in bacteria[J]. Nucleic Acids Res,2014,42(1):W124-129.
[24] WRIGHT P R, GEORG J, MANN M, et al. CopraRNA and IntaRNA:predicting small RNA targets, networks and interaction domains[J]. Nucleic Acids Res,2014,42(1):W119-123.
[25] YING X M, CAO Y, WU J Y, et al. sTarPicker: a method for efficient prediction of bacterial sRNA targets based on a two-step model for hybridization[J]. Plos One,2011,6(7):e22705.
[26] MELAMED S, FAIGENBAUM-ROMM R, PEER A, et al. Mapping the small RNA interactome in bacteria using RIL-seq[J]. Nat Protoc,2018,13(1):1-33.
[27] MERCIER N, PR?VOST K, MASS?E, et al. MS2-affinity purification coupled with RNA sequencing in gram-positive bacteria[J]. J Vis Exp,2021,168(6):1-13.
[28] PENG Y, SOPER T J, WOODSON S A. RNase footprinting of protein binding sites on an mRNA target of small RNAs[J]. Methods Mol Biol,2012,905:213-224.
[29] NILSEN T W. Toeprinting [J]. Cold Spring Harb Protoc,2013,2013(9):896-899.
[30] LIU M Y, GUI G J, WEI B D, et al. The RNA molecule CsrB binds to the global regulatory protein CsrA and antagonizes its activity in Escherichia coli[J]. J Biol Chem,1997,272(28):17502-17510.
[31] SONNLEITNER E, HAAS D. Small RNAs as regulators of primary and secondary metabolism in Pseudomonas species[J]. Appl Microbiol Biotechnol,2011,91(1):63-79.
[32] WASSARMAN K M. 6S RNA:a regulator of transcription[J]. Mol Microbiol,2007,65(6):1425-1431.
[33] WILLKOMM D K, HARTMANN R K. 6S RNA-an ancient regulator of bacterial RNA polymerase rediscovered[J]. Biol Chem,2005,386(12):1273-1277.
[34] WASSARMAN K M. 6S RNA, a global regulator of transcription[J/OL]. Microbiol Spectr,2018,6(3):10.1128/microbiolspec.RWR-0019-2018.
[35] TROTOCHAUD A E, WASSARMAN K M. A highly conserved 6S RNA structure is required for regulation of transcription[J]. Nat Struct Mol Biol,2005,12(4):313-319.
[36] BARRICK J E, SUDARSAN N, WEINBERG Z, et al. 6S RNA is a widespread regulator of eubacterial RNA polymerase that resembles an open promoter[J]. RNA,2005,11(5):774-784.
[37] SMIRNOV A, F?RSTNER K U, HOLMQVIST E, et al. Grad-seq guides the discovery of ProQ as a major small RNA-binding protein[J]. Proc Natl Acad Sci USA,2016,113(41):11591-11596.
[38] DARFEUILLE F, UNOSON C, VOGEL J, et al. An antisense RNA inhibits translation by competing with standby ribosomes[J]. Mol Cell,2007,26(3):381-392.
[39] VOGEL J, ARGAMAN L, WAGNER E G, et al. The small RNA IstR inhibits synthesis of an SOS-induced toxic peptide[J]. Curr Biol,2004,14(24):2271-2276.
[40] JAHN N, PREIS H, WIEDEMANN C, et al. BsrG/SR4 from Bacillus subtilis-the first temperature-dependent type I toxin-antitoxin system[J]. Mol Microbiol,2012,83(3):579-598.
[41] HERTEL R, MEYERJ?RGENS S, VOIGT B, et al. Small RNA mediated repression of subtilisin production in Bacillus licheniformis[J]. Sci Rep,2017,7(1):5699.
[42] GR?TZNER J, REMES B, EISENHARDT K M H, et al. sRNA-mediated RNA processing regulates bacterial cell division[J]. Nucleic Acids Res,2021,49(12):7035-7052.
[43] FR?HLICH K S, PAPENFORT K, FEKETE A, et al. A small RNA activates CFA synthase by isoform-specific mRNA stabilization[J]. Embo J,2013,32(22):2963-2979.
[44] M?LLER T, FRANCH T, UDESEN C, et al. Spot 42 RNA mediates discoordinate expression of the E. coli galactose operon[J]. Genes Dev,2002,16(13):1696-1706.
[45] SALVAIL H, CARON M P, B?LANGER J, et al. Antagonistic functions between the RNA chaperone Hfq and an sRNA regulate sensitivity to the antibiotic colicin[J]. EMBO J,2013,32(20):2764-2778.
[46] WADLER C S, VANDERPOOL C K. A dual function for a bacterial small RNA:SgrS performs base pairing-dependent regulation and encodes a functional polypeptide[J]. Proc Natl Acad Sci USA,2007,104(51):20454-20459.
[47] BRONESKY D, WU Z, MARZI S, et al. Staphylococcus aureus RNAIII and its regulon link quorum sensing, stress responses, metabolic adaptation, and regulation of virulence gene expression[J]. Annu Rev Microbiol,2016,70(1):299-316.
[48] KAITO C, SAITO Y, IKUO M, et al. Mobile genetic element SCCmec-encoded psm-mec RNA suppresses translation of agrA and attenuates MRSA virulence[J]. PLoS Pathog,2013,9(4):e1003269.
[49] MANGOLD M, SILLER M, ROPPENSER B, et al. Synthesis of group A streptococcal virulence factors is controlled by a regulatory RNA molecule[J]. Mol Microbiol,2004,53(5):1515-1527.
[50] GIMPEL M, BRANTL S. Dual-function sRNA encoded peptide SR1P modulates moonlighting activity of B. subtilis GapA[J]. RNA Biol,2016,13(9):916-926.
[51] MIYAKOSHI M, CHAO Y, VOGEL J. Cross talk between ABC transporter mRNAs via a target mRNA-derived sponge of the GcvB small RNA[J]. EMBO J,2015,34(11):1478-1492.
[52] ACU?A L G, BARROS M J, PE?ALOZA D, et al. A feed-forward loop between SroC and MgrR small RNAs modulates the expression of eptB and the susceptibility to polymyxin B in Salmonella Typhimurium[J]. Microbiology,2016,162(11):1996-2004.
[53] DURAND S, CALLAN-SIDAT A, MCKEOWN J, et al. Identification of an RNA sponge that controls the RoxS riboregulator of central metabolism in Bacillus subtilis[J]. Nucleic Acids Res,2021,49(11):6399-6419.
[54] CHAREYRE S, MANDIN P. Bacterial iron homeostasis regulation by sRNAs[J]. Microbiol Spectr,2018,6(2):1-11
[55] MIKA F, HENGGE R. Small regulatory RNAs in the control of motility and biofilm formation in E. coli and Salmonella[J]. Int J Mol Sci,2013,14(3):4560-4579.
[56] DE LAY N, GOTTESMAN S. A complex network of small non-coding RNAs regulate motility in Escherichia coli[J]. Mol Microbiol,2012,86(3):524-538.
[57] MANDIN P, GOTTESMAN S. Integrating anaerobic/aerobic sensing and the general stress response through the ArcZ small RNA[J]. Embo J,2010,29(18):3094-3107.
[58] J?RGENSEN M G, NIELSEN J S, BOYSEN A, et al. Small regulatory RNAs control the multi-cellular adhesive lifestyle of Escherichia coli[J]. Mol Microbiol,2012,84(1):36-50.
[59] HOLMQVIST E, REIMEGRD J, STERK M, et al. Two antisense RNAs target the transcriptional regulator CsgD to inhibit curli synthesis[J]. Embo J,2010,29(11):1840-1850.
[60] MIKA F, BUSSE S, POSSLING A, et al. Targeting of csgD by the small regulatory RNA RprA links stationary phase, biofilm formation and cell envelope stress in Escherichia coli[J]. Mol Microbiol,2012,84(1):51-65.
Biological Characteristics and Regulation Mechanisms
of Bacterial Non-coding Small RNAWANG Haiyan,HE Tingting,LI Haoyang(College of Life Sciences, Sichuan University, Chengdu 610064, Sichuan)
AbstractBacterial non-coding small RNAs (sRNA) exist as relatively short transcripts (50-500 nt) that are transcribed but do not encode proteins. sRNA plays a post-transcriptional regulatory role on target genes mostly by imperfect base-pairing with target mRNA, and rarely by binding target protein. sRNA is involved in the regulation of many important physiological processes, such as amino acid metabolism and transport, stress response and virulence, etc., forming a multi-level regulatory network with traditional transcription factors to enable bacteria responding quickly to environmental stress. Due to the important role of sRNA in bacterial regulatory network, the discovery of new sRNAs and revealing their regulatory mechanisms have become a hotspot in prokaryotic gene expression regulation research. This review summarizes the advance in screening, identification, regulatory mechanism and biological function of bacterial sRNA, hoping to provide a more comprehensive perspective for researchers of related fields.
Keywords:bacteria; noncoding sRNA; post-transcriptional regulation
(編辑 郑月蓉)

