上海地区猪萨佩罗病毒的检测及遗传进化分析
李本强,刘惠莉,刘佳佳,陶 洁,程靖华,石 迎,乔长涛,沈晓晖*
(1 上海市农业科学院畜牧兽医研究所∕上海市农业遗传育种重点实验室,上海 201106;2 上海市种猪工程技术中心,上海 201106;3 上海海洋大学,上海 201306)
猪萨佩罗病毒(Porcine sapelovirus,PSV)属小RNA 病毒科,是微球形、无囊膜、单股正链RNA 病毒的一种,为肠道病毒属II 群、猪肠道病毒8 型[1]。 通过对肠道病毒基因组及进化关系进一步研究,TSENG等[2]将该病毒划为萨佩罗病毒属。 PSV 基因组为单股正链RNA,全长约7 400 bp,包含1 个大的开放阅读框架,编码1 个多聚蛋白前体,在5’端有一个Vpg 区,3’末端连有Poly A 结构[3-4]。
PSV 于20 世纪60 年代在英国首先发现[5],随后在北美、日本、澳大利亚等地先后出现[6-10]。 PSV在饲养猪群中感染率很高,多呈散发,不同年龄的猪均有易感性[4,11],幼龄猪易感性较强,病猪、康复猪和隐性感染猪为主要传染源。 感染PSV 后可导致猪肠道、呼吸道、生殖器官疾病以及脑脊髓炎等多系统综合征[12-13],还可能呈现亚临床感染。 但无论其以何种形式爆发,都会给养猪业带来潜在威胁。 目前对PSV 的研究主要集中于病原分离与鉴定、基因组与结构蛋白分析方面。 本研究通过监测上海地区猪场PSV 感染情况,并对其中2 个PSV 分离株进行全基因组测定与分析,旨在进一步明确PSV 在腹泻疫情中的作用。
1 材料与方法
1.1 材料
1.1.1 样品采集
于2016—2020 年从上海地区11 个猪场采集具有腹泻症状的猪粪便样品1 446 份,用PBS 液将样品按照1∶10 的比例稀释,-80 ℃冰箱冻存。
1.1.2 主要试剂及仪器
Trizol、M-MLV RNA 聚合酶、PCR 试剂及pMD-18T 载体等均购自TaKaRa 公司;5’RACE kit 试剂盒购自北京百泰克生物技术有限公司;pEASY-Blunt Zero Cloning Kit、DNA 凝胶回收试剂盒、Trans1-T1 Phage Resistant Chemically Competent Cell 购自北京全式金生物公司;Phanta Max Super-Fidelity DNA Polymerase购自诺唯赞生物公司。
PCR 仪和核酸电泳仪为美国Bio-Rad 公司生产。
1.2 方法
1.2.1 样品的处理及总RNA 的提取
将-80 ℃保存的样品漩涡震荡5 min,取250 μL 溶液,采用Trizol 法提取总RNA,具体操作方法按照TaKaRa 产品说明书进行。
1.2.2 引物的设计及合成
根据Genbank 中HQ875059、KJ821021、KJ821020 和KX574284 序列,在3D 保守区域设计1 对PSV 特异检测引物(PSV-F:5’-GATGTGGCGCATGCTCTT-3’,PSV-R:5’-TGCTGCCTCCTGTGTTGTTAT-3’),扩增片段长度约为624 bp[14]。 采用实验室已公布的引物用于扩增PSV 全基因组序列,引物序列由铂尚生物技术(上海)有限公司合成。
1.2.3 临床样品的RT-PCR 检测
以提取的样品RNA 为模板,按照Reverse Transcriptase M-MLV 的说明书进行反转录,合成cDNA。RT-PCR 反应体系为:cDNA 1 μL,10 × PCR mix 2 μL,PSV-F(0. 5 μmol∕L)和PSV-R(0. 5 μmol∕L)各0.5 μL,用水补至20 μL;反应程序为:94 ℃4 min;94 ℃30 s;52 ℃30 s;72 ℃35 s,30 个循环;72 ℃8 min;4 ℃结束反应。
1.2.4 病毒的增殖及鉴定
选取生长状态良好的PK15 细胞,胰酶消化后铺于6 孔板中。 第2 天,弃去细胞上清,用PBS 洗涤2遍,加入含0.5 μg∕mL 胰酶的DMEM 病毒培养液;吸附2 h 后,弃去上清,加入含胰酶的DMEM 培养液。置于37 ℃培养箱,观察细胞病变情况,若无病变,则5 d 后收毒;冻融2 次后,重复上述过程,进行病毒的连续传代培养。 不同代次的细胞培养物各取250 μL,进行RT-PCR 检测,验证病毒传代成功与否。
1.2.5 间接免疫荧光检测
将传至第5 代的PSV 接种PK15 细胞,吸附1 h 后,吸掉病毒液,用DMEM 洗涤2 次。 加入含有0.5 μg∕mL TPCK 的DMEM 培养基于37 ℃培养72 h 后,弃去细胞上清,PBST 洗1 遍,70%乙醇冰上固定15 min;PBST 洗3 遍并甩干,加入1∶1 000 稀释的PSV 单抗,37 ℃湿盒中孵育1 h;重复洗涤,加入1∶3 000稀释的FITC 标记羊抗鼠IgG,37 ℃孵育30 min;洗涤后,置于倒置荧光显微镜(Axio Observer)下观察。
1.2.6 PSV 全基因组序列扩增及序列分析
取第5 代PSV 的病毒液,扩增全基因组序列。 反应体系参照Phanta Max Super-Fidelity DNA Polymerase 说明书进行PCR 反应。 反应程序:94 ℃4 min;94 ℃30 s,52 ℃30 s,72 ℃60 s,35 个循环;72 ℃8 min;4 ℃结束反应。
PSV 的5’末端序列扩增采用5’RACE 方法进行[15]。 将目的片段与pEASY-Blunt Zero Cloning Kit 载体进行连接,随后用Trans1-T1 Phage Resistant Chemically Competent Cell 进行克隆,挑取阳性重组质粒进行测序。
采用Lasergene 分析软件对各测定片段进行拼接,获得PSV 全基因组序列并进行基因分析,利用MEGA 6.0软件制作进化树。
2 结果与分析
2.1 PSV 流行情况调查
对2016—2020 年采集的1 416 份粪便样品进行RTPCR 检测,结果显示,共有256 份PSV 阳性样品,总阳性率 为 17.7%。 各 年 PSV 阳 性 率 分 别 为 18.26%(65∕356)、3.03% (3∕99)、18.72% (44∕235)、21.58%(126∕584)和10.47%(18∕172)(图1),表明PSV 在猪群中普遍存在。
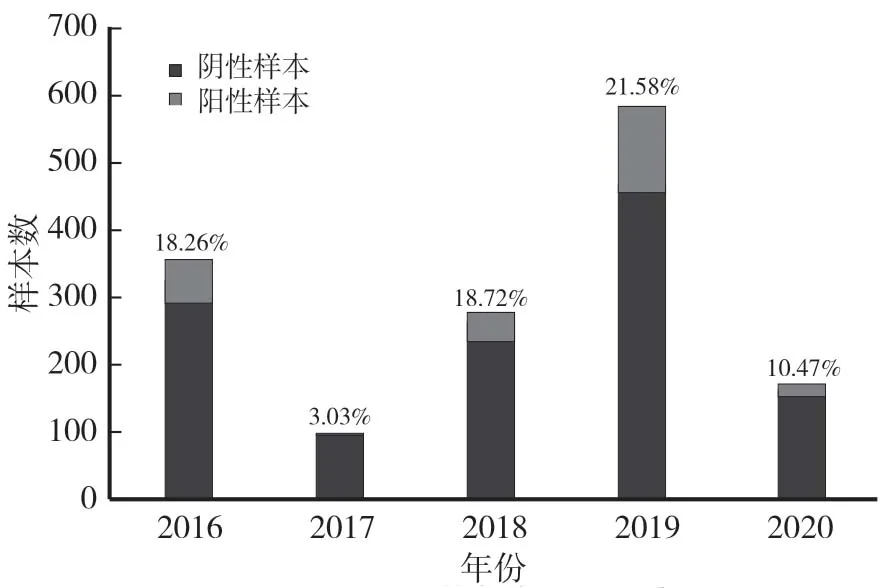
图1 猪群中PSV 的年度流行情况Fig.1 Prevalence trends of PSV infection in pig herds
2.2 PSV 病毒分离
将PCR 检测为阳性的PSV 样品接种PK15 细胞,连续传5 代后,2 株PSV 上出现细胞病变。 PCR 检测显示其第1 代次、第2 代次的扩增条带都较弱,从第3 代开始,病毒增殖比较稳定。 将成功分离的2 株PSV 毒株分别命名为CM2019008 和CM2019337(图2)。
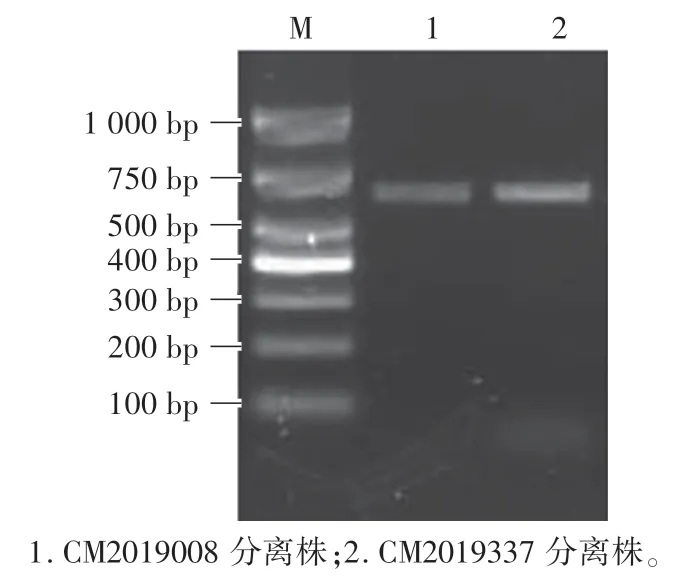
图2 分离株的PCR 鉴定Fig.2 PCR identification of the isolated strain
2.3 病毒间接免疫荧光鉴定
将第5 代的CM2019008 和CM2019337 病毒分别接种于PK15 细胞,间接免疫荧光检测显示,2 株分离病毒均能与PSV 自制单抗发生免疫反应,出现特异性绿色荧光(图3),表明这两个毒株在PK15 细胞上增殖成功。

图3 PSV 分离株的间接免疫荧光检测(200 ×)Fig.3 Indirect immunofluscent test of PSV isolates(200 ×)
2.4 PSV 基因组遗传分析
2.4.1 PSV 全基因组的遗传进化分析
利用Mega 6.0 软件对2 株PSV 和国内外其他代表性毒株进行全基因组遗传进化分析。 结果表明,分离株CM2019337 与上海PSV 株MN685785.1 进化关系最近,分离株CM2019008 与SHPD202150 在中国PSV 的一个大的分支上。 中国PSV 分离株与日本2 株PSV 分离株同处一个大的分支上,遗传进化关系最为接近(图4)。
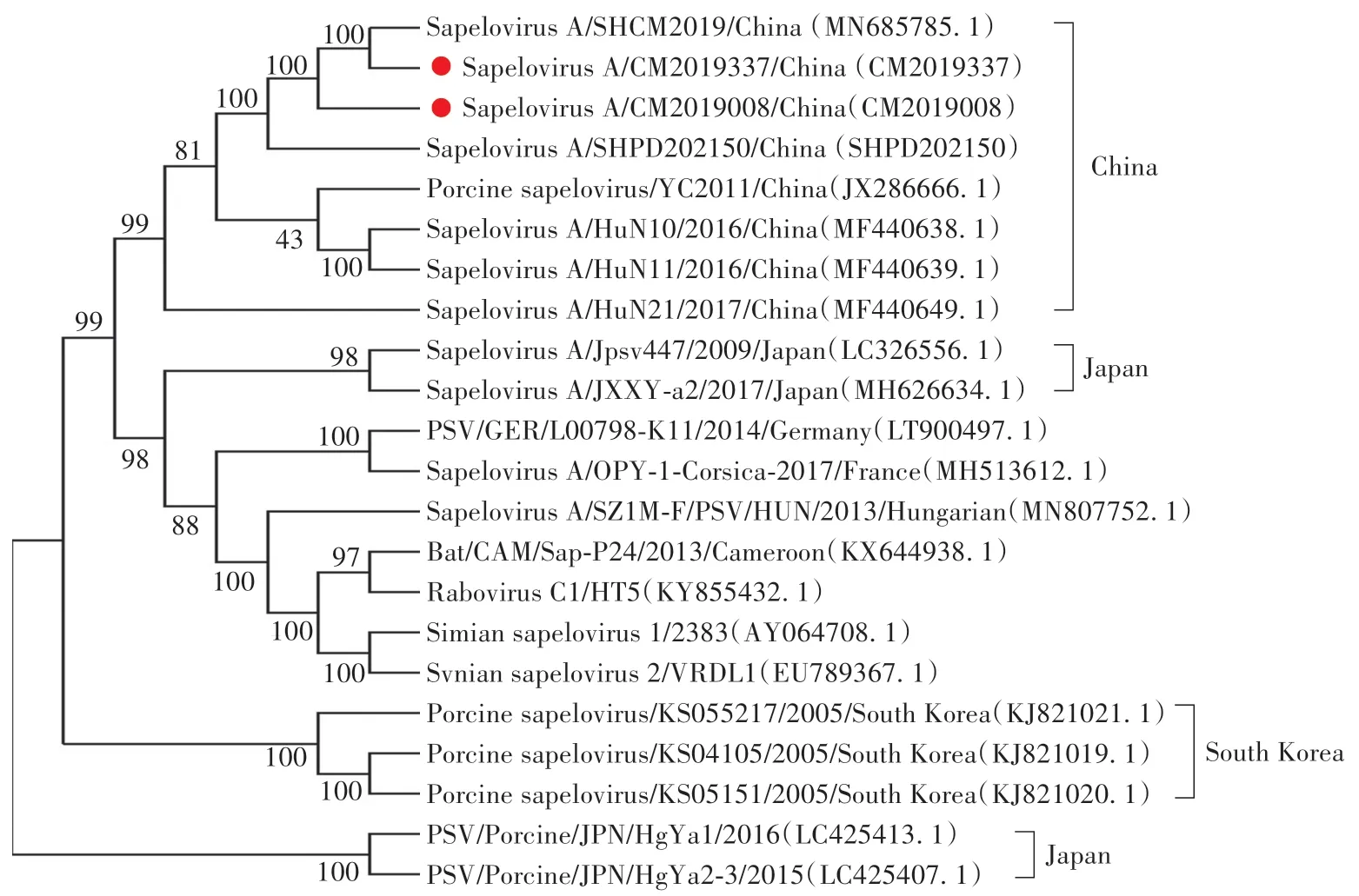
图4 PSV 全基因组序列构建的系统进化树Fig.4 The phylogenetic tree constructed based on the whole gene sequence of PSV
进一步利用DNAStar 软件对上述毒株的核苷酸相似性进行分析,发现CM2019008 和CM2019337 的核苷酸相似性为94.8%,其次是MN685785.1,两者核苷酸相似性为94.4%;CM2019337 和MN685785.1的核苷酸相似性为99.5%,其次是JX286666.1,核苷酸相似性为89.8%。 中国的PSV 毒株与韩国PSV 毒株核苷酸相似性为87%—88.5%,与欧洲PSV 毒株核苷酸相似性为84.9%—87.3%(图5)。

图5 蛋白编码区序列相似性比较Fig.5 Homology comparison of sequence in protein coding region
2.4.2 分离株蛋白编码区的重组分析
在SimPlot 3.5.1 软件中CM2019008 和CM2019337 作为独立的重组事件进行标准相似节点分析以查找有无重组事件的发生,结果所示,CM2019008 和CM2019337 分离株在VP1、VP2 和VP3 区域与HeBO4核苷酸序列高度相似,而在3D 区域与ISU-SHIC 高度相似(图6A)。 利用RDP、Chimaera、Bootscan、3Seq、Maxchi、Phylpro、GENECONV、LARD 及SISCAN 九种算法对重组毒株进行分析以寻找重组毒株的潜在断裂点。 Bootscan 分析结果所示,CM2019008 和CM2019337 分离株在3D 区域存在重组断点(6B)。 利用邻接法构建重组毒株的进化树,结果如图6C 所示,CM2019008 和CM2019337 分离株的重组区域位于4 696—5 945 bp 处。
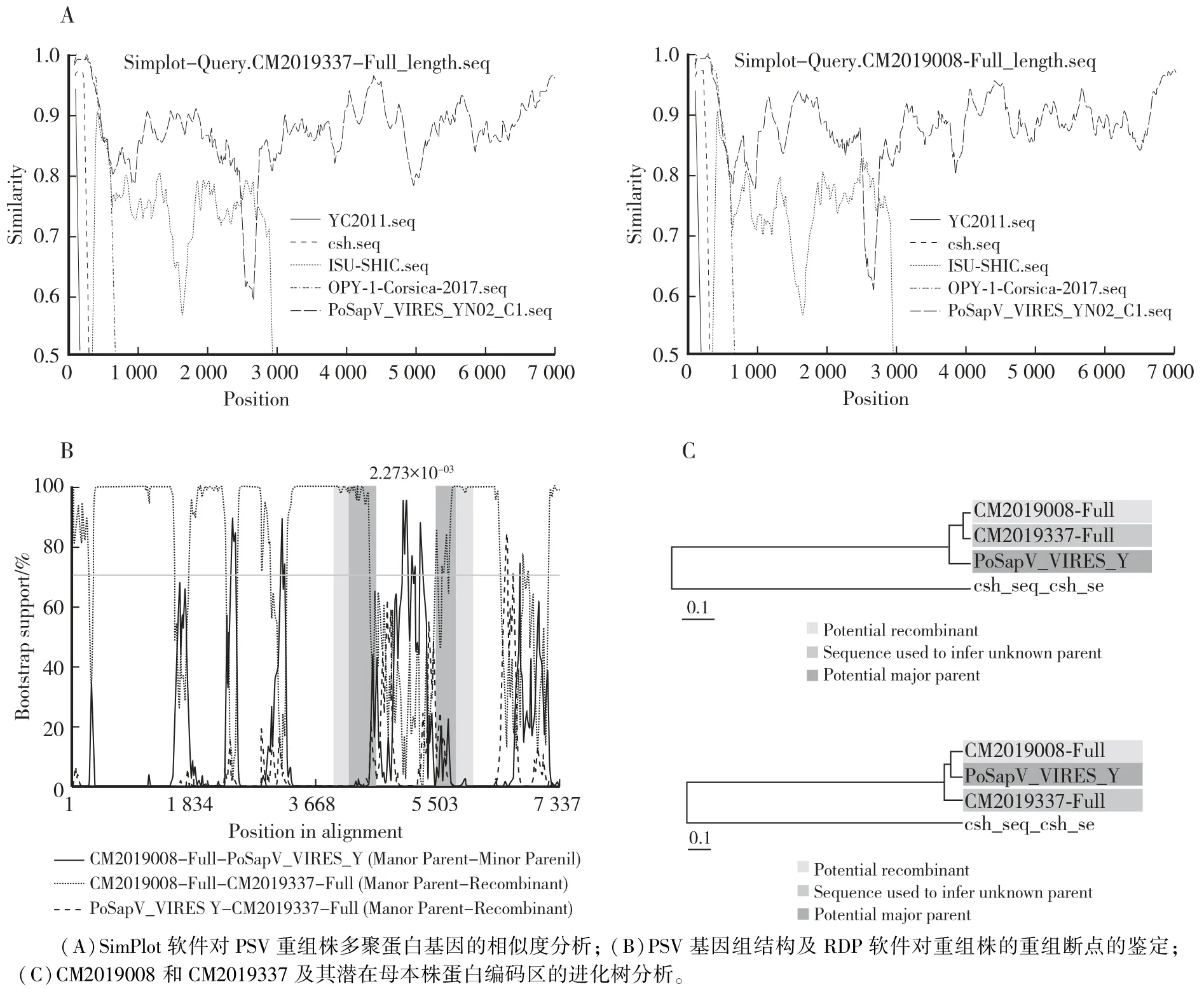
图6 潜在重组毒株多聚蛋白重组事件Fig.6 Recombinant events identified according to the polyprotein gene of potential recombinant strains
2.4.3 分离株VP1 基因的氨基酸序列分析
采用MegAlign 和GeneDoc 2.7 软件对VP1 基因对应的氨基酸序列进行分析,结果如图7 所示。 与PSV 经典株相比,CM2019008 共存在6 个氨基酸位点的突变,分别为第28、88、98、223、229、271 位,且突变位点多集中在VP1 氨基酸的两端序列;CM2019337 共存在5 个氨基酸位点的突变,分别为第28、94、95、218、236 位。 CM2019008 和CM2019337 中间氨基酸99—217 位点相对保守。

图7 PSV VP1 基因氨基酸突变位点的分析Fig.7 Analysis of amino acid mutation sites of PSV VP1 gene
2.4.4 VP1 蛋白的潜在糖基化位点和抗原表位的分析
运用在线分析软件NetNGlyc 1.0 Server 和GeneDoc 2.7 对VP1 蛋白的潜在糖基化位点进行分析,结果表明所有PSV 分离株和参考株都含有潜在NSTS 糖基化位点,且都含173 位潜在糖基化位点。 其中分离株CM2019008 和CM2019337 分别含有1 个和2 个潜在糖基化位点;日本PSV 分离株LC326556.1 含有2 个糖基化位点,德国PSV 分离株LT900497.1 和法国PSV 分离株MH513612.1 分别含有2 个和1 个糖基化位点。
采用在线分析软件BepiPred 1.0 Server 和DNAStar Protean 对分离株VP1 蛋白的抗原表位进行分析,发现中国分离株CM2019008 和CM2019337 与中国已有的分离株及国外分离株的抗原表位差异不大,抗原表位均匀分散在整个VP1 蛋白分子表面。
3 结论与讨论
本研究利用RT-PCR 方法对上海地区腹泻猪群中PSV 的携带情况进行检测,发现PSV 总体阳性率为17.7%,与Lan 等[16]在中国华东地区猪粪便中17.2%的PSV 检出率、Chen 等[17]在中国华南地区猪粪便中18.21%的PSV 检出率、Li 等[18]在四川地区猪粪便中20.4%的PSV 检出率相近。 表明PSV 在上海地区猪场普遍存在。
本研究从阳性样品中分离得到两株PSV。 对两株PSV 全基因组序列进行分析,发现其与日本部分流行PSV 毒株之间亲缘关系最近,与韩国的分离株亲缘关系较远。 我国的PSV 分离株呈簇聚集在同一分支中,说明我国的流行毒株可能存在独立进化。 核苷酸相似性分析发现PSV 分离株与韩国PSV 毒株核苷酸相似性为87%—88.5%,与欧洲PSV 毒株核苷酸相似性为84.9%—87.3%。 病毒突变和重组是病毒进化的主要方式,在进化过程中扮演着至关重要的角色。 本研究发现,CM2019008 和CM2019337 分离株的重组区域位于4 696—5 945 bp 处,此区域编码病毒特异性RNA 聚合酶,具有合成病毒RNA 能力。
VP1 衣壳蛋白是PSV 最外部的蛋白,是诱导机体产生中和抗体的主要蛋白质,也是诱导猪场产生体液免疫应答的主要成分。 目前,关于VP1 基因的氨基酸突变对生物学特性的意义还未见报道。 本研究对PSV 分离株和已报道的PSV 毒株进行了序列分析,发现PSV 分离株VP1 基因的氨基酸较少发生突变,同时整个VP1 基因的氨基酸序列未发现插入和缺失现象,该结果对病毒防治具有指导意义。
蛋白质糖基化修饰与很多重要的功能蛋白或酶相关,特别是与蛋白质的空间构想、生物活性、运输和定位密切相关,参与很多复杂的生理生化反应,如:分子识别、细胞通信、信号转导等,因此糖基化位点分析对研究病毒机制具有十分重要的意义[19]。 本研究发现分离株PSV 和参考PSV 都含有潜在NSTS 糖基化位点,其中分离株CM2019008 和法国分离株MH513612.1 含有1 个潜在糖基化位点,CM2019337 和日本LC326556.1 及德国分离株LT900497.1 含有2 个潜在糖基化位点。 而所有的PSV 都含有173 NSTS 糖基化位点。 通过对分离株VP1 蛋白的抗原表位分析,发现分离株CM2019008 和CM2019337 与中国目前已有的分离株及国外分离株的抗原表位差异不大,这可能是VP1 蛋白保持良好抗原性的重要原因之一,研究结果对进一步研究PSV 高感染率及分子机制具有指导意义。

