LKA_Unet:将基于大卷积核的空间注意力机制应用于Unet
蔡一民 杨威
摘 要: 医学图像分割技术能够自动勾勒出医学图像中人体组织结构和病变区域,进而辅助医生诊疗,减少误诊率。然而大多数现有医学图像分割模型使用的是感受野固定且很小的卷积核,不能完美地拟合图像中的全局特征信息。针对这个问题,我们提出了一个具有超大感受野的空间注意力机制模块。在DentalPanoramicXrays数据集上的实验表明,该模块加入到Unet网络后,分割精度提升了约2个百分点。
关键词: 深度学习; 医学图像分割; 空洞卷积; 空间注意力机制
中图分类号:TP391.1 文献标识码:A 文章编号:1006-8228(2023)05-81-04
LKA_Unet: applying spatial attention mechanism based on large
convolutional kernel to Unet
Cai Yimin, Yang Wei
(Guizhou University, Guiyang, Guizhou 550025, China)
Abstract: Medical image segmentation technology can automatically outline the human tissue structure and lesion areas in medical images, thus assisting doctors in diagnosis and treatment and reduce misdiagnosis. However, most existing medical image segmentation models use convolutional kernels with fixed and small receptive fields, which cannot perfectly fit the global feature information in images. To address this problem, we propose a spatial attention mechanism module with a large field of perception. Experiments on the DentalPanoramicXrays dataset show that the segmentation accuracy is improved by about 2 percentage points after the module is added to the Unet network.
Key words: deep learning; medical image segmentation; atrous convolution; spatial attention mechanism
0 引言
图像分割技术是人工智能中一个重要分支。医学图像分割在肿瘤、心血管等领域应用广泛。然而由于医学图像结构复杂且存在大量非结构化信息(如图像阴影),医学图像分割任务往往是非常困难的。现有的医学图像分割模型考虑到模型参数量以及训练速度等因素,卷积核的大小通常都是[3×3]、[5×5]、[7×7]、[9×9]等。这些模型并不能完美地拟合学习图像中的长距离依赖信息。受VAN[1]的启发,本文提出一个基于超大卷积核的空间注意力機制模块,并探索了该模块对经典Unet模型分割精度的提升程度。
1 相关工作
深度学习是人工智能领域的一个重要分支,利用大量数据进行深度学习以获得较高的分类、分割和目标检测的精度。深度学习的出现改变了人工智能的发展历程,其优势在于,能快速准确地训练出强大的模型,其本质是通过对大量数据进行训练。在经过近几年的发展后,深度学习已经从图像分类领域延伸到了图像分割、文本识别和语音语义识别等领域。在过去十年中,大量科研工作者投入到了深度学习技术研究工作中,包括最初的AlexNet网络[2],以及后续的Resnet网络[3]、VIT网络[4]和Swin Transformer[5]网络等。深度学习中广泛使用的方法是卷积神经网络(Convolutional Neural Network, CNN),其基本思想是利用多个卷积和不同大小的池化层将图像表示为一个个独立且相似度较高的值,将这些值合并后生成最终输出。通过多次卷积运算产生网络训练样本从而提高模型性能。CNN算法简单易用,且能在较短时间内得到高精度图像分割结果。
在各种医学图像分割任务上,U-Shape架构的网络,也被称为类U-Net[6]的网络,已经取得了较大的成功并成为分割任务的标杆。在Unet的基础上,衍生出了各种魔改模型,包括但不限于V-Net[7],Unet++[8],Unet3+[9],Swin Unet[10],Res Unet[11]等。还有人提出了R-shape架构,该类模型包括UnetR[12],Swin UnetR[13]和UNetR++[14]等。上述这些模型在不同分割任务上取得了巨大的成功,并且在商业应用上也取得了令人瞩目的成就。但是,在这些模型内部使用的卷积核几乎完全是固定的。然而,在图像分割整个过程中,图像中的信息密度是会发生变化的。从直观上而言,高分辨率的医学图像中特征信息比较分散,而低分辨率的医学图像中特征信息比较密集。固定大小的卷积核并不能很好地拟合密度不同的特征信息。
在本文中,我们提出了一个新的空间注意力机制模块,该模块被添加到经典的Unet网络中以试图缓解上述问题。在Unet网络的不同阶段中这个注意力机制模块的感受野大小是可变的。当图像处于高空间分辨率的阶段我们用大感受野的卷积核去提取特征,当图像处于低空间分辨率的时候我们使用小感受野的卷积核去提取特征。实验结果表明,在经典的Unet模型中添加了我们提出的空间注意力机制模块,分割精度提高了将近2个百分点。
2 模型设计细节
2.1 模型总览
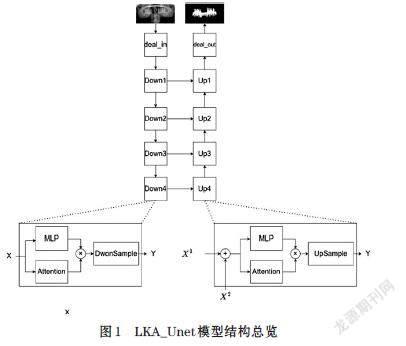
LKA_Unet模型架构如图1所示,该模型由四个编码器(Down1、Down2、Down3以及Down4)和四个解码器(Up1、Up2、Up3、Up4)组成。每个编码器由一个MLP子模块、一个Attention子模块和一个DownSample子模块组成。每个解码器由一个MLP模块、一个Attention子模块和一个UpSample子模块组成。我们将在2.2节对每个子模块的内部结构以及主要功能进行说明。
2.2 子模块介绍
2.2.1 MLP模块
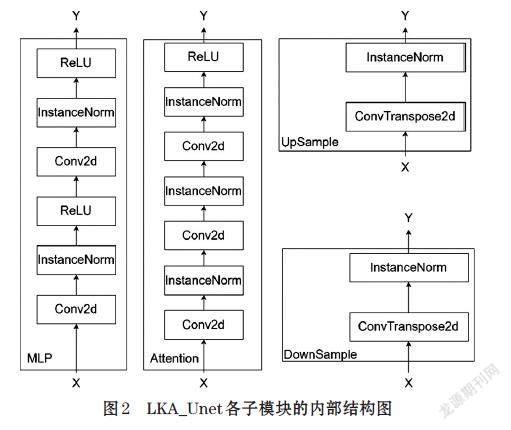
如图2所示,按照卷积层,实例归一化层和一个ReLU激活层的顺序堆叠两次后构成了MLP子模块。MLP模块并不会改变特征图的空间分辨率,它改变的是特征图的通道数目。不同的通道可以被视为对不同特征的响应,每个通道都有一个对应的卷积核,这些卷积核的参数是可学习的。
2.2.2 Attention模块
Attention模块是LKA_Unet模型的主要亮点。针对不同空间分辨率的图像,Attention模块的感受野的大小是变化的。具体来说,在Down1中,图像的空间分辨率和原始图像相同(deal_in模块作为一个中间模块,只会改变原始输入图像的通道数,以便于模型在代码实现方面更为简洁),因此它里面的Attention模块的感受野大小为31×31。在Down2中,此时图像被下采样到远空间分辨率1/4,因此在Down2中,Attention模块的感受野大小为15×15。在Down3和Down4中,输入图像的空间分辨率分别相当于原始图像的1/8和1/16,因此它俩内部的Attention模块感受野大小分别为7×7大小和3×3。
由于图像经过在不同的编码器中,空间分辨率也不一样。直观上看,空间分辨率较高的图像需要使用较大的卷积核去提取特征,而对于空间分辨率较小的图像需要用较小的卷积核来进行特征提取。如果使用固定大小的卷积核,卷积核过小,则无法提取高分辨率图像中的长距离依赖信息,若卷积过大,则会在提取低分辨率图像中的特征时导致无用计算过多,甚至造成因卷积核过多零填充导致模型的输出异常(比如输出中含有过多的nan或者inf)。
在实现方面,针对较大的卷积核,我们是通过堆叠多层空洞卷積来实现的。对于一个堆叠了多层空洞卷积的Attention模块,其感受野的大小可以由如下递推公式计算出来:
[Kn'=Kn+(Kn-1)×(dn-1)][F1=K1'][Fn]
[=Fn-1+Kn'-1][=Fn-1+(Kn-1)×dn]
其中,[Kn]表示第n个的卷积核大小,[dn]表示第n个卷积核的空洞大小,[Fn]表示经过堆叠n个卷积核之后Attention模块感受野的大小。Attention是一个空间注意力机制模块,因此,它不会改变输入图像的空间分辨率,它惟一的作用是提取图像特征。借助Attention模块的输入和输出之间的相等关系以及上述公式,就可以设计出堆叠的卷积核个数n,以及每个卷积核的大小[Kn]和空洞参数[dn]。
2.2.3 DownSample子模块和UpSample子模块
DownSample子模块主要用于降低图像空间分辨率,即下采样。UpSample子模块主要用于恢复图像空间分辨率,即上采样。DownSample由一个卷积层和一个实例归一化层组成,UpSample由一个转置卷积层和一个实例归一化层组成。在代码实现方面也可以通过bilinear插值的方式替代UpSample模块里面的转置卷积层。在Down1中和Up1中将卷积核大小k和步长s均设置为4,分别用于4倍下采样图像和4倍上采样图像。在Down2、Down3、Down4和Up4、Up3,以及Up2中卷积核大小k和步长s分别设置为2,分别用于2倍下采样和2倍上采样。从Down1到Down4阶段,下采样倍数总计为32倍,和经典Unet完全一样。从Up4到Up1,上采样倍数总计为32,完成恢复到原图像的空间分辨率。
3 实验细节
3.1 实验数据集简介
实验中使用的数据集是DentalPanoramicXrays[15,16],数据下载链接为:https://github.com/SerdarHelli/Segmentation-of-Teeth-in-Panoramic-X-ray-Image-Using-U-Net。该数据集一共包含116个口腔CBCT全景片,分割目标为所有牙齿区域。该数据集总共包含116个病例,为了读取和处理方便,作者已经将X射线图像转换成了png格式的图像并给出了对应的Ground Truth。
3.2 实验环境和实验细节
我们的实验室基于Monai0.9.1[17]和Pytorch-lighting1.7.5[18]完成的,实验使用的电脑是一台小型工作站,该机器的操作系统为Windows10,CPU型号为Intel(R) Xeon(R) CPU E5-2690,显卡为英伟达RTX2080Ti。
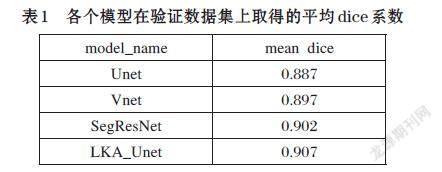
所有图像尺寸被缩放到[512×512],使用了固定随机种子42初始化Pytorch[19]环境和Cuda环境,并使用该种子划分训练集和验证集。划分比例为0.8:0.2,其中验证集不参与训练。数据增强方面,我们使用了Monai[17]提供的随机水平翻转和垂直翻转,以及随机强度缩放和平移。每个模型最少训练20个epoch,并使用Pytorch-lightning[18]提供的EarlyStop技术防止模型过拟合。我们的模型和其他对比模型在验证集上取得的最高平均dice系数如表1所示,训练过程中各个模型在验证集上取得的平均dice系数变化曲线如图3所示。
3.3 实验结果分析
各個模型在实验使用到的验证数据集上取得的平均dice系数如表1所示。相比较于Unet,LKA_Unet在验证数据集上取得的dice系数提高了2个百分点左右。相比较于SegResNet,LKA_Unet的分割性能虽然提升不是很大,但是收敛速度明显快于SegResNet。
4 结论
在本文中我们提出了一个新的空间注意力机制模块,该模块被添加到经典的Unet网络后能够促进Unet在不同阶段更好地提取图像特征信息。在DentalPanoramicXrays[15,16]数据集上进行的实验表明,我们提出的空间注意力机制模块确实在一定程度上提高了Unet网络的分割精度。
参考文献(References):
[1] Guo M, Lu C, Liu Z, et al. Visual attention network[J].
arXiv preprint arXiv:2202.09741,2022
[2] Krizhevsky A, Sutskever I, Hinton G E. Imagenet
classification with deep convolutional neural networks[J]. Communications of the ACM,2017,60(6):84-90
[3] He K, Zhang X, Ren S, et al. Deep residual learning for
image recognition[C],2016
[4] Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is
worth 16x16 words: Transformers for image recognition at scale[J]. arXiv preprint arXiv:2010.11929,2020
[5] Liu Z, Lin Y, Cao Y, et al. Swin transformer: Hierarchical
vision transformer using shifted windows[C],2021
[6] Ronneberger O, Fischer P, Brox T. U-net: Convolutional
networks for biomedical image segmentation[C].Springer,2015
[7] Milletari F, Navab N, Ahmadi S. V-net: Fully convolutional
neural networks for volumetric medical image segmentation[C].IEEE,2016
[8] Zhou Z, Siddiquee M M R, Tajbakhsh N, et al. Unet++:
Redesigning skip connections to exploit multiscale features in image segmentation[J]. IEEE transactions on medical imaging,2019,39(6):1856-1867
[9] Huang H, Lin L, Tong R, et al. Unet 3+: A full-scale
connected unet for medical image segmentation[C].IEEE,2020
[10] Cao H, Wang Y, Chen J, et al. Swin-unet: Unet-like
pure transformer for medical image segmentation[J]. arXiv preprint arXiv:2105.05537,2021
[11] Diakogiannis F I, Waldner F C C O, Caccetta P, et al.
ResUNet-a: A deep learning framework for semantic segmentation of remotely sensed data[J]. ISPRS Journal of Photogrammetry and Remote Sensing,2020,162:94-114
[12] Hatamizadeh A, Tang Y, Nath V, et al. Unetr:Transformers for 3d medical image segmentation[C], 2022
[13] Hatamizadeh A, Nath V, Tang Y, et al. Swin unetr: Swin
transformers for semantic segmentation of brain tumors in mri images[C].Springer,2022
[14] Shaker A, Maaz M, Rasheed H, et al. UNETR++:
Delving into Efficient and Accurate 3D Medical Image Segmentation[Z]. arXiv,2022
[15] HELL I S, Hamamci A. Tooth Instance Segmentation on
Panoramic Dental Radiographs Using U-Nets and Morphological Processing[J]. D{\"u}zce {\"U}niversitesi Bilim ve Teknoloji Dergisi,2022,10(1):39-50
[16] Abdi A, Kasaei S. Panoramic dental X-rays withsegmented mandibles[J]. Mendeley Data, v2,2020[17] Consortium M. MONAI: Medical Open Network for AI[Z].2020
[18] Falcon W, Team T P L. PyTorch Lightning[Z].2019
[19] Paszke A, Gross S, Massa F, et al. Pytorch: An
imperative style, high-performance deep learning library[J]. Advances in neural information processing systems,2019,32

