与结肠腺癌相关的细胞周期生物标志物的识别
俞秋奕 贺平安

摘 要: 結直肠癌是一种致死率较高的癌症,结肠腺癌是其最常见的病理亚型。挖掘与结肠腺癌相关的细胞周期相关基因、生长因子和激素,对研究结肠腺癌的发病机制以及诊断治疗都具有重要价值。利用加权基因共表达网络分析方法,根据TCGA和GTEx数据库中结肠腺癌样本的转录组数据构建基因共表达网络;利用模块特征相关系数,筛选出与结肠腺癌相关的重要模块;通过基因差异表达分析和富集分析,得到重要模块的差异基因和模块功能信息;利用生存分析识别与结肠腺癌相关的细胞周期生物标志物;进一步根据蛋白互作网络推断诱导结肠腺癌发生的内源性因素。结果表明:结肠腺癌相关的7个细胞周期生物标志物(CDC45、EREG、PBK、TOP2A、PER1、SNCG、NGFR)和4个内源性因素(ANP、BNP、FGF、NGF),这些生物标志物和内源性因素可作为结肠腺癌诊断和治疗的依据。
关键词: 结肠腺癌;细胞周期;加权基因共表达网络分析;基因差异表达分析;蛋白互作网络
中图分类号: O29
文献标志码: A
文章编号: 1673-3851 (2023) 09-0628-10
引文格式:俞秋奕,贺平安. 与结肠腺癌相关的细胞周期生物标志物的识别[J]. 浙江理工大学学报(自然科学),2023,49(5):628-637.
Reference Format: YU Qiuyi, HE Ping′an. Identification of cell cycle-related biomarkers in colon adenocarcinoma[J]. Journal of Zhejiang Sci-Tech University,2023,49(5):628-637.
Identification of cell cycle-related biomarkers in colon adenocarcinoma
YU Qiuyi, HE Ping′an
(School of Science, Zhejiang Sci-Tech University, Hangzhou 310018, China)
Abstract: Colorectal cancer is a kind of cancer with a high fatality rate, and colon adenocarcinoma is its most common pathological subtype. The exploration of cell cycle-related genes, growth factors and hormones related to colon adenocarcinoma is of great value for the study of pathogenesis, diagnosis and treatment of colon adenocarcinoma. Weighted gene co-expression network analysis was used to construct gene co-expression network for the colon adenocarcinoma samples from TCGA and GTEx databases. Significant modules related to colon adenocarcinoma were selected by using the correlation coefficient of module eigengene and sample characteristics. Differentially expressed genes and module function information of significant modules were obtained by differential expression gene analysis and enrichment analysis. Survival analysis was used to identify cell cycle-related biomarkers of colon adenocarcinoma. Furthermore, the endogenous factors inducing colon adenocarcinoma were inferred based on the protein-protein interaction network. Finally, seven cell cycle biomarkers (CDC45, EREG, PBK, TOP2A, PER1, SNCG, and NGFR) and four endogenous factors (ANP, BNP, FGF, and NGF) related to colon adenocarcinoma were identified. These biomarkers and endogenous factors can be used as the basis for diagnosis and treatment of colon adenocarcinoma.
Key words: colon adenocarcinoma; cell cycle; weighted gene co-expression network analysis; differential expression gene analysis; protein-protein interaction network
0 引 言
结直肠癌是全世界发病率最高的癌症之一。2022年中国和美国的结直肠癌确诊病例数分别为59.22万例和16.02万例[1],分别占该年新增癌症确诊病例的12.3%和6.8%。尽管通过癌症预防筛查、早期诊断以及高质量治疗使得结直肠癌的发病率和死亡率有所下降,但其仍然是一个主要的全球公共卫生问题。据统计,全球结直肠癌发病年龄在过去十年趋于年轻化[2],其原因尚不清楚。在中国,年龄在40到44岁之间的结直肠癌患者人数迅速上升[3],造成了极大的医疗和社会负担。因此,迫切需要对结直肠癌的病理机制进行研究;结肠腺癌作为结直肠癌最常见的病理亚型,已成为该领域的主要研究对象之一。
目前,结肠腺癌大部分研究重点集中在从分子层面上寻找与结肠腺癌发生、发展相关的各类生物标志物,这些生物标志物大致可分为如下几类:第一类与RNA相关,包括非编码RNA和编码RNA,如长链非编码RNA[4](lncRNA)、微小RNA[5](miRNA)、环状RNA[6](circRNA)等,或信使RNA[7](mRNA)等;第二类是具有某种特定功能的基因,如免疫相关基因[8]、代谢相关基因[9]、糖酵解相关基因[10]和线粒体相关基因[11]等。此外,构建生物标志物调控网络可以更进一步探索相关肿瘤的发生发展机理。杨海鸥等[5]对发掘的结直肠癌相关miRNA构建了信号通路调控网络,通过该网络研究了结直肠癌发生机制以及优化治疗策略。
细胞周期是一个复杂过程,包括细胞生长、遗传物质复制和细胞增殖等过程。一个完整的细胞周期过程可分为G1期、S期、G2期和M(有丝分裂)期4个阶段,每个阶段都受相关基因的严格调控。研究表明,细胞周期调控异常基本上存在于所有肿瘤类型中,是肿瘤发生的驱动力[12]。对细胞周期相关基因的分析有助于揭示癌症的发病机理,从而为癌症诊断和治疗提供帮助。Li等[13]对编码细胞周期蛋白(Cyclin)家族中的24个cyclin基因进行研究,利用它们的表达水平构建出结肠癌分期预测模型,结果显示该模型比传统的肿瘤TNM分期方法精度更高,并由此推断cyclin基因可作为一种结肠癌诊断和预后的生物标志物。闫鸣等[14]实验发现,在人结直肠癌组织中高表达的细胞周期蛋白依赖性激酶1(Cyclin-dependent kinases 1,CDK1),可作为结直肠癌早期诊断及靶向药物治疗的生物标志物。然而,相关研究只关注了几类直接参与细胞周期调控的基因,而对其他间接参与细胞周期调控并在细胞周期过程中发挥重要作用的基因缺乏探讨。
生长因子和激素在癌症发生发展中具有重要作用,生长因子和激素可通过与细胞表面或细胞内特定受体结合作用于细胞,是癌细胞异常增殖的诱因[15]。朱红珍等[16]发现,表皮生长因子及其受体、胰岛样生长因子和结合蛋白在结直肠癌中表达异常,且与结直肠癌的分期密切相关,在结直肠癌患者中具有较高检测价值。钱颖超等[17]研究了雌激素及其受体在结直肠癌中发挥的作用,发现雌激素水平升高能降低结直肠癌的发病风险,而雌激素受体表达水平降低与结直肠癌的转移相关,为结直肠癌的早期诊断及治疗提供了依据。
本文针对结肠腺癌基因转录组数据,利用生物信息学方法识别与细胞周期相关的生物标志物,通过构建蛋白互作(Protein-protein interaction, PPI)网络,推断可能诱导结肠腺癌发生的内源性因素。应用加权基因共表达网络分析(Weighted gene co-expression network analysis, WGCNA)方法,构建结肠腺癌基因的共表达网络;利用模块特征相关系数,筛选与结肠腺癌高度相关的重要模块;运用基因差异表达分析和富集分析,得到重要模块中差异基因和模块功能信息;對重要模块中与细胞周期相关的差异基因进行生存分析,识别出可作为结肠腺癌潜在生物标志物的关键细胞周期相关基因;进一步构建关键细胞周期相关基因的PPI网络,识别调控部分关键细胞周期相关基因的潜在受体,推断与结肠腺癌发生发展密切相关的生长因子和激素等内源性因素。
1 数据和方法
1.1 数据及数据预处理
本文所选用的结肠腺癌基因转录组数据下载自UCSC Xena(http://xena.ucsc.edu/)数据库[18],下载的数据共639个样本,其中,TCGA含有290个患病样本和41个正常样本;GTEx中包含308个正常样本,弥补了TCGA数据库中所含正常样本不足的缺点。每个样本都包含58581个基因的表达数据,数据采用FPKM标准化方法。
由于下载数据集的基因表达形式为log2(xFPKM+0.001)(xFPKM为每个基因表达水平的FPKM标准化数值),导致数据中包含负数不利于后续分析,因此需要对数据进行预处理。对原数据基因表达单位进行反运算,还原为FPKM形式,此时所有表达为负数的基因表达量可等同为0,故全部赋值为0。进一步对数据进行背景校正和分位数归一化,设置同一个基因在所有样本中的平均表达水平阈值为5,筛去低于该阈值的低丰度基因,最终得到一个由639个结肠腺癌样本共11035个基因的表达值构成的数据集X=(xij)11035×639,其中xij表示第i个基因在第j个样本中的表达值。为识别细胞周期相关基因和受体,本文分别在Gene ontology(GO)数据库和BaderLab细胞相互作用数据库中,搜集了2388个细胞周期相关基因和1851个受体基因。
1.2 构建基因共表达网络
基因共表达网络是基于基因表达数据,通过计算基因间的相关性来构建基因网络,用于分析基因调控关系的方法。该网络通常显示为节点(基因)和边(作用关系)的图形,可以揭示基因之间的功能和相互作用。本文利用WGCNA算法构建基因共表达网络。
对输入数据构建样本聚类树,根据聚类树高度去除离群样本点,通过计算任意两个基因间的相关系数,得到相关性矩阵S=(sij),其中:sij=|cor(xi,xj)|=|CAB|为基因i和基因j间的皮尔森相关系数;
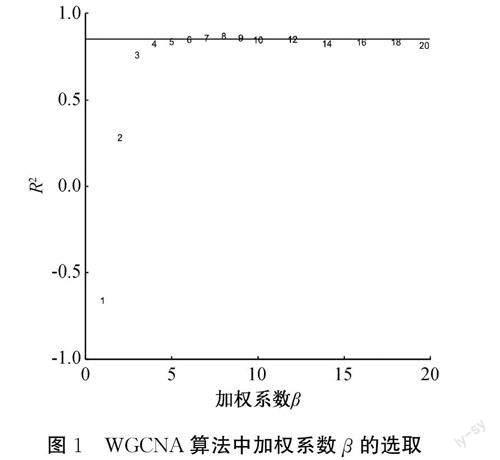
为进一步区分基因间的相关性,WGCNA算法将相关性矩阵S转化为邻接矩阵A=(αij),其中:αij=sβij为任意基因对(i,j)的相关性指标;β为加权系数。定义连通度ki=∑ni=1i≠jαij,表示基因i与其他基因α值的和,n为基因总数。基于无尺度分布假设,ki的对数值log10(ki)与其出现概率的对数值log10(p(ki))呈负相关关系,通过一元线性回归方法可求解最佳β值,同时要求R2大于0.8。
为描述基因间的相似性,该算法引入基因的间接相关性指标Iij=∑mαimαmj,从而将邻接矩阵A转化为拓扑重叠矩阵(Topological overlap matrix, TOM)Ω=(ωij),
将TOM矩阵的相似性度量转化为节点相异度dωij=1-ωij,根据dωij值将表达模式相似的基因划分为相同的模块。为获得合适的模块,本文设置基因模块最小规模为30。根据WGCNA的定义,模块特征基因(Module eigengene, ME)是模块的第一主成分,可被看作是基因转录组数据的模块表示。通过计算ME与临床特征向量间的皮尔森相关系数,能确定与疾病相关的重要临床模块。
本文根据样本组织来源创建了一个635维的疾病特征向量,其中1代表患病,0代表正常,计算出模块特征相关系数a,设定阈值|a|>0.5和显著性检验值p<0.05,筛选与结肠腺癌相关的重要模块。
1.3 基因差异表达分析
通过比较不同条件下基因表达的差异,能够识别出与条件有关的基因,揭示基因调控网络和相关的生物过程,为疾病的病理机制和治疗提供线索。本文运用基因差异表达分析,计算每个基因在患病样本和正常样本中表达量的差异倍数(Fold change, FC),通常采用对数形式,根据结果的正负值,分为上调基因和下调基因,并设定阈值|log2xFC|>1(xFC为每个基因在患病样本和正常样本中表达量的差异倍数值),经过错误发现率(False discovery rate, FDR)校正过后的差异显著性检验值p<0.05,筛选患病样本和正常样本中具有差异表达的基因。
1.4 富集分析
富集分析是一种常用的基因功能注释方法,其作用在于通过比较基因组数据中不同基因的富集情况,来寻找与特定生物过程、细胞功能或疾病相关的生物学通路、功能模块或分子机制等信息[19]。富集分析的统计原理是用超几何分布模型来检验一组基因中某个功能类的显著性,假设一组输入基因的数目为n,其中有k个基因富集于某种特定生物学功能或信号通路,则该通路在这组基因中的显著性检验p值可通过(5)式计算:
其中:N表示所有基因的数目;K表示所有基因中注释为某特定生物学功能或信号通路的背景基因集中的基因数目。p值结果采用Benjamini-Hochberg方法进行多重检验校正,本文设定p<0.05为富集结果显著。
1.5 生存分析
生存分析是一种非参数统计方法[20],常用于研究不同组的癌症患者在一种或多种因素作用下其生存概率随时间发展而发生的变化趋势,在评估治疗效果、预测疾病进程、研究疾病发生机制和筛选生物标志物等方面具有重要作用。
假设在某因素作用下个体的生存时间为T,则其生存时间超过t(t≥0)的概率可用S(t)=P(T>t)表示,其中S(t)也称为生存函数。通过对S(t)的估计可绘制生存曲线,从而反映个体生存概率随时间发展的变化趋势。
本文采用Kaplan-Meier方法估计S(t),即使用乘积极限法将S(t)写成递推式的形式。假设时间t1的生存函数值为S(t1),则时间t2(t2>t1)的生存函數值可表示为S(t2)=P(δ1)×S(t1),其中:P(δ1)=1-d/n为个体从t1存活到t2的概率;在时间t1到时间t2内,d和n分别表示实际发生事件的个体数和有可能发生事件的个体总数。该方法通过Log-rank检验来比较两组癌症患者之间的生存曲线是否存在差异,并获得显著性检验p值。
本文将总体生存期(Overall survival, OS)作为生存时间,关注的事件是结肠腺癌发病导致患者的死亡。从OncoLnc网站上下载了TCGA中所有结肠腺癌病人的OS数据以及每一个细胞周期相关基因的表达数据,取上下四分位数作为高低表达的阈值进行生存分析,保留具有生存差异(p<0.05)的细胞周期相关基因并绘制其KaplanMeier生存曲线,作为关键细胞周期相关基因进行后续分析。
1.6 构建PPI网络
PPI网络能反映基因所对应蛋白间的相互作用关系,为识别细胞周期相关基因的潜在受体提供了一定的线索。STRING数据库是构建PPI网络的常用工具,整合了大量已知和预测的蛋白质之间的联系,包括物理相互作用和功能联系等[21]。从WGCNA构建的共表达基因网络中筛选出细胞周期相关基因直接相关的基因子集,根据共表达基因间的相关系数进行排序,选取上四分位数以上的基因,并从中挑出受体基因作为构建PPI网络的候选基因;接着将候选基因上传到STRING数据库,设置参数置信度大于0.4,并选出其中主要的相互作用作进一步分析。
2 结果与讨论
2.1 数据聚类结果及重要模块识别
利用WGCNA算法,输入数据集X中的数据,首先根据样本聚类树高度共舍去4个异常值,此时患病样本290个,正常样本345个。计算任意两个基因间的皮尔森相关系数得到相关性矩阵,再根据无尺度网络确定加权系数β的最佳取值,结果如图1所示。图1表明:当β=7时,R2=0.8558,首次超过0.85,此时满足无尺度网络特征,因此本文将β=7作为加权系数构造邻接矩阵;通过计算节点相异度,将635个结肠腺癌样本中的11035个基因聚类到了10个不同的共表达模块,这10个模块分别被记为M1-M10;通过计算10个模块的ME值与疾病特征向量间的皮尔森相关系数,识别与结肠腺癌发生发展相关的重要模块。
各个模块的模块基因数及模块特征相关系数见表1。M1、M2、M4和M9与结肠腺癌有较高关联性,其中M1模块的模块特征相关系数达到了0.93且所含基因数最多,可能提示该模块中的某些基因是导致结肠腺癌发生发展的重要因素,M2、M4和M9模块的模块特征相关系数分别为-0.68、-0.76和0.68(见表1)。
2.2 差异基因筛选结果
通过基因差异表达分析,对4个重要模块中的基因分别做差异分析,筛选结肠腺癌患者与正常人之间的具有明显差异的基因以及其中的细胞周期相关差异表达基因(见表2)。由表2可以看出:M1和M2两个模块中含有较多的差异基因,分别有279和214个表达差异的基因;在M1模块的差异基因中,上调基因与下调基因的数量相差不大,为138∶141;该模块中与细胞周期相关的差异基因明显分化,上调的细胞周期相关基因个数为49,而下调的仅为10;类似的,在M2模块中,尽管上调基因的个数少于下调基因的个数,它们的个数比为90∶124,然而细胞周期相关基因的上调基因数量多于下调基因,为10∶7;细胞周期相关基因的上调表明细胞分裂速度加快,可能是导致细胞周期调控异常促进结肠腺癌发生发展的主要原因;对于其余2个模块,M4模块中所含差异基因较少且只有一个下调的细胞周期相关基因,而M9模块中则不含细胞周期相关基因。
2.3 富集分析结果
将M1模块中的279个和M2模块中的214个差异基因进行GO和KEGG富集分析,以此推断其可能参与的生物过程及所在的信号通路。其中GO富集分析主要从3个层面对基因进行描述:生物学过程(Biological process, BP)、分子功能(Molecular function, MF)和细胞成分(Cellular component, CC)。基因比例(Gene Ratio)为差异基因中与该通路相关的基因数与整个差异基因总数的比值,可以反映一组基因在某一特定通路中的富集程度。由于M9和M4两个模块拥有的差异基因较少,不能得到富集结果,故略去它们的富集分析结果。M1模块中主要的GO富集结果如图2(a)所示,结果显示279个差异基因显著富集在生物学过程中的“organelle fission”,“nuclear division”,“mitotic sister chromatid segregation”等过程,均与细胞周期M期进程密切相关,同时也解释了在该模块中可以筛选出更多的细胞周期相关基因。KEGG富集结果显示,279个差异基因高度富集在“Cytokine-cytokine receptor interaction”“Chemokine signaling pathway”“Cell cycle”等通路,表明该模块中差异基因参与受体相互作用(见图2(b))。
M2模块中的差异基因在KEGG中无显著通路,主要GO富集结果如图3所示。214个差异基因主要富集在生物学过程中的“muscle system process”“regulation of heart contraction”等过程,参与调节肌肉系统和心脏收缩功能(见图3)。
2.4 生存分析结果
使用Kaplan-Meier生存分析方法,利用4个重要模块中筛选出的共77个细胞周期相关基因的OS数据,按表达值取大于等于上四分位数的为高表达组,小于等于下四分位数的为低表达组,通过Log-rank检验比较两组的生存曲线是否存在显著差异。取差异显著性检验值p<0.05,筛选出M1和M2两个模块中共7个对结肠腺癌生存影响显著的关键细胞周期相关基因。M1模块中共得到6个关键细胞周期相关基因,分别为PBK(p=0.0036),EREG(p=0.0180),SNCG(p=0.0220),PER1(p=0.0410),CDC45(p=0.0460)和TOP2A(p=0.0480),其Kaplan-Meier生存曲线如图4所示。
M2模块中得到一个关键细胞周期相关基因,NGFR(p=0.027),图5为其Kaplan-Meier生存曲线。7个关键细胞周期相关基因中上调的有4个且全部在M1模块,分别为:PBK、EREG、CDC45和TOP2A。其中PBK编码生成一种丝氨酸/苏氨酸蛋白激酶,参与有丝分裂调控及肿瘤进程,可作为多种肿瘤的生物标志物[22],PBK在结直肠癌组织和细胞系中过表达,可将正常细胞系转化为致瘤细胞系,促进肿瘤发生[23]。PBK在结肠腺癌中高表达且使得结肠腺癌患者生存率显著降低(见图4(a)),
说明该基因对结肠腺癌的发生发展有着促进作用。EREG是表皮生长因子家族的一员,编码生成一种表皮生长因子受体的配体,与癌症发展密切相关,可作为癌症治疗的潜在药物靶点[24]。EREG的异常激活已被证明与结直肠癌的不良预后相关,并促进肿瘤的恶化[25]。图4(b)显示,高表达的EREG在结肠腺癌患者中显示出较差的生存结果,说明其过表达加重结肠腺癌的恶化。CDC45编码产生启动DNA复制所必需的蛋白质,与多种癌症发生相关,可作为结直肠癌的生物标志物[26],高表达的CDC45在结肠腺癌中显示出较差的生存结果(见图4(e)),说明其高表达促进结肠腺癌的恶化。TOP2A可编码产生参与染色体分离的核蛋白,在细胞分裂、防止DNA过度复制和染色体结构维护等方面起重要作用[27]。研究发现,TOP2A在结肠癌中过表达,通过下调TOP2A表达,能够显著抑制细胞增殖、迁移和侵袭,促进癌细胞凋亡[28]。该发现与TOP2A在结肠腺癌中的生存曲线一致(见图4(f)),提示TOP2A过表达加速了结肠腺癌细胞的增殖,发挥着促癌作用。
下调基因共3个,分别为M1模块中的SNCG和PER1,以及M2模块中的NGFR。其中SNCG编码的蛋白是突触核蛋白家族的成员之一,在细胞增殖中起重要作用[29]。研究发现SNCG与多种消化系统肿瘤的发生相关[30],高表达的SNCG可作为结直肠癌肝转移的生物标志物[31]。在结肠腺癌中,SNCG下调且随着生存时间的增加表现出较好的生存结果(见图4(c)),说明低表达的SNCG抑制结肠腺癌细胞增殖,发挥着抑癌作用。PER1是调控机体昼夜节律的核心基因,有助于维持和优化结肠的功能[32]。研究发现,过表达的PER1可使癌细胞DNA损伤诱导其凋亡,而抑制PER1则可以减缓细胞凋亡 [33]。在结肠腺癌中,低表達的PER1显著降低了结肠腺癌患者的生存率(见图4(d)),说明低表达的PER1减缓了结肠腺癌细胞的凋亡,促进结肠腺癌进一步恶化。
NGFR编码的蛋白是肿瘤坏死因子受体超家族的成员,也是消化系统肿瘤中唯一的抑癌因子[34]。研究发现NGFR在结直肠癌中过表达,能够通过诱导细胞周期阻滞抑制细胞增殖和侵袭[35],可作为预测结直肠癌患者化疗敏感性的标志物[36]。NGFR在结肠腺癌中下调,且高表达的NGFR显著提升了结肠腺癌患者的生存率(见图5),提示其是抑制结肠腺癌发生发展的重要基因。
综合图4和图5结果分析显示:CDC45、EREG、PBK、TOP2A、PER1、SNCG和NGFR与结肠腺癌的发生发展密切相关,可作为结肠腺癌的潜在生物标志物。
2.5 PPI网络
为进一步寻找这些生物标志物相关的生长因子、激素等内源性因素,本文利用PPI网络得到了与其密切相关的受体蛋白,通过搜索与这些受体相关的生长因子和激素,以此推断与结肠腺癌发生发展相关的内源性因素。针对M1模块中6个关键细胞周期相关基因,利用WGCNA构建的基因共表达网络筛选出只与这些关键细胞周期相关基因有直接相关的基因,以两个基因间的相关性权重值的上四分位数作为阈值,高于该值的基因作为候选基因;通过这些基因的表达状态和基因类型筛选具有差异表达的受体基因。这主要是考虑到受体表达水平的变化可能是结合了某种生长因子或激素导致的,因此本文选用差异表达的受体构建互作网络,共筛选出18个受体,其中5个上调,13个下调(见表3)。
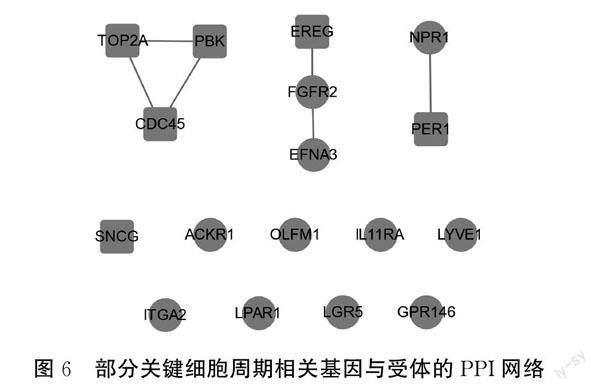
将选定的18个受体和6个关键细胞周期相关基因作为候选基因,上传到STRING数据库中构建PPI网络,方形节点为关键细胞周期相关基因,圆形节点为受体,识别出PER1的潜在受体NPR1和EREG的潜在受体FGFR2(见图6)。受体NPR1基因产物为一种膜结合的鸟苷酸环化酶,可作为心房钠肽(ANP)和脑钠肽(BNP)的主要受体,这两种肽在心肌中对预防心肌加速纤维化具有重要作用[37]。受体基因FGFR2编码的蛋白是成纤维细胞生长因子(FGF)受体家族的成员之一,能与FGF等多种信号相互作用驱动肿瘤发生[38]。ANP、BNP和FGF在结肠腺癌发生和发展的过程中可能具有重要的作用。Chen等[39]在溃疡性结肠炎患者中观察到ANP及其受体水平下降,并发现ANP可能改变结肠屏障功能,提出ANP治疗能作为溃疡性结肠炎的一种潜在治疗方法。卢晓华等[40]发现BNP的表达水平与结直肠癌患者术后深静脉血栓形成(DVT)之间有密切联系。Matsuda等[41]研究发现FGFR2及其亚型在结直肠癌中过度表达,并实验推测FGFs/FGFRs可能以自分泌方式调节结直肠癌细胞的生长。M2模块中NGFR本身是神经生长因子(NGF)的细胞表面受体,通过与NGF结合共同参与炎症反应,可导致溃疡性结肠炎的发生[42],NGF能够促进结直肠癌的发展和且高表达的NGF与结直肠癌的不良预后相关[43]。而在结肠腺癌病人中,ANP、BNP、FGF和NGF所对应的受体均下调(见图6),表明肿瘤细胞可能长期受到这些内源性因素的刺激,导致受体逐渐适应且敏感度降低[44]。因此,ANP、BNP、FGF和NGF可能是诱导结肠腺癌发生发展的内源性因素。
3 结 论
本文针对结肠腺癌的TCGA和GTEx联合基因转录组数据,使用WGCNA算法、基因差异表达分析、富集分析、生存分析和PPI网络分析等方法,得到了与结肠腺癌发生发展相关的细胞周期生物标志物和相关内源性因素,结论如下:
a)识别出7个具有生存差异的与细胞周期相关的关键细胞周期相关基因:CDC45、EREG、PBK、TOP2A、PER1、SNCG和NGFR,这些基因在结肠腺癌的发生发展中发挥着重要作用,可作为结肠腺癌的生物标志物。
b)识别出与结肠腺癌发生发展相关的受体基因NPR1、FGFR2和NGFR,其中:NPR1为ANP和BNP的主要受体,FGFR2为FGF的主要受体,NGFR为NGF的主要受体。这些生长因子和激素与结肠腺癌密切相关,推测ANP、BNP、FGF和NGF可能是诱导结肠腺癌发生发展的内源性因素。
参考文献:
[1]Xia C F, Dong X S, Li H, et al. Cancer statistics in China and United States, 2022: Profiles, trends, and determinants[J]. Chinese Medical Journal , 2022, 135(5):584-590.
[2]Scheurlen K M, Billeter A T, O′Brien S J, et al. Metabolic dysfunction and early-onset colorectal cancer-how macrophages build the bridge[J]. Cancer Medicine, 2020, 9(18):6679-6693.
[3]周雄, 胡明, 李子帥等. 2020年全球及中国结直肠癌流行状况分析[J]. 海军军医大学学报, 2022, 43(12):1356-1364.
[4]童曼曼, 刘熙称, 孙晓宇等. 长链非编码RNA相关的竞争性内源性RNA网络中结肠腺癌潜在生物标志物的研究进展[J]. 癌症进展, 2022, 20(18):1869-1871.
[5]杨海鸥, 王欣怡, 左梦思, 等. 结直肠癌相关miRNA及其信号通路调控网络综合分析[J]. 肿瘤综合治疗电子杂志, 2020, 6(2):102-108.
[6]李建祺, 张晶, 甄亚男, 等. 结直肠癌组织中差异表达cirRNA的筛选及功能分析[J]. 山东医药, 2019, 59(8):13-16.
[7]欧阳清, 康海仙, 姚运红, 等. NDRG1 mRNA在结直肠癌中的表达及意义[J]. 世界复合医学, 2022, 8(1):1-3.
[8]李伟华, 赵鹏宇, 黎鸿坚, 等. 基于免疫基因的RNA-seq数据构建结直肠癌预后生存预测模型[J]. 中华结直肠疾病电子杂志, 2021, 10(6):585-590.
[9]王婷, 李春晓, 南鹏, 等. 基于多数据库分析代谢相关基因DLAT在结直肠癌中的表达及其临床意义[J]. 解放军医学杂志, 2019, 44(4):311-317.
[10]何耀明, 李尚仁, 李晓平, 等. 基于糖酵解风险基因老年结直肠癌患者预后风险模型构建[J]. 河南医学高等专科学校学报, 2022, 34(3):259-266.
[11]Gao H L, Xing F. A novel signature model based on mitochondrial-related genes for predicting survival of colon adenocarcinoma[J]. BMC Medical Informatics and Decision Making, 2022, 22(1):277.
[12]Suski J M, Braun M, Strmiska V, et al. Targeting cell-cycle machinery in cancer[J]. Cancer Cell, 2021, 39(6):759-778.
[13]Li J L, Zhou L Y, Liu Y, et al. Comprehensive analysis of cyclin family gene expression in colon cancer[J]. Frontiers in Oncology, 2021, 11:674394.
[14]閆鸣, 白日星, 程石, 等. 细胞周期蛋白依赖性激酶1在人结直肠癌组织中的表达意义[J]. 中国临床医生杂志, 2021, 49(9):1080-1082.
[15]Gonzlez G, Lage A. Cancer vaccines for hormone/growth factor immune deprivation: A feasible approach for cancer treatment[J]. Current Cancer Drug Targets, 7(3):229-241.
[16]朱红珍, 沈建东, 俞志忠. 血清表皮生长因子及受体、胰岛素样生长因子和结合蛋白在结直肠癌患者中的表达及其与疾病分期的关系[J]. 中国医学创新, 2021, 18(31):147-150.
[17]钱颖超, 曾思恩. 雌激素/雌激素受体在结直肠癌发生发展中的研究进展[J]. 华夏医学, 2021, 34(1):176-180.
[18]Goldman M J, Craft B, Hastie M, et al. Visualizing and interpreting cancer genomics data via the Xena platform[J]. Nature Biotechnology, 2020, 38(6):675-678.
[19]Mubeen S, Tom K A, Hofmann A M, et al. On the influence of several factors on pathway enrichment analysis[J]. Briefings in Bioinformatics, 2022, 23(3):bbac143.
[20]Indrayan A, Tripathi C B. Survival analysis: Where, why, what and how?[J]. Indian Pediatrics, 2022, 59(1):74-79.
[21]Szklarczyk D, Gable A L, Nastou K C, et al. The STRING database in 2021: Customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets[J]. Nucleic Acids Research, 2021, 49(D1): D605-D612.
[22]Wen H, Chen Z, Li M, et al. An integrative pan-cancer analysis of PBK in human tumors[J]. Frontiers in Molecular Biosciences, 2021, 8:755911.
[23]Zhu F, Zykova T A, Kang B S, et al. Bidirectional signals transduced by TOPK-ERK interaction increase tumorigenesis of HCT116 colorectal cancer cells[J]. Gastroenterology, 2007, 133(1):219-231.
[24]Cheng W L, Feng P H, Lee K Y, et al. The role of EREG/EGFR pathway in tumor progression[J]. International Journal of Molecular Sciences, 2021, 22(23):12828.
[25]Li X D, Miao S Y, Wang G L, et al. Amphiregulin and epiregulin expression in colorectal carcinoma and the correlation with clinicopathological characteristics[J]. Onkologie, 2010, 33(7):353-358.
[26]Hu Y, Wang L, Li Z, et al. Potential prognostic and diagnostic values of CDC6, CDC45, ORC6 and SNHG7 in colorectal cancer[J]. Oncotargets and Therapy, 2019, 12:11609-11621.
[27]Uusküla-Reimand L, Wilson M D. Untangling the roles of TOP2A and TOP2B in transcription and cancer[J]. Science Advances, 2022, 8(44):eadd4920.
[28]Zhang R, Xu J, Zhao J, et al. Proliferation and invasion of colon cancer cells are suppressed by knockdown of TOP2A[J]. Journal of Cellular Biochemistry, 2018, 119(9):7256-7263.
[29]Liu J, Spence M J, Zhang Y L, et al. Transcriptional suppression of synuclein gamma (SNCG) expression in human breast cancer cells by the growth inhibitory cytokine oncostatin M[J]. Breast Cancer Research Treat, 2000, 62(2):99-107.
[30]魏祎, 曾妍. SNCG在消化系统肿瘤中的相关研究进展[J]. 农垦医学, 2021, 43(5):432-435.
[31]郭春光, 孙力超, 刘骞, 等. SNCG在结直肠癌肝转移中的表达及其临床意义[J]. 中华胃肠外科杂志, 2012, 15(6): 625-628.
[32]Polidarov L, Houdek P, Sldek M, et al. Mechanisms of hormonal regulation of the peripheral circadian clock in the colon[J]. Chronobiology International, 2017, 34(1):1-16.
[33]Liu Y, Hao J, Yuan G, et al. PER1 as a tumor suppressor attenuated in the malignant phenotypes of breast cancer cells[J]. International Journal of General Medicine, 2021, 14:7077-7087.
[34]Blondy S, Christou N, David V, et al. Neurotrophins and their involvement in digestive cancers[J]. Cell Death and Disease, 2019, 10(2):123.
[35]Yang Z, Chen H, Huo L, et al. Epigenetic inactivation and tumor-suppressor behavior of NGFR in human colorectal cancer[J]. Molecular Cancer Research, 2015, 13(1):107-119.
[36]Chen H, Huang J, Chen C, et al. NGFR increases the chemosensitivity of colorectal cancer cells by enhancing the apoptotic and autophagic effects of 5-fluorouracil via the activation of S100A9[J]. Frontiers in Oncology, 2021, 11:652081.
[37]Nakagawa Y, Nishikimi T, Kuwahara K. Atrial and brain natriuretic peptides: Hormones secreted from the heart[J]. Peptides, 2019, 111:18-25.
[38]Ferguson H R, Smith M P, Francavilla C. Fibroblast growth factor receptors (FGFRs) and noncanonical partners in cancer signaling[J]. Cells, 2021, 10(5):1201.
[39]Chen C, Zhang Y, Tao M, et al. Atrial natriuretic peptide attenuates colitis via inhibition of the cGAS-STING pathway in colonic epithelial cells[J]. International Journal of Biological Sciences, 2022, 18(4):1737-1754.
[40]盧晓华, 田红梅, 王冲, 等. 凝血四项、血浆D-D、NT-proBNP水平与结直肠癌患者术后DVT的关系[J]. 分子诊断与治疗杂志, 2022, 14(3):495-498.
[41]Matsuda Y, Ueda J, Ishiwata T. Fibroblast growth factor receptor 2: Expression, roles, and potential as a novel molecular target for colorectal cancer[J]. Pathology Research International, 2012, 2012:574768.
[42]余建杰, 张德奎. 神经生长因子及其受体在溃疡性结肠炎中的作用研究进展[J]. 中国全科医学, 2012, 15(33):3809-3812.
[43]雷雲, 杨铃, 张涛. 神经生长因子对结直肠癌发展和不良预后的影响[J]. 成都医学院学报, 2021, 16(2):148-152.
[44]Avraam J, Wu Y, Richerson G B. Perinatal nicotine reduces chemosensitivity of medullary 5-HT neurons after maturation in culture[J]. Neuroscience, 2020, 446:80-93.
(责任编辑:廖乾生)
收稿日期: 2023-02-22网络出版日期:2023-05-05
基金项目: 国家自然科学基金项目(61772027)
作者简介: 俞秋奕(1998-),女,江苏无锡人,硕士研究生,主要从事生物信息学方面的研究。
通信作者: 贺平安,E-mail:pinganhe@zstu.edu.cn

