调控玉米株高和穗位高候选基因Zmdle1的定位
周文期,张贺通,何海军,龚佃明,杨彦忠,刘忠祥,李永生,王晓娟,连晓荣,周玉乾,邱法展
调控玉米株高和穗位高候选基因的定位
周文期1,张贺通2,何海军1,龚佃明2,杨彦忠1,刘忠祥1,李永生1,王晓娟1,连晓荣1,周玉乾1,邱法展2
1甘肃省农业科学院作物研究所,兰州 730070;2华中农业大学作物遗传改良全国重点实验室,武汉 430070
【目的】株高是玉米株型育种的重要目标性状之一,不仅与玉米籽粒的机械化收获及抗倒伏相关,也与玉米产量密切相关。因此,挖掘玉米株高QTL/基因并解析其功能具有重要的理论和育种价值,定位一个新的玉米矮秆基因,阐明其生物学功能,为加速改良玉米的株型提供重要的理论依据和基因资源。【方法】利用化学诱变剂甲基磺酸乙酯(EMS)诱变甘肃省农业科学院作物研究所自育玉米骨干自交系LY8405,M2后代分离获得一个单基因调控隐性遗传的玉米矮秆低穗位突变体,M3、M4后代能稳定遗传,命名为(),通过与Mo17杂交构建F2分离群体,借助极端性状混池测序分析法(BSA-seq)及目标区段重组交换鉴定的方法,基于Mo17参考基因组对目标区段内的基因进行挖掘和功能注释,定位候选基因。【结果】开展了表型鉴定,突变体苗期表型与对照LY8405无显著性差异,成熟期植株株高和穗位高较LY8405分别降低87.2和55.4 cm,为35.0%和62.9%,差异极显著。细胞形态学观察表明,穗下节间数减少及节间细胞长度缩短是导致的株高和穗位高显著降低的主要原因。利用F2﹕3遗传群体,进行突变基因的遗传分析,后代野生型和突变体植株分离比例符合3﹕1(χ2=2.854),说明突变基因为细胞核遗传的单隐性基因。因此,根据BSA-seq结果,将候选基因初步定位在玉米第1染色体Bin1.09-1.10区段约15 Mb区间内,进一步利用Mo17和重测序结果开发多态性分子标记,通过图位克隆手段精细定位目标基因,最终将候选基因定位到约600 kb大小区间,该区间内有16个候选基因。比对重测序数据,发现在第2 062位置碱基G变成A,导致氨基酸由甘氨酸变成丝氨酸,且转录水平表达比LY8405极显著降低,第223位置碱基由T变成C,导致第75位氨基酸由丝氨酸变成脯氨酸,转录水平无显著差异,通过对该候选区段群体关联分析及功能注释发现,和均与玉米生长发育相关。【结论】定位到第1染色体末端Bin1.09区段候选基因,有效调控玉米株高和穗位高,精细定位缩小目标区段至600 kb,区间内与株高显著关联。
玉米;株高;穗位高;BSA-seq;基因定位;关联分析
0 引言
【研究意义】玉米(L.)是集粮、经、饲于一体的三元作物,是国内外种植面积最大的农作物之一,玉米生产对维护粮食安全、促进畜牧业发展、满足工业原料需求等发挥举足轻重的作用,对提高人们生活水平和国民经济收入具有非常重要的意义[1-2]。玉米株高(plant height,PH)和穗位高(ear height,EH)是影响产量和对某些非生物胁迫耐受性的重要农艺性状,因此,了解PH和EH的遗传控制对阐明玉米生长发育的调控具有重要意义。【前人研究进展】PH和EH是与植物光合效率、种植密度、抗倒性和产量密切相关的重要农艺性状,纵观农业发展史,粮食作物整体株高的降低对世界农业产生了深远的影响[3]。从1960年以来,小麦和水稻半矮秆品种的大面积种植和推广,以及后来“绿色革命”基因()和()的发掘,促进了小麦和水稻产量迅速增加,其中,参与了GA合成通路中GA20氧化酶()的生物合成[4-5],而作为GA信号通路上的抑制因子,编码GA信号转导途径的关键调控元件DELLA蛋白[6],二者的发现有效地将高秆变为矮秆和半矮秆。然而,在玉米中这种能有效降低株高且对产量性状没有负效应的半矮化基因已有应用,但基因资源相对匮乏,亟待更多优异等位基因的挖掘和利用。玉米育种的理想株型包括株型、叶型、穗型和生育期等,理想的株型能充分利用太阳能,最大程度提高玉米产量,现阶段提高玉米种植密度已经成为产量增加的最有效途径之一,研究表明,PH与EH是直接影响玉米种植密度和产量的2个关键因子,并且还影响到玉米的抗倒伏性和收获指数[7]。PH是由节间数目和节间长度构成,因此,矮秆玉米主要通过减少节间数目和缩短节间长度来实现[8]。2003年,Multani等[9]利用转座子标签法克隆了玉米矮秆基因(),编码一个调控玉米茎秆中生长素极性运输的糖蛋白,由于糖蛋白的缺失,导致玉米突变体穗下节间显著缩短,PH和EH极显著降低,茎秆变粗,雄穗和雌穗及叶片无显著差异,因此,优良的综合性状使在矮秆育种过程中发挥了重要作用,其等位基因在生产上被广泛应用[10]。前人研究已经克隆了一些控制玉米PH的基因,也对玉米矮秆相关的突变体表型、控制PH和EH遗传机制进行了阐述[11-12]。如,已经公布的第1染色体有影响生长素极性运输的[9-10];与油菜素内酯合成酶基因()[13]、植物G蛋白基因()[14]、赤霉素运输相关DELLA蛋白基因()[15]、()[16]、玉米中叶绿体基因()[17]、株高降低基因()[18]和转录因子基因()[19];第2染色体有()[20]和矮化基因[21];第3染色体的()[22]、()[23]、()[24]、[25];第5染色体的矮小植株()[26]、[27]、()[28]、[29];第6染色体的()[30]、()[31];第8染色体有通过生长素合成途径参与玉米株高调控的基因()[32]、微管运动相关基因()[33]、BR信号转导相关基因()[34]、第9染色体有与赤霉素合成相关的[35-36]、与生长素转运相关的()[37];第10染色体有与激酶调控相关的[38]等。以上基因大多数与激素合成、代谢和信号转导相关,比如通过赤霉素合成途径调控玉米PH和EH的基因有、、和等[17, 22, 36, 39],通过赤霉素信号转导途径参与调控的有和[29];通过生长素合成途径参与调控的基因有[32],通过生长素极性运输调控的基因有、和等[9, 26, 37]。通过油菜素内酯生物合成途径和信号转导途径分别参与玉米株高调控的相关基因有、、等[13, 24, 27]和[34]。在玉米育种中,PH和EH是影响玉米株型进而影响产量的重要农艺性状之一,矮秆玉米株型紧凑,穗位适中,更适合高密度种植,它能使作物具有较强的抗倒伏能力和较高的光能利用率。【本研究切入点】虽然前人对控制玉米株高的QTL及基因进行了大量挖掘和克隆,但大多数突变体因过度矮化或不具备实用性的农艺性状,对玉米产量构成因素产生不良效果,育种利用价值较小,因此,挖掘具有理想农艺性状的矮秆资源,克隆能降低玉米PH和EH,且对产量无负效应的关键基因,对选育耐密植、宜机收新品种具有应用价值。【拟解决的关键问题】本研究拟克隆玉米候选基因,解析调控玉米株高的遗传机理并评估其育种价值,为加速矮化株型改良提供理论依据。
1 材料与方法
1.1 试验材料
玉米骨干自交系LY8405(甘肃省农业科学院自育自交系,以美国杂交种先玉335为基础材料,经过连续8代以上的自交选择育成纯系,属于SS类型),花粉经EMS诱变处理(未处理的LY8405为对照),诱变处理参考周文期等[40-41],收获M0代,在甘肃省农业科学院张掖试验场和海南省农业科学院乐东县试验基地,夏季和冬季分别交替种植,M2后代筛选分离到矮秆低穗位核隐性遗传突变株,M3、M4后代能稳定遗传,命名为(),本研究对突变体和突变体进行等位性检测,发现导致矮化的可能是一个不同于的新基因。因此,将与LY8405回交,获得BC2F2代植株,进行候选基因定位。
1.2 BC2F2遗传分离群体的构建
前期研究经过正反交测定,表型是核单基因隐性遗传突变引起。利用与LY8405进行2代回交,纯化突变基因,获得BC2材料,再与不同基因型自交系Mo17杂交,F1代自交,构建BC2F2分离群体,从F2中分离纯合株,用于候选基因的定位。
1.3 候选基因的初步定位
利用二代测序技术,根据BSA极端性状混池测序原理,提取亲本LY8405、和Mo17亲本各3株,选择Mo17×2子代池株高和穗位最高的植株30株,株高和穗位最低的植株30株,混池提取其RNA,建库进行转录组测序,DNA水平的全基因组测序提取了PH和EH极高和极低植株各100株。在分离群体搭建过程中,子代会根据表型进行选择,筛选出突变型子代池和野生型子代池,根据遗传连锁交换定律,子代池的基因型会和表型产生共分离,反映在物理图谱层面,与表型连锁的染色体区段会和不连锁的染色体区间产生稳定的SNP-index差异。差异大小范围为0—1。若该参数为0,代表子代所有测到的Reads都来自野生亲本;若该参数为1,代表子代所有Reads都来自突变亲本;该参数为0.5,则代表此子代混池中SNP来自2个亲本基因组的频率一致[42-43]。
1.4 基因型的鉴定及候选基因的精细定位
利用图位克隆和全基因组高通量测序技术相结合,确定候选基因。在玉米抽穗期,用取样器进行取样,叶片面积约1 cm2,采用CTAB法提取叶片DNA。采用NanoDro2000检测提取的DNA质量和浓度。将提取的DNA稀释到合适浓度进行PCR扩增,引物序列见表1。扩增的PCR产物用全自动毛细管电泳系统(大片段分析仪)或者聚丙烯酰胺凝胶电泳进行基因型分型。SSR、InDel标记根据差异带型分别标记为A、B、H,与LY8405带型一致的记为A,与Mo17带型一致的记为B,杂合带型记为H,缺失带型记为—,N表示样品数。根据以下公式计算突变基因重组交换率,交换率越低,说明分子标记距离候选基因位置越近。
交换率=[(2×B+1×H+0×A)/N×2]×100%。

表1 本研究使用的部分引物序列
2 结果
2.1 Zmdle1与LY8405苗期表型无显著差异
在LY8405诱变材料中,M2代分离到1个株型矮化、穗位高度极显著降低突变株,M2分离比例符合3﹕1(野生型﹕突变株),M3和M4矮秆低穗位性状纯合且稳定遗传,该突变体苗期表型与对照LY8405无明显差异(图1-A—F),浸种发芽后3 d,LY8405平均株高6.9 cm,根长6.0 cm,平均株高6.8 cm,根长6.1 cm,无差异。统计第7、14、21、28和35天生长的LY8405和茎叶长度,均无显著差异(图1-G)。田间种植表型观察,植株第六片叶展开,第七片叶露出叶心时期逐渐表现出株高差异,后期越来越明显,成熟期植株PH和EH较对照LY8405分别降低了87.2和55.4 cm,为35.0%和62.9%,差异极显著(<0.01),将其命名为矮秆低穗位突变体(,,表2和图2)。

A:萌发后3 d的植株,株高及根长无显著差异,标尺=5 cm;B—F:分别表示种子萌发后生长7、14、21、28和35 d的植株表型;G:苗期株高,无显著差异。标尺=10 cm
2.2 Zmdle1与LY8405成熟期PH和EH的差异性
野生型LY8405成熟期株高平均为248.9 cm,穗位高为88.1 cm;株高为161.7 cm,与LY8405相比,平均降低了87.2 cm,约下降35.0%,穗位高仅为32.7 cm,平均降低了55.4 cm,约下降62.9%,差异极显著(<0.01)(表2、图2-A—B和图2-D);分别与Mo17和B73杂交,F2后代分离到表型稳定的(图2-C)。对和LY8405植株节间数及节间长度进行统计分析,发现LY8405和总节间数相同,都有15个节间(部分共14节),但是LY8405穗上共8节,穗下7节,而穗上9节或8节,穗下6节,穗位降低一个节间,且穗位节、穗上3节和穗下3节长度均极显著减小(图2-E,穗上节橙色标注极显著性差异,穗下节蓝色标注极显著性差异),说明穗下节间数减少及节间长度缩短是导致的PH和EH显著降低的主要原因。
为了更直观地看到PH和EH降低的原因,将整株植株进行节间剖析,发现穗位节第7节降低,与LY8405相比,穗位节节下部分降低最为显著(图3-A,1—7节),穗上节1—2节差异较为显著,其他节间长度差异不明显;随后利用不同生育期的植株切片观察,统计每个节间的细胞形态及数目,数据显示节间细胞总数目并未减少,茎节细胞长度变短导致节间长度缩短,从而降低PH和EH(图3-C—E)。另外,株型矮小,与LY8405相比,其根系显著变小,须根没有对照发达(图3-B)。

A、B:Zmdle1突变体比LY8405株型矮小,穗位更低;C:LY8405×Mo17,F2分离出Zmdle1纯合表型,矮秆低穗位;D:PH和EH统计;E:LY8405和Zmdle1节间数及每一节的长度,共15节,穗位节及穗上三节(橙色)和穗下三节(蓝色)均差异极显著。标尺=30 cm。**表示差异极显著(P<0.01)
2.3 Zmdle1和LY8405其他农艺性状调查对比
LY8405与雌雄穗发育、授粉结实均正常,说明PH和EH降低的同时,未影响果穗的正常结实(图4),穗长及穗粗没有显著变化(=0.16),秃尖稍长(图4-A)。统计测量了雌穗穗长、穗粗、轴粗、穗行数和行粒数,除轴粗有显著差异外,其他农艺性状均无显著性变化(表2),但是粒型性状发生了明显变异,相比LY8405,粒长变短,粒宽变宽,差异极显著(<0.01)(表2和图4-G—H);而千粒重平均为300 g,无显著差异(表2、图4-C和图4-E)。与LY8405总叶片数目相同,均为20—21片。与LY8405相比,倒3叶变长、变窄,差异显著(<0.01)(表2和图4-B);雄穗穗长无变化,但雄穗分枝数较LY8405减少(表2和图4-F),开花期提前5—7 d。上述农艺性状的调查结果表明,除PH和EH降低,其他农艺性状如生育期、籽粒及根等也发生了一些改变,但是该突变体区别其他已报道的株高极度矮化突变体,其单株产量及千粒重并没有下降,是一个新类型的株高突变体。
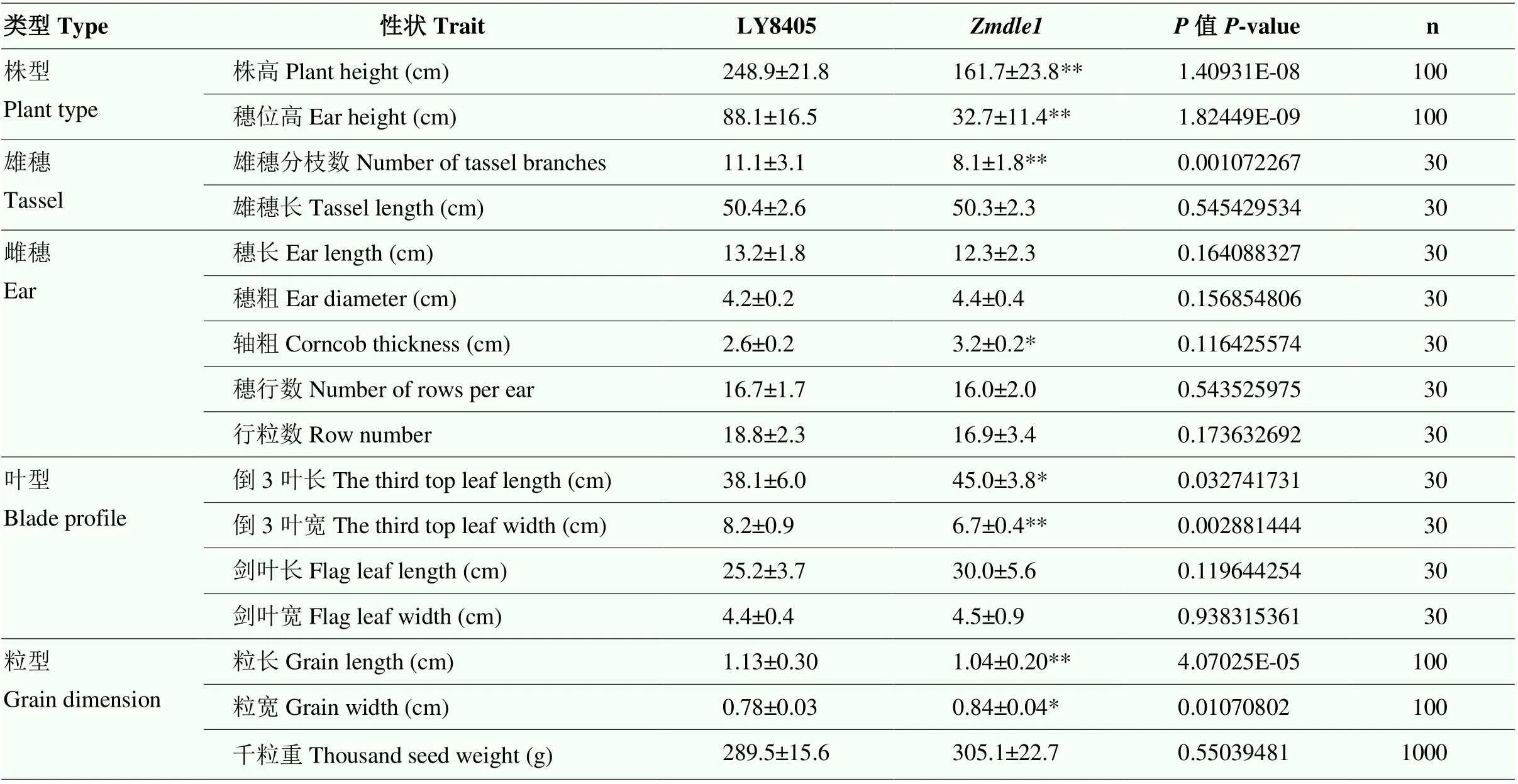
表2 突变体Zmdle1和对照LY8405的农艺性状数据分析
*表示差异显著(<0.05),**表示差异极显著(<0.01)。下同
* mean significant difference with the control (<0.05), ** mean very significant difference (<0.01). The same as below
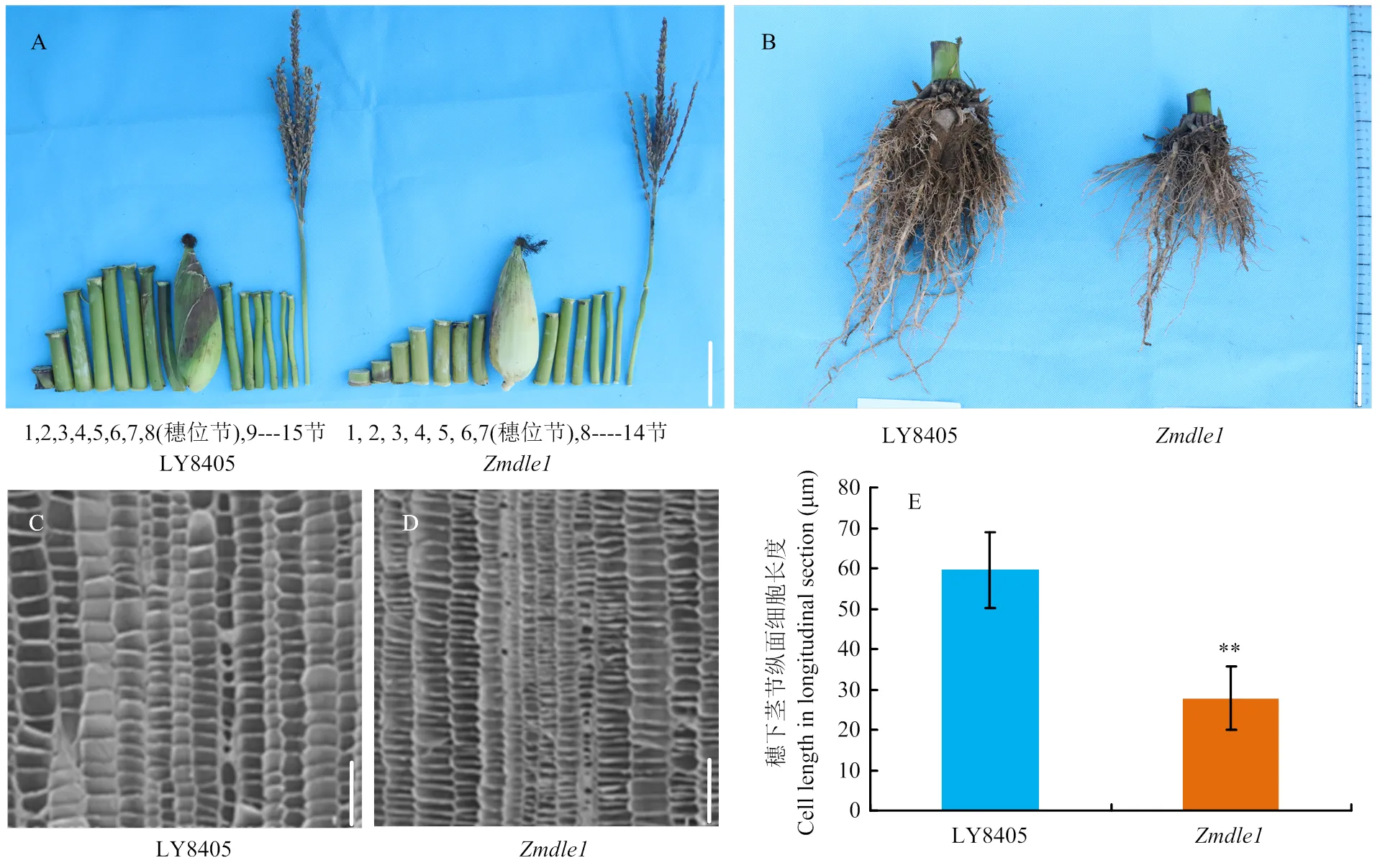
A:Zmdle1与LY8405每节节间图,Zmdle1穗下节及穗上2节均较LY8405短;B:Zmdle1的根较LY8405变小;C、D:LY8405与Zmdle1穗下茎细胞的电子扫描形态图;E:穗下茎节纵面细胞长度。A、B标尺=5 cm;C、D标尺=200 μm

A:Zmdle1与LY8405果穗,结实良好;B:从左往右,分别是剑叶、倒2叶和倒3叶,Zmdle1倒3叶叶片变长,变窄;C、E:穗部和籽粒性状;D:Zmdle1与LY8405幼穗发育正常,无差异;F:LY8405和Zmdle1雄穗发育正常,Zmdle1分枝数明显减少2—4个,花期提前一周;G、H:Zmdle1籽粒粒长较LY8405变短,粒宽变宽,n=20。A、B、D—H:标尺=5 cm;C:标尺=2 cm
2.4 Zmdle1遗传群体构建及分离规律分析
将分别与Mo17和B73杂交,构建了两套F2遗传群体,对F2后代分别进行遗传统计分析,发现×Mo17 F2群体中,野生型和突变体植株分离比例符合3﹕1(χ2=2.854)(表3),说明突变基因为细胞核遗传的单隐性基因。但是×B73 F2群体中分离到纯合表型植株明显减少,分析其原因是LY8405自交系中含有20%—30%的B73基因型,突变体与B73杂交,后代亲缘关系较近,基因型未能发生充分重组交换及分离,因此,本研究利用×Mo17杂交F2:3遗传群体,进行候选基因精细定位。

表3 F2群体中正常植株和矮秆植株的分离比检测
2.5 转录组测序及候选基因初定位
利用BSA及BSR极端分离群体混池测序的原理,对F2后代PH和EH极高和极矮2种极端性状植株,分别选取30株,取倒3叶叶片(叶片变长变窄的表型与株高表型连锁,且有差异显著性),等量混合提取RNA,2个亲本(Mo17、)各取3株,设置3个重复,进行了亲本及子代的转录组测序。通过Trimmomatic软件对12个样本的原始数据进行过滤截短,去除低质量碱基得到用于后续分析的过滤后数据,并且使用一种基于java的软件fastqc进行数据质控分析。对于每个SNP,使用经验贝叶斯方法来估计SNP标记与突变体库中的基因之间没有重组的条件概率,给定SNP等位基因的特异性计数。而为了获得最有可能与给定基因相关的区域,通过用固定数量的SNP(N=50)和步长为5个SNP的滑动窗口扫描整个基因组。结果表明,主要在第1染色体末端和第3染色体中部有显著效应位点,散点位置ΔSNP-index值大于0.5,说明候选基因可能位于该2个区间的一个之内,推测控制PH和EH的主效基因可能位于第1染色体末端Bin1.08—1.10区间,或第3染色体Bin3.07—3.08位置(图5)。
2.6 Zmdle1基因组重测序及候选基因连锁定位分析
分析转录组测序数据,散点不集中,定位区间不理想,因此,后续研究扩大分离群体样本,种植约4 000株,再次采集了100株F2后代PH和EH极高、极低2个极端植株的倒3叶叶片,混池提取DNA,亲本取样3株,3个重复,进行了全基因组高通量测序,定位候选基因,分析数据时,选取ΔSNP-index定位,在超出置信线的染色体区域内筛选候选基因,定位到2个区间,即第1染色体末端位置(图6,黑色方框标出)和第5染色体,置信区间更高。因此,结合RNA转录组和DNA的高通量测序数据,第1染色体末端区间在2次测序结果中均有定位,且物理位置完全吻合,确定为候选基因区段。通过对第1染色体Bin1.07、Bin1.08、Bin1.09及Bin1.10位置多态性分子标记筛选,约65对引物可利用LY8405和Mo17亲本区别的多态性标记,其中BKN2-8和umc2510标记位置交换频率约为15%(表4),因此,将候选基因区间初步定位在Bin1.09、BKN2-8和umc2510标记间,约为6.5 Mb。

表4 初步定位标记与表型的交换频率
2.7 候选基因ZmDLE1精细定位
以BC2F2分离群体为材料,结合转录组测序及重测序初步定位结果,将第1染色体末端区间的多态性分子标记用于F2分离群体的基因型验证和分析,确保了该位点的准确性,定位到1 Mb区间。进一步开发并加密新的分子标记,利用BSA测序结果对LY8405、和Mo17及其子代DNA测序的大数据,设计和筛选有较多碱基差异(>10 bp)的多态性分子标记(表1),利用4 000株F2纯合表型单株,将目标基因精细定位在InDel-25和InDel-26之间约600 kb(255.26—255.86 Mb)的区间内(图7),区间内有16个候选基因。利用重测序数据分析,以及功能注释和关联群体的SNP关联分析,结果表明,的cDNA全长为2 145 bp,在第2 062位置碱基G变成A,导致第688位置的氨基酸由甘氨酸(G)变成丝氨酸(S),且转录水平检测基因表达量比LY8405极显著降低(表4),的cDNA全长共1 416个碱基,第223位置碱基由T变成C,导致第75位氨基酸由丝氨酸(S)变成脯氨酸(P),转录水平无显著差异(表5)。提取513份关联群体中定位区段内的多态性数据(SNP和Indel标记)和株高表型数据,使用Tassel 5软件的GLM方法进行候选区段关联分析。结果显示,存在于株高显著关联的SNP位点(图8)。综上,确定主要候选基因是和,编码扩展蛋白Expansin-4蛋白,编码生长素相关PINOID蛋白激酶,均可能通过植物细胞扩张、激素调节等影响植物的生长发育,确定为重点基因,下一步将进行测序验证、生物信息学分析和功能解析,确定功能基因。

黑色方框标出可能存在的候选基因区间Black boxes indicate possible candidate gene intervals
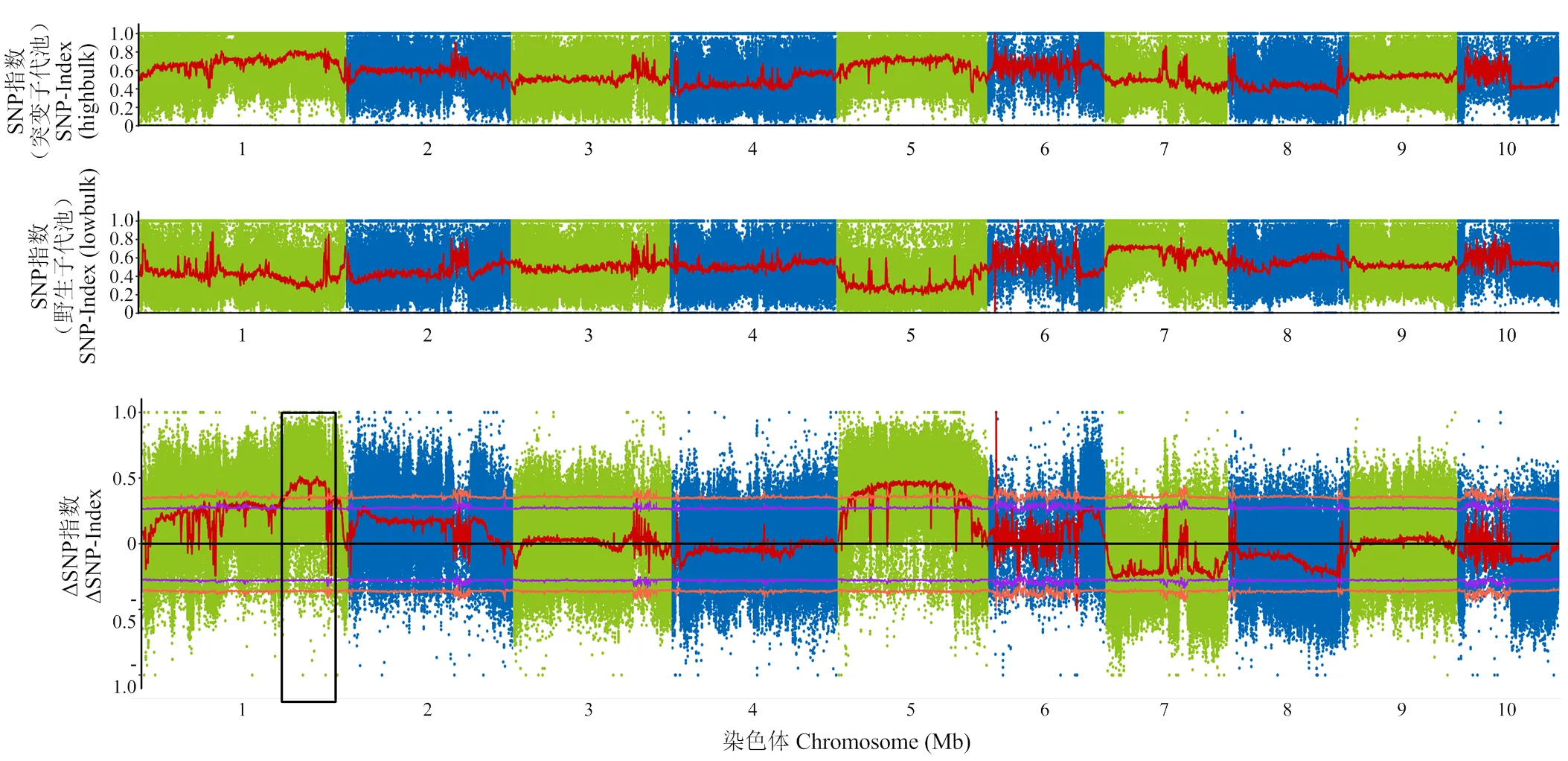
ΔSNP-index为该群体的连锁定位图。黑色方框标出可能存在的候选基因区间。绿色点和蓝色点为SNP-index或ΔSNP-index;红色线为SNP-index或ΔSNP-index划窗后的拟合线;紫色线为95%置信线;橙黄色线为99%置信线;SNP-index (highbulk)为突变子代的连锁定位图;SNP-index(lowbulk)为野生子代的连锁定位图

图7 ZmDLE1候选基因的精细定位图

表5 区段内候选基因

株高性状关联位点基因是Zm00001d033231 The gene associated with plant height trait was Zm00001d033231
3 讨论
3.1 Zmdle1株高和穗位降低主要原因是穗下节间数目的减少和茎节间细胞缩短
矮秆基因是导致株高矮化的直接原因,表现在作物细胞学和形态学的变化。从形态上观察,植株矮化直接表现在节间数目的减少或者节间长度的变短,或两者同时变少,导致株高降低;从细胞水平观察,株高矮化作物茎节间单位面积内细胞数目减少以及单个细胞的纵向平均长度减小,或两者同时变小,均会导致玉米植株的矮化[8]。因此,通过改变总节间数量和单个节间长度可产生株高差异,而独立的茎节长度又与茎秆细胞的纵向长度和单位面积的细胞数目密切相关[44-45]。Tang等[44]研究说明玉米株高的变化主要因为平均茎秆节间长度的改变。Salvi等[45]在对玉米穗上部、穗下部节间数目的研究分析发现,穗下节间数的差异与PH显著相关,茎秆节间数目的变化导致了PH的升高或降低。Lv等[46]研究发现玉米突变体茎秆纵切面单个茎节细胞大小没有显著差异,但突变体茎秆节间细胞排列紧凑,数目减少,导致PH明显降低。以上研究结论同本文的研究结果一致。本研究中,与LY8405相比,穗下节间数减少,且细胞学观察表明,与LY8405纵向细胞数目不变,细胞长度显著变短,可能是突变体中细胞伸长受到了抑制。茎的伸长过程受赤霉素、生长素和油菜素内酯等植物激素的调控,生长素合成受阻或信号传导中断最显著的表现就是植株矮化。同时,根细胞的伸长可能受到不同程度抑制,与LY8405相比,突变体的根系变小,根系发达程度降低,可能会存在潜在倒伏风险,但因为PH和EH极显著降低,尤其是平均穗位高仅有33 cm,使整个植株重心下移,不同生态环境轮替田间种植了6年,未发生倒伏现象,与其他自交系组配的杂交种,田间种植3年以上,也未发生倒伏。
3.2 植物激素调控玉米株高相关基因研究进展
目前研究表明调控玉米株高涉及到的植物激素主要有赤霉素(gibberellin,GA)、油菜素内酯(brassinolide,BR)及生长素(Auxin)[47]。GA是一类广泛存在的植物激素,其化学结构属于四环二萜类化合物,对植物生长发育、开花结果、发育及成熟等都有作用,尤其对于茎秆伸长发挥关键作用。鉴别获得如、、、、、等多个突变体与GA合成相关,此类突变体中活性赤霉素含量大幅减少,植株表现出矮化现象,如是一个与赤霉素合成相关的基因,阻止GA20向GA1的转化,编码细胞色素P450类蛋白,参与GA合成早期阶段GA12向GA53的转化,从而影响玉米株高[3, 22, 35]。AN1参与GA生物合成途径中贝壳杉烯的合成,影响赤霉素的合成途径,最终抑制茎间细胞的伸长,导致玉米植株矮化[17]。DELLA蛋白是GA信号通路的重要负调控因子,可阻断GA诱导的基因表达,玉米中与“绿色革命”基因高度同源的和基因显性突变均可引起玉米株高显著降低[29, 48]。BR是一类多羟基的类固醇植物激素,也是促进植物节间伸长的内源激素之一,调控植株高矮。突变体和都涉及到BR合成途径,突变体严重矮化,节间距很短,茎节不伸长,没有成熟的花粉,根数减少,编码BR合成途径中的C-6氧化酶[13]。()突变体中株高降低,节数不变,株高的降低是由于节间长的缩短导致的,叶片长度为野生型的60%,是拟南芥的同源突变,发生了终止突变,导致油菜素内酯合成受阻[24]。与拟南芥同源,突变体极端矮化,雄穗结粒雌穗小花等表型,在还原24-methyhloesterol的C-24过程中存在缺陷[27]。Auxin合成、分布、代谢及其极性运输和不平均分布影响植物的生长发育,可促进细胞扩增及伸长,从而影响到植物的茎秆伸长。玉米短轴突变体,节间显著缩短,尤其果穗下部节间位点缩短导致PH和EH极显著降低,参与调控生长素极性运输,调节玉米茎的生长发育[9]。Li等[37]发现表达产生PIN- FORMED(PIN)蛋白影响IAA的极性运输,将IAA从植株茎秆中运输到根部,促使玉米侧根增加,抑制地上组织茎的节间伸长,过表达会降低株高、节间长和穗位高。而本研究的突变体具有典型的生长素调控基因类型的表型,突变体株高矮化,穗位高降低显著。
3.3 关键候选基因功能预测和同源基因分析
本研究将控制株高基因初步确定为候选基因为和,(Chr.1:255266246-255275500)编码Expansin-B4,同源性分析发现,该基因在拟南芥中的同源基因是,该蛋白通过调控细胞壁的松弛程度参与植物激素调节、细胞扩张等植物生长过程[49]。前人研究发现扩展蛋白(Expansins)是一类细胞壁蛋白,Expansin蛋白可分为4个亚家族-expansin A(EXPA)、EXPB、expansin-like A(EXLA)和EXLB,能够通过打断细胞壁多聚物之间的氢键诱导酸依赖的细胞壁延展和压力松弛[50-51]。在植物细胞的伸展及一系列涉及细胞壁修饰的生命活动中,Expansin蛋白起着关键作用。例如在水稻中,细胞壁松弛蛋白OsEXPA4的表达对茎秆节间的伸长起着关键作用,在植物组织伸长区特异性表达,过量表达或者抑制其表达都会影响组织伸长区的变化进而造成节间长度差异,过表达通过增加细胞大小来刺激植物生长,而敲除该基因则减缓植物生长[52]。此外,至少6个EXPANSIN蛋白基因在负责节间伸长的分生区和伸长区之间表达,分别是、、、、和这些扩展蛋白基因的表达都会造成水稻的节间差异[49, 53]。本研究电镜观察发现茎节细胞长度缩短,与细胞壁松弛蛋白作用机理非常相似。
(Chr.1:255293638-255295053)编码PINOID蛋白激酶,在拟南芥中的同源基因是,编码一种蛋白丝氨酸/苏氨酸激酶,该激酶可能作为细胞生长素外排的正调节因子。生长素输出载体PIN是一类膜蛋白,和生长素极性运输相关,在拟南芥中,pin-formed()和p-glycoprotein()突变体,因为体内生长素极性运输受阻,表现为植株矮化[54-55]。水稻与拟南芥丝氨酸苏氨酸蛋白激酶PID同源,可能参与控制水稻生长素极性转运,通过分析在地上部分生组织的分布,说明该基因可能在形态建成和器官发生的过程中起重要作用[56],OsPID通过磷酸化OsPIN1a和OsPIN1b而调控生长素的运输,改变生长素的极性分布,调控水稻花器官的形成和发育,OsPID与LAX1/LAX2互作,通过控制水稻的分枝和分蘖等性状[57]。介导生长素从地上部向根茎结合部转运,调控分蘖角度和株高性状[58]。前人研究表明玉米和通过生长素极性运输参与调控玉米PH和EH发育,株高显著降低。Xing等[10]研究表明QTL的SNP变异使RIL88()的节间纵向细胞数目和株高降低了20%,并且比已知的突变体表型更弱,对产量影响不显著。与表型非常相似,其节间缩短,细胞长度变短,基因功能缺陷引起生长素运输不足,影响细胞壁各组分的生物合成、物质运输和代谢等,突变型和野生型植株在发育早期表型无显著差异[9, 26]。ZmPIN1a蛋白在调节生长素时空不对称分布中起重要作用,影响植物多个器官的生长发育,在玉米中过表达可以降低节间长度、PH和EH[37]。综上,PINOID蛋白激酶功能保守,通过调控生长素运输控制植株形态建成和花的发育,可能调控玉米PH和EH发育[59]。候选基因及功能仍需进一步验证,更深入研究包括候选基因表达模式分析、亚细胞定位、遗传互补试验、超表达及CRISPR/Cas 9载体构建及遗传转化,解析候选基因的生物学功能及调控株高的遗传机理。
4 结论
确定了2个调控玉米株高和穗位高的关键候选基因和,将为玉米株高性状的遗传改良提供重要的基础材料和基因选择位点。
[1] 周文期, 周玉乾, 刘忠祥, 连晓荣, 王晓娟, 何海军, 杨彦忠, 寇思荣.12C6+重离子束辐照玉米后代的生物学效应. 核农学报, 2019, 33(12): 2311-2318.
ZHOU W Q, ZHOU Y Q, LIU Z X, LIAN X R, WANG X J, HE H J, YANG Y Z, KOU S R. Biological effects of offspring of maize irradiated by12C6+heavy ion beam. Journal of Nuclear Agricultural Sciences, 2019, 33(12): 2311-2318. (in Chinese)
[2] 周文期, 王晓娟, 寇思荣, 李艳春, 何海军. 玉米叶夹角形成的分子调控机理研究. 土壤与作物, 2019, 8(3): 339-348.
ZHOU W Q, WANG X J, KOU S R, LI Y C, HE H J. Molecular regulation mechanism of leaf angle formation in maize. Soils and Crops, 2019, 8(3): 339-348. (in Chinese)
[3] TENG F, ZHAI L H, LIU R X, BAI W, WANG L Q, HUO D A, TAO Y S, ZHENG Y L, ZHANG Z X. ZmGA3ox2, a candidate gene for a major QTL, qPH3.1, for plant height in maize. The Plant Journal, 2013, 73(3): 405-416.
[4] SASAKI A, ASHIKARI M, UEGUCHI-TANAKA M, ITOH H, NISHIMURA A, SWAPAN D, ISHIYAMA K, SAITO T, KOBAYASHI M, KHUSH G S, KITANO H, MATSUOKA M. Green revolution: a mutant gibberellin-synthesis gene in rice. Nature, 2002, 416(6882): 701-702.
[5] SPIELMEYER W, ELLIS M H, CHANDLER P M.(), “green revolution” rice, contains a defective gibberellin 20-oxidase gene. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(13):9043-9048.
[6] PENG J, RICHARDS D E, HARTLEY N M, MURPHY G P, DEVOS K M, FLINTHAM J E, BEALES J, FISH L J, WORLAND A J, PELICA F, SUDHAKAR D, CHRISTOU P, SNAPE J W, GALE M D, HARBERD N P. “Green revolution” genes encode mutant gibberellin response modulators. Nature, 1999, 400(6741): 256-261.
[7] PEIFFER J A, ROMAY M C, GORE M A, FLINT-GARCIA S A, ZHANG Z W, MILLARD M J, GARDNER C A C, MCMULLEN M D, HOLLAND J B, BRADBURY P J, BUCKLER, E S. The genetic architecture of maize height. Genetics, 2014, 196(4):1337-1356.
[8] CUI F, LI J, DING A W, ZHAO C H, WANG L, WANG X Q, LI S S, BAO Y G, LI X F, FENG D S, KONG L R, WANG H G. Conditional QTL mapping for plant height with respect to the length of the spike and internode in two mapping populations of wheat. Theoretical and Applied Genetics, 2011, 122(8): 1517-1536.
[9] MULTANI D S, BRIGGS S P, CHAMBERLIN M A, BLAKESLEE J J, MURPHY A S, JOHAL G S. Loss of an MDR transporter in compact stalks of maizeand sorghummutants. Science, 2003, 302(5642): 81-84.
[10] XING A Q, GAO Y F, YE L F, ZHANG W P, CAI L C, CHING A, LLACA V, JOHNSON B, LIU L, YANG X H, KANG D M, YAN J B, LI J S. A rare SNP mutation in Brachytic2 moderately reduces plant height and increases yield potential in maize. Journal of Experimental Botany, 2015, 66(13): 3791-3802.
[11] 周文期, 连晓荣, 刘忠祥, 周玉乾,赵小强. 玉米株高和穗位高的调控机理研究. 分子植物育种, 2021, 19(23): 7965-7976.
ZHOU W Q, LIAN X R, LIU Z X, ZHOU Y Q, ZHAO X Q. Research progress on the mechanism of controlling maize plant height and ear height. Molecular Plant Breeding, 2021, 19(23): 7965-7976. (in Chinese)
[12] 马雅杰, 高悦欣, 李依萍, 龙艳, 董振营, 万向元. 玉米株高和穗位高的遗传基础与分子机制. 中国生物工程杂志, 2021, 41(12): 61-73.
MA Y J, GAO Y X, LI Y P, LONG Y, DONG Z Y, WAN X Y. Progress on genetic analysis and molecular dissection on maize plant height and ear height. China Biotechnology, 2021, 41(12): 61-73. (in Chinese)
[13] Makarevitch I, Thompson A, Muehlbauer G J, Springer N M.gene in maize encodes a brassinosteroid C-6 oxidase. PLoS ONE, 2012, 7(1): e30798.
[14] Bommert P, Je B I, Goldshmidt A, Jackson D. The maize Gα gene COMPACT PLANT2 functions in CLAVATA signalling to control shoot meristem size. Nature, 2013, 502(7472): 555-558.
[15] Thornsberry J M, Goodman M M, Doebley J,Kresovich S, Nielsen D, Buckler E S. Dwarf8 polymorphisms associate with variation in flowering time. Nature Genetics, 2001, 28(3): 286-289.
[16] Stein O L. Rates of leaf initiation in two mutants of,and. American Journal of Botany, 1955, 42(10): 885-892.
[17] Bensen R J, Johal G S, Crane V C, Tossberg J T, Schnable P S, Meeley R B, Briggs S P. Cloning and characterization of the maizegene. The Plant Cell, 1995, 7(1): 75-84.
[18] Li W, Ge F H, Qiang Z Q, Zhu L, Zhang S S, Chen L M, Wang X Q, Li J S, Fu Y. Maize ZmRPH1 encodes a microtubule- associated protein that controls plant and ear height. Plant Biotechnology Journal, 2020, 18(6): 1345-1347.
[19] Bolduc N, Hake S. The maize transcription factor KNOTTED1 directly regulates the gibberellin catabolism gene ga2ox1. The Plant Cell, 2009, 21(6): 1647-1658.
[20] Bajguz A, Tretyn A. The chemical characteristic and distribution of brassinosteroids in plants. Phytochemistry, 2003, 62(7): 1027-1046.
[21] Katsumi M, Phinney B O, Jefferies P R, Henrick C A. Growth response of theandmutants of maize to some kaurene derivatives. Science, 1964, 144(3620): 849-850.
[22] Chen Y, Hou M M, Liu L J, Wu S, Shen Y, Ishiyama K, Kobayashi M, McCarty D R, Tan B C. The maize DWARF1 encodes a gibberellin 3-oxidase and is dual localized to the nucleus and cytosol. Plant Physiology, 2014, 166(4): 2028-2039.
[23] Guan J C, Koch K E, Suzuki M, Wu S, Latshaw S, Petruff T, Goulet C, Klee H J, McCarty D R. Diverse roles of strigolactone signaling in maize architecture and the uncoupling of a branching-specific subnetwork. Plant Physiology, 2012, 160(3): 1303-1317.
[24] Hartwig T, Chuck G S, Fujioka S, Klempien A, Weizbauer R, Potluri D P V, Choe S, Johal G S, Schulz B. Brassinosteroid control of sex determination in maize. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(49): 19814-19819.
[25] Fujioka S, Yamane H, Spray C R, Gaskin P, Macmillan J, Phinney B O, Takahashi N. Qualitative and quantitative analyses of gibberellins in vegetative shoots of normal,,,, andseedlings ofL.. Plant Physiology, 1988, 88(4): 1367-1372.
[26] Avila L M, Cerrudo D, Swanton C, Lukens L. Brevis plant1, a putative inositol polyphosphate 5-phosphatase, is required for internode elongation in maize. Journal of Experimental Botany, 2016, 67(5): 1577-1588.
[27] Best N B, Hartwig T, Budka J, Fujioka S, Johal G, Schulz B, Dilkes B P.encodes a maize ortholog of thebrassinosteroid biosynthesis gene DWARF1, identifying developmental interactions between brassinosteroids and gibberellins. Plant Physiology, 2016, 171(4): 2633-2647.
[28] Bommert P, Lunde C N, Nardmann J, Vollbrecht E, Running M, Jackson D, Hake S, Werr W. Thick tasselencodes a putative maize ortholog of theCLAVATA1 leucine-rich repeat receptor-like kinase. Development, 2005, 132(6): 1235-1245.
[29] Lawit S J, Wych H M, Xu D P, Kundu S M, Tomes D T. Maize DELLA proteins dwarf plant8 and dwarf plant9 as modulators of plant development. Plant and Cell Physiology, 2010, 51(11): 1854-1868.
[30] Cleary A L, Smith L G. Thegene is required for spatial control of cytoskeletal arrays associated with cell division during maize leaf development. The Plant Cell, 1998, 10(11): 1875-1888.
[31] Jiang F K, Guo M, Yang F, Duncan K, Jackson D, Rafalski A, Wang S C, Li B L. Mutations in an AP2 transcription factor-like gene affect internode length and leaf shape in maize. PLoS ONE, 2012, 7(5): e37040.
[32] Phillips K A, Skirpan A L, Liu X, Christensen A, Slewinski T L, Hudson C, Barazesh S, Cohen J D, Malcomber S, McSteen P. Vanishing tassel2 encodes a grass-specific tryptophan aminotransferase required for vegetative and reproductive development in maize. The Plant Cell, 2011, 23(2): 550-566.
[33] Lau K H. Co-orthologs of KATANIN1 impact plant morphology and show differential evolution in maize[D]. Purdue University, 2016: 37-38.
[34] Kir G, Ye H, Nelissen H, Neelakandan A K, Kusnandar A S, Luo A, D Inzé D, Sylvester A W, Yin Y H, Becraft P W. RNA interference knockdown of BRASSINOSTEROID INSENSITIVE1 in maize reveals novel functions for brassinosteroid signaling in controlling plant architecture. Plant Physiology, 2015, 169(1):826-839.
[35] Helliwell C A, Chandler P M, Poole A, Dennis E S, Peacock W J. The CYP88A cytochrome P450, ent-kaurenoic acid oxidase, catalyzes three steps of the gibberellin biosynthesis pathway. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(4):2065-2070.
[36] Winkler R G, Helentjaris T. The maizegene encodes a cytochrome P450-mediated early step in Gibberellin biosynthesis. The Plant Cell, 1995, 7(8): 1307-1317.
[37] Li Z X, Zhang X R, Zhao Y J, Li Y J, Zhang G F, Peng Z H, Zhang J R. Enhancing auxin accumulation in maize root tips improves root growth and dwarfs plant height. Plant Biotechnology Journal, 2018, 16(1): 86-99.
[38] Becraft P W, Stinard P S, McCarty D R. CRINKLY4: aTNFR-like receptor kinase involved in maize epidermal differentiation. Science, 1996, 273(5280): 1406-1409.
[39] Harris L J, Saparno A, Johnston A, Prisic S, Xu M, Allard S, Kathiresan A, Ouellet T, Peters R J. The maizegene is induced by Fusarium attack and encodes an ent-copalyl diphosphate synthase. Plant Molecular Biology, 2005, 59(6): 881-894.
[40] 周文期, 寇思荣, 连晓荣, 杨彦忠, 刘忠祥, 王晓娟, 何海军, 周玉乾. 水稻和玉米叶表皮突变体的筛选和鉴定. 植物生理学报, 2020, 56(2): 189-199.
Zhou W Q, Kou S R, Lian X R, YANG Y Z, LIU Z X, WANG X J, HE H J, ZHOU Y Q. Screening and identification of leaf epidermal mutants in rice and maize. Plant Physiology Journal, 2020, 56(2): 189-199. (in Chinese)
[41] 周文期, 连晓荣, 周玉乾, 王兴荣, 杨彦忠, 刘忠祥, 王晓娟, 何海军, 寇思荣. EMS诱变玉米自交系种质创新应用. 玉米科学, 2020, 28(6): 31-38.
Zhou W Q, Lian X R, Zhou Y Q, WANG X R, YANG Y Z, LIU Z X, WANG X J, HE H J, KOU S R. Innovative application of EMS mutagenesis germplasm of maize inbred lines. Journal of Maize Sciences, 2020, 28(6): 31-38. (in Chinese)
[42] Abe A, Kosugi S, Yoshida K, Natsume S, Takagi H, Kanzaki H, Matsumura H, Yoshida K, Mitsuoka C, Tamiru M, Innan H, Cano L, Kamoun S, Terauchi R. Genome sequencing reveals agronomically important loci in rice using MutMap. Nature Biotechnology, 2012, 30(2): 174-178.
[43] 周文期, 刘忠祥, 王晓娟, 何海军, 周玉乾, 杨彦忠, 连晓荣, 李永生. 利用BSA-Seq方法快速定位作物农艺性状QTL/基因概述. 甘肃农业科技, 2022, 53(4): 1-10.
Zhou W Q, Liu Z X, Wang X J, HE H J, ZHOU Y Q, YANG Y Z, LIAN X R, LI Y S. Rapid mapping of QTL/gene for agronomic traits in crops using BSA-Seq method. Gansu Agricultural Science and Technology, 2022, 53(4): 1-10. (in Chinese)
[44] Tang J H, Teng W J, YAN J B, Ma X Q, Meng Y J, Dai J R, Li J S. Genetic dissection of plant height by molecular markers using a population of recombinant inbred lines in maize. Euphytica, 2007, 155(1/2): 117-124.
[45] Salvi S, Corneti S, Bellotti M, Carraro N, Sanguineti M C, Castelletti S, Tuberosa R. Genetic dissection of maize phenology using an intraspecific introgression library. BMC Plant Biology, 2011, 11: 4.
[46] Lv H K, Zheng J, Wang T Y, Fu J J, Huai J L, Min H W, ZHANG X, TIAN B H, SHI Y S, WANG G Y. The maize d2003, a novel allele of VP8, is required for maize internode elongation. Plant Molecular Biology, 2014, 84(3): 243-257.
[47] 孙强, 任姣姣, 徐晓明, 李宗泽, 黄博文, 陈占辉, 吴鹏昊. 玉米株高和穗位高QTL定位和全基因组选择探究. 玉米科学, 2022, 30(4): 40-47.
Sun Q, Ren J J, Xu X M, LI Z Z, HUANG B W, CHEN Z H, WU P H. QTL mapping and genomic selection for plant height and ear height in maize. Journal of Maize Sciences, 2022, 30(4): 40-47. (in Chinese)
[48] Cassani E, Bertolini E, Cerino Badone F, Landoni M, Gavina D, Sirizzotti A, Pilu R. Characterization of the first dominant dwarf maize mutant carrying a single amino acid insertion in the VHYNP domain of the dwarf8 gene. Molecular Breeding, 2009, 24(4): 375-385.
[49] Lee Y, Choi D S, Kende H. Expansins: ever-expanding numbers and functions. Current Opinion in Plant Biology, 2001, 4(6): 527-532.
[50] Cosgrove D J. Nanoscale structure, mechanics and growth of epidermal cell walls. Current Opinion in Plant Biology, 2018, 46: 77-86.
[51] Cosgrove D J. Non-enzymatic action of expansins. The Journal of Biological Chemistry, 2020, 295(19): 6782.
[52] Choi D, Lee Y, Cho H T, Kende H. Regulation of expansin gene expression affects growth and development in transgenic rice plants. The Plant Cell, 2003, 15(6): 1386-1398.
[53] Lee Y, Choi D. Biochemical properties and localization of the beta–expansin OsEXPB3 in rice (L.). Molecules and Cells, 2005, 20(1): 119-126.
[54] PetrÁsek J, Friml J. Auxin transport routes in plant development. Development, 2009, 136(16): 2675-2688.
[55] Peer W A, Blakeslee J J, Yang H B, Murphy A S. Seven things we think we know about auxin transport. Molecular Plant, 2011, 4(3): 487-504.
[56] Morita Y, Kyozuka J. Characterization of OsPID, the rice ortholog of PINOID, and its possible involvement in the control of polar auxin transport. Plant and Cell Physiology, 2007, 48(3):540-549.
[57] Wu H M, Xie D J, Tang Z S, Shi D Q, Yang W C. PINOID regulates floral organ development by modulating auxin transport and interacts with MADS16 in rice. Plant Biotechnology Journal, 2020, 18(8):1778-1795.
[58] Chen Y N, Fan X R, Song W J, Zhang Y L, Xu G H. Over-expression of OsPIN2 leads to increased tiller numbers, angle and shorter plant height through suppression of OsLAZY1. Plant Biotechnology Journal, 2012, 10(2): 139-149.
[59] Yue R Q, Tie S G, Sun T, Zhang L, Yang Y J, Qi J S, Yan S F, Han X H, Wang H Z, Shen C J. Genome-wide identification and expression profiling analysis of ZmPIN, ZmPILS, ZmLAX and ZmABCB auxin transporter gene families in maize (L.) under various abiotic stresses. PLoS One, 2015, 10(3): e0118751.
candidate gene Localization ofgene regulating plant height and ear height in maize

1Crops Research Institute, Gansu Academy of Agricultural Sciences, Lanzhou 730070;2National Key Laboratory of Crop Genetic Improvement, Huazhong Agricultural University, Wuhan 430070
【Objective】Plant height is one of the important target traits in maize plant type breeding, which isclosely related not only to mechanized grain harvesting and lodging resistance, but also to maize yield. Therefore, it is of great theoretical and breeding value to isolate QTL/gene of maize plant height and analyze its function. This study aims to locate a novel maize dwarf gene, clarify its biological function, and provide important theoretical basis and gene resources for accelerating the improvement of maize plant type. 【Method】A single recessive mutant was derived in maize inbred line LY8405, from Crop Research Institute of Gansu Academy of Agricultural Sciences, by chemical mutagenic agent Ethyl Methyl Sulfonate (EMS). A maize dwarf and low ear mutant was isolated from the M2progeny, and the M3and M4progeny could stably inherit, which was named(). The F2population was constructed by hybridization with Mo17 and was identified by bulked segregate analysis-sequencing (BSA-seq) and target segment recombination exchange. Based on the Mo17 reference genome, the genes in the target region were obtained and functionally annotated to locate candidate genes.【Result】Phenotypic identification ofwas carried out, and the phenotype ofat seedling stage was not significantly different from that of the control LY8405. The plant height and ear height ofat mature stage were reduced by 87.2 cm and 55.4 cm, respectively, accounting for about 35.0% and 62.9%, difference is extremely significant. Morphological observation showed that the decrease of internode number and the shortening of internode cell length were the main reasons for the significant decrease of plant height and ear height of. The genetic analysis of the mutant gene was conducted by using the F2:3genetic populations. Themutant is inherited in a 3﹕1 (χ2=2.854) ratio and is a single recessive gene. Therefore, according to the results of BSA-seq, the candidate genewas initially located in the 15 Mb region of Bin1.09-1.10 on chromosome 1 of maize. The polymorphism molecular markers were further developed using the re-sequencing results of Mo17 and, and the target gene was accurately cloned by map-based cloning. Finally, the candidate genes were mapped to the size range of 600 kb, and there were 16 candidate genes in this range. By comparing the re-sequencing data, it was found thatgene changed into A at the 2062 position G, which resulted in amino acid changing from glycine to serine, and the transcription level expression was significantly reduced compared with LY8405.changed from T to C at the 223rd position leading to the 75th amino acid changed from serine to proline, and there was no significant difference in transcription level. Through association analysis of natural populations and the predicted genes for functional annotation, it was found thatandwere related to the growth and development of maize. 【Conclusion】The candidate genewas genetic locus significantly associated with plant height within the target region.
maize; plant height; ear height; BSA-seq; fine mapping; association analysis

10.3864/j.issn.0578-1752.2023.05.002
2022-10-26;
2022-12-25
国家自然科学基金(32160490)、作物遗传改良全国重点实验室开放课题资助、甘肃省重大专项(21ZD11NA005,21ZD10NF003)、甘肃省农业科学院生物育种专项共同资助(2022GAAS04)
周文期,E-mail:zhouwenqi850202@163.com。通信作者周玉乾,E-mail:yuqianzhou2008@163.com。通信作者邱法展,E-mail:qiufazhan @mail.hzau.edu.cn
(责任编辑 李莉)

