利用大刍草渗入系群体定位玉米株高和穗位高QTL
柳志华,肖仁杰,徐莹,罗红兵,2,黄成,2*
利用大刍草渗入系群体定位玉米株高和穗位高QTL
柳志华1,肖仁杰1,徐莹1,罗红兵1,2,黄成1,2*
(1.湖南农业大学农学院,湖南 长沙 410128;2.湖南省玉米工程技术研究中心,湖南 长沙 410128)
利用玉米自交系W22与大刍草杂交衍生得到的包含866个家系的渗入系群体,结合均匀覆盖玉米全基因组的19 838个SNP分子标记,采用R/qtl的多QTL模型对玉米株高和穗位高进行高精度的QTL定位分析。结果表明:玉米株高和穗位高存在广泛的遗传变异,属于典型的数量性状,由微效多基因控制;共检测到4个控制株高的QTL,分别位于第1、5和8染色体上,单个株高QTL表型贡献率变幅为2.33%~4.85%,加性效应的变幅为2.33~6.01 cm;共检测到10个控制穗位高的QTL,分别位于第1、2、3、5、6、7和8染色体上,单个穗位高QTL表型贡献率的变幅为1.77%~6.15%,加性效应的变幅为1.75~6.25 cm。
玉米;大刍草渗入系;株高;穗位高;QTL定位
株高和穗位高是构成玉米理想株型的2个重要农艺性状。研究表明,株高和穗位高过高,会降低种植密度、抗倒伏性和收获指数;过低,则影响田间通透性,提高病虫害感染率,降低生物产量[1]。进一步解析玉米株高和穗位高的遗传基础,对选育具有理想株型且高产的玉米品种具有重要意义。
玉米株高和穗位高是典型的数量性状,由微效多基因控制。随着测序成本的逐渐降低和分子标记技术的快速发展,国内外研究者利用连锁群体和关联群体对玉米株高和穗位高开展了大量的定位研究[2–7],并检测到许多控制玉米株高和穗位高的数量性状基因座(quantitative trait locus,QTL)和关联位点,但仅有少数基因被克隆。主要在玉米茎尖表达,提高的表达,促进GA1的积累,增加细胞长度和节间长,表现出更高的株高[8]。[9]、[10]、[11]、[12]和[13]可通过赤霉素途径调控玉米株高。[14]和[15]是调控玉米生长素极性运输的关键基因,与野生型相比,的突变体植株和的过表达植株均显著降低了株高和穗位高。尽管目前对玉米株高和穗位高的研究较多,但利用玉米–大刍草渗入系群体解析玉米株高和穗位高遗传基础的研究还鲜有报道。
本研究中,利用包含866个家系的玉米–大刍草渗入系群体,结合均匀覆盖全基因组的19 838个SNP分子标记,对玉米株高和穗位高进行高精度的QTL定位分析,旨在为进一步克隆玉米株高和穗位高QTL和分子标记辅助选择育种提供理论依据。
1 材料与方法
1.1 材料
以玉米自交系W22作为受体亲本和轮回亲本,以大刍草(ssp.)作为供体亲本,通过杂交1次、回交2次和自交3次衍生而成BC2S3渗入系群体,共包含866个家系。该群体已经利用GBS(genotyping-by-sequencing)简化基因组测序技术获得了均匀覆盖玉米全基因组的19 838个SNP分子标记。
1.2 方法
1.2.1试验设计
2019年春季在湖南省浏阳市(28.2°N, 113.6°E)国家农作物品种区域试验站种植BC2S3渗入系群体。田间试验采用扩增式不完全随机区组设计。每个小区种植2行,每行种植15株,株距25 cm。每垄种植2个家系。垄高15 cm,垄宽70 cm,沟宽30 cm。田间管理同当地生产管理。
1.2.2表型调查
散粉10 d后调查株高和穗位高,每个家系从第3株开始,连续调查8个单株。株高即从地面至植株顶叶叶环的高度。穗位高即从地面至最上部雌穗着生节的高度。
1.2.3数据分析
利用SAS软件计算每个家系株高和穗位高的最佳线性无偏估计值(BLUP);运用R软件对表型数据进行正态分布检验,计算变异系数、偏度、峰度,并分析株高和穗位高的相关性。
1.2.4QTL定位
参照HUANG等[16]的方法,利用R/qtl的多QTL模型进行QTL定位分析。应用Haley–Knott回归进行QTL简单区间定位分析,并采用置换检验(permutation test)10 000次的方法确定各性状QTL的LOD阈值(= 0.05)。将简单区间定位得到的QTL模型进行多QTL模型拟合,并利用R/qtl的refineqtl命令优化每个QTL的位置。进一步利用addqtl命令检测基因组是否存在其他显著改善模型的QTL,如果检测到新的QTL,则重新拟合多QTL模型和优化QTL位置。重复此过程,直至检测不到新的QTL。最后利用fitqtl命令计算所有QTL解释的总表型贡献率及单个QTL的加性效应和表型贡献率。参照MCCOUCH等[17]在1997年提出的方法进行QTL命名。
2 结果与分析
2.1 株高和穗位高表型值统计分析
运用R软件对玉米–大刍草渗入系群体的株高和穗位高表型值进行统计分析,结果(表1)表明:渗入系群体的株高和穗位高存在广泛的遗传变异,株高和穗位高的变异范围分别为113.2~192.3 cm和28.0~81.5 cm,变异系数分别为8.35%和18.85%,穗位高的变异程度大于株高;株高和穗位高的偏度和峰度绝对值均小于1,说明渗入系群体的株高和穗位高表型值均服从正态分布,呈现典型的数量性状遗传特征,符合QTL定位要求。

表1 株高和穗位高表型值的统计分析结果
2.2 株高和穗位高的频率分布及相关性分析
频率分布分析结果(图1–a,图1–b)表明,渗入系群体的株高和穗位高表型值均呈现连续性变异,且频数分布图与正态分布曲线拟合较好,表明株高和穗位高属于典型的由多基因控制的数量性状。相关性分析结果(图1–c)表明,株高和穗位高之间存在极显著的正相关,Pearson相关系数为0.66(< 0.01),株高和穗位高的拟合方程为=0.89+109.36,株高和穗位高呈线性的正相关关系,表明株高和穗位高可能具有相似的遗传基础,具有部分相同的调控基因和代谢途径。

图1 株高和穗位高的频率分布及相关性分析结果
2.3 株高和穗位高QTL定位分析
利用R/qtl的多QTL模型对渗入系群体的株高和穗位高进行QTL定位分析,采用置换检验10 000次的方法,确定株高和穗位高QTL的LOD阈值(分别为4.22和4.16),共检测到14个QTL,分别位于第1、2、3、5、6、7和8染色体上(图2)。
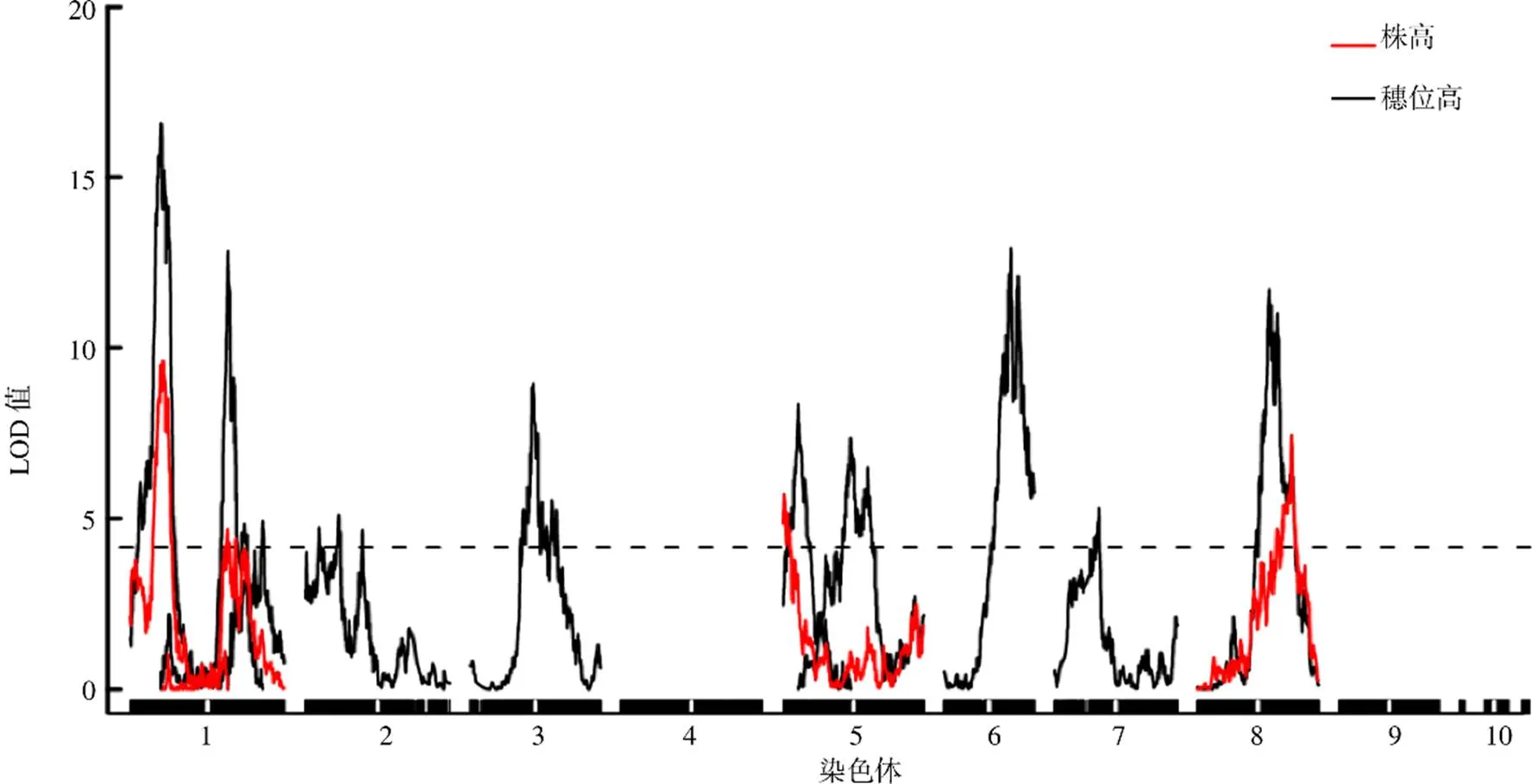
图2 株高和穗位高QTL图谱
2.3.1株高QTL
共检测到4个玉米株高QTL,分别为、、和,位于第1、5和8染色体上,两侧分子标记分别为m00824与m01009、m06413与m06822、m25156与m25315、m45071与m45142,单个QTL的LOD峰值为4.77~9.77,加性效应大小为2.33~6.01 cm,表型贡献率为2.33%~4.85%,且4个QTL的增效等位基因均来自大刍草(表2)。

表2 株高和穗位高QTL定位结果
“△”示大刍草等位基因的显性效应减少的性状表型值。
2.3.2穗位高QTL
共检测到10个玉米穗位高QTL,分别为、、、、、、、、和,位于第1、2、3、5、6、7和8染色体上,两侧分子标记分别为m00817与m00912、m06496与m06617、m07716与m07838、m08456与m10331、m16893与m17099、m25461与m25542、m28055与m29030、m35223与m35631、m36519与m38032、m43919与m44688,单个QTL的LOD峰值为4.92~16.58,加性效应大小为1.75~6.25 cm,表型贡献率为1.77%~6.15%,且10个QTL中有9个QTL的增效等位基因来自大刍草,只有的增效等位基因来自玉米自交系W22(表2)。
进一步分析发现,位于第1染色体上的株高QTL(35.0~44.0 cM)与穗位高QTL(35.0~39.6 cM)的置信区间存在重叠,位于第1染色体上的株高QTL(113.3~125.2 cM)与穗位高QTL(117.9~122.3 cM)的置信区间也存在重叠,且增效等位基因均来自大刍草,表明这2个区段可能存在紧密连锁的控制玉米株高和穗位高的不同基因,或是同一基因的一因多效作用。
3 结论与讨论
本研究中,玉米株高和穗位高之间存在极显著的正相关关系,表明株高和穗位高可能具有相似的遗传基础,且一般株高较高的植株穗位高也相应较高,这与前人的研究结果相一致[1–2]。QTL定位分析发现:在第1染色体上的2个区段均检测到置信区间重叠的株高和穗位高QTL,表明这2个区段可能存在紧密连锁的控制玉米株高和穗位高的不同基因,或是同一基因的一因多效作用;在第5和8染色体上,同时检测到了株高和穗位高QTL,但QTL的置信区间没有重叠;大部分株高和穗位高QTL的置信区间没有重叠,甚至位于不同的染色体上,在玉米第2、3、6和7染色体上仅检测到穗位高QTL,而没有检测到株高QTL。以上结果表明,控制玉米株高和穗位高的基因大多是不同的,即株高和穗位高具有相对独立的遗传基础和不同的遗传调控机制;因此,在今后的育种实践中,可以单独改良玉米株高和穗位高。
[1] 兰进好,褚栋.玉米株高和穗位高遗传基础的QTL剖析[J].遗传,2005,27(6):925–934. LAN J H,CHU D.Study on the genetic basis of plant height and ear height in maize(L.)by QTL dissection[J].Hereditas (Beijing),2005,27(6):925–934.
[2] 郑德波,杨小红,李建生,等.基于SNP标记的玉米株高及穗位高QTL定位[J].作物学报,2013,39(3):549–556. ZHENG D B,YANG X H,LI J S,et al.QTL identification for plant height and ear height based on SNP mapping in maize(L.)[J].Acta Agronomica Sinica,2013,39(3):549–556.
[3] 何坤辉,常立国,崔婷婷,等.多环境下玉米株高和穗位高的QTL定位[J].中国农业科学,2016,49(8):1443–1452. HE K H,CHANG L G,CUI T T,et al.Mapping QTL for plant height and ear height in maize under multi- environments[J].Scientia Agricultura Sinica,2016,49(8):1443–1452.
[4] LI X P,ZHOU Z J,DING J Q,et al.Combined linkage and association mapping reveals QTL and candidate genes for plant and ear height in maize[J].Frontiers in Plant Science,2016,7:833.
[5] WANG B B,LIU H,LIU Z P,et al.Identification of minor effect QTLs for plant architecture related traits using super high density genotyping and large recombinant inbred population in maize()[J]. BMC Plant Biology,2018,18(1):17.
[6] WENG J F,XIE C X,HAO Z F,et al.Genome-wide association study identifies candidate genes that affect plant height in Chinese elite maize(L.) inbred lines[J].PLoS One,2011,6(12):e29229.
[7] YANG N,LU Y L,YANG X H,et al.Genome wide association studies using a new nonparametric model reveal the genetic architecture of 17 agronomic traits in an enlarged maize association panel[J].PLoS Genetics,2014,10(9):e1004573.
[8] TENG F,ZHAI L H,LIU R X,et al.,a candidate gene for a major QTL,.1,for plant height in maize[J].The Plant Journal,2013,73(3):405–416.
[9] WINKLER R G,HELENTJARIS T.The maizegene encodes a cytochrome P450-mediated early step in gibberellin biosynthesis[J].The Plant Cell,1995,7(8):1307–1317.
[10] THORNSBERRY J M,GOODMAN M M,DOEBLEY J,et al.polymorphisms associate with variation in flowering time[J].Nature Genetics,2001,28(3):286–289.
[11] WINKLER R G,FREELING M.Physiological genetics of the dominant gibberellin-nonresponsive maize dwarfs,and[J].Planta,1994,193(3):341–348.
[12] BENSEN R J,JOHAL G S,CRANE V C,et al.Cloning and characterization of the maizegene[J].The Plant Cell,1995,7(1):75–84.
[13] 白文钦,胡明瑜,潘晓雪,等.玉米赤霉素受体基因的克隆及功能研究[J].安徽农业大学学报,2019,46(1):133–140. BAI W Q,HU M Y,PAN X X,et al.Cloning and functional study ofgene in maize[J].Journal of Anhui Agricultural University,2019,46(1):133–140.
[14] MULTANI D S,BRIGGS S P,CHAMBERLIN M A,et al.Loss of an MDR transporter in compact stalks of maizeand sorghummutants[J].Science,2003,302:81–84.
[15] LI Z X,ZHANG X R,ZHAO Y J,et al.Enhancing auxin accumulation in maize root tips improves root growth and dwarfs plant height[J].Plant Biotechnology Journal,2018,16(1):86–99.
[16] HUANG C,CHEN Q Y,XU G H,et al.Identification and fine mapping of quantitative trait loci for the number of vascular bundle in maize stem[J].Journal of Integrative Plant Biology,2016,58(1):81–90.
[17] MCCOUCH S R,CHO Y G,YANO M,et al.Report on QTL nomenclature[J].Rice Genetics Newsletter,1997,14:11–13.
QTL mapping of plant height and ear height in maize using a teosinte introgression line population
LIU Zhihua1, XIAO Renjie1, XU Ying1, LUO Hongbing1,2, HUANG Cheng1,2*
(1.College of Agronomy, Hunan Agricultural University, Changsha, Hunan 410128, China; 2.Maize Engineering Technology Research Center of Hunan Province, Changsha, Hunan 410128, China)
Using a large population of 866 maize-teosinte introgression lines that has been genotyped into 19,838 SNP markers, we performed a high resolution QTL mapping of the plant height and ear height. The results showed that there are wide genetic variations in plant height and ear height, which belong to typical quantitative traits and are controlled by the genes of small effect. Totally, 4 QTL of the plant height were located on chromosome 1, 5 and 8, and the phenotypic contribution rate and additive effect of each QTL ranged from 2.33% to 4.85% and from 2.33 cm to 6.01 cm, respectively. In addition, 10 QTL of the ear height were located on chromosome 1, 2, 3, 5, 6, 7 and 8, and the phenotypic contribution rate and additive effect of each QTL ranged from 1.77% to 6.15% and from 1.75 cm to 6.25 cm, respectively.
maize; teosinte introgression line; plant height; ear height; QTL mapping

S513.01
A
1007-1032(2020)04-0381-05
10.13331/j.cnki.jhau.2020.04.001
柳志华,肖仁杰,徐莹,罗红兵,黄成.利用大刍草渗入系群体定位玉米株高和穗位高QTL [J].湖南农业大学学报(自然科学版),2020,46(4):381–385.
LIU Z H, XIAO R J, XU Y, LUO H B, HUANG C. QTL mapping of plant height and ear height in maize using a teosinte introgression line population[J]. Journal of Hunan Agricultural University(Natural Sciences), 2020, 46(4): 381–385.
http://xb.hunau.edu.cn
2019–10–17
2019–11–27
国家重点研发计划(2016YFD0101803);国家自然科学基金项目(31801319);湖南省教育厅优秀青年项目(18B117)
柳志华(1993—),女,甘肃平凉人,硕士研究生,主要从事玉米种质创新与利用研究,liuzhihua317@stu.hunau.edu.cn;
,黄成,博士,讲师,主要从事玉米遗传学及功能基因组学研究,hc66@hunau.edu.cn
责任编辑:毛友纯
英文编辑:柳正

