OsNAC2d基因编辑水稻突变体的创建及其对干旱胁迫的响应
李兆伟 莫祖意 孙聪颖 师 宇 尚 平 林伟伟 范 凯 林文雄,*
基因编辑水稻突变体的创建及其对干旱胁迫的响应
李兆伟1,2莫祖意1,2孙聪颖1,2师 宇1,2尚 平2,3林伟伟1,2范 凯2,3林文雄1,2,*
1福建农林大学生命科学学院, 福建福州 350002;2福建农林大学/ 福建省农业生态过程与安全监控重点实验室, 福建福州 350002;3福建农林大学农学院, 福建福州 350002
为探究转录因子OsNAC2d的生物学功能及其对水稻耐旱性的影响, 本研究利用CRISPR/Cas9编辑技术对粳稻中花11中的基因进行突变, 并考察突变体在田间种植下的农艺性状, 以及突变体幼苗在干旱胁迫下的生长情况和基因表达水平。结果表明,基因主要在水稻成熟籽粒、叶片和花药中表达, 在根系和茎中的表达量较低, 并且受干旱胁迫诱导, 在10株阳性基因突变株系的T2代植株中, 筛选出6种纯合突变体, 田间试验调查表明,突变体与野生型中花11相比, 株高、有效穗、穗长、穗粒数、结实率、千粒重和单株产量等农艺性状无显著差异。在干旱胁迫时,突变体幼苗的根系与植株生长、根系和地上部生物量的积累均受到抑制, 且在突变体中的表达量维持在较低水平, 而野生型水稻的表达则受干旱胁迫诱导而增强, 植株生长与生物量积累未受到显著抑制, 表明转录因子OsNAC2d正调控水稻响应干旱胁迫。创建的突变体材料为进一步揭示OsNAC2d的生物学功能及其响应干旱胁迫的精细调控机制提供了优质种质资源。
水稻; 干旱胁迫; 基因编辑;基因
水稻作为世界主要粮食作物之一, 占全球粮食总产量的四分之一, 是全球近一半以上人口的主粮, 尤其在亚洲地区, 约90%的人口以稻米为主食[1]。我国的水稻产量约占粮食总产量的1/3, 是全国2/3人口的主粮, 维持水稻产量稳定对保障国家粮食安全具有重要现实意义[2]。然而, 随着全球生态环境恶化, 极端气候频发, 降水区域分布不均匀和环境高温持续出现, 农业灌溉水资源日益匮乏, 农田旱灾现象变得日趋严重, 干旱已成为制约水稻产量潜力发挥和造成稻谷减产的最主要环境胁迫因素之一, 严重威胁着国家的粮食安全[3-4]。为有效缓解农田干旱现象, 除大力兴修农田水利设施、开发节水灌溉栽培技术等措施外, 选育对干旱胁迫耐性较强的水稻品种是缓解农田水资源短缺的有效措施之一[5]。进入21世纪以来, 随着现代分子遗传学和生物技术的快速发展, 大量水稻新基因的遗传调控功能被相继发掘, 以关键农艺性状功能基因为基础, 制定高效、精确的分子设计育种策略, 将水稻功能基因组学成果应用于遗传育种改良, 能有效弥补传统水稻育种手段周期长、效率低、预见性差等缺陷[1,6]。
NAC是近二十多年来发现的一类植物特有的转录因子, 依据矮牵牛()的()基因、拟南芥()的基因和()基因的首字母命名[7-8], 氨基酸序列包含1个特异的N端NAC结构域和C端转录调控结构域, NAC结构域由150~160个氨基酸形成5个亚结构域(A~E), 其中A、C、D亚结构域序列高度保守, 并含核定位信号, B和E亚结构域序列的变异频率较高, 赋予NAC转录因子不同功能, C端的转录调控结构域的氨基酸组分呈现多样化, 起对靶基因的转录激活功能[9], 在植物响应生物和非生物胁迫反应的激素调控、信号转导、器官构建、免疫应激等过程中发挥重要调节作用[10]。水稻中已经发现151个NAC转录因子[11], 其中多个NAC转录因子参与植株对干旱胁迫的响应调节, 如干旱胁迫能诱导在水稻叶片保卫细胞中表达, 过表达的转基因植株通过调节叶片气孔关闭, 减少水分丢失, 提高对土壤干旱胁迫的抗性[12]; 过表达能扩大转基因植株的根系中柱和通气组织, 并改善植株的根型结构, 增强对干旱胁迫的抗性, 显著提高在干旱条件下的稻谷产量[13]; 转录因子OsNAC5能特异结合耐旱功能基因()的启动子, 增加表达丰度, 从而增强水稻的抗旱性和耐盐性[14]; 过表达显著增加植株的根皮层和根中柱细胞数目, 使根系直径增大, 根系变粗, 显著提升植株的抗旱性, 并提高转基因植株在干旱条件下的田间产量[15]; 在过表达的转基因植株中, 转录因子OsNAC3通过清除干旱、高温等胁迫造成的细胞内多余活性氧积累, 维持细胞内活性氧稳态, 提高转基因植株的抗旱性和耐高温能力[16]。
本课题组前期通过转录组测序分析, 发现了1个在水稻叶片衰老过程显著差异表达基因[17],该基因的表达水平受干旱胁迫诱导, 基因ID号为, 位于水稻3号染色体, 编码区含有1503个核苷酸, 无内含子, 氨基酸序列包含一个典型的NAC结构域, 属于水稻NAC转录因子家族, 然而该基因的在水稻生长发育阶段的生理作用和分子调控模式等生物学功能, 目前还未见相关报道。本研究通过CRISPR/Cas9编辑技术构建了突变体, 并从T2代中分离到纯合突变植株, 调查突变植株的田间农艺性状, 考察突变体幼苗在干旱条件下的生长情况和基因响应干旱的表达模式。研究结果对系统揭示转录因子OsNAC2d应答干旱逆境的分子调控机理有积极的理论价值。
1 材料与方法
1.1 试验材料
以粳稻中花11 (Zhonghua 11, ZH11,subsp. Keng)为遗传背景材料, 进行重组载体转化。转化用的农杆菌为EHA105菌种, 原核感受态为大肠杆菌DH5α菌株。pYL-U6a-gRNA、pYL-U6b- gRNA中间载体以及pYLCRISPR/Cas9-MT表达载体, 均由福建农林大学海峡联合研究院朱强教授惠赠, 最早由华南农业大学刘耀光院士赠予。试验材料种植于福建省福州市福建农林大学试验农场的转基因材料管理专区。
试验用TRIzol试剂、反转录试剂盒、T4连接酶、qPCR试剂盒等购买于宝生物工程(大连)有限公司,I限制性内切酶购买于NEB (北京)有限公司, KOD-Plus-Neo高保真酶购买于东洋纺(上海)生物科技有限公司, 基因编辑靶点序列和引物合成、以及测序服务均由生工生物工程(上海)股份有限公司完成。
1.2 OsNAC2d表达模式分析
野生型ZH11水稻种子经浸泡、催芽、播种后, 采用溶液培养至四叶一心时, 剪取叶、茎、根组织, 液氮速冻后用于RNA提取, 检测在不同组织中的表达情况; 另选取生长到二叶一心的植株, 在完全营养液中添加聚乙二醇(PEG-6000)至终浓度为15%, 模拟环境干旱胁迫, 分别于胁迫第0、0.5、3、6、12和24 h剪取叶片, 液氮速冻后提取样品RNA,用于检测在干旱胁迫不同时期的表达量。
1.3 靶点设计与CRISPR/Cas9-OsNAC2d-T12载体构建
依据(LOC_Os03g39100)基因序列特征, 利用CCTop-CRISPR/Cas9在线软件在基因反义链的上游和下游位置各设计1个20 bp的编辑靶点(图2-A), PAM序列分别为GGG和AGG, 参照李兆伟等[18]方法, 在靶点1与靶点2序列端头添加gccg/aaac与gttg/aaac连接接头序列, 使其分别能与pYL-U6a-gRNA和pYL-U6b-gRNA中间载体经I酶切后形成互补黏性末端。
参照Ma等[19]的方法构建双靶点pYLCRISPR/ Cas9-OsNAC2d-T12表达载体, 先将2个靶点分别与pYL-U6a-gRNA和pYL-U6b-gRNA中间载体连接, 再经过2次PCR扩增获得含特异性接头的U6a- OsNAC2d-T1和U6b-OsNAC2d-T2表达盒, 依据图2-B方式, 将U6a-OsNAC2d-T1和U6b-OsNAC2d-T2表达盒与pYLCRISPR/Cas9-MT载体连接, 并转化DH5α感受态, 平板培养, 单菌落测序后采用农杆菌介导法转化ZH11愈伤, 经培养获得转基因株系, 利用表1中的潮霉素基因检测引物, PCR扩增筛选出阳性转基因株系。
1.4 编辑植株的突变类型检测鉴定
为检测编辑植株中基因的突变情况, 分别在2个靶位点两端设计引物OsNAC2d-T1-F/ OsNAC2d-T1-R和OsNAC2d-T2-F/OsNAC2d-T2-R (表1), 提取编辑突变水稻植株的T2代幼苗叶片DNA, 作为模板, 进行PCR扩增, 2个检测靶点的扩增产物大小分别为373 bp和415 bp, 扩增产物送生工生物工程(上海)股份有限公司进行测序, 以野生型ZH11水稻DNA为模板的扩增产物片段测序序列为参照, 分析编辑植株的基因突变类型。
1.5 osnac2d突变体的农艺性状考查
编辑突变植株幼苗经测序鉴定后, 单本插秧移栽到福建农林大学小试验农场转基因材料管理专区,依据当地常规稻田管理方式进行水肥管理、病虫草害防除。待水稻成熟时, 分别于不同变异类型突变体中选取10株长势基本一致的植株, 考察株高、有效穗、穗长、穗粒数、结实率、千粒重和单株产量等田间农艺性状。利用SPSS13.0软件计算平均值与标准差, 采用单因素方差分析比较指标的种间差异, 以≤0.05为显著性差异。

表1 本研究采用的核苷酸序列及相应引物
1.6 osnac2d突变体耐旱性分析
选取和突变体与野生型ZH11为试验材料, 种子经浸泡、催芽后, 利用完全营养液培养14 d至幼苗长至二叶一心, 更换新鲜营养液, 并添加PEG-6000至终浓度为15%, 进行干旱模拟培养, 胁迫培养3 h后, 剪取叶片提取总RNA, 用于分析在各个基因型材料中的表达水平, 每个突变体材料分别设置3组重复处理, 胁迫培养14 d后, 分别从每个处理选取5株用于观察幼苗表型, 并测定根长、苗长、干重等外观指标, 衡量突变体植株对干旱胁迫的耐受性。
2 结果与分析
2.1 OsNAC2d基因的组织表达模式与干旱响应模式
对O在野生型ZH11水稻的根、茎、叶、花药和成熟籽粒中的表达量检测发现,在ZH11水稻不同组织中的表达量存在较大差异, 其中在成熟籽粒中的表达量最高, 其次为叶片与花药, 在根系中的表达水平显著低于在籽粒、叶片和花药中的表达水平, 而在茎中表达水平最低(图1-A); 通过15%的PEG-6000胁迫处理发现, 叶片和根系中O的表达量在PEG处理3 h后显著提高, 在处理3~12 h期间, 均显著高于未处理时, 而在PEG处理24 h后, 叶片和根系中的O表达水平开始降低到接近于未处理前水平(图1-B), 表明基因在水稻叶片和根系对干旱逆境胁迫响应中起作用。
2.2 OsNAC2d基因编辑靶点设计与pYLCRISPR/ Cas9-OsNAC2d-T12载体构建
为进一步探究的生物学功能, 借助CRISPR/Cas9技术编辑突变ZH11的基因, 根据基因编码区含1503个碱基且无内含子的序列结构特征, 以基因CDS编码区+133 ~ +135 bp的GGG作为PAM序列, +136 ~ +155 bp区域的20 bp碱基片段作为靶点1序列, 编码区下游+1090 ~ +1092 bp处的AGG作为靶点2的PAM序列, 从+1093 ~ +1112 bp间的20 bp碱基序列为靶点2编辑位点(图2-A), 以增加基因的突变频率。
为构建pYLCRISPR/Cas9-OsNAC2d-T12表达载体, 首先将双链靶点1与pYL-U6a-gRNA中间载体连接, 靶点2与pYL-U6b-gRNA中间载体连接, 使2个靶点序列分别由OsU6a和OsU6b启动子驱动, 再通过PCR扩增得到含特异连接接头的U6a启动子-gRNA1表达盒和U6b启动子-gRNA2表达盒, 根据图2-B连接模式, 将2个表达盒与pYLCRISPR/ Cas9-MT载体经酶切连接, 获得pYLCRISPR/Cas9- OsNAC2d-T12重组载体, 测序表明, 重组载体的2个靶序列核苷酸(图3)与设计靶点序列完全一致(表1), 表明2个靶点均准确连接到了pYLCRISPR/Cas9- MT载体上, 重组pYLCRISPR/Cas9-OsNAC2d-T12表达载体可用于转化水稻愈伤组织。
2.3 T0代阳性植株筛选
将重组pYLCRISPR/Cas9-OsNAC2d-T12载体通过农杆菌介导法侵染转化ZH11愈伤组织, 经组织培养获得11株再生转化植株, 提取再生植株叶片的基因组DNA, 利用PCR扩增潮霉素基因, 琼脂糖凝胶电泳检测发现, 共10株转基因株系的基因组DNA能扩增出280 bp的潮霉素基因条带(图4), 为阳性再生株系, 其中1个转基因株系没有检测到潮霉素标记基因, 为阴性植株。

图1 水稻OsNAC2d的表达量分析
A:在野生型ZH11水稻的根、茎、叶、花药和籽粒中的表达水平; B: 叶片和根系中在15%的PEG-6000胁迫下的动态表达分析, 取样时间分别为处理后0、0.5、3、6、12和24 h; 不同字母表示在< 0.05水平的显著性差异。
A: the differential expression level ofgene in the root, stem, leaf, anther, and grain of wild type ZH11; B: the temporal expression ofin leaves and roots of rice incubated in the nutrient solution including 15% PEG-6000, and samples were collected at 0, 0.5, 3, 6,12, and 24 hours after treatment, respectively. Different letters above the columns are significantly different at the 0.05 probability level.

图2 OsNAC2d的gRNA靶点和pYLCRISPR/Cas9-OsNAC2d-T12表达载体重组示意图
A:的2个gRNA靶点位置和碱基序列; B: 2个靶点gRNA表达盒与pYLCRISPR/Cas9-MT重组示意图。
A: the position and nucleotide sequences of two gRNA targets in thegene locus; B: the recombining diagram of two gRNA cassettes and pYLCRISPR/Cas9-MT vector.

图3 pYLCRISPR/Cas9-OsNAC2d-T12重组表达载体的2个靶点测序检测
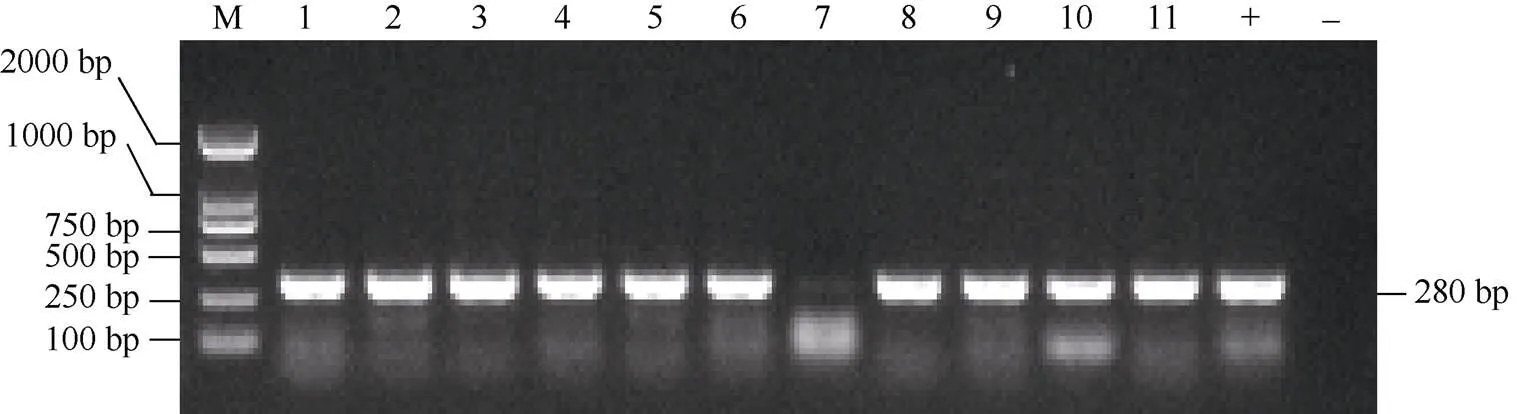
图4 阳性转基因株系鉴定
-: 阴性对照, 为等体积的水代替模板; +: 阳性对照, 为等体积阳性质粒作模板; 1~11: 转基因株系; M: 2 kb DNA ladder。
-: the negative control is an equal volume of water instead of the template; +: the positive control is an equal volume of the positive plasmid as a template; 1–11: transgenic lines; M: 2 kb DNA ladder.
2.4 纯合osnac2d突变体植株的筛选与鉴定
为尽快获得突变性状稳定遗传的纯合突变植株,本研究直接对转基因株系的T2代植株基因组的2个靶点区域碱基进行PCR扩增和测序, 并与野生型序列进行比较分析。测序分析结果表明, 在10个阳性株系的T2代植株中, 共筛选鉴定出6种在2个靶点均产生纯合突变的植株, 由图5可见,突变体在靶点1插入1个A碱基, 导致靶点1与靶点2之间的碱基位移, 在靶点2处的GC碱基突变为1个A碱基, 造成1个碱基缺失;突变体在2个靶点分别缺失21与3个碱基,突变体在靶点1插入1个A碱基, 在靶点2处则缺失1个G碱基, 并且和纯合突变体是来源于同一个T0代杂合突变体的分离后代; 在T0代杂合突变株系的2个纯合T2代突变植株中,突变体在靶点1缺失含36个碱基的小片段, 在靶点2则插入1个T碱基,突变体则在2个靶点区域分别缺失7和3个碱基;突变体在T0代即为纯合突变, 在靶点1插入1个A碱基, 在靶点2缺失GCA碱基。
2.5 osnac2d突变体的农艺性状
纯合突变植株经田间种植试验, 对其农艺性状调查发现, 除的株高、的穗粒数和的结实率低于野生型ZH11外, 其余突变体植株的株高、穗粒数、结实率等指标均与野生型ZH11无显著差异, 此外, 6种突变体的有效穗数、穗长、千粒重和单株产量等指标也与野生型ZH11无显著差异, 表明基因突变未影响正常田间种植模式下的植株农艺性状和产量构成参数。
2.6 osnac2d突变体耐旱性调查
鉴于突变体在靶点1插入1个A碱基, 在靶点2缺失3个碱基,突变体在2个靶点分别缺失7个和3个碱基, 均引起碱基移码突变, 因此选这2个基因型突变体进行耐旱性调查。在正常营养液中培养14 d后的和突变体经15%的PEG-6000胁迫处理14 d后, 植株的根系和地上部分均受到抑制, 植株变得矮小、瘦弱; 而同样处理条件下的野生型ZH11则未产生明显外观可见的受抑制表型(图6)。对植株生长情况的测定分析表明,和突变体受PEG胁迫后的根系长度、植株地上部苗长以及植株总长度均显著低于其在完全营养液中的长势, 而野生型ZH11各部位的生长长度与未处理组无显著差异(图7)。对植株生物量的测定发现, PEG胁迫引起、突变体与野生型ZH11的根系生物量均较其在完全营养液中显著降低(图8-A); 此外,、突变体在PEG胁迫下的地上部生物量显著低于其在完全营养液中生长时的生物量, 而野生型ZH11在PEG处理与完全营养液中的地上部生物量的差异未达到显著性(图8-B); 就植株总生物量而言,、突变体与野生型ZH11在PEG胁迫下的植株总生物量均显著低于其在完全营养液中培养时的生物量, 表明所有供试材料的物质合成与植株生长均受到了PEG胁迫抑制, 而在同一PEG胁迫处理条件下,和突变体的植株总生物量则显著低于野生型ZH11 (图8-C), 表明突变体水稻幼苗受PEG抑制的程度较野生型ZH11更为严重, 突变体变得对干旱胁迫更敏感, 苗期长势较容易受到环境干旱影响。

图5 osnac2d突变体与野生型植株中的OsNAC2d基因序列比对分析
蓝色字母表示靶点序列, 黄色高亮为PAM序列, 删除线代表缺失碱基, 红色小写字母为插入碱基, -表示缺失, +表示插入, WT代表野生型(中花11)。
The blue letters are the target genome sequence, the yellow highlighted letters denote PAM, dashes strikethrough indicate the deleted bases, and insertion nucleotides are shown in red lowercase letters. -: deletion; +: insertion; WT: wild type (ZH11).

表2 不同类型osnac2d突变体植株及其野生型水稻(WT)的农艺性状
表中数据为平均值±标准误; 同列数据后相同字母表示在0.05水平上差异不显著, 不同字母表示在0.05水平上差异显著。
Data in the table are presented as average values ± standard deviation; values followed by the same letter within the same column are not significantly different at the 0.05 probability level, and values followed by the different letter within the same column are significantly different at the 0.05 probability level.
2.7 osnac2d突变体中OsNAC2d响应干旱胁迫的表达情况
进一步对的表达量检测发现, 在完全营养液培养条件下,与突变体中的表达量显著低于野生型ZH11, 当处于PEG胁迫条件下时,与突变体中的表达量较其在完全营养液培养时未发生显著变化, 表达水平维持在较低水平, 并显著低于野生型ZH11, 而野生型ZH11中的表达量则被PEG胁迫诱导增强, 较其在完全营养液中的表达水平显著提升(图9)。

图6 osnac2d突变体与野生型水稻在含15%的PEG-6000营养液胁迫下的表型
A和D为野生型水稻中花11, B和E为突变体, C和F为突变体, 对照组为完全营养液培养, PEG-6000处理时长为14 d。
A and D denote wild type ZH11, B and E denotemutant, C and F denotemutant. The control group is the seedlings incubated in the complete nutrient solution. The duration of PEG-6000 treatment is 14 days.

图7 osnac2d突变体与野生型ZH11在含15%的PEG-6000营养液中的长势情况
A为根系长度, B为地上部长度, C为植株总长度, 对照组为完全营养液培养, PEG-6000处理时长为14 d, 不同字母表示在< 0.05水平的显著性差异。
A: the length of root; B: the length of seedling; C: the length of whole plant. The control group is the seedlings incubated in the complete nutrient solution. The duration of PEG-6000 treatment is 14 d. Different lowercase letters above the columns are significantly different at the 0.05 probability level.
3 讨论
随着水资源短缺与全球降雨不均匀分布, 干旱已成为制约农业生产的最大、最广泛的非生物胁迫因子, 严重影响农作物的生长和发育, 限制农作物增产, 并造成严重的产量损失, 制约着农田生态系统的有效产出[4,20]。农作物耐旱性研究与抗旱品种选育, 是在有限水资源条件下缓解农作物产量损失, 并保障农作物产量稳定的有效途径之一。作物耐旱性相关新基因的发掘及功能研究, 对通过分子标记辅助育种、新种质资源的精确设计选育等具有积极的实践指导价值。
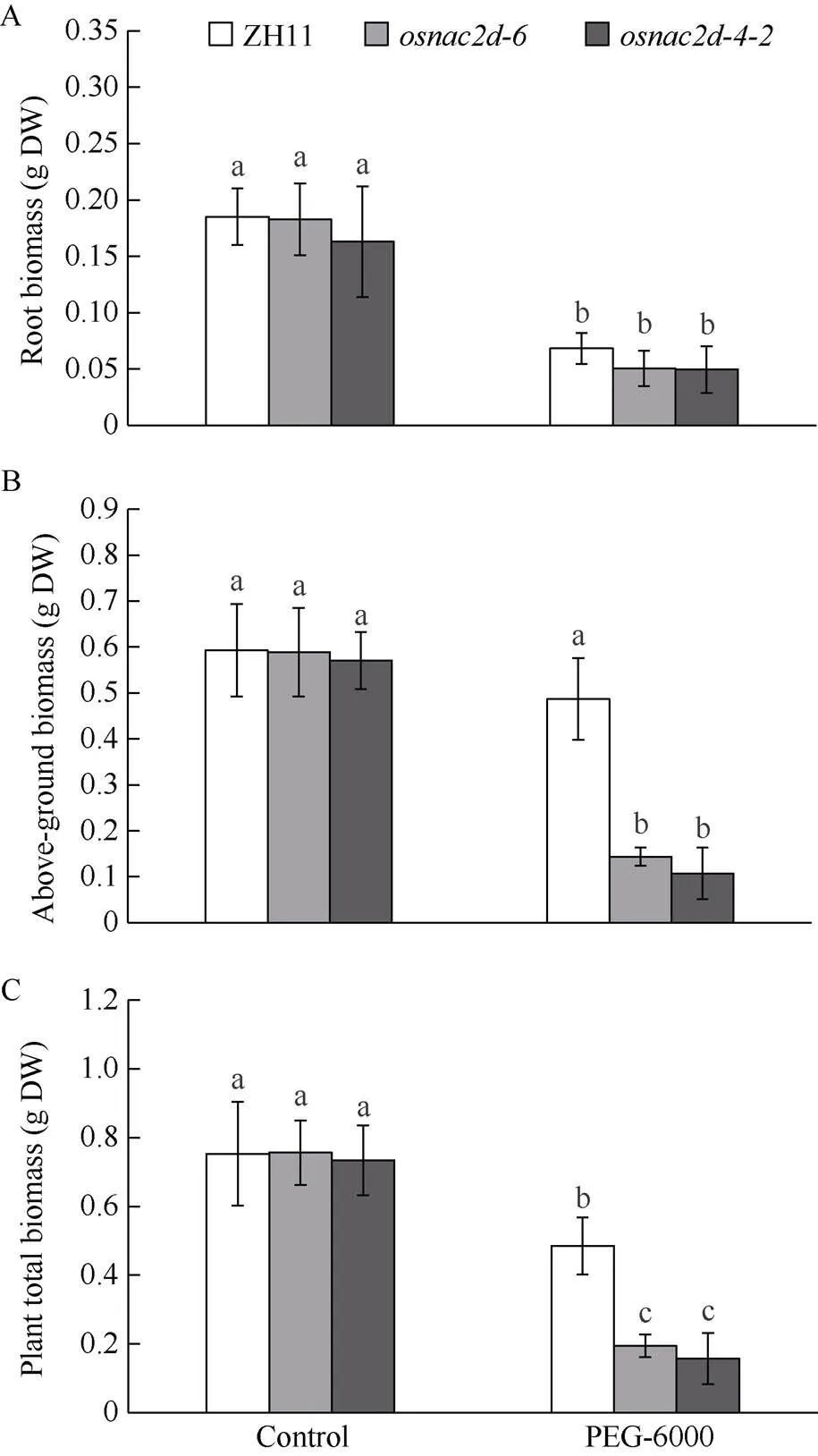
图8 osnac2d突变体与野生型ZH11在含15%的PEG-6000营养液中的生物量
A为根系生物量, B为地上部生物量, C为植株总生物量, 对照组为完全营养液培养, PEG-6000处理时长为14 d, 不同字母表示在< 0.05水平的显著性差异。
A: the root biomass; B: the above-ground biomass; C: the total biomass of whole plant. The control group is the seedlings incubated in the complete nutrient solution. The duration of PEG-6000 treatment is 14 days. Different lowercase letters above the columns are significantly different at the 0.05 probability level.
CRISPR/Cas9编辑系统是近十多年来发展的对靶基因进行快速、准确剪切修复的编辑技术, 能快速高效地对目标性状基因进行编辑突变, 获得性状改良并可稳定遗传的纯合突变株系, 极大地缩短了育种周期, 已在多种作物突变体种质资源创建中得到应用[21-25]。在本研究中, 为探索的生物功能, 借助CRISPR/Cas9编辑系统, 设计双编辑靶点, 突变粳稻ZH11中的基因, 共获得10株T0代阳性转化再生植株(图4), 通过对T2代植株进行测序分析, 得到6种在2个靶点区域均产生纯合突变的后代分离植株(图5), 并获得了在靶点1区域呈短片段碱基缺失的、、等3个纯合突变体, 其中突变体与是T0代杂合突变体在T2代的分离纯合后代。此外, 突变体、、在靶点1处插入1个A碱基, 但在靶点2处分别缺失1或3个碱基, 其中和突变体在T0代即为纯合突变株系, 在T2代未检测到新的分离后代。与单靶点编辑方式多产生单碱基插入或缺失突变相比[26-27],双靶点编辑植株的突变类型较为丰富, 增加了基因突变频率, 较易从后代分离植株中筛选到呈片段化缺失的突变类型, 并且通过对T2代分离后植株目标基因直接进行扩增测序, 消除了T0代植株中的杂合突变、双等位突变等的干扰, 能快速获得可稳定遗传的纯合突变植株。

图9 osnac2d-6、osnac2d-4-2突变体和野生型ZH11中OsNAC2d基因在15%的PEG-6000胁迫下的表达水平
对照组为完全营养液培养, 不同字母表示在< 0.05水平的显著性差异。
The control group is the seedlings incubated in the complete nutrient solution. Different letters above the columns are significantly different at the 0.05 probability level.
NAC转录因子在植物响应干旱、高低温和高盐等非生物逆境胁迫过程中起重要调节作用[28], 尤其在干旱条件下, NAC转录因子能显著影响植株对干旱胁迫的耐受性[29], 如转录因子ANAC19、ANAC55和ANAC72的基因过量表达能改善拟南芥的耐旱性[30]; 番茄转录因子SINAC35依赖于ABA途径调控生长素代谢关键响应因子ARF1、ARF2、ARF8 (auxin response factor, ARF)的表达水平, 促进根系生长发育,进而提高番茄的抗旱性[31]; 在水稻中, 转录因子OsNAC3[16]、OsNAC5[15]、OsNAC6[32]、OsNAC9[13]、OsNAC22[33]等的调控基因过表达均能提高转基因植株的抗旱能力, 是干旱胁迫的正调控响应因子; 相反, 转录因子OsNAC2表达量则受干旱胁迫抑制, 并且RNAi植株表现出较强的抗干旱能力, 而基因过表达则未改善植株的抗旱性, 表明OsNAC2是一个干旱胁迫的负调控转录因子[34]。在本研究中,突变体株系在田间正常种植条件下的株高、有效穗、穗长、穗粒数、结实率、千粒重、单株产量等农艺性状均与野生型ZH11无显著差异(表2), 表明转录因子OsNAC2d并未对植株正常种植调节下的生长发育产生影响。在干旱胁迫条件下,-和--突变体幼苗的生长发育受到显著抑制, 植株变得矮小、瘦弱(图6), 突变体植株的根长、苗长、根系生物量、地上部生物量等均显著低于其在正常营养液中的长势(图7和图8), 突变体植株中表达量较低, 且未被干旱胁迫诱导, 而野生型的表达水平则被干旱诱导提高到约2.5倍(图9), 表明水稻基因表达被抑制后, 植株的耐干旱能力下降, 转录因子OsNAC2d以正调控方式响应环境干旱胁迫。图7-A结果表明, 干旱胁迫显著抑制和突变体的根系延伸, 而野生型ZH11的根系伸长未受到抑制, 但干旱胁迫引起、突变体和野生型ZH11的根系生物量均较其在完全营养液中显著降低(图8-A), 表明根系作为感受土壤水分的直接器官, 更易遭受干旱胁迫损伤, 其中基因变异导致突变体水稻根系的伸长生长与物质积累均被抑制, 而野生型水稻为保持植株生长发育的水分需求, 根系维持缺水胁迫下的伸长生长, 以提升对水分的吸收。饶玉春等[35]认为, 在遭受干旱胁迫时, 水稻根系加速伸长生长以促进水分吸收, 但根系伸长生长消耗较多的碳氮同化物质, 而随着干旱胁迫程度加剧, 必然导致根系的生物量积累减少。此外, 田间干旱试验表明, 水稻早期遭遇干旱胁迫, 会导致秧苗质量下降, 分蘖发生推迟; 在分蘖至开花期遭遇干旱胁迫, 引起根系对土壤肥料吸收减少, 叶片扩展减缓, 并降低叶片的光合作用, 减少有效分蘖数和有效穗, 最终影响稻谷产量[36]。本研究通过水培试验进一步证实, 在秧苗期干旱胁迫时,、突变体等干旱胁迫敏感水稻根系的伸长和干物质积累受到抑制, 并影响了地上部组织生长。
水稻OsNAC转录因子调节干旱胁迫响应的作用机制存在一定差异, 如转录因子OsSNAC1调控叶片气孔关闭, 以减少叶片细胞的水分丢失[10]; OsNAC3能清除干旱、高温等胁迫引起的组织细胞内活性氧积累, 维持逆境胁迫下的细胞内活性氧稳态[14]; OsNAC5通过启动植株耐旱基因表达, 并调控根皮层与中柱细胞数目, 促使根系变粗, 提高抗旱能力[12-13]; OsNAC9则通过调节根系的中柱与通气组织结构, 进而改善植株的根型构造, 以维持植株的耐旱性[11]。在本研究中,基因主要在籽粒中大量表达, 其次为叶片和花药, 在根系与茎中表达量则显著低于籽粒、叶片和花药(图1-A), 并且受干旱胁迫诱导(图1-B)。Zhang等[37]报道玉米转录因子ZmNAC128和ZmNAC130在种子胚乳中高表达, 调节灌浆期种子中的淀粉和蛋白质积累过程; 水稻转录因子OsNAC127和OsNAC129在籽粒中特异性表达, 通过调控碳水化合物转运和钙信号途径而调节籽粒在干旱、高温等逆境胁迫下的灌浆过程[38], 本研究发现籽粒中高表达的转录因子OsNAC2d可能在籽粒灌浆阶段的逆境响应与种子成熟脱水过程中发挥重要调节作用。此外, 在干旱胁迫下,突变体株系的地上部生物量显著低于野生型(图8-B), 而根系生物量与野生型并无显著差异(图8-A), 表明转录因子OsNAC2d通过参与调控叶片的组织活动与生理代谢而响应环境干旱胁迫。研究结果对水稻干旱逆境适应性品种的分子改良与选育具有积极意义, 并为进一步揭示OsNAC2d响应干旱胁迫的生物学调控机制提供了合理的突变种质资源, 我们将在后续研究中进一步探究转录因子OsNAC2d的生物学功能及其在籽粒灌浆阶段的逆境响应作用。
4 结论
借助CRISPR/Cas9编辑与测序技术, 得到6种基因型纯合突变植株。突变体植株与野生型相比, 在田间正常生长条件下的株高、有效穗、穗长、穗粒数、结实率、千粒重和单株产量等农艺性状无显著差异。在干旱胁迫时,突变体植株的根系与幼苗生长、以及植株生物量积累均受到抑制, 且转录因子OsNAC2d的基因表达维持在较低水平, 而野生型水稻中的表达量则因干旱诱导而增加, 植株的生长与生物量积累未被显著抑制, 表明转录因子OsNAC2d正调控水稻的干旱胁迫响应过程。
[1] 张海淼, 李洋, 刘海峰, 孔令广, 丁新华. 水稻重要农艺性状调控基因及其育种利用研究进展. 生物技术通报, 2020, 36(12): 155–169.
Zhang H M, Li Y, Liu H F, Kong L G, Ding X H. Research progress on regulatory genes of important agronomic traits and breeding utilization in rice., 2020, 36(12): 155–169 (in Chinese with English abstract).
[2] 张洪程, 胡雅杰, 杨建昌, 戴其根, 霍中洋, 许轲, 魏海燕, 高辉, 郭保卫, 邢志鹏, 胡群. 中国特色水稻栽培学发展与展望. 中国农业科学, 2021, 54: 1301–1321.
Zhang H C, Hu Y J, Yang J C, Dai Q G, Huo Z Y, Xu K, Wei H Y, Gao H, Guo B W, Xing Z P, Hu Q. Development and prospect of rice cultivation in China., 2021, 54: 1301–1321 (in Chinese with English abstract).
[3] Bernier J, Atlin G, Serraj R, Kumar A, Spaner D. Breeding upland rice for drought resistance., 2008, 88: 927–939.
[4] 王英, 张浩, 马军韬, 张丽艳, 邓凌伟, 王永力, 高晶, 张国民.水稻抗旱研究进展与展望. 热带作物学报, 2018, 39: 1038–1043.
Wang Y, Zhang H, Ma J T, Zhang L Y, Deng L W, Wang Y L, Gao J, Zhang G M. Advances in drought tolerance of rice., 2018, 39: 1038–1043 (in Chinese with English abstract).
[5] Zhang Q F. Strategies for developing green super rice., 2017, 104: 16402–16409.
[6] 郭韬, 余泓, 邱杰, 李家洋, 韩斌, 林鸿宣. 中国水稻遗传学研究进展与分子设计育种. 中国科学: 生命科学, 2019, 49: 1185–1212.
Guo T, Yu H, Qiu J, Li J Y, Han B, Lin H X. Advances in rice genetics and breeding by molecular design in China., 2019, 49: 1185–1212 (in Chinese with English abstract).
[7] Aida M, Ishida T, Fukaki H, Fujisawa H, Tasaka M. Genes involved in organ separation in: an analysis of the cup-shaped cotyledon mutant., 1997, 9: 841–857.
[8] Souer E, Houwelingen A V, Kloos D, Mol J, Koes R. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries., 1996, 85: 159–170.
[9] 柳展基, 邵凤霞, 唐桂英. 植物NAC转录因子的结构功能及其表达调控研究进展. 西北植物学报, 2007, 27: 1915–1920.
Liu Z J, Shao F X, Tang G Y. The research progress of structure, function and regulation of plant NAC transcription factors., 2007, 27: 1915–1920 (in Chinese with English abstract).
[10] 李鹏, 黄耿青, 李学宝. 植物NAC转录因子. 植物生理学通讯, 2010, 46: 294–300.
Li P, Huang G Q, Li X B. Plant NAC transcription factors., 2010, 46: 294–300 (in Chinese).
[11] Nuruzzaman M, Manimekalai R, Sharoni A M, Satoh K, Kondoh H, Ooka H, Kikuchi S. Genome-wide analysis of NAC transcription factor family in rice., 2010, 465: 30–44.
[12] Hu H H, Dai M Q, Yao J L, Xiao B Z, Li X H, Zhang Q F, Xiong L Z. Overexpressing a NAM, ATAF, and CUC (NAC) transcrition factor enhances drought resistance and salt tolerance in rice., 2006, 103: 12987–12992.
[13] Redillas M C, Jeong J S, Kim Y S, Jung H, Bang S W, Choi Y D, Ha S H, Reuzeau C, Kim J K. The overexpression ofalters the root architecture of rice plants enhancing drought resistance and grain yield under field conditions., 2012, 10: 792–805.
[14] Takasaki H, Maruyama K, Kidokoro S, Ito Y, Fujita Y, Shinozaki K, Yamaguchi-Shinozaki K, Nakashima K. The abiotic stress-responsive NAC-type transcription factorregulates stress-inducible genes and stress tolerance in rice., 2010, 284: 173–183.
[15] Jeong J S, Kim Y S, Redillas M C Jang G, Jung H, Bang S W, Choi Y D, Ha S H, Reuzeau C, Kim J K.overexpression enlarges root diameter in rice plant leading to enhanced drought tolerance and increased grain yield in the field., 2013, 11: 101–114.
[16] Fang Y J, Liao K F, Du H, Xu Y, Song H Z, Li X H, Xiong LZ. A stress-responsive NAC transcription factorconfers heat and drought tolerance through modulation of reactive oxygen species in rice., 2015, 66: 6803–6817.
[17] Li Z, Pan X, Guo X, Fan K, Lin W. Physiological and transcriptome analyses of early leaf senescence formutant rice (L.) during the grain-filling stage., 2019, 20: 1098.
[18] 李兆伟, 零东兰, 孙聪颖, 曾慧玲, 刘凯基, 蓝颖珊, 范凯, 林文雄. 利用CRISPR/Cas9技术定向编辑水稻. 中国农业科学, 2021, 54: 2699–2709.
Li Z W, Ling D L, Sun C Y, Zeng H L, Liu K J, Lan Y S, Fan K, Lin W X. CRISPR/Cas9 targeted editing ofin rice., 2021, 54: 2699–2709 (in Chinese with English abstract).
[19] Ma X L, Zhang Q, Zhu Q, Liu W, Chen Y, Qiu R, Wang B, Yang Z, Li H, Lin Y, Xie Y, Shen R, Chen S, Wang Z, Chen Y, Guo J, Chen L, Zhao X, Dong Z, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants., 2015, 8: 1274–1284.
[20] 沈少炎, 吴玉香, 郑玉善. 植物干旱胁迫响应机制研究进展——从表型到分子. 生物技术进展, 2017, 7(3): 169–176.
Shen S Y, Wu Y X, Zheng Y S. Review on drought response in plants from phenotype to molecular., 2017, 7(3): 169–176 (in Chinese with English abstract).
[21] Belhaj K, Chaparro-Garcia A, Kamoun S, Patron N J, Nekrasov V. Editing plant genomes with CRISPR/Cas9., 2015, 32: 76–84.
[22] Bhatta B P, Malla S. Improving horticulture cropsCRISPR/ Cas9: current successes and prospects.(Basel), 2020, 9: 1360.
[23] Yimam Y T, Zhou J, Akher S A, Zheng X, Qi Y, Zhang Y. Improving a quantitative trait in rice by multigene editing with CRISPR-Cas9., 2021, 2238: 205–219.
[24] Rao MJ, Wang L. CRISPR/Cas9 technology for improving agronomic traits and future prospective in agriculture., 2021, 254: 68.
[25] Ren C, Liu Y, Guo Y, Duan W, Fan P, Li S, Liang Z. Optimizing the CRISPR/Cas9 system for genome editing in grape by using grape promoters., 2021, 8: 52.
[26] 黄忠明, 周延彪, 唐晓丹, 赵新辉, 周在为, 符星学, 王凯, 史江伟, 李艳锋, 符辰建, 杨远柱. 基于CRISPR/Cas9技术的水稻温敏不育基因突变体的构建. 作物学报, 2018, 44: 844–851.
Huang Z M, Zhou Y B, Tang X D, Zhao X H, Zhou Z W, Fu X X, Wang K, Shi J W, Li Y F, Fu C J, Yang Y Z. Construction ofmutants in rice based on CRISPR/Cas9 technology., 2018, 44: 844–851 (in Chinese with English abstract).
[27] 季新, 李飞, 晏云, 孙红正, 张静, 李俊周, 彭廷, 杜彦修, 赵全志. 基于CRISPR/Cas9系统的水稻光敏色素互作因子基因编辑. 中国农业科学, 2017, 50: 2861–2871.
Ji X, Li F, Yan Y, Sun H Z, Zhang J, Li J Z, Peng T, Du Y X, Zhao Q Z. CRISPR/Cas9 system-based editing of phytochrome- interacting factor., 2017, 50: 2861–2871 (in Chinese with English abstract).
[28] 黄前量, 徐庆国, 殷绪明. 水稻NAC转录因子基因的克隆与功能初步分析. 分子植物育种, 2022, 20: 5229–5235.
Huang Q L, Xu Q G, Yin X M. Cloning and functional analysis of, a NAC transcription factor gene from rice., 2022, 20: 5229–5235.
[29] 王春雨, 张茜. 植物NAC转录因子功能研究进展. 生物技术通报, 2018, 34(11): 8–14.
Wang C Y, Zhang Q. Research progress on plant NAC transcription factors., 2018, 34(11): 8–14 (in Chinese with English abstract).
[30] 张慧珍, 白雪芹, 曾幼玲. 植物NAC转录因子的生物学功能. 植物生理学报, 2019, 55: 915–924.
Zhang H Z, Bai X Q, Zeng Y L. Biological functions of plant NAC transcription factors., 2019, 55: 915–924 (in Chinese with English abstract).
[31] Tran L S, Nakashima K, Sakuma Y, Simpson S D, Fujita Y, Maruyama K, Fujita M, Seki M, Shinozaki K, Yamaguchi-Shinozaki K. Isolation and functional analysis ofstress inducible NAC transcription factors that bind to a drought responsive-element in the early responsive to dehydration stress 1 promoter., 2004, 16: 2481–2498.
[32] Wang G, Zhang S, Ma X, Wang Y, Kong F, Meng Q. A stress-associated NAC transcription factor (SlNAC35) from tomato plays a positive role in biotic and abiotic stresses., 2016,158: 45–64.
[33] Nakashima K, Tran L P, Nguyen D V, Fujita M, Maruyama K, Todaka D, Ito Y, Hayashi N, Shinozaki K, Yamaguchi-Shinozaki K. Functional analysis of a NAC-type transcription factor OsNAC6 involved in abiotic and biotic stress-responsive gene expression in rice., 2007, 51: 617–630.
[34] Hong Y, Zhang H, Huang L, Li D, Song F. Overexpression of a stress-responsive NAC transcription factor geneimproves drought and salt tolerance in rice., 2016, 7: 4.
[35] 罗莉琼, 吕波, 陈旭, 李捷, 明凤.通过ABA依赖途径负调控水稻的多种非生物胁迫反应. 复旦学报(自然科学版), 2016, 55(1): 89–96.
Luo L Q, Lyu B, Chen X, Li J, Ming F.negatively regulates various abiotic stress through ABA-dependent pathway in rice.(Nat Sci Edn), 2016, 55(1): 89–96 (in Chinese with English abstract).
[36] 饶玉春, 戴志俊, 朱怡彤, 姜嘉骥. 水稻抗干旱胁迫的研究进展. 浙江师范大学学报(自然科学版), 2020, 43(4): 417–429.
Rao Y C, Dai Z J, Zhu Y T, Jiang J J. Advances in research of drought resistance in rice.(Nat Sci Edn), 2020, 43(4): 417–429 (in Chinese with English abstract).
[37] 梅德勇, 王士梅, 朱启升, 陈秀晨, 龚存力, 韩云芳, 于智坤. 水稻抗旱性遗传生理机制及育种研究进展. 安徽农学通报, 2016, 22(22): 10–14.
Mei D Y, Wang S M, Zhu Q S, Chen X C, Gong C L, Han Y F, Yu Z K. Progress on the genetic and physiological mechanism of rice drought resistance and breeding strategies., 2016, 22(22): 10–14 (in Chinese with English abstract).
[38] Zhang Z, Dong J, Ji C, Wu Y, Messing J. NAC-type transcription factors regulate accumulation of starch and protein in maize seeds., 2019, 116: 11223–11228.
[39] Ren Y, Huang Z, Jiang H, Wang Z, Wu F, Xiong Y, Yao J. A heat stress responsive NAC transcription factor heterodimer plays key roles in rice grain filling., 2021, 72: 2947–2964.
Construction of rice mutants by gene editing ofand their response to drought stress
LI Zhao-Wei1,2, MO Zu-Yi1,2, SUN Cong-Ying1,2, SHI Yu1,2, SHANG Ping2,3, LIN Wei-Wei1,2, FAN Kai2,3, and LIN Wen-Xiong1,2,*
1College of Life Sciences, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;2Fujian Provincial Key Laboratory of Agroecological Processing and Safety Monitoring, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;3College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China
The main objective of this study is to explore the biological function of transcription factor OsNAC2d and its effect on drought resistance in rice. The sequence ofwas firstly edited invariety Zhonghua 11 through the CRISPR/Cas9 technology. The agronomic traits ofmutants were investigated in normal field cultivation, and the growth status and relative expression level ofgenes were performed in the young mutant seedlings under the drought condition. There was the higher expression level ofin grain, leaf, and anther than those in root and stem, and the relative expression ofwas enhanced by drought stress. Six homozygous T2lines were identified in two editing target sites ofgenomic sequence through the nucleotide sequencing technology. There was no significant difference betweenmutants and wild type in the agronomic traits including plant height, effective panicle number per plant, panicle length, grain number per panicle, seed setting rate, 1000-grain weight, and grain yield per plant. Under the drought condition, the growth of roots and seedling ofmutants were depressed, and the biomass of roots and above-ground were also decreased. Meanwhile, the relative expression ofin themutant retained as extremely low level as those in the normal cultivating condition. However, the relative expression ofin wild type was enhanced by drought stress, and the growth and biomass accumulation of wild type were not evidently hindered by drought stress. These results indicated that OsNAC2d positively regulated the drought response in rice. Themutants in this present study provided a precious genetic resource for deeply revealing the biological function of OsNAC2d and its fine regulating mechanism in response to drought stress.
rice; drought stress; gene editing;gene
10.3724/SP.J.1006.2023.12076
本研究由福建省自然科学基金项目(2021J01098, 2022J01142)资助。
This study was supported by the Natural Science Foundation of Fujian Province (2021J01098, 2022J01142).
林文雄, E-mail: wenxiong181@163.com
E-mail: lizw197@163.com
2021-11-05;
2022-06-07;
2022-07-08.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220707.1349.008.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/)

