祁连山黄参叶片转录组测序及生物信息学分析
张春梅,闫 芳,宋 海,张喜峰1,,陈 叶1,
(1. 河西学院 农业与生态工程学院,甘肃 张掖 734000;2. 甘肃省河西走廊特色资源利用重点实验室,甘肃 张掖 734000;3. 河西学院 生态与绿洲农业研究院,甘肃 张掖 734000)
黄参Sphallerocarpusracills,伞形科Apiaceae迷果芹属Sphallerocarpus的单种植物[1],在我国零星分布于西北、东北、华北地区,尤其分布于祁连山、焉支山(甘肃张掖市境内),像张掖市山丹县这样大面积形成群落优势的情况很罕见[2]。研究黄参对祁连山区水土保持、遏制草场沙漠化和维持生物多样性具有重要的生态和经济意义。《本草纲目》中记载,黄参具有补气养血、滋补肝肾、通经活络等功效,其肉质根富含人体必需的16种氨基酸,被誉为“小人参”[3],作为营养丰富、经济、药用价值极高的天然珍品,黄参成为甘肃特产、西部开发交易会指定产品,在食品、医药、化工等领域具有极大的应用潜力与开发前景。由于生态环境恶化及当地百姓的掠夺式经营,野生黄参种群数量骤减[3]。
绝大部分非模式生物缺乏基因组数据,因此,获得转录组学信息尤为重要[4-7]。转录组学是获取基因序列的首选方式[8-9],非常有利于研究无参考基因组的非模式植物[10-11],研究转录组学有助于揭示生物体的基因表达、研究结构变异、新基因以及植物的优良性状及功能基因的定位[12-15]。
目前,濒危野生黄参种质的功能基因组、基因序列信息及遗传背景匮乏,对黄参的抢救性保护及其遗传结构研究迫切需要基因组资源。关于黄参转录组信息尚未见报道,本研究采用BGISEQ-500平台,对黄参幼嫩叶片进行转录组测序,研究黄参转录组,并结合生物信息学对获得的Unigene(转录组数据库序列)开展功能注释、代谢通路和EST-SSR分析,获得的转录组信息将为今后黄参分子标记的开发和关键基因的克隆以及功能分析等提供科学数据,为黄参基因组水平的研究奠定基础。
1 材料和方法
1.1 实验材料
材料采自甘肃省张掖市山丹县军马场。野生黄参,于2018年5月进行单株取样,采集当年刚生长出的幼嫩叶片,迅速放入液氮速冻后,保存于-80 ℃冰箱中。
1.2 主要试剂及仪器
1.2.1 主要仪器
超级工作台(苏州净化工作台设备有限公司);多样品组织研磨仪(上海净信实业发展有限公司);SANYO制冰机(济南金茂科创科技有限公司);冷冻离心机(CL-21R,Thermo,美国);超声破碎仪(FS-150,Ultrasonic processor,中国);震荡仪(Shaker,Thermo,美国);金属浴(GL-150,其林贝尔,中国);分析天平(BSA224S,Sartorius,德国);Bio-Rad凝胶成像系统(麦克森公司);NanoDrop 2000微量分光亮度计(Thermo Scientific, USA);Agilent 2100 Bioanalyzer(美国安捷伦公司);NovaseqTM6000(美国Illumina公司)。
1.2.2 主要试剂
TRIzol®Reagent购自Invitrogen公司;氯仿、异丙醇、乙醇等化学试剂购自天津市大茂化学试剂厂,分析纯;NovaSeqTM6000 v1.5试剂盒(美国Illumina公司)。
1.3 实验方法及步骤
1.3.1 黄参叶片RNA 提取及转录组测序
采集当年刚生长出的幼嫩叶片,液氮速冻并保存于-80 ℃冰箱中,以保证RNA的相态保持在刚采摘时的状态。采用试剂盒法,Trizol试剂,TIANGEN提取方法参照试剂说明。叶肉细胞移入1.5 mL 离心管中,加入1 mL Trizol,混匀,室温静置5 min;加入0.2 mL氯仿,振荡15 s, 静置2 min;4 ℃12 000 g离心15 min,裂解液分层成水相和有机相,取上清;加入0.5 mL异丙醇,将管中液体轻轻混匀,室温静置10 min;4 ℃ 12 000 g 离心10 min,弃上清,水相转移后,加入1 mL体积分数为75%乙醇,轻轻洗涤沉淀。4 ℃ 7 500 g离心5 min,短暂离心,吸去残留液体,弃上清;晾干3~5 min;将获得的RNA沉淀溶于30 μL的DEPC(焦炭酸二乙酯)水中。DEPC是RNA酶的强抑制剂,是一种潜在的致癌物质,操作中应在通风条件下进行,并避免接触皮肤。
1.3.2 cDNA 文库构建及转录组序列组装
取一定量的RNA 样品,使用 oligo(dT)磁珠富集mRNA。加入试剂盒中提供的打断试剂,适温反应一定时间后mRNA片段化。合成一链、二链cDNA。配制反应体系,使接头与cDNA连接。PCR反应及产物回收、扩增。PCR产物变性,充分混匀,得到单链环形产物,PCR产物变性,即得到文库。文库质量委托陕西致研生物科技有限公司使用 Agilent 2100 Bioanalyzer 检测,检测合格后采用BGISEQ-500测序。利用Trinity软件对reads进行序列组装,使用BUSCO软件对组装序列进行质量评估,评估基因组装完整性。
1.3.3 功能注释及SSR检测
利用生物信息学分析获得的黄参Unigene。为获得全面的基因功能信息,对组装得到的Unigene进行7大功能数据库注释,包括NR(NCBI non redundant protein sequences)、NT(NCBI nucleotide sequences)、KOG/COG(clusters of orthologous groups of proteins/eu-Karyotic ortholog groups)、GO (gene ontology,基因本体)、KEGG(kyoto encyelopedia of genes and genomes)、SwissProt (reviewed protein sequence database)和Pfam(protein family)。使用MISA对Unigene进行检测,软件参数为1-12、2-6、3-5、4-5、5-4、6-4、100,150,其中:1-12代表单碱基重复至少12次才算SSR,100表示2个SSR之间的间隔碱基数大于100 bp,150表示SSR位点距离两端侧翼序列大于150 bp。双碱基6次,三碱基5次,以此类推,重复单元最多有6个碱基,2个微卫星之间的距离小于100 bp。随机选取10对引物,委托陕西致研生物科技有限公司合成。
2 结果与分析
2.1 高通量测序、de novo组装
测序共获得70.24 M原始读数,去除低质量的reads,得到68.48 M clean reads,最终获得6.85 Gb。质量评估结果见表1,转录本和Unigene组装统计结果见表2。由表可见,测序得到的黄参数据质量较高,可以满足后续的生物信息学分析。其中,长度为200~300 bp的有25 343条Unigene(40.66%),长度为>300~500 bp的有16 463条Unigene(26.42%),长度为>500~3 000 bp的有15 276条Unigene(24.51%),5 241条Unigene(10.01%)的长度大于3 000 bp,由此可知,随着基因长度增加,基因数量下降。使用Transdecoder检测出62 323个CDS(见表2)。

表1 黄参测序后的质量评估Tab. 1 Quality evaluation of-sequencing output data in Sphallerocarpus racills
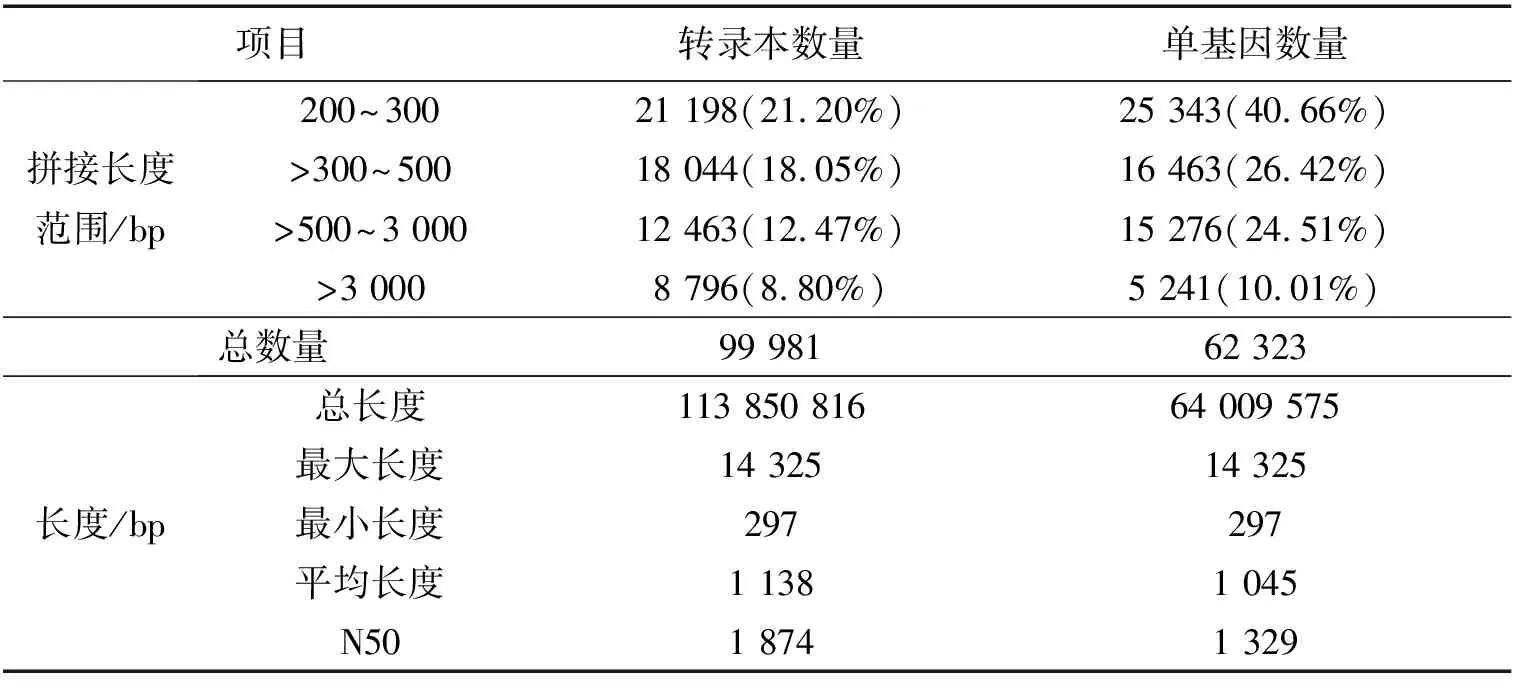
表2 黄参测序后的转录本和Unigene组装统计Tab. 2 Data assembly for transcript and unigene in transcriptome of Sphallerocarpus racills
2.2 黄参转录组基因总体注释情况
对黄参转录组基因进行7大功能数据库注释,结果见表3。结果显示,注释成功的Unigene数目最多的是NR(e≤10-5),有66 451条,占总Unigene的66.46%;NT(e≤10-5)有49 390条,占49.40%;Swissprot(e≤10-5)有48 281条,占48.29%;KEGG(e≤10-10)有51 479条,占51.49%; KOG(e≤10-3)有61 116条,占61.13%;Pfam(e≤0.01)有55 859条,占55.87%。GO(e≤10-3)数据库注释到的基因最少,有36 958条,占36.97%;比对结果显示,在7大数据库中均能成功注释的Unigene有17 074条,占总Unigene的17.08%。
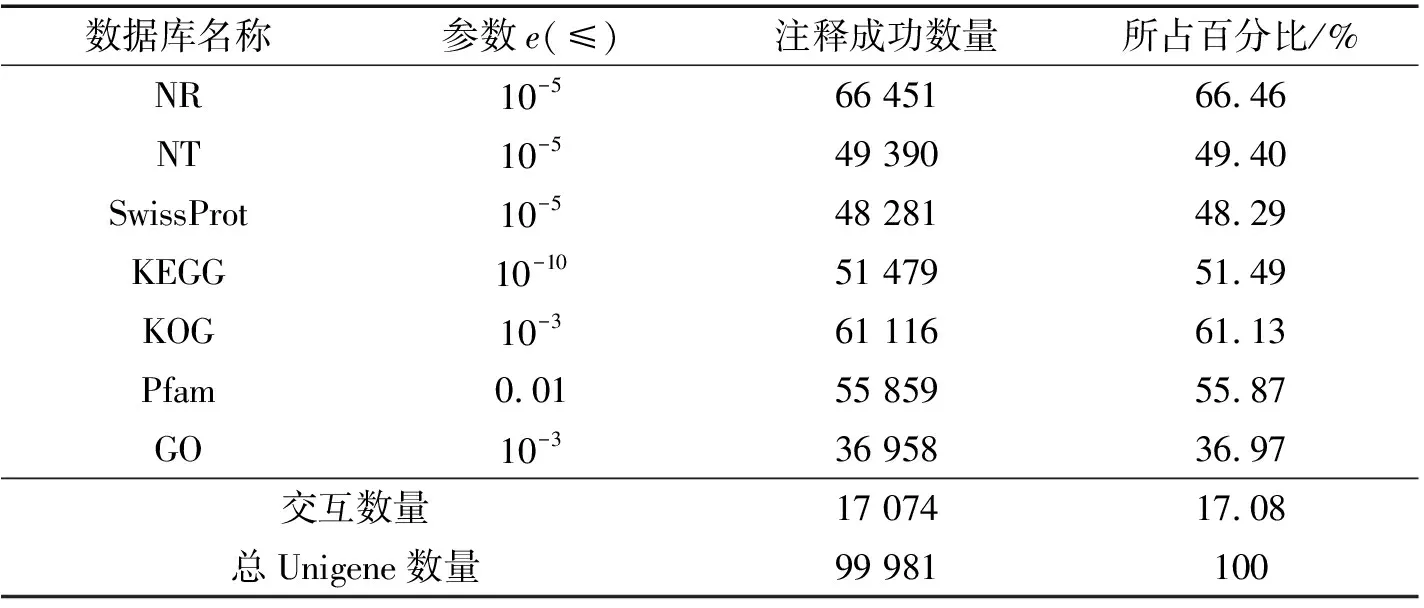
表3 黄参转录组基因注释情况统计Tab. 3 Statistics of gene annotation of Sphallerocarpus racills transcriptome
2.2.1 NR功能注释
NR库注释结果的物种分布统计结果见图1。图1显示,匹配最多的物种是胡萝卜Daucuscarotasubsp.sativus,注释到的基因数量最多,共有45 239条,占比为68.08%,同源性最高;其余依次为大麦Hordeumvulgaresubsp.vulgare、向日葵Helianthusannuus、蓝隐藻GuillardiathetaCCMP2712、轮藻Klebsormidiumnitens,分别有963、711、638、524条Unigene,占比分别为1.45%、1.07%、0.96%、0.85%,黄参与这4种植物的同源性均相对较低,同源序列都不足2%,剩下近30%分布于其他物种,共有18 333条Unigene(占27.59%)属于其他序列。
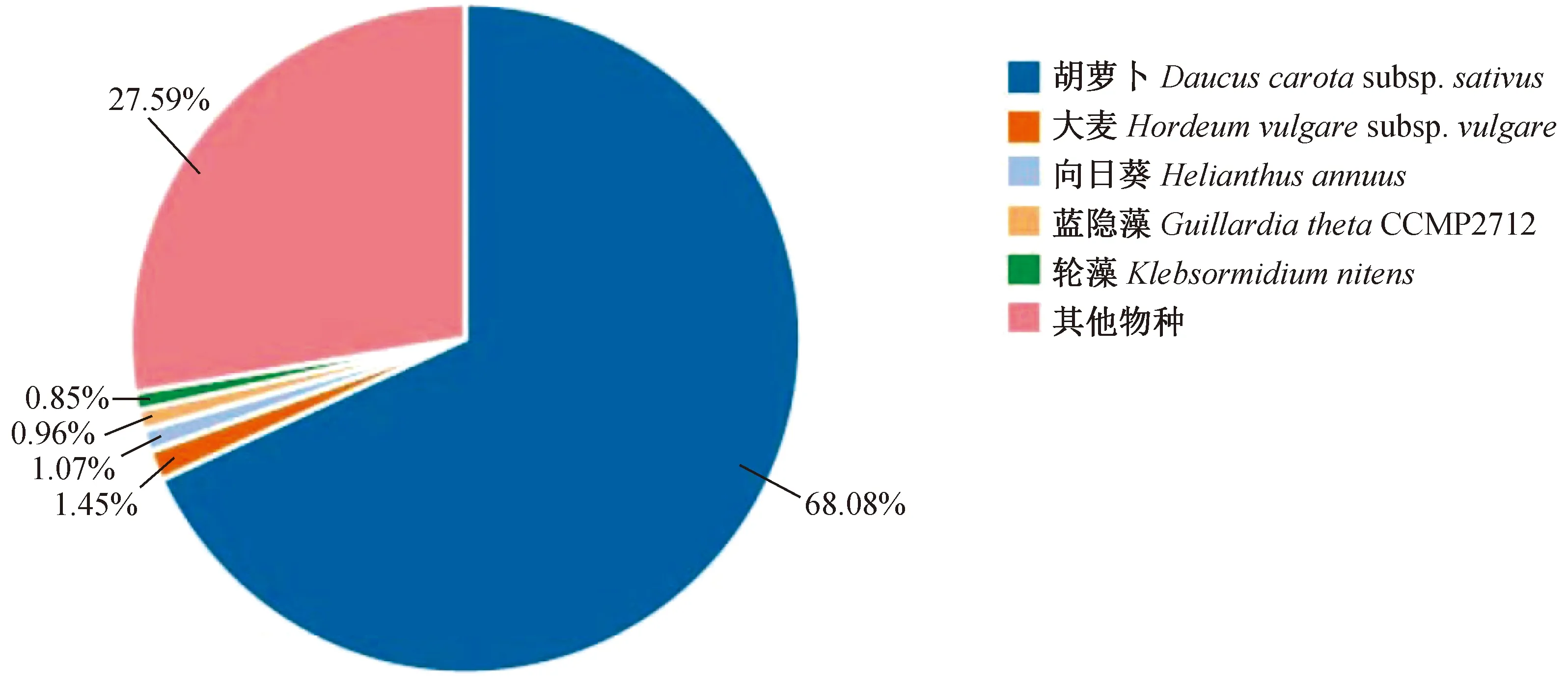
图1 根据NR注释结果统计注释不同物种的分布Fig. 1 Statistics of species distribution of Unigenes annotation in NR database for Sphallerocarpus racills
2.2.2 GO功能注释分类
注释到生物学过程大类(共15个亚类)的基因有18 317条,其中,占比最高的亚类为细胞过程(cellular process),注释数量为11 026,其次为生物调节过程(biological regulation),注释数量为3 710,有关细胞增殖、碳利用等的基因表达甚少。
注释到细胞组分大类(共11个亚类)的基因有25 108条,其中,细胞(cell)和细胞膜组分(membrane part)占比最高,分别为11 062和10 341条,再次是细胞器部分(organelle part),基因为3 512条,而定位于病毒核心、细胞器的基因几乎未表达。
注释到分子功能大类(共14个亚类)的基因为34 615条,其中,最具有代表性的是参与分子结合功能(binding),注释数量最多(18 119),其次是与催化活性(catalytic activity)相关的基因,注释数量为16 074,而与蛋白质标签、分子载体活性、分子转运活性、养分库活动、毒素活性相关的基因几乎未表达(见图2)。
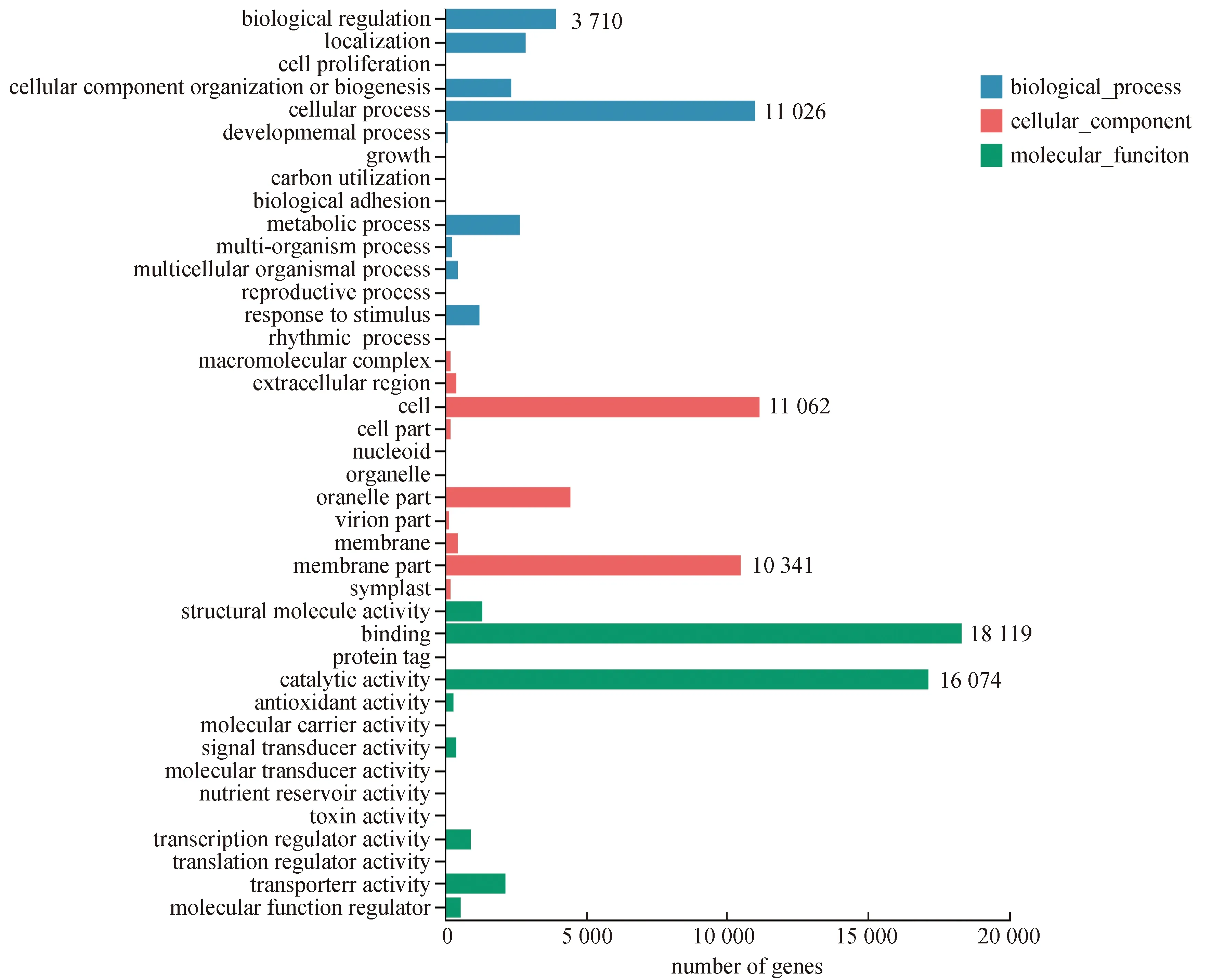
图2 Unigene的GO功能分类Fig. 2 GO function classification of Unigene
2.2.3 代谢通路分析
KEGG注释结果见图3,注释到51 479条Unigene,占总Unigene的51.49%。注释成功的所有Unigene归属到5大类代谢通路中的20条通路。11条与代谢有关的通路,有15 162个Unigene,占29.45%:以全局和总览图代谢通路(global and overview maps)在所有代谢通路中所占比例最高(11 346个,22.04%)。其次为碳水化合物代谢通路(4 137个,8.04%)和脂类代谢通路(2 618个,5.09%)。与遗传信息处理相关的代谢通路有4条,其中以翻译过程(translation,4 341,8.43%)所占比例最高,其次是折叠、分类与降解(folding, sorting and degradation,3 851,7.49%)代谢通路。与环境信息处理相关的通路有2条——信号转导(signal transduction)和膜运输(membrane transport),分别为2 489、446条。细胞过程和生物系统的相关通路最少,各有1条,占4.67%和3.30%。

图3 黄参转录组Unigene的代谢通路功能分布统计Fig. 3 KEGG classification of Sphallerocarpus racills transcriptome
2.2.4 KOG注释分类
KOG数据库包含了7个完整基因组真核生物的直系同源家族蛋白质。黄参转录组有61 116条Unigeine获得了注释,占总Unigene的61.13%,分为26大类功能区,包括功能预测、碳水化合物运输与代谢、翻译后修饰、蛋白质运输、分子加工、信号转导代谢等蛋白质家族。在不同的功能分类中,基因数量存在明显差异,一般功能预测类基因数量最多(11 205,18.33%),其次是信号转导机制(7 618,12.46%)、翻译后修饰、蛋白质运输(5 306,8.68%)、未知功能(5 019,8.21%)、转录(4 006,6.56%)、翻译、核糖体结构和生物发生(3 694,6.04%)、胞内转运、分泌和囊泡运输(3 616,5.92%)、RNA加工和修饰(3 412,5.59%)。除此之外,负责碳水化合物运输与代谢有2 510个Unigene(4.11%),负责脂类运输和代谢有2 391个Unigene(3.91%),同时1 984个Unigene负责能源生产与转换。氨基酸转运与代谢、细胞骨架、次生代谢产物生物合成与转运、复制、重组与修复、细胞壁/膜/包膜生物发生、细胞分裂、核苷酸转运和代谢、防御机制及辅酶转运与代谢匹配数目较少(均小于3%)(图4)。只有极少数Unigene负责细胞运动、细胞核结构(均小于0.13%)。由此可见,黄参中功能预测的基因最多,信号转导、翻译、修饰及蛋白质运输参与的基因次之,细胞运动、细胞核结构最少。
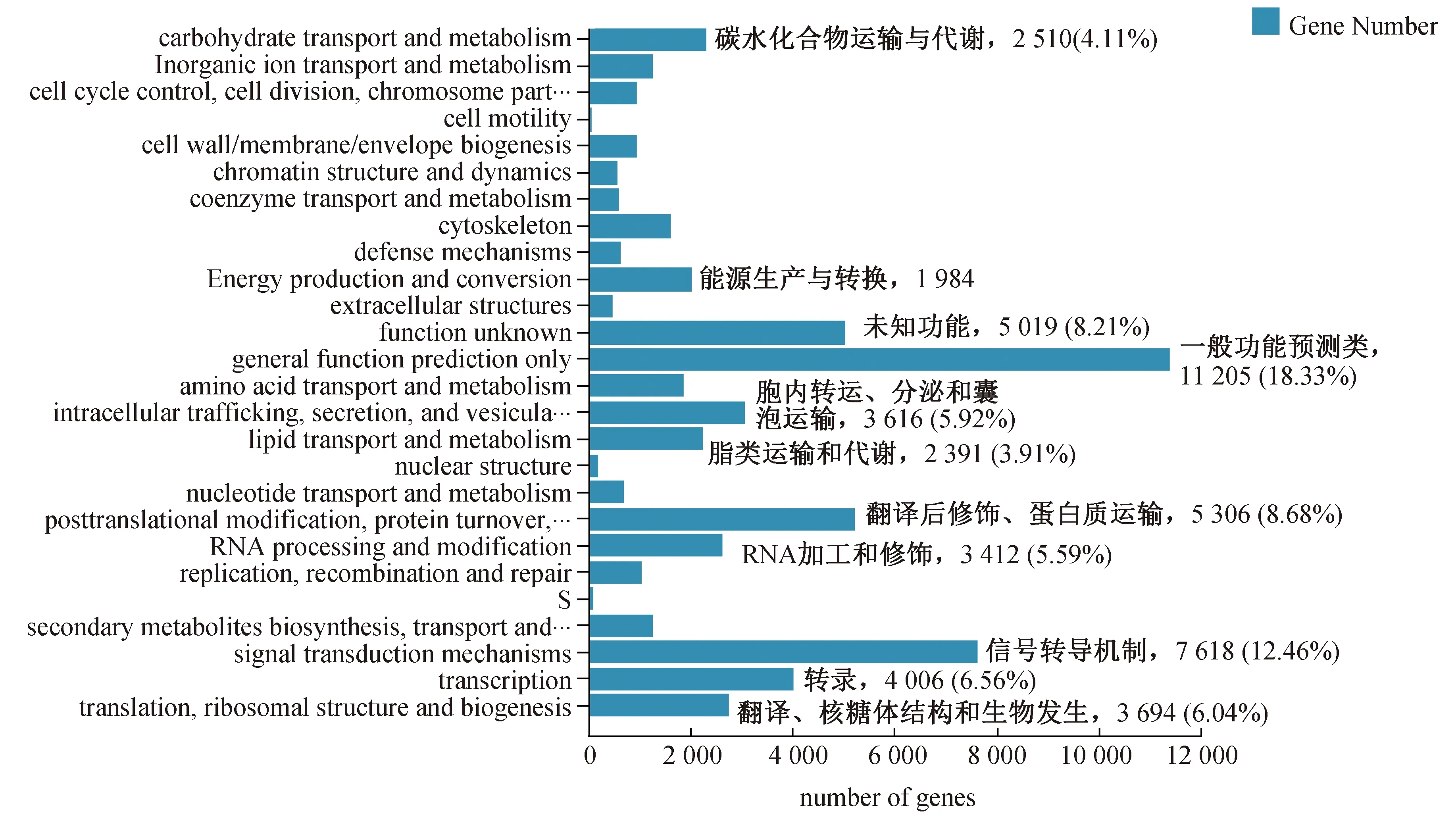
图4 黄参转录组Unigene的KOG注释分类Fig. 4 KOG classification of Sphallerocarpus racills
2.3 基因转录因子分析
植物转录因子(transcription factor,TF)也称为反式作用因子,是在转录过程中发挥重要作用的蛋白质,与应答生物和非生物胁迫密切相关。与逆境胁迫相关的转录因子主要有MYB类、bZIP类、WRKY类、AP2/EREBP类和NAC类5个大家族[16]。对黄参转录因子家族进行分类统计,结果见图5,共预测到2 370个编码转录因子的Unigene,分布在57个转录因子家族中。由图5可知,C2H2属于最大家族,Unigene数量为268个,占总Unigene数量的11.31%;其次是MYB、WRKY、BHLH、C3H、AP2-EREBP、NAC等转录因子家族较多,Unigene数量分别为220、154、145、130、121、110,分别占9.28%、6.49%、6.10%、5.49%、5.06%、4.64%。转录因子的分析可从功能基因组的水平上为进一步开展黄参研究提供数据支持。

图5 黄参转录因子家族分布Fig. 5 Transcription factor family distributionin Sphallerocarpus racills
2.4 黄参转录组SSR检测分析
对黄参转录组Unigene的简单序列重复SSR(simple sequence repeat)进行检测,结果显示17 308个SSR分布于13 256个Unigene中。二碱基重复的数量最多(6 721,38.83%),其次是三碱基重复(6 302,23.21%),其余为单碱基重复(3 378,19.52%)、五碱基重复(363,2.10%),最少的是四碱基和六碱基重复,均为272,占1.57%(见表4)。二碱基SSR中,重复频率最高是TA、AT和TC,最低是GC;三碱基重复总共60种,发生频率最高的是CAA、TGTTTG,最低的是CGA和CGG;四碱基重复频率最高是AAAT、CACT和TTTG,最低是ACAG。重复次数越多,多态性越高,多态性位点较多的是二、三碱基重复,去除SSR位点靠前或靠后的序列,将序列输入Primer3引物设计软件,对其中的10对引物进行设计,结果见表5。
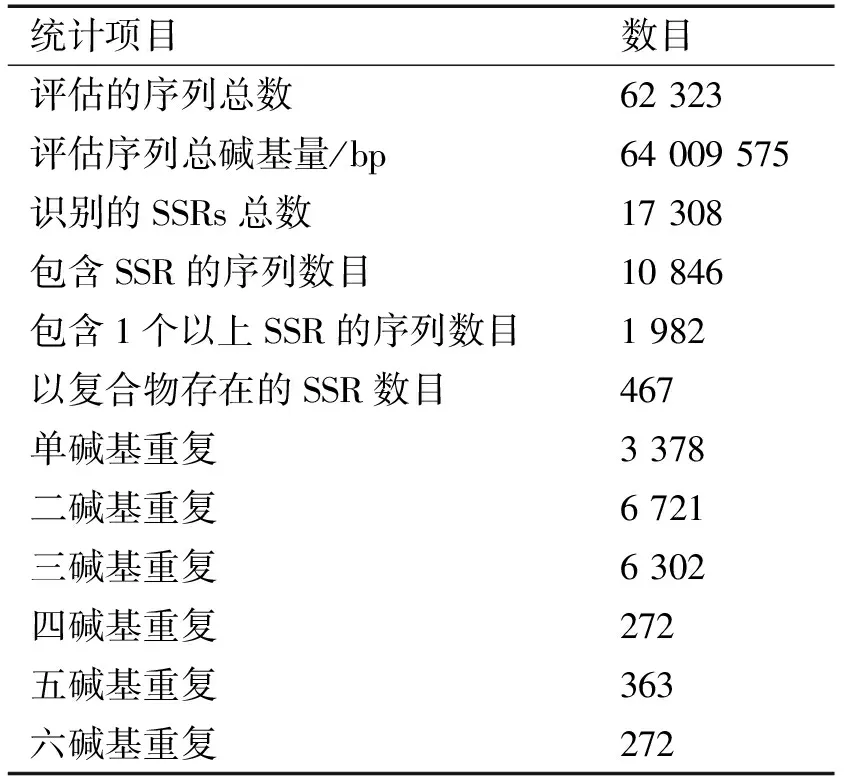
表4 黄参SSR分析结果统计Tab. 4 Summary of simple sequence repeat (SSR) in Sphallerocarpus racills
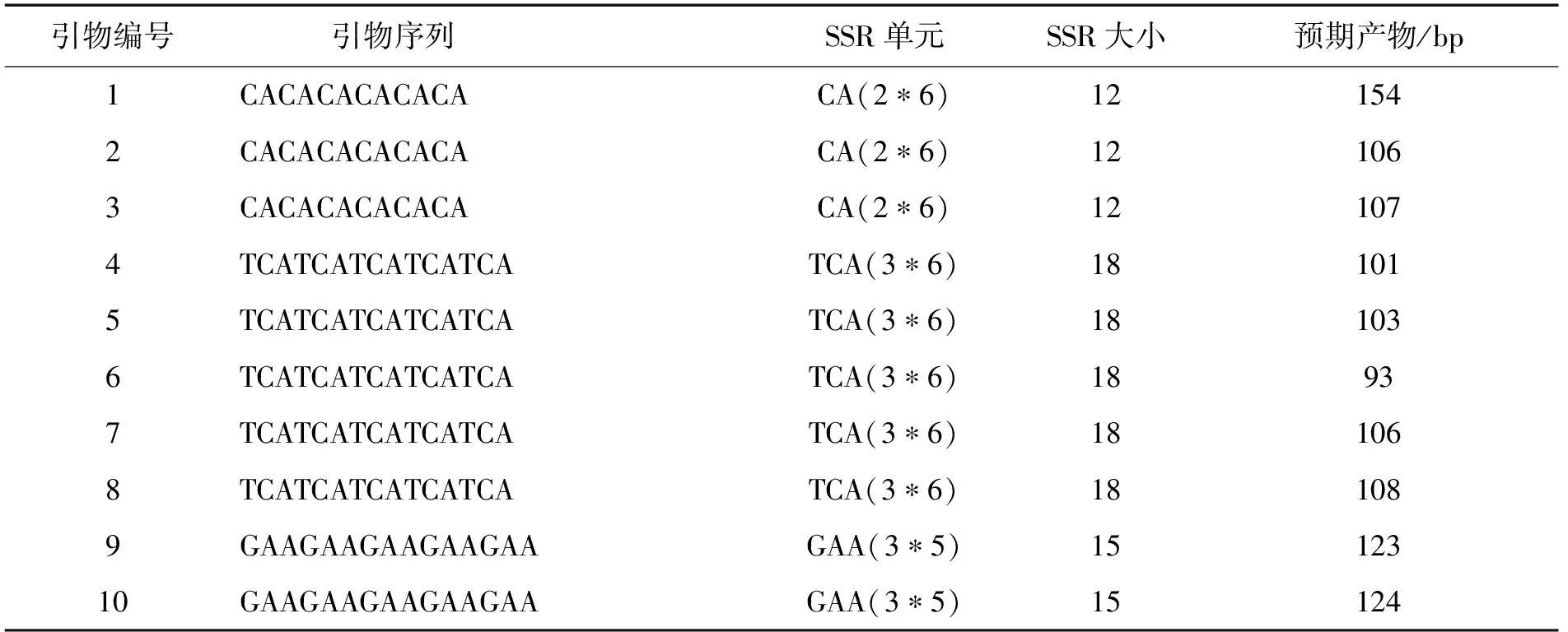
表5 黄参SSR部分引物(10对)设计表Tab. 5 Information of partial primers of development in Sphallerocarpus racills (10 pairs)
3 讨论与结论
高通量测序深受研究者欢迎,并越来越多地应用第二代测序技术来解决生物学问题。例如,在基因组水平上对还没有参考序列的物种进行从头测序,获得该物种的参考序列。该技术已广泛应用到植物特殊功能基因的挖掘与鉴定[17],为后续研究和分子育种奠定基础。本研究采用BGISEQ-500平台对黄参叶片进行转录组测序,获得了野生黄参功能基因组信息。全长非嵌合序列中存在大量的冗余序列,将其聚类到一起进行去冗余,得到新的一致性序列,然后将非全长序列比对到一致性序列上进行校正,最终得到准确度大于99%的高质量序列,即为转录本。本研究最终得到总有效碱基数6.85 Gb,得到99 981个Unigene,转录本总长度113 850 816 bp,平均长度1 138 bp,N50的长度1 874 bp,GC含量39.93%。N50是评价组装序列完整性的重要指标,N50越长,代表组装的完整性越好。本研究结果显示,黄参Q20(碱基正确识别率达99%)序列占96.42%,Q30(碱基正确识别率达99.9%)高质量序列占92.09%,碱基错误率为0.01%,低于1%,表明所获得的黄参转录组序列质量较高。
将黄参Unigene比对到7大功能数据库进行注释,其中:1)黄参Unigene比对到NR数据库共有66 451条注释成功,与胡萝卜有较高同源性,而与其他物种的同源性较低。2)GO分析显示,黄参有78 040条Unigene得到注释,按功能分为生物过程、细胞组分、分子功能3大类,分别有15、11、14个亚类,最富集的通路主要是在生物学过程中。3)在KOG数据库比对分析结果中,黄参共有61 116条Unigeine获得了注释。26个可能的功能大类中,黄参功能预测的基因最多,信号转导、翻译、修饰及蛋白质运输参与的基因次之,细胞运动、细胞核结构最少。4)KEGG是系统分析基因功能、基因组信息的数据库,是进行生物体内代谢分析、代谢网络研究的强有力工具,涵盖了药物开发(drug development)、细胞过程(cellular processes)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)、人类疾病,仅限动物(human diseases)、代谢(metabolism)、生物系统(organismal systems)等方面。本研究中黄参转录组数据注释到KEGG数据库的有富集在20条代谢通路中的51 479个Unigenes,与代谢相关的通路最多,第2位是碳水化合物代谢基因。该发现有助于揭示黄参淀粉及药用物质合成途径,功能基因的分析、代谢通路的注释等,为后期开展黄参的代谢组学、功能基因组研究奠定了基础,这部分内容将在后续工作中做进一步研究。
转录因子也称为反式作用因子,是指能够与真核基因的顺式作用元件发生特异性相互作用,并对基因的转录有激活或抑制作用的DNA结合蛋白。转录因子能调控多个与抗逆相关基因的表达,使植物的抗逆性得到改善,可为基础研究及生产应用提供理论依据,因此,也逐渐成为植物抗逆机制研究的核心内容。植物的抗逆性状是多基因控制的数量性状,多个转录因子家族均与植物的抗逆性有重要关系。目前已从高等植物中分离鉴定出数百种转录因子与植物抗逆性密切相关,可调控植物体感受干旱、高盐、低温和病原等信号的相关基因的表达[18]。C2H2型锌指蛋白主要涉及生长发育和环境胁迫应答反应,调控植物抗逆境胁迫、抗病和生长发育方面的生物学功能[19];MYB转录因子响应非生物胁迫逆境[20];WRKY转录因子是近几年研究比较热的与植物胁迫应答相关的转录因子[21],参与转录重编程的调控[22],在多种生物和非生物胁迫及诸如水杨酸[23]、赤霉素[24]等植物激素信号转导中起重要作用。目前,越来越多的研究表明WRKYs是ABA应答信号网络的关键节点[24]。BHLH是第二大类转录因子,不仅影响植物生长发育,还参与调控信号转导和激素合成[25],结构高度保守,它和NAC一样,是一类植物特有的转录因子,响应不同的生物胁迫和低温、干旱及高盐等非生物逆境[25]。生长在祁连山中的黄参抗逆性强,通过研究这些转录因子,将有利于进一步揭示这些转录因子如何调控黄参更好地适应逆境胁迫。
简单序列重复SSR标记是目前最理想的分子标记,广泛用于开展遗传多样性、基因定位、遗传图谱构建和比较基因组学研究[15]。本试验的SSR分析结果为野生黄参的分子标记及遗传学研究提供了一定的理论基础。
本次转录组测序结果揭示,黄参具有丰富的基因表达,并通过生物信息学分析获得了黄参基因的注释信息及代谢通路,获得的转录组信息将为后续黄参分子标记的开发和关键基因的克隆及功能分析等研究提供科学数据。

