亚麻生氰糖苷合成关键酶CYP79基因家族的鉴定及表达分析
齐燕妮 李闻娟 赵丽蓉 李 雯 王利民 谢亚萍 赵 玮 党 照 张建平,*
亚麻生氰糖苷合成关键酶CYP79基因家族的鉴定及表达分析
齐燕妮1李闻娟1赵丽蓉2李 雯2王利民1谢亚萍1赵 玮1党 照1张建平1,*
1甘肃省农业科学院作物研究所, 甘肃兰州 730070;2甘肃农业大学农学院 / 甘肃省干旱生境作物重点实验室 / 甘肃省作物遗传改良与种质资源创新重点实验室, 甘肃兰州 730070
CYP79蛋白是生氰糖苷合成关键酶, 但关于亚麻基因的研究鲜有报道。本研究对包括亚麻在内的9种作物的CYP79基因家族进行了鉴定, 并分析了亚麻基因的序列特征、复制事件、共线性关系、系统进化、顺式作用元件及表达模式。结果表明, 在亚麻、白亚麻、毛果杨、木薯、芝麻、高粱、大豆、葡萄及水稻中分别鉴定到9、9、3、2、5、7、6、16和4个CYP79家族成员; 系统进化分析显示,基因的进化具有物种特异性;不均匀分布在4条染色体上, 具有1~3个外显子, 其启动子区含大量激素与逆境响应相关元件; 共克隆到8个亚麻CYP79基因的全长DNA序列及5个成员的全长cDNA序列; LuCYP79蛋白序列长度为282~565 aa, 等电点为5.84~9.14, 分子量为31.56~62.86 kD, 均为亲水性蛋白, 定位于内质网; 共有5对基因发生复制事件, 占全部基因的77.8%, 全部经历了强烈的纯化选择, 其中和在拟南芥和木薯中均具有同源基因。表达分析表明, LuCYP79家族成员具有组织特异性, 且各成员在不同遗传背景下表达模式不同, 其中、、和在4个品种中的表达差异显著。相关分析表明, 50 d时的/与成熟亚麻籽中生氰糖苷含量呈极显著正相关, 20 d时的/及()/分别与成熟亚麻籽中生氰糖苷含量呈极显著正相关, 初步推测其可能是亚麻籽生氰糖苷合成的关键基因。研究结果对进一步阐明亚麻CYP79蛋白的功能具有积极意义, 并为培育低生氰糖苷亚麻品种提供了理论参考。
亚麻; CYP79基因家族; 生氰糖苷; 生物信息学; 表达分析
亚麻(L.), 是一种一年生的自花授粉的二倍体作物(2= 2= 30)[1]。因其油用、纤用、饲用及工业等价值, 至今仍被广泛种植[2-3]。亚麻籽富含α-亚麻酸、木酚素(3%)、维生素E、膳食纤维等多种营养物质, 已经应用于奶制品、烤制品、肉制品等多种功能性食品[4]。亚麻籽中α-亚麻酸含量是油料作物中最高的(55%以上), 是人类从膳食中摄取α-亚麻酸的重要来源之一。此外, 亚麻籽还被添加于动物饲料中, 以改善其健康状况并提高其营养价值[3]。然而, 亚麻籽中还含有剧毒化学物质, 即氰化物生氰糖苷。人和动物如果长期摄入含生氰糖苷的食物会引起氰化物慢性中毒, 主要症状为生长发育迟缓和甲状腺肿大[5-7]。生氰糖苷的存在对亚麻籽食用或加工品质具有极大的影响, 导致亚麻及其相关产业发展严重受限, 因此将亚麻籽生氰糖苷含量控制在合理的水平具有非常重要的意义。
在高等植物中, 生氰糖苷的合成是以氨基酸为底物, 由CYP79、CYP71及糖基转移酶催化的串联反应, 其中CYP79催化氨基酸转化成相应的醛肟, 这是生氰糖苷合成的起始限速步骤[8]。1995年, 首次在高粱中发现CYP79A1, 催化L-酪氨酸转化为p-羟基苯乙醛肟, 最终产生蜀黍苷[9], 随后在拟南芥[10]、木薯[11]、毛果杨[12]及玉米[13]等多个物种中鉴定到CYP79蛋白, 这说明CYP79广泛存在于高等植物中。Bark等[14]通过对白芥CYP79B1、高粱CYP79A1、拟南芥CYP79B2及旱金莲、番木瓜、油菜的CYP79氨基酸序列进行分析发现, CYP79家族相当保守, 同源性很高, 而且都含有CYP79家族特有的FXP (E/D)RH及亚铁血红素结合序列SFSTG (K/R)RGC(A/I)A, 表明不同植物中催化第一步反应的酶都是由保守的CYP79家族同源基因编码。研究发现, 表达了高粱基因的拟南芥, 其硫代葡萄糖苷含量比对照增加了3.5~4.5倍[15]。拟南芥和突变体中, 最终产物短链芥子油甙和长链芥子油甙大幅度减少甚至完全消失[16-17]。此外, CYP79具有底物特异性, 从而产生不同种类的防御物质[18]。拟南芥中CYP79B2与CYP79B3以L-色氨酸为底物分别参与吲哚类硫苷和植保素的形成[19], CYP79A2催化苯丙氨酸生成苯基乙醛肟[20], 而CYP79F1和CYP79F2分别以短链和长链甲硫氨酸衍生物为底物[16-17]。木薯CYP79D1和CYP79D2分别以L-缬氨酸和L-异亮氨酸为底物, 最终产物分别为亚麻苦苷和百脉根苷[21]。欧洲紫衫CYP79A118优先催化L-酪氨酸, 最终产生紫衫氰醇苷[22]。
近年来, 随着亚麻全基因组测序工作的完成(登录号为QMEI02000000), 利用生物信息学方法进行基因家族分析已经成为挖掘亚麻功能基因的重要手段。目前已经在拟南芥[23]和玉米[12]中分别鉴定到7个和4个基因, 但亚麻中关于基因的研究鲜有报道。本研究拟鉴定亚麻基因组中CYP79基因家族成员并对其进行克隆, 通过对序列特征、系统进化、共线性关系及时空表达模式等方面进行全面系统的分析, 以期为进一步解析亚麻家族各成员的功能奠定基础, 为利用分子手段定向控制亚麻生氰糖苷的含量提供新的途径。
1 材料与方法
1.1 试验材料
本研究选取4个亚麻品种, 陇亚10号、R2、匈牙利3号和张亚2号, 均种植于甘肃省农业科学院试验基地。出苗后10 d、20 d、30 d、40 d分别采集4个品种的根、茎、叶; 开花初期采集根、茎、叶、花; 并且采集开花后10 d、20 d、30 d、40 d、50 d的根、茎、叶、种子。样品于液氮中速冻后储存在-80℃, 用于DNA和RNA提取。
1.2 CYP79基因家族的鉴定
亚麻和野生白亚麻基因组相关数据来自本研究室(亚麻基因组序列已上传至NCBI, http://www.ncbi. nlm.nih.gov/, 登录号为QMEI02000000; 亚麻和白亚麻CDS序列, 蛋白质序列及gff文件已分别上传至https://doi.org/10.6084/m9.figshare.13614311.v3和https://doi.org/10.6084/m9.figshare.19119737.v1)。拟南芥、毛果杨、木薯、高粱、大豆、葡萄及水稻基因组序列和注释文件从植物基因组数据库(phytozome v9.0, http://www.phytozome.net/)下载, 芝麻全基因组蛋白序列, 拟南芥、木薯及玉米等已知CYP79蛋白序列从NCBI下载。利用Blastp, 将已知CYP79蛋白序列与亚麻、野生白亚麻、毛果杨、木薯、芝麻、高粱、大豆、葡萄及水稻全基因组蛋白数据库分别进行比对, 之后用Pfam和NCBI 保守结构域数据库(CDD)进行结构域确认, 最后确认候选序列是否含有CYP79蛋白特有的FXP(E/D)RH位点和血氧红素结合位点SFSTG(K/R)RGC(A/I)A。
1.3 DNA、RNA提取及基因克隆
用试剂盒Plant Easy Spin RNA Miniprep Kit(BIOMIGA, 美国)提取各样品RNA, 随后利用NanoDrop2000分光光度计和凝胶电泳对RNA的浓度和纯度进行检测。采用PrimeScriptRT Reagent Kit with gDNA Eraser (Perfect Real Time; TaKaRa)进行基因组DNA去除和cDNA合成。使用CP Plant Miniprep Kit (BIOMIGA, 美国)提取出苗后20 d的叶片DNA。
根据从陇亚10号基因组获取的序列, 利用Primer Premier 5.0设计基因全长扩增引物(附表1)。分别以DNA和cDNA为模板, 利用KOD FX高保真酶(TOYOBO)进行PCR扩增。PCR产物纯化后克隆到pENTR/D-TOPO克隆试剂盒(Invitrogen, 美国)提供的载体上并进行测序。用MEGA6.0进行序列分析, 最后将所有序列上传至GenBank数据库。
1.4 LuCYP79基因家族序列分析
利用GSDS (http://gsds.cbi.pku.edu.cn/)进行基因结构分析及可视化[24]。利用ExPASy ProtParam (http://web.wxoasy.org/protparam/)[25]和Plant-mPloc (http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)分别对LuCYP79蛋白的理化性质和亚细胞定位进行预测; 同时使用ExPASy ProtScale (https:// web.expasy.org/protscale/)预测LuCYP79蛋白的亲疏水性。分别利用SOPMA (https://npsa-prabi.ibcp.fr/cgi- bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)和SWISS-MODEL(https://swissmodel.expasy.org/ interactive)预测LuCYP79蛋白的二级和三级结构。利用MEME在线软件(http://alternate.meme-suite.org/ tools/meme)分析LuCYP79蛋白的保守基序[26]。
1.5 LuCYP79家族成员在染色体上的分布及共线性分析
利用在线软件Map Gene to Chromosome (http://mg2c.iask.in/mg2c_v2.0/)将基因定位到染色体。采用MCScanX, 在默认参数下分析基因在亚麻基因组中的串联复制和片段复制事件[27], 利用TBtools分析亚麻基因组内及亚麻与木薯、拟南芥、葡萄、芝麻、水稻、高粱之间的共线性并进行可视化。利用MGEA6.0将发生复制事件的基因的CDS序列转化为氨基酸序列, 进行MUSCLE比对之后重新转化为核苷酸序列。将比对文件导入DnaSP6, 计算非同义替换率(Ka)和同义替换率(Ks), 利用Ka/Ks比值进行选择压力分析。一般来讲, Ka/Ks > 1、= 1 和 < 1 分别表示正向选择、中性选择和纯化选择。对发生复制的每一个基因对, 用Mya (million years ago) = Ks/(2×6.1×10–9)×10–6计算分歧时间[28]。
1.6 LuCYP79家族成员启动子分析
从亚麻基因组序列中提取基因起始位置上游2000 bp的序列, 通过PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析顺式作用元件[29]。
1.7 CYP79家族的系统进化分析
利用MEGA6.0软件的ClustalW对亚麻、白亚麻、拟南芥、毛果杨、木薯、芝麻、高粱、大豆、葡萄、玉米及水稻等11个物种CYP79蛋白序列进行多序列比对, 随后采用邻接法(Neighbor-Joining method)构建系统进化树, Bootstrap校验参数为1000次[30]。
1.8 qRT-PCR分析
引物设计、qRT-PCR反应体系及程序参考Zhang等[31]的方法, 引物见附表2。试验仪器为Eco Real-Time PCR System (Illumine), 以为内参基因[32]。每个样品3次重复, 采用2–ΔΔCt法计算基因的相对表达量[33]。
1.9 生氰糖苷含量测定及数据分析
参考寇向龙等[34]方法测定陇亚10号、R2、匈牙利3号和张亚2号4个品种成熟籽粒中生氰糖苷含量。采用SPSS 19.0进行显著性差异分析和相关性分析。
2 结果与分析
2.1 LuCYP79家族的鉴定与克隆
通过Blastp比对, CDD和Pfam数据库进行结构域分析以及CYP79家族保守位点的确认, 在亚麻、野生白亚麻、毛果杨、木薯、芝麻、高粱、大豆、葡萄及水稻基因组中分别鉴定到9、9、3、2、5、7、6、16和4个CYP79成员(表1和附表3)。栽培亚麻与野生白亚麻基因数目相同, 但不同物种间CYP79家族成员的数量具有显著差异。根据亚麻基因在染色体上的位置, 将其依次命名为~。
以陇亚10号的DNA和cDNA分别作为模板, 对基因进行扩增。除, 其余基因均可得到完整的DNA序列(图1-A), 但只有、、、和获得了完整的CDS序列(图1-B)。基因DNA序列长度在1096~2116 bp之间, CDS长度在849~1698 bp之间, 从而推测出蛋白序列长度在282~565 aa之间(表1)。将序列提交至 NCBI 数据库, GenBank登录号分别为OM688195、OM688196、OM688197、OM688198、OM688199、OM688200、OM688201、OM688202和OM688203。
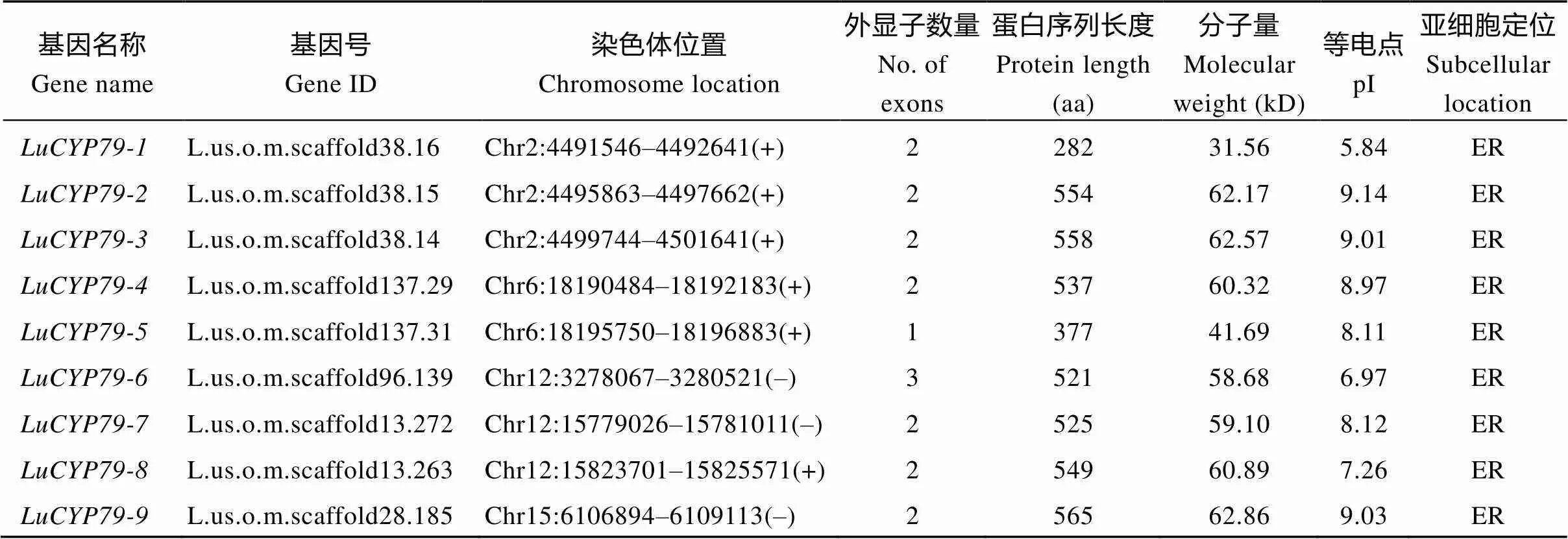
表1 亚麻CYP79基因家族主要特征
ER: 内质网。ER: endoplasmic reticulum.
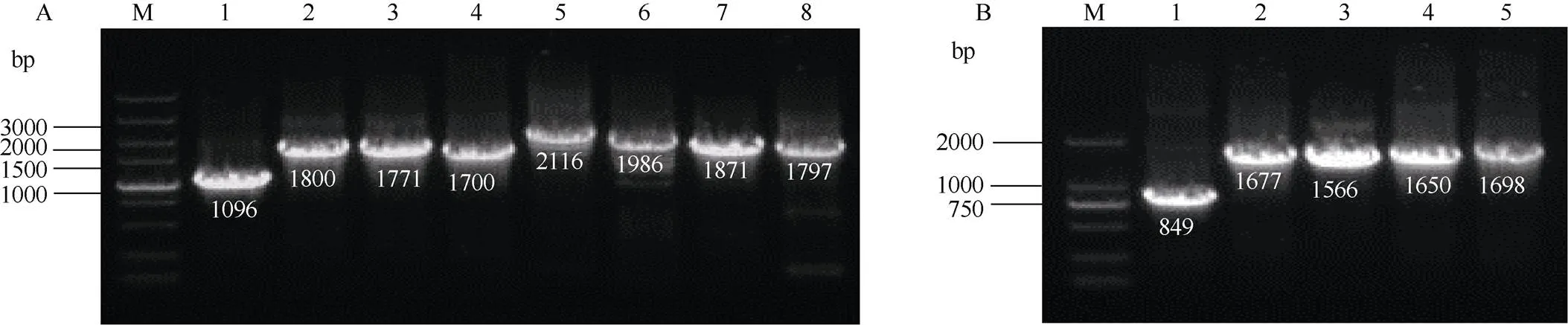
图1 LuCYP79基因全长DNA扩增(A)和cDNA扩增(B)
(A) M: DL 5000 marker; 1–8:,,,,,,, and, respectively. (B) M: DL 2000 marker; 1–5:,,,, and, respectively.
2.2 LuCYP79家族序列特征分析
基因结构分析结果表明, 除-5和分别具有1个和3个外显子外, 其余基因均具有2个外显子(表1和图2)。理化性质分析结果表明, LuCYP79蛋白分子量在31.56~62.86 kD之间; 等电点在5.84~9.14之间, 只有LuCYP79-1和LuCYP79-6为酸性蛋白, 其余均为碱性蛋白; 所有LuCYP79蛋白为亲水性蛋白(附图1)。亚细胞定位结果表明, 所有LuCYP79蛋白均定位在内质网。对LuCYP79蛋白二级结构进行预测发现, α-螺旋含量最多, 其次为无规则卷曲和延伸链, β-转角含量最少(附表4)。LuCYP79蛋白三级结构预测结果与二级结构一致(附图2)。对LuCYP79蛋白进行保守基序分析发现, 9个LuCYP79蛋白共预测到14个保守基序(Motif), 其中有5个成员具有14个Motif, LuCYP79-1含有的Motif最少(图2和附图3)。Motif 1、Motif 4和Motif 8存在于所有LuCYP79成员, 而且Motif 1和Motif 4分别含有CYP79家族高度保守的血氧红素结合位点SFSTG(K/R)RGC(A/I)A和FXP(E/D)RH位点(图3)。此外, 除Motif14, 其余10个Motif存在于8个LuCYP79蛋白。
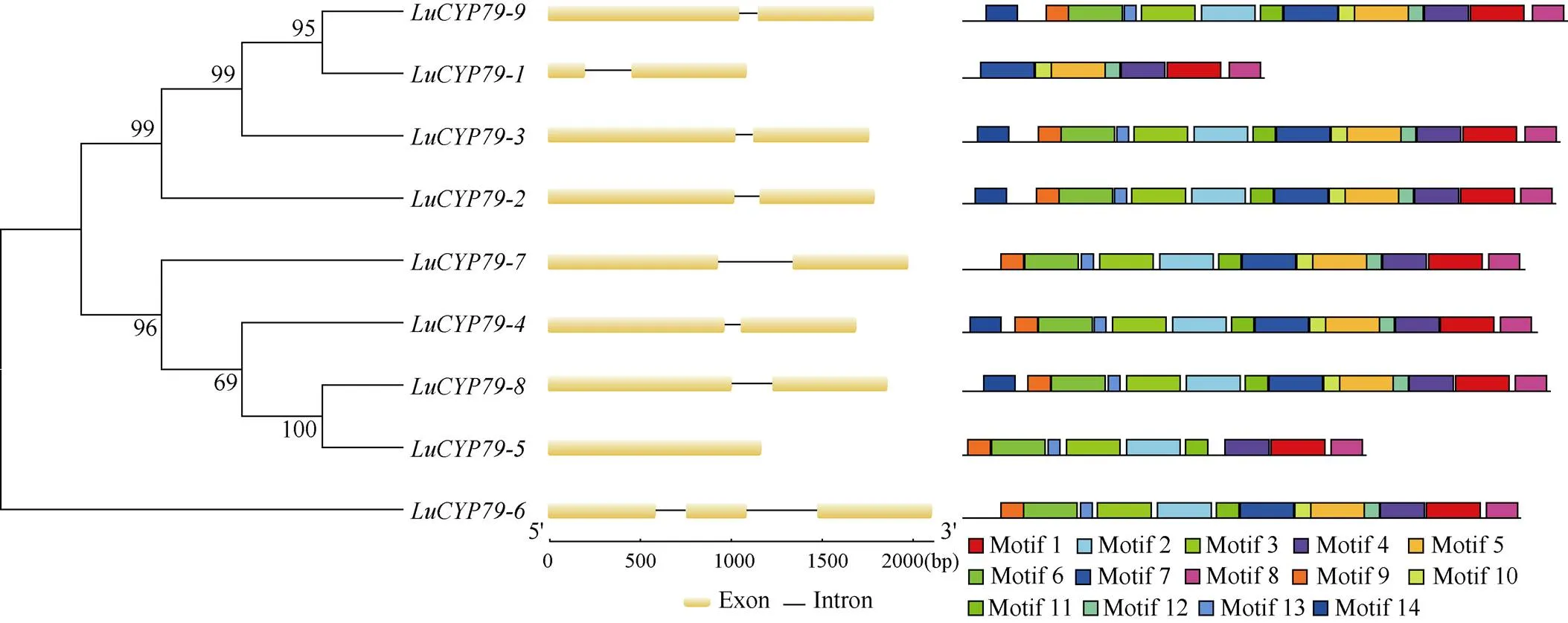
图2 LuCYP79系统进化、基因结构及Motif分析

图3亚麻LuCYP79家族SFSTG(K/R)RGC(A/I)A和FXP(E/D)RH保守位点
2.3 LuCYP79在染色体上的分布及共线性分析
LuCYP79家族成员分布在4条染色体上, 其中、和分布在2号染色体,和分布在6号染色体,、和分布在12号染色体, 15号染色体仅有分布(图4)。亚麻基因组内部共线性分析显示, 2对基因(/、/)存在共线性关系, 分布在4条染色体上(图5-A)。进一步分析发现, 有5对基因发生复制事件, 其中3对为串联复制(//、/), 2对发生片段复制(/、/) (图4和表2)。串联复制基因占全部基因的55.6%, 说明串联复制事件在LuCYP79基因家族扩张中起主要作用。通过Ka/Ks比值判断基因有无选择压力。复制基因的Ka/Ks比值全部小于1, 说明所有发生复制的基因经历了纯化选择(表2)。有4对基因的Ka/Ks比值在0.1左右, Ka/Ks平均值为0.1548, 说明基因进化非常保守。对复制事件发生的分歧时间进行计算发现,发生串联复制的分歧时间最短和最长分别在5412万和9053万年前, 发生片段复制的分歧时间最短和最长分别在134万和8796万年前。为进一步了解亚麻家族的进化机制, 分别分析其与拟南芥、木薯、芝麻、葡萄、高粱、水稻之间的共线性关系, 3个基因与拟南芥存在共线性关系,,和在亚麻中有1~2个同源基因; 2个与木薯存在共线性关系, MeCYP79D2在亚麻中有2个同源基因(图5-B); 亚麻基因与葡萄、芝麻及2个单子叶植物没有共线性。
2.4 LuCYP79家族成员启动子分析
为进一步研究基因的转录调控机制和潜在功能, 提取起始密码子上游2000 bp预测顺式作用元件。如表3所示,启动子区除含有真核生物基本元件(TATA-box、CAAT-box)外, 均含有厌氧诱导必需的响应元件(ARE)和光响应元件(G-box、GT1-motif、Box 4和GATA-motif等)。启动子区还含有4类激素响应元件, 分别为脱落酸响应元件(ABRE)、茉莉酸甲酯响应作用元件(TGACG-motif和CGTCA-motif)、生长素响应元件(TGA-element、AuxRR-core和AuxRE)和赤霉素响应元件(GARE-motif和P-box)。所有基因启动子区都含有ABRE; 除外, 其余8个基因都含有TGACG-motif和CGTCA-motif; 有6个基因含有不同类型的生长素响应元件。启动子还含有与逆境胁迫相关的作用元件, 其中有5个基因含有低温响应元件(LTR), 4个基因含有干旱响应元件(MBS)。此外,启动子还含有种子调控元件(RY-element)、分生组织相关调控元件(CAT-box)和玉米醇溶蛋白代谢相关的调控元件(O2-site)等。

图4 LuCYP79基因在亚麻染色体的分布
灰色线条和红色矩形分别代表片段复制和串联复制。
Gray line and red rectangle indicates segmental and tandem duplication, respectively.
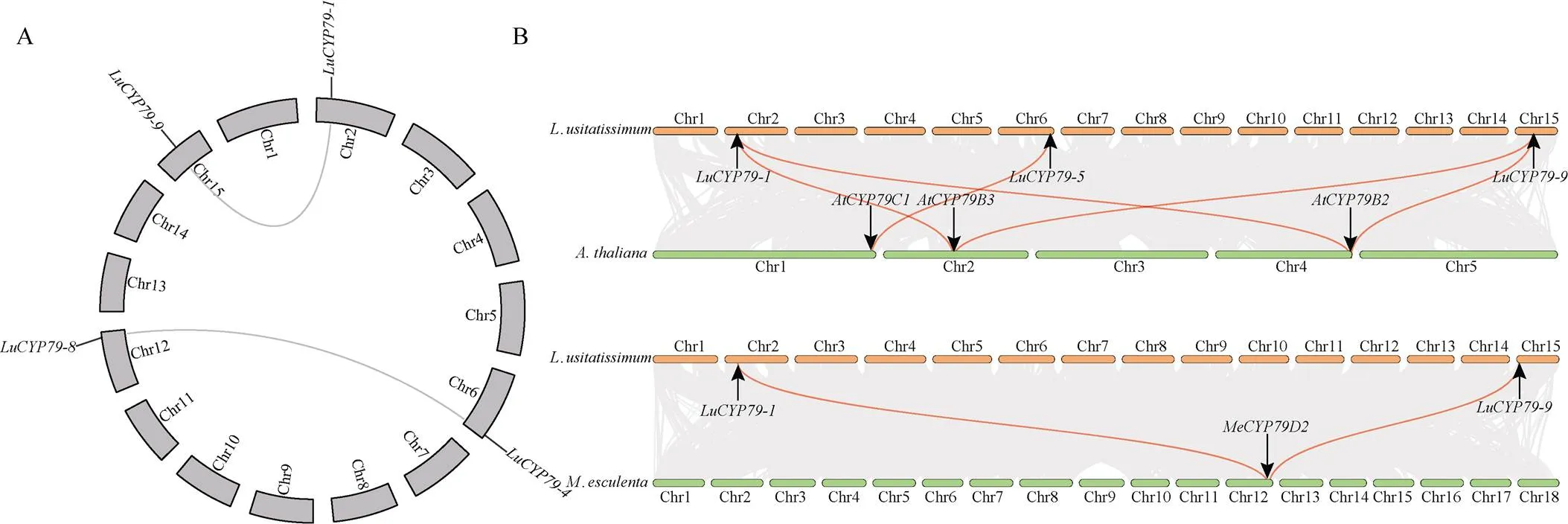
图5 亚麻基因组内LuCYP79基因共线性及其与拟南芥和木薯间的共线性
A: 灰色线条表示具有共线性的基因对。B: 灰色线条表示亚麻与其他物种的共线性区域, 彩色线条表示与不同物种具有共线性的基因对。
A: the gray lines indicate the colineargene pairs. B: the gray lines show the collinear regions between flax and other species, and the color lines represent the collineargene pairs between different species.
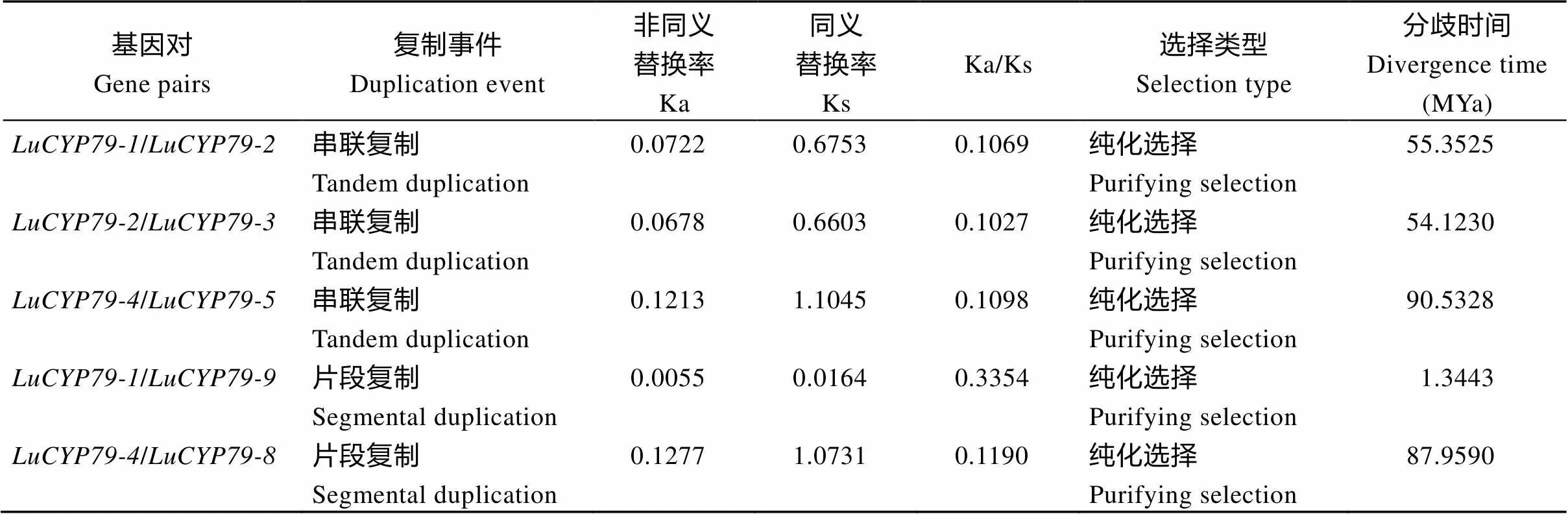
表2 LuCYP79基因复制事件、选择压力及分歧时间

表3 LuCYP79启动子顺式作用元件
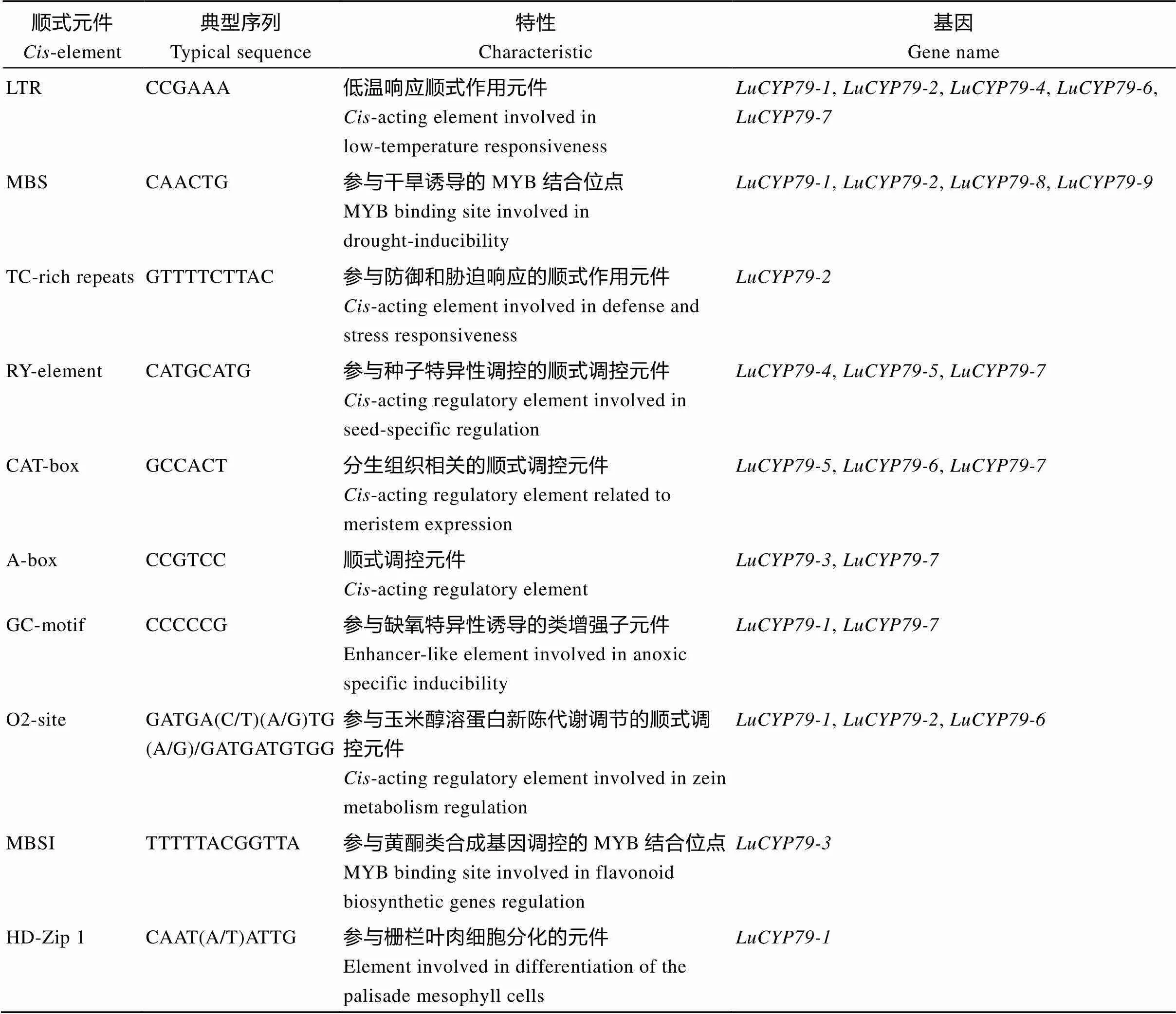
(续表3)
2.5 CYP79家族系统进化分析
为明确不同物种CYP79家族成员间的进化关系, 本研究利用8个双子叶植物(亚麻、野生白亚麻、拟南芥、大豆、葡萄、芝麻、木薯、毛果杨)及3个单子叶植物(水稻、玉米和高粱)的全长蛋白序列构建了系统进化树。如图6所示, CYP79蛋白可分为五大支。第1支含有亚麻、白亚麻、木薯全部CYP79以及拟南芥AtCYP79B2和AtCYP79B3; 第2支含有毛果杨和大豆全部CYP79; 第3支包括芝麻全部CYP79及拟南芥AtCYP79C1、AtCYP79C2、AtCYP79F1和AtCYP79F2; 第4支含有高粱、玉米、水稻等单子叶植物全部CYP79; 第5支为葡萄CYP79。结果表明, LuCYP79与LbCYP79亲缘关系最近, 同时它们与木薯MeCYP79D1和MeCYP79D2具有相当近的亲缘关系, 而且与拟南芥AtCYP79B2和AtCYP79B3亲缘关系密切, 而其他物种的CYP79大多分别聚在一起, 表明CYP79具有物种特异性。
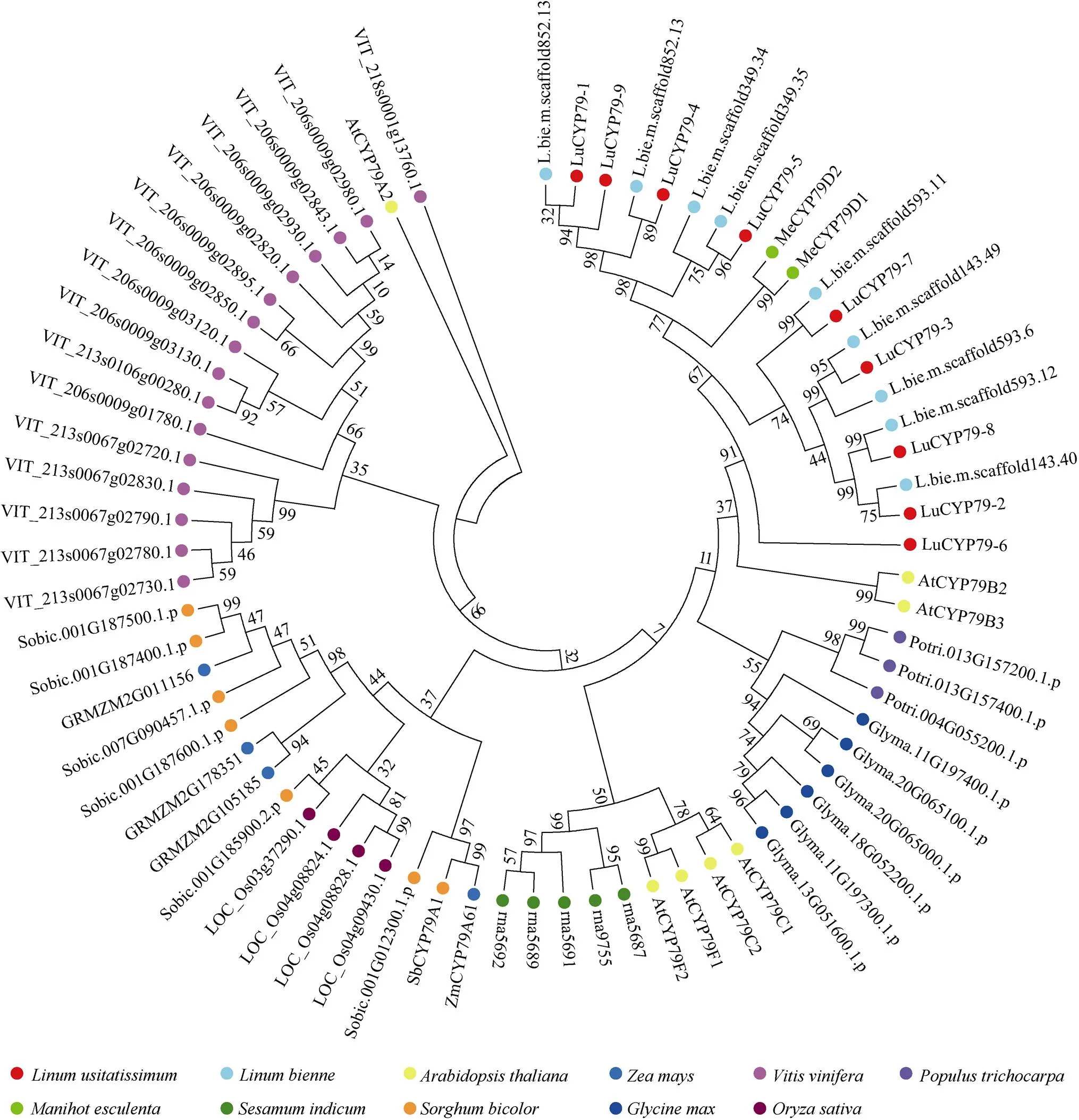
图6 CYP79家族系统进化树
2.6 LuCYP79基因的表达分析
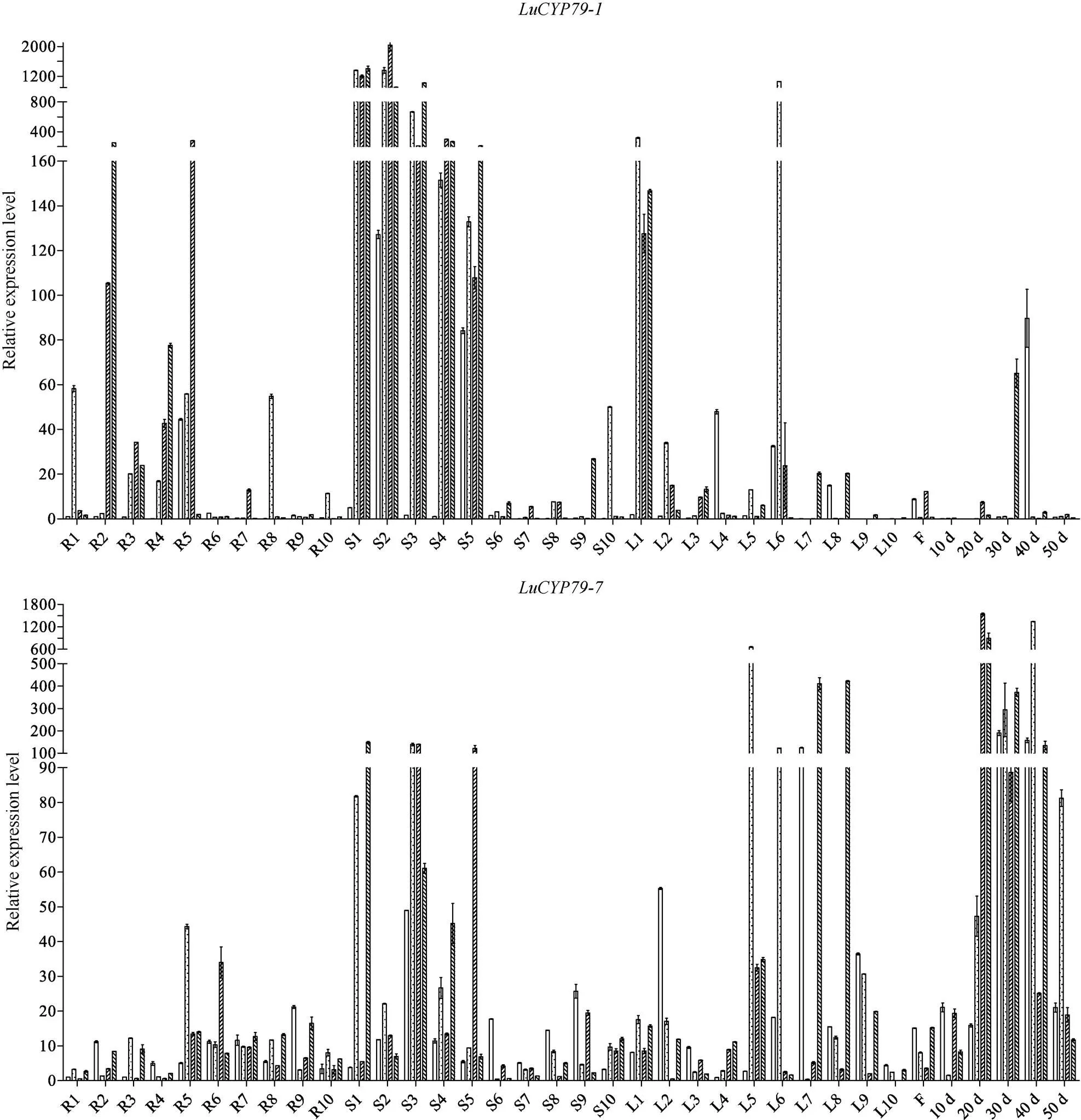
利用qRT-PCR分析9个LuCYP79家族成员在4个亚麻品种不同发育阶段种子中的表达模式发现, 各基因在不同品种中具有不同的表达模式, 如、和在陇亚10号开花后40 d的种子中表达量最高, 但在R2中的表达量在开花后20 d达到最高, 而在匈牙利3号中, 其在开花后30 d的种子中表达量最高;在陇亚10号、张亚2号、R2及匈牙利3号中的表达量分别在开花后50 d、40 d、20 d及30 d的种子中达到最高;在4个品种种子中的表达量分别在50 d、40 d、50 d、20 d达到最高;在不同品种种子中的表达量高峰分别为10 d、50 d、20 d、20 d;的表达量高峰分别在30 d、40 d、20 d、20 d;分别在30 d、40 d、20 d、30 d的种子中表达量较高(图7)。总的来说, 各基因在张亚2号不同发育阶段种子中的表达量普遍低于其他3个品种。除表达模式外, 不同发育阶段各基因的累积表达量在不同品种中具有明显差异(图8), 其中、、和在4个品种中的差异最明显, 最高表达量是最低表达量的4倍以上, 差异最大的可达到28倍。

图7 LuCYP79家族在4个亚麻品种不同发育阶段种子中的表达模式

图8 LuCYP79家族在4个亚麻品种种子发育阶段的累积表达量
为进一步解析、、和的功能, 本研究分析了它们在不同品种、不同组织、不同发育阶段的表达模式。结果表明, 各基因在不同组织均有表达, 呈现出组织特异性表达, 且各基因表达模式不同(图9)。在茎中的表达量总体高于其他组织, 且在苗期茎中的表达量高于发育后期茎中的表达量;在茎、叶及种子中都有较高的表达量, 茎中的表达量苗期高于发育后期, 但在叶中的表达量发育后期高于苗期;和在根和茎中的表达量较高, 而且都是营养生长阶段的表达量总体高于生殖生长阶段。
2.7 LuCYP79基因表达与亚麻籽生氰糖苷含量的相关分析
本研究对陇亚10号、张亚2号、R2和匈牙利3号4个亚麻品种成熟籽粒中生氰糖苷含量进行测定发现, 这4个亚麻品种成熟籽粒中生氰糖苷含量差异显著(≤0.05) (图10)。相关分析表明,与在开花后50 d亚麻籽中基因表达量的比值与成熟亚麻籽中生氰糖苷含量呈极显著正相关(≤0.01) (图11-A); 在开花后20 d的亚麻籽中,与及+与基因表达量的比值分别与成熟籽粒中生氰糖苷含量表现为极显著正相关 (≤0.01) (图11-B, C)。
(图9)
R1~R4: 出苗后10 d、20 d、30 d、40 d根; R5: 开花初根; R6~R10: 开花后10 d、20 d、30 d、40 d、50 d根; S1~S4: 苗期10 d、20 d、30 d、40 d茎; S5: 开花初茎; S6~S10: 开花后10 d、20 d、30 d、40 d、50 d茎; L1~L4: 苗期10 d、20 d、30 d、40 d叶; L5: 开花初叶; L6~L10: 开花后10 d、20 d、30 d、40 d、50 d叶; F: 花; 10~50 d: 开花后10~50 d种子。
R1–R4: 10 days, 20 days, 30 days, and 40 days root at seeding stage; R5: root at early blooming stage; R6–R10: 10 days, 20 days, 30 days, 40 days, and 50 days root after flowering; S1–S4: 10 days, 20 days, 30 days, and 40 days stem at seeding stage; S5: stem at early blooming stage; S6–S10: 10 days, 20 days, 30 days, 40 days, and 50 days stem after flowering; L1–L4: 10 days, 20 days, 30 days, and 40 days leaf at seeding stage; L5: leaf at early blooming stage; L6–L10: 10 days, 20 days, 30 days, 40 days, and 50 days leaf after flowering; F: flower; 10–50 d: 10–50 days seeds after flowering.
3 讨论
3.1 CYP79基因家族在进化过程中具有一定的保守性
随着生活质量的提高, 人们对食品安全问题日益重视, 亚麻籽中有毒氰化物生氰糖苷的存在已成为限制亚麻相关产业发展的重要原因之一。亚麻基因组的释放使亚麻分子生物学研究进入一个新的阶段, 从分子水平上控制生氰糖苷的合成或加速其代谢是降低亚麻籽中生氰糖苷含量的有效途径之一。CYP79家族是生氰糖苷合成的限速酶[8], 在植物中广泛存在, 目前已经在拟南芥[16-17,35]、木薯[11]、毛果杨[12]、玉米[13]、青花菜[36]等物种中相继被克隆, 但关于亚麻基因的结构特征及功能尚不清楚。

图10 4个亚麻品种成熟籽粒中生氰糖苷含量
不同小写字母表示在0.05水平具有显著性差异。
Different lowercase letters mean significant differences at the 0.05 probability level.
本研究在亚麻、白亚麻、毛果杨、木薯、芝麻、高粱、大豆、葡萄及水稻等9个物种中分别获得9、9、3、2、5、7、6、16和4个CYP79成员, 显示不同物种间CYP79家族成员数量存在显著差异, 这与不同物种进化中基因的缺失及扩张有关。但栽培亚麻与其祖先种白亚麻数量相同, 表明在亚麻驯化过程中CYP79家族的进化较为保守。本研究并未克隆到全部基因, 这可能是因为LuCYP79家族成员序列相似性较高, 难以进行特异性扩增, 或是表达量较低, 无法获得足够的扩增产物进行克隆。对LuCYP79蛋白进行保守基序分析发现, Motif 1、Motif 4和Motif 8存在于所有成员, 而且Motif 1和Motif 4分别含有该家族保守位点SFSTG(K/R)RGC(A/I)A和FXP(E/D)RH, 说明这3个Motif是LuCYP79家族的特征性基序。亚细胞定位显示该蛋白在内质网, 表明LuCYP79在内质网中起作用。系统进化分析结果显示,与其野生种亲缘关系最近, 基本成对聚在一起, 其次为木薯和拟南芥, 这与物种间亲缘关系不尽一致[31], 而且其他物种基本单独聚在一起, 说明在各物种中的进化具有特异性。所有单子叶植物的单独聚为一支, 表明在单子叶植物与双子叶植物分化后独立进化。
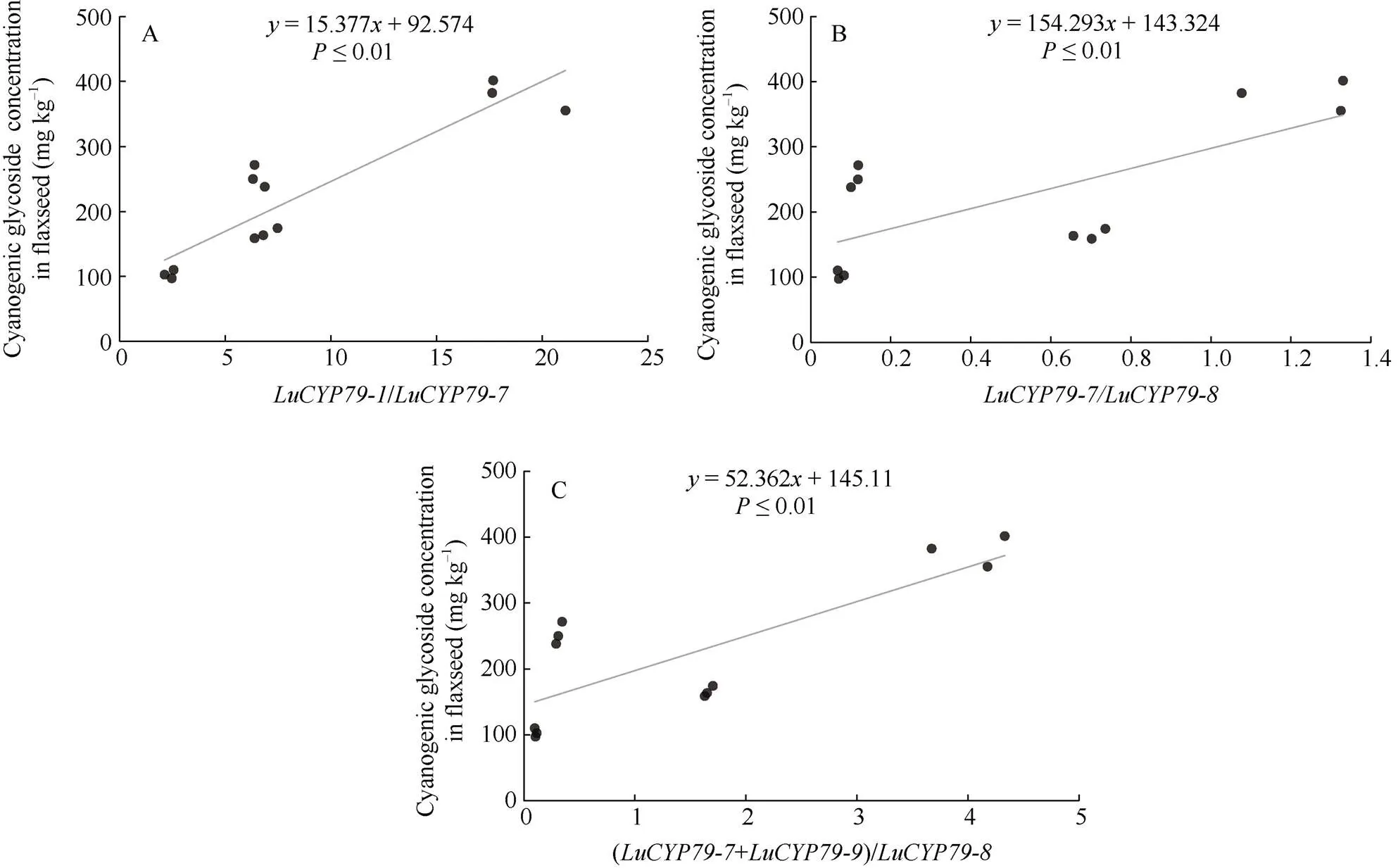
图11 LuCYP79基因表达量与成熟亚麻籽中生氰糖苷含量的相关性
Y: 成熟亚麻籽中生氰糖苷含量; A、B及C中的分别代表与、与及与基因表达量的比值。
Y: cyanogenic glycoside concentration in mature flaxseed;in A, B, and C represents the gene expression ratio ofto,to, and+to, respectively.
基因复制事件是基因家族扩张及功能分化的重要原因, 主要有全基因组复制/片段复制和串联复制2种方式[37-38]。在进化中也发生了复制事件, 有3对基因发生了串联复制, 2对基因发生了片段复制, 发生复制的基因占全部基因的77.8%, 说明串联复制和片段复制在LuCYP79基因家族扩张中起关键作用。的Ka/Ks比值都小于1, 平均Ka/Ks比值为0.1548, 说明LuCYP79家族经历了非常强烈的纯化选择, 进一步说明在进化过程中的保守性。共线性分析结果显示, 分别有3个和2个亚麻基因在拟南芥与木薯中具有同源基因, 其中和在2个物种中都有同源基因, 说明这2个基因在进化中比较保守; 但与葡萄、芝麻及单子叶植物并无共线性, 这与系统进化分析结果相符, 进一步说明基因在各物种中的进化具有特异性。
3.2 LuCYP79基因参与调控亚麻对激素及逆境胁迫的响应
基因表达受其上游区顺式作用元件的调控, ABRE为脱落酸响应的作用元件[39]; LTR是响应低温诱导的作用元件[40]; MYB转录因子结合顺式元件参与植物对干旱的应答[41]。通过分析LuCYP79家族的顺式作用元件, 可以进一步了解LuCYP79家族的调控机理及其可能参与的生物过程。本研究发现,启动子区含有ABRE、TGACG-motif、TGA-element、GARE-motif等多个激素响应元件以及逆境胁迫(干旱、低温)相关元件MBS和LTR, 这表明LuCYP79除催化生氰糖苷合成外, 还参与调控亚麻对激素及非生物胁迫的响应等多个生理过程, 同时还说明基因的表达受多种因素调控。
3.3 LuCYP79基因参与调控亚麻籽生氰糖苷合成
基因表达模式对深入研究基因功能具有非常重要的作用。为进一步了解LuCYP79家族各成员的功能, 本研究在不同品种、不同组织、不同发育时期对基因进行了表达分析。基因在陇亚10号、张亚2号、R2及匈牙利3号4个品种亚麻籽中具不同的表达趋势, 这可能与各品种的遗传背景相关。此外, 各基因在张亚2号中的表达量普遍较低, 这与生氰糖苷含量测定结果相一致, 张亚2号成熟籽粒中生氰糖苷含量显著低于其他3个品种。对各基因在种子不同发育阶段的累积表达量进行分析发现, 各成员的累积表达量在籽粒生氰糖苷含量具有显著差异的4个亚麻品种中差异极大, 其中、、和在4个品种中的差异达到4倍以上, 差异最大的可达到28倍。且本研究发现, 花后50 d籽粒中与基因表达量的比值, 花后20 d籽粒中与、与基因表达量的比值分别与成熟籽粒中生氰糖苷含量呈极显著正相关(≤0.01), 说明这几个基因可能在亚麻籽生氰糖苷合成中起关键作用。进一步分析、、和在不同组织中的表达模式发现, 这几个基因呈现出不同的组织特异性表达, 且具有不同的表达趋势, 此外, 同一个基因在不同品种中的时空表达模式也不尽相同, 说明这些基因可能在不同基因型亚麻品种生氰糖苷合成或是其他生理过程中发挥不同的作用, 但各成员具体的功能还需深入探究。
4 结论
本研究在亚麻基因组中共鉴定到9个LuCYP79家族成员, 均含有CYP79家族特有的保守位点, 并对其理化性质、基因结构、染色体定位、复制事件、共线性关系、进化特征等进行了分析, 预测到基因启动子区含有多种激素及逆境胁迫相关元件。在不同遗传背景下LuCYP79基因家族各成员具有不同的时空表达模式,、、和可能是调控亚麻籽生氰糖苷合成的关键基因。
附表和附图 请见网络版: 1) 本刊网站http://zwxb.chinacrops.org/; 2) 中国知网http:// www.cnki.net/; 3) 万方数据http://c.wanfangdata. com.cn/Periodicalzuowxb.aspx。
[1] Zohary D. Monophyletic vs. polyphyletic origin of the crops on which agriculture was founded in the near east., 1999, 46: 133–142.
[2] Oomah B D. Flaxseed as a functional food source., 2001, 81: 889–894.
[3] Turner T D, Mapiye C, Aalhus J L, Beaulieu A D, Patience J F, Zijlstra R T. Flaxseed fed pork: n-3 fatty acidenrichment and contribution to dietary recommendations., 2014, 96: 541–547.
[4] Goyal A, Sharma V, Upadhyay N, Gill S, Sihag M. Flax and flaxseed oil: an ancient medicine & modernfunctional food., 2014, 9: 1633–1653.
[5] Oomah B D, Mazza G, Kenaschuk E O. Cyanogenic compounds in flaxseed., 1992, 40: 1346–1348.
[6] Conn E E. Cyanogenic glycosides., 1981, 7: 479–500.
[7] Nelson L. Acute cyanide toxicity mechanisms and manifestations., 2006, 32: 8–10.
[8] Ganjewala D, Kumar S, Devi A, Ambika K. Advances in cyanogenic glycosides biosynthesis and analyses in plants., 2010, 54: 1–14.
[9] Sibbesen O, Koch B, Halkier B A, Moller B L. Cytochrome P-450TYR is a multifunctional heme-thiolate enzyme catalyzing the conversion of L-tyrosine to p-hydroxyphenylacetaldehyde oxime in the biosynthesis of the cyanogenic glucoside dhurrin in(L.) Moench., 1995, 270: 3506–3511.
[10] Wang C W, Dissing M M, Agerbirk N, Crocoll C, Halkier B A. Characterization ofCYP79C1 and CYP79C2 by glucosinolate pathway engineering inshows substrate specificity toward a range of aliphatic and aromatic amino acids., 2020, 11: 57.
[11] Andersen M D, Busk P K, Svendsen I, Møller B L. Cytochromes P-450 from cassava (Crantz) catalyzing the first steps in the biosynthesis of the cyanogenic glucosides linamarin and lotaustralin: cloning, functional expression in, and substrate specificity of the isolated recombinant enzymes., 2000, 275:1966–1975.
[12] Irmisch S, McCormick A C, Boeckler G A, Schmidt A, Reichelt M, Schneider B, Block K, Schnitzler J, Gershenzon J, Unsicker S B, Köllner T G. Two herbivore-induced cytochrome P450 enzymes CYP79D6 and CYP79D7 catalyze the formation of volatile aldoximes involved in poplar defense., 2013, 25: 4737–4754.
[13] Irmisch S, Zeltner P, Handrick V, Gershenzon J, Köllner T G. The maize cytochrome P450 CYP79A61 produces phenylacetaldoxime and indole-3-acetaldoxime in heterologous systems and might contribute to plant defense and auxin formation., 2015, 15: 128–139.
[14] Bak S, Nielsen H L, Halkier B A. The presence of CYP79 homologues in glucosinolate-producing plants shows evolutionary conservation of the enzymes in the conversion of amino acid to aldoxime in the biosynthesis of cyanogenic glucosides and glucosinolates., 1998, 38: 725–734.
[15] Petersen B L, Andreasson E, Bak S, Agerbirk N, Halkier B A. Characterization of transgenicwith metabolically engineered high levels of p-hydroxybenzylglucosinolate., 2001, 212: 612–618.
[16] Reintanz B, Lehnen M, Reichelt M, Gershenzon J, Kowalczyk M, Sandberg G, Godde M, Uhl R, Palme K. Bus, a bushyCYP79F1 knockout mutant with abolished synthesis of short-chain aliphatic glucosinolates., 2001, 13: 351–367.
[17] Chen S X, Glawischnig E, Jørgensen K, Naur P, Jørgensen B, Olsen C E, Hansen C H, Rasmussen H, Pickett J A, Halkier B A. CYP79F1 and CYP79F2 have distinct functions in the biosynthesis of aliphatic glueosinolates in., 2003, 33: 923–937.
[18] Bak S, Paquette S M, Morant M, Morant V M, Saito S, Bjarnholt N, Zagrobelny M, Jorgensen K, Osmani S, Simonsen H T, Perez R S, Heeswijck T B, Jorgensen B, Moller B L. Cyanogenic glycosides a case study for evolution and application of cytochromes P450., 2006, 5: 309–329.
[19] Hull A K, Vij R, Celenza J L.cytochrome P450s that catalyze the first step of tryptophan-dependent indole-3-acetic acid biosynthesis., 2000, 97: 2379–2384.
[20] Wittstock U, Halkier B A. Cytochrome P450 CYP79A2 fromL. catalyzes the conversion of l-phenylalanine to phenylacetaldoxime in the biosynthesis of benzylglucosinolate., 2000, 275:14659–14666.
[21] Jorgensen K, Morant A V, Morant M, Jensen N B, Olsen C E, Kannangara R, Motawia M S, Moller B L, Bak S. Biosynthesis of the cyanogenic glucosides linamarin and lotaustralin in cassava isolation biochemical characterization and expression pattern of CYP71E7 the oxime-metabolizing cytochrome P450 enzyme., 2011, 155: 282–292.
[22] Luck K, Jia Q D, Huber M, Handrick V, Wong G K, Nelson D R, Chen F, Gershenzon J, Köllner T G. CYP79 P450 monooxygenases in gymnosperms: CYP79A118 is associated with the formation of taxiphyllin in., 2017, 95: 169–180.
[23] Halkier B A, Hansen C H, Naur P, Mikkelsen M D, Wittstock U. The Role of Cytochromes P450 in Biosynthesis and Evolution of Glucosinolates. Amsterdam: Elsevier Science Press, 2002. pp 223–248.
[24] Hu B, Jin J P, Guo A Y, Zhang H, Luo J C, Gao G. GSDS 2.0: anupgraded gene feature visualization server., 2015, 31: 1296–1297.
[25] Wilkins M R, Gasteiger E, Bairoch A, Sanchez J C, Williams K L, Appel R D, Hochstrasser D F. Protein identification and analysis tools on the ExPASy server., 1999, 112: 531–552.
[26] Bailey T L, Johnson J, Grant C E, Noble W S. The MEME suite., 2015, 43: W39–W49.
[27] Wang Y P, Li J P, Paterson A H. MCScanX-transposed: detecting transposed gene duplications based on multiple collinearity scans., 2013, 29: 1458–1460.
[28] Lynch M, Conery J S. The evolutionary fate and consequences of duplicate genes., 2000, 290: 1151–1155.
[29] Lescot M, Dehais P, Thijs G, Marchal K, Moreau Y, Van de Peer Y, Rouze P, Rombauts S. PlantCARE, a database of plantacting regulatory elements and a portal to tools for in silico analysis of promoter sequences., 2002, 30: 325–327.
[30] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: molecular evolutionary genetics analysis version 6.0., 2013, 30: 2725–2729.
[31] Zhang J P, Qi Y N, Wang L M, Wang L L, Yan X C, Dang Z, Li W J, Zhao W, Pei X W, Li X M, Liu M, Tan M L, Wang L, Long Y, Wang J, Zhang X W, Dang Z H, Zheng H K, Liu T M. Genomic comparison and population diversity analysis provide insights into the domestication and improvement of flax., 2020, 23: 100967.
[32] Huis R, Hawkins S, Neutelings G. Selection of reference genes for quantitative gene expression normalization in flax (L.)., 2010, 10: 71.
[33] Antonov J, Goldstein D R, Oberli A, Baltzer A, Pirotta M, Fleischmann A, Altermatt H J, Jaggi R. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization., 2005, 85: 1040–1050.
[34] 寇向龙, 徐美蓉, 张建平, 赵宾宾, 韩舜愈. 响应面法优化亚麻籽氢氰酸提取条件. 食品工业科技, 2016, 37(21): 201–204.
Kou X L, Xu M R, Zhang J P, Zhao B B, Han S Y. Optimization of extraction conditions for hydrocyanic acid in flaxseed by response surface methodology., 2016, 37(21): 201–204 (in Chinese with English abstract).
[35] Mikkelsen M D, Hansen C H, Wittstock U, Halkier B A. Cytochrome P450 CYP79B2 fromcatalyzes the conversion of tryptophan to indole-3-acetaldoxime, a precursor of indole glucosinolates and indole-3-acetic acid., 2000, 275: 33712–33717.
[36] 李占省, 刘玉梅, 方智远, 杨丽梅, 庄木, 张扬勇, 吕红豪. 青花菜 P450全长克隆、表达及其与不同器官中莱菔硫烷含量的相关性分析. 中国农业科学, 2018, 51: 2357–2367.
Li Z S, Liu Y M, Fang Z Y, Yang L M, Zhuang M, Zhang Y Y, Lyu H H. Full length cloning, expression and correlation analysis of P450gene with sulforaphane content in different broccoli organs., 2018, 51: 2357–2367 (in Chinese with English abstract).
[37] Ren R, Wang H F, Guo C C, Zhang N, Zeng L P, Chen Y M, Ma H, Qi J. Widespread whole genome duplications contribute to genome complexity and species diversity in angiosperms., 2018, 11: 414–428.
[38] Zhu Y, Wu N N, Song W L, Yin G J, Qin Y J, Yan Y M, Hu Y K. Soybean () expansin gene superfamily origins: segmental and tandem duplication events followed by divergent selection among subfamilies., 2014, 14: 93.
[39] 华静静, 陈晓静. 植物体中WD40蛋白的研究进展. 黑龙江农业科学, 2015, (5): 153–156.
Hua J J, Chen X J. Progress of WD40 proteins in plant., 2015, (5): 153–156 (in Chinese with English abstract).
[40] Mehlgarten C, Jablonowski D, Wrackmeyer U, Tschitschmann S, Sondermann D, Jäger G, Gong Z, Byström A S, Schaffrath R, Breunig K D. Elongator function in tRNA wobble uridine modification is conserved between yeast and plants., 2010, 76: 1082–1094.
[41] 杨琳, 王宇, 杨剑飞, 李玉花. 花青素积累相关负调控因子的研究进展. 园艺学报, 2014, 41: 1873–1884.
Yang L, Wang Y, Yang J F, Li Y H. Research advances on negative regulators of anthocyanin accumulation., 2014, 41: 1873–1884 (in Chinese with English abstract).
Identification and expression analysis of CYP79 gene family, a key enzyme for cyanogenic glycoside synthesis in flax
QI Yan-Ni1, LI Wen-Juan1, ZHAO Li-Rong2, LI Wen2, WANG Li-Min1, XIE Ya-Ping1, ZHAO Wei1, DANG Zhao1, and ZHANG Jian-Ping1,*
1Institute of Crop Sciences, Gansu Academy of Agricultural Sciences, Lanzhou 730070, Gansu, China;2College of Agronomy, Gansu Agricultural University / Key Laboratory of Arid Land Crop Science in Gansu Province / Gansu Key Laboratory of Crop Improvement and Germplasm Enhancement, Lanzhou 730070, Gansu, China
CYP79 is a key enzyme in the synthesis of cyanogenic glycoside. However, there is no systematical study ofgenes in flax. In this study, we identifiedgene family in 9 crops including flax, and focused on the sequence characteristics, duplication events, collinearity, evolution,-acting elements and expression patterns ofgenes. The results revealed that a total of 9, 9, 3, 2, 5, 7, 6, 16, and 4 CYP79 family members were identified in flax, pale flax, poplar, cassava, sesame, sorghum, soybean, grape, and rice, respectively. Phylogenetic analysis showed that the evolution ofgenes was species-specific.were distributed unevenly on four chromosomes and had 1–3 exons. The promoter region ofcontained lots of elements involved in response to hormone and stress. 8 full-length DNA sequences and 5 full-length cDNA sequences of flaxgenes were cloned. LuCYP79 proteins contained 282–565 amino acid residues with molecular weight of 31.56–62.86 kD, and isoelectric point of 5.84–9.14. All LuCYP79 members were hydrophilic protein and located in endoplasmic reticulum. There were five pairs ofgenes with duplication events, accounting for 77.8% of all genes, and all the duplication genes underwent strong purification selection. Bothandhad homologous genes inand cassava. The relative expression levels showed that LuCYP79 family members were tissue-specific and had different expression patterns under different genetic backgrounds. There were significantly different in the relative expression of,,, andin the four cultivars. Moreover, correlation analysis showed that/in 50 days flaxseed was significantly positively correlated with the concentration of cyanogenic glycosides in mature flaxseed./and ()/in 20 days flaxseed were significantly positively correlated with the concentration of cyanogenic glycosides in mature flaxseed, respectively. It was preliminarily speculated that they might be the key genes in the synthesis of cyanogenic glycoside in flaxseed. These results have positive significance for further elucidating the function of CYP79 proteins in flax and provide theoretical references for breeding flax varieties with low cyanogenic glycoside.
flax;gene family; cyanogenic glycoside; bioinformatics; the relative expression level
10.3724/SP.J.1006.2023.24042
本研究由甘肃省农业科学院现代生物育种项目(2020GAAS08, 2022GAAS04), 财政部和农业农村部国家现代农业产业技术体系建设专项(油料), 国家自然科学基金项目(31760426), 甘肃省知识产权计划项目(21ZSCQ026)和甘肃省科技计划项目(21JR7RA722, 21JR1RA354)资助。
This study was supported by the Modern Biology Breeding Project of Gansu Academy of Agricultural Sciences (2020GAAS08, 2022GAAS04), the China Agriculture Research System (Oil) of MOF and MARA, the National Natural Science Foundation of China (31760426), the Intellectual Property Planning project of Gansu Province (21ZSCQ026), and the Science and Technology Project of Gansu Province (21JR7RA722, 21JR1RA354).
通信作者(Corresponding author):张建平, E-mail: zhangJPzw3@gsagr.ac.cn
E-mail: xbsdqyn@126.com
2022-02-21;
2022-07-21;
2022-08-19.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20220818.1625.006.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
——以多重共线性内容为例

