结核分枝杆菌PE_PGRS29蛋白结构与功能的生物信息学分析
吴 珊,周海金,徐广贤*
(1. 西安市人民医院(西安市第四医院)检验科,陕西 西安 710000;2. 广东医科大学 医学技术学院医学检验系,广东 东莞 523000)
结核病是机体感染结核分枝杆菌(Mycobacteriumtuberculosis,Mtb)后发生的一种慢性传染病,严重危害人类健康,据2019年世界卫生组织(WHO)统计,全球估算结核病死亡数约为140×104个,是单一病菌感染致死率最高的传染病之一[1]。牛结核分枝杆菌引起的牛结核病是一种全年都可能发生的慢性人畜共患病,不仅影响牛的健康和农场的经济效益, 还影响养牛人员的身体健康及牛奶、牛肉等相关食品的质量安全[2]。结核病潜伏期较长,目前尚缺乏特效的治疗方法。结核病的难以治疗和根除障碍与Mtb在宿主细胞内生活方式复杂多变密切有关,因而探索Mtb病原体-宿主细胞之间的相互作用,对结核病的诊疗有重要指导作用。
自噬(Autophagy)是细胞内的一种“自食(Self-eating)”现象,是普遍存在于真核细胞中的一种高度保守的代谢过程,也是细胞内物质降解的主要途径[3]。泛素介导的异种吞噬是一种选择性自噬,在宿主防御包括结核分枝杆菌(Mtb)在内的细胞内病原体中具有至关重要的作用[4]。然而,宿主泛素靶标侵入微生物引发异种吞噬的确切机制仍不清楚。Chai等[5]研究发现泛素可以识别Mtb表面蛋白PE_PGRS29,这是一种以前未发现的泛素结合蛋白,含有真核生物样泛素化相关(UBA)结构域,UBA介导的泛素可与PE_PGRS29直接结合,随后招募自噬受体p62将分枝杆菌递送至LC3相关的自噬体,PE_PGRS29-泛素相互作用的破坏减弱了巨噬细胞中Mtb的异种吞噬清除,并增加了炎症反应升高小鼠的细菌负荷。作为新发现的泛素结合蛋白,其生物学功能及可能存在蛋白与蛋白之间相互作用的网络尚未完全清楚。本试验采用生物信息学分析方法对PE_PGRS29蛋白的结构和功能进行预测分析[6-9],为进一步了解泛素靶标入侵微生物引发异种吞噬的作用和调节机制提供理论依据,有助于更好的预防和控制结核病。
1 材料与方法
1.1 材料
Mtb H37Rv株全基因组,从美国国立生物技术信息中心(NCBI)GenBank数据库获取PE_PGRS29蛋白的基因序列及氨基酸序列等信息。PE_PGRS29蛋白的Gene ID为886556,基因组序列为NC_000962.3,蛋白序列为YP_177814.1。
1.2 方法
1.2.1 PE_PGRS29基本信息及序列分析 在NCBI中获取PE_PGRS29蛋白的基因序列,通过ORF Finder分析其开放阅读框。
1.2.2 PE_PGRS29蛋白基本理化性质分析 PE_PGRS29蛋白的理化特性如分子式、分子质量、等电点、不稳定指数等通过Expasy提供的在线软件Protparam进行预测分析。
1.2.3 PE_PGRS29蛋白的亲疏水性、跨膜结构、信号肽、结构域及磷酸化位点分析 蛋白质的折叠情况可由氨基酸的亲疏水性来反映,疏水区可能出现潜在的跨膜结构区[10]。蛋白的亲疏水性和跨膜区域分别用ProtScale和TMHMM Server v.2.0在线分析软件分析;利用信号肽服务器SignalP 4.0预测PE_PGRS29蛋白的信号肽的有无及数量;用NetPhos 3.1 Server预测蛋白的磷酸化位点。
1.2.4 PE_PGRS29蛋白二级结构分析与三级结构建模 用SOPMA预测分析PE_PGRS29蛋白的二级结构,然后在SWISS-MODEL软件中输入蛋白的氨基酸序列,建立PE_PGRS29蛋白的三级结构模型。
1.2.5 PE_PGRS29蛋白的B细胞抗原表位分析 登录IEDB网站,输入该蛋白的氨基酸序列,通过 Karplus & Schulz分析柔韧性、Kolaskar8. Tongaonkar预测抗原性、Emini预测抗原表面可及性、Parker预测亲水性及Chou & Fasman法预测氨基酸编码蛋白质的β转角[8],联合对B细胞抗原表位进行分析。
1.2.6 PE_PGRS29蛋白的Th细胞抗原表位分析 通过SYFPEITHI和RANKPEP对PE_PGRS29抗原蛋白Th表位进行综合预测。进入SYFPEITHI首页,选择HLA-DRE1*0101、0401、0701、0801、0901、1101、1501这7种HLA基因分型并选取得分>18分的氨基酸序列进行辅助T细胞表位预测分析;进入RANKPEP主页,选择SYFPEITHI所预测的HLA基因分型,找出红色标记的结果,确定SYFPEITHI和RANKPEP的综合预测结果,筛选出分析结果均较好的辅助T细胞表位。
1.2.7 PE_PGRS29蛋白的细胞毒性T淋巴细胞抗原表位分析 通过SYFPEITHI、NetMHCpan对PE_PGRS29抗原蛋白的细胞毒性T淋巴细胞表位进行综合预测。
1.2.8 PE_PGRS29蛋白的相互作用网络 与PE_PGRS29蛋白相互作用的其他蛋白信息使用STRING数据库进行检索。
1.2.9 PE_PGRS29蛋白与人类蛋白同源性分析 使用EXPASY对PE_PGRS29蛋白氨基酸序列与人类蛋白的同源性进行BLAST分析。
1.2.10 PE_PGRS29蛋白的同源性预测和进化树构建 运用在线 uniprot蛋白数据库的Blast工具,将PE_PGRS29基因序列与同源性较高的核苷酸进行比,并用MEGA-X软件构建进化树。
2 结果与分析
2.1 PE_PGRS29蛋白的基因基本信息及序列分析
Rv1468c基因(ID:886556)位于Mtb(H37Rv)的全基因组(NC_000962.3)1 655 609~1 656 721区域,全长1 113 bp。PE_PGRS29蛋白在NCBI GenBank数据库中氨基酸序列的登录号为YP_177814.1,由370个氨基酸构成,包含18种氨基酸,其中以甘氨酸(34.6%)、丙氨酸(19.2%)、天冬氨酸(7.0%)、丝氨酸(5.7%)的组成比例较高。经ORF Finder工具分析,PE_PGRS29蛋白含有5个开放阅读框,最长的一个开放阅读框显示PE_PGRS29基因被通读,全长1 113 bp(图1)。
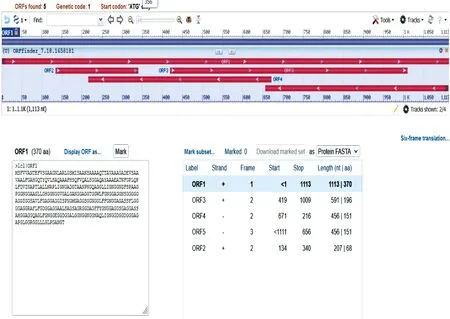
图1 PE_PGRS29蛋白的开放阅读框分析Fig.1 Open reading box analysis of the PE_PGRS29 protein
2.2 PE_PGRS29蛋白基本理化性质
PE_PGRS29蛋白分子式为C1375H2111N431 O465S3,原子总数为4 385,相对分子质量为322.1567×103,消光系数为9 970,280 nm处的吸光度为0.309,脂肪族氨基酸指数为66.81。PE_PGRS29蛋白带负电荷的残基(Asp+Glu)12个,带正电荷的残基(Arg+Lys)5个,其理论等电点pI为4.66。PE_PGRS29蛋白的不稳定性指数为15.60,提示该蛋白为稳定蛋白。
2.3 PE_PGRS29蛋白的亲疏水性、跨膜结构、信号肽、保守域及磷酸化位点
ProtScale预测PE_PGRS29蛋白为亲水性蛋白,第331位氨基酸亲水性得分最高为-1.533,第51为氨基酸疏水性得分最高为2.378(图2)。PE_PGRS29蛋白信号肽的分析结果显示(图3),标准值D值为0.166,小于阈值0.450,C、S、Y评分均在阈值之下,推测PE_PGRS29蛋白无信号肽。应用TMHMM分析该蛋白的跨膜结构(图4),其370个氨基酸残基跨膜概率值均未突破阈值线(图4粉色横线),表明该蛋白1~370位氨基酸序列均属于膜外侧(outside),不存在跨膜螺旋(TMhelix),预测该蛋白位于Mtb细胞壁和细胞膜上。用NCBI数据库BLAST软件分析显示(图5),PE_PGRS29蛋白有1个结构域,属于PE蛋白超家族。用NetPhos 3.1 Server软件分析显示(图6),PE_PGRS29蛋白含有10个丝氨酸磷酸化位点,2个苏氨酸磷酸化位点和1个酪氨酸磷酸化位点。
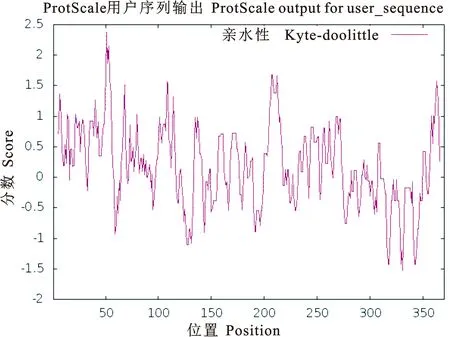
图2 PE_PGRS29蛋白的亲(疏)水性分析Fig. 2 Hydrophilic (hydrophobic) analysis ofPE_PGRS29 protein
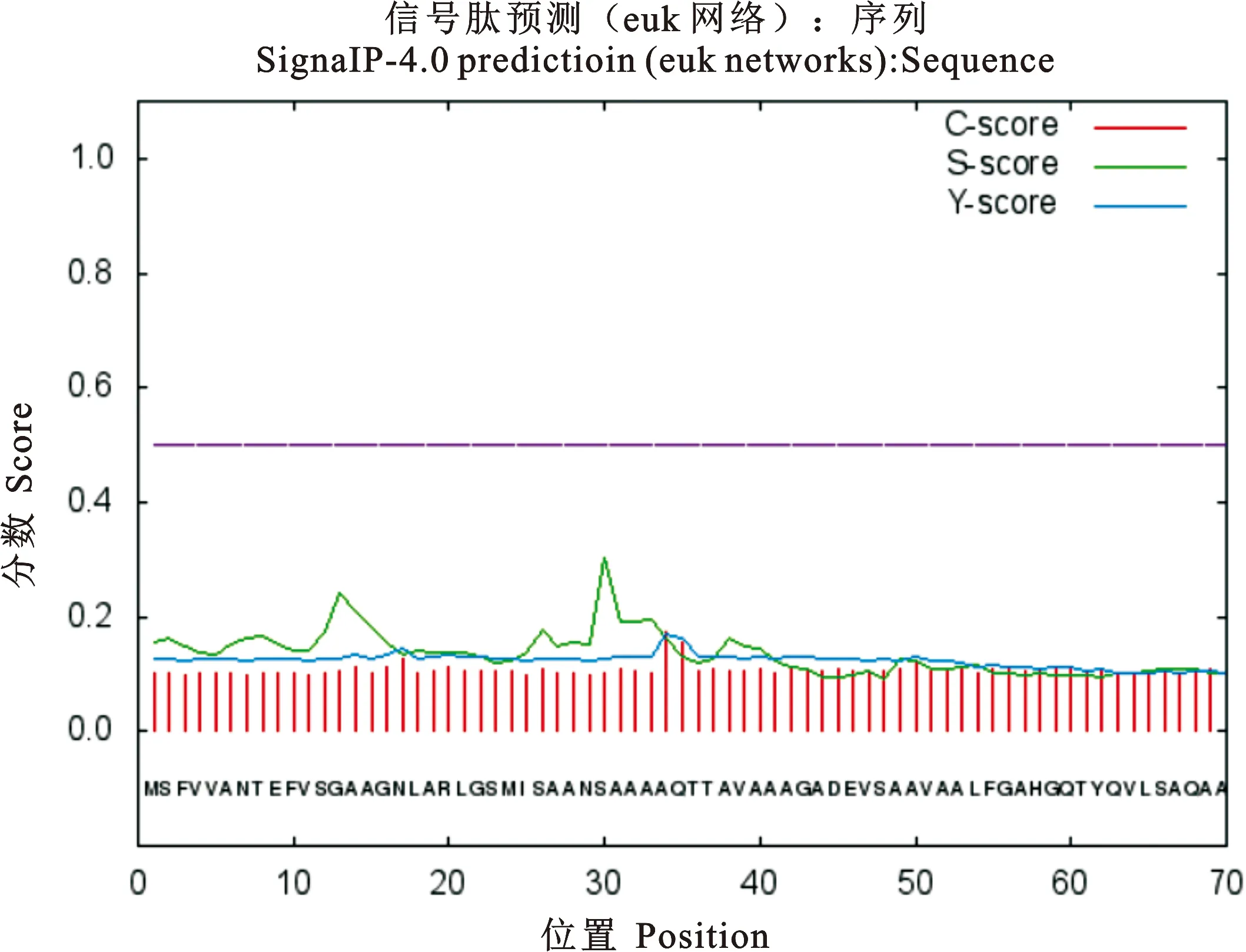
图3 PE_PGRS29蛋白信号肽预测Fig. 3 PE_PGRS29 protein signaling peptide prediction
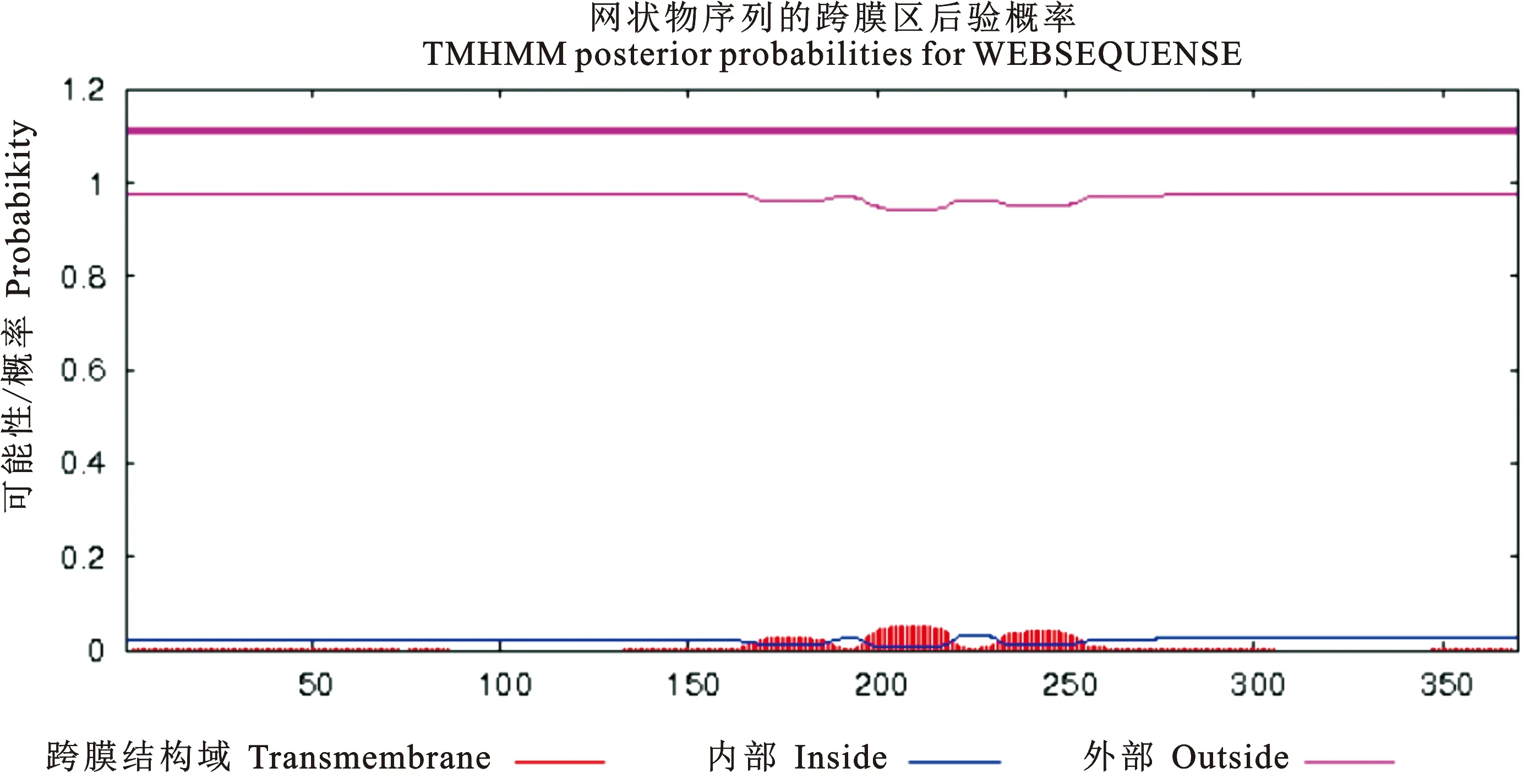
图4 TMHMM Server, v. 2.0预测PE_PGRS29蛋白跨膜螺旋Fig. 4 TMHMM Server, v. 2.0 predicts PE_PGRS29 protein transmembrane helix

图5 NCBI BLAST预测PE_PGRS29蛋白结构域Fig. 5 NCBI BLAST predicts the PE_PGRS29 protein domain
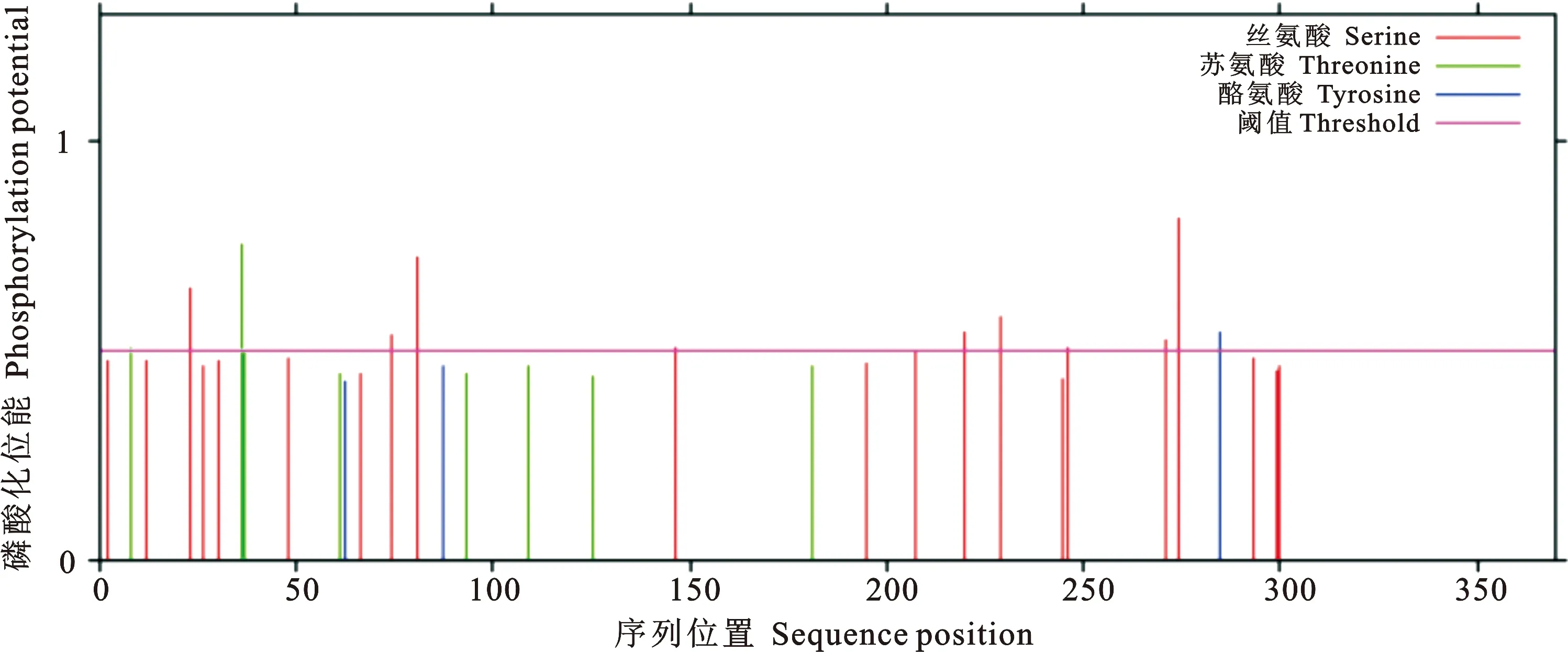
图6 NetPhos 3.1 预测PE_PGRS29蛋白磷酸化位点Fig. 6 NetPhos 3.1 predicts the phosphorylation sites of the PE_PGRS29 protein
2.4 PE_PGRS29蛋白二级结构分析与三级结构建模
通过SOPMA预测PE_PGRS29蛋白的二级结构,α-螺旋(Hh)81个,占21.89%;β-转角 (Tt)39个,占10.54%;β-折叠(Ee)81个,占21.89%;无规则卷曲(Cc)169个,占45.68%(图7)。PE_PGRS29蛋白的三级结构模型通过SWISS MODEL蛋白质分析软件进行构建,结果见图8。

图7 PE_PGRS29蛋白二级结构预测A.二级结构柱状图;B.二级结构序列图;C.二级结构峰图Fig. 7 Prediction of secondary structure of PE_PGRS29 proteinA. Bar chart of secondary structure; B. Secondary structure sequence diagram; C. Secondary structure peak map

图8 PE_PGRS29蛋白三级结构同源建模Fig. 8 Homologous modeling of the tertiarystructure of PE_PGRS29 protein
2.5 PE_PGRS29蛋白的B细胞抗原表位预测
PE_PGRS29蛋白的B细胞抗原表位通过IDB软件进行预测,柔韧性参数以基线1.018为参照,大于1.018的氨基酸区段柔韧性强,柔韧性越强越有利与抗体结合,是易形成抗原表位的区域。另外,结合线性表位、β转角、可塑性、表面可及性、亲疏水性、抗原等6个方面(图9)来筛选B细胞的抗原表位,结果见表1。
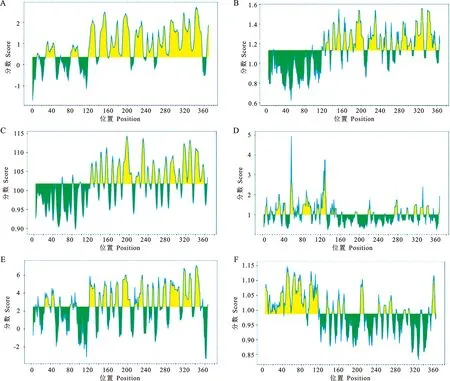
图9 IEDB预测PE_PGRS29蛋白的B细胞表位A. 线性表位预测; B. β转角预测; C. 可塑性预测; D. 表面可及性预测; E. 亲疏水性预测; F. 抗原预测Fig.9 Predicrion of B cell epitopes of Rv1733e protein in IEDBA. Linear epitope prediction; B. β-rotation angle prediction; C. Plasticity prediction;D. Surface accessibility prediction; E. Hydrophlicity prediction; F Antigenicity prediction
2.6 PE_PGRS29蛋白的Th细胞抗原表位
在线预测PE_PGRS29蛋白的限制性Th细胞表位,其中HLA-DRBI*0101、HLA-DRB1*0401表型具有最多的表位;潜在的限制性Th细胞抗原表位结果见表2。对该2种方法预测分值较高的表位作进一步统计分析,DRBl*0801和DRBl*0901表型的表位数量少。在分析 SYFPEITH和 RANKPEI的预测结果后,对于某个表型分值均较高的氨基酸序列进行筛选,以作为候选Th表位,结果见表3。HLA-DRB*0101、0401亚型的共同表位分布在59~66位,HLADRBl*0101、0401和1101亚型的共同表位分布在59~62位。
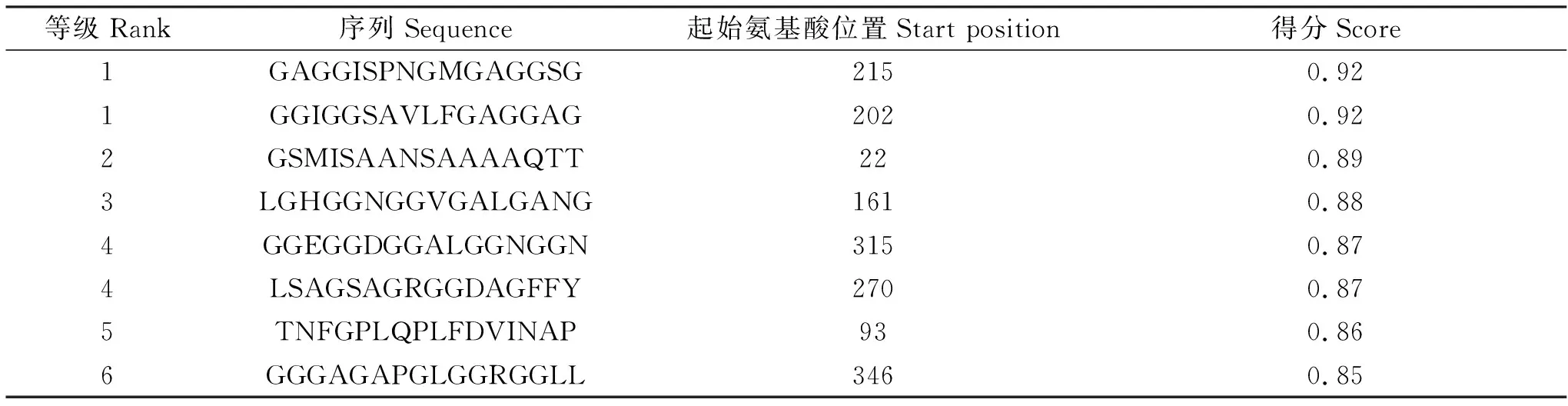
表1 PE_PGRS29蛋白B细胞抗原表位预测结果Table 1 Epitope prediction of PE_PGRS29 protein B cell

表2 不同软件预测的PE_PGRS29蛋白Th细胞表位信息Table 2 Epitope information of PE_PGRS29 protein Th cell predicted by different software
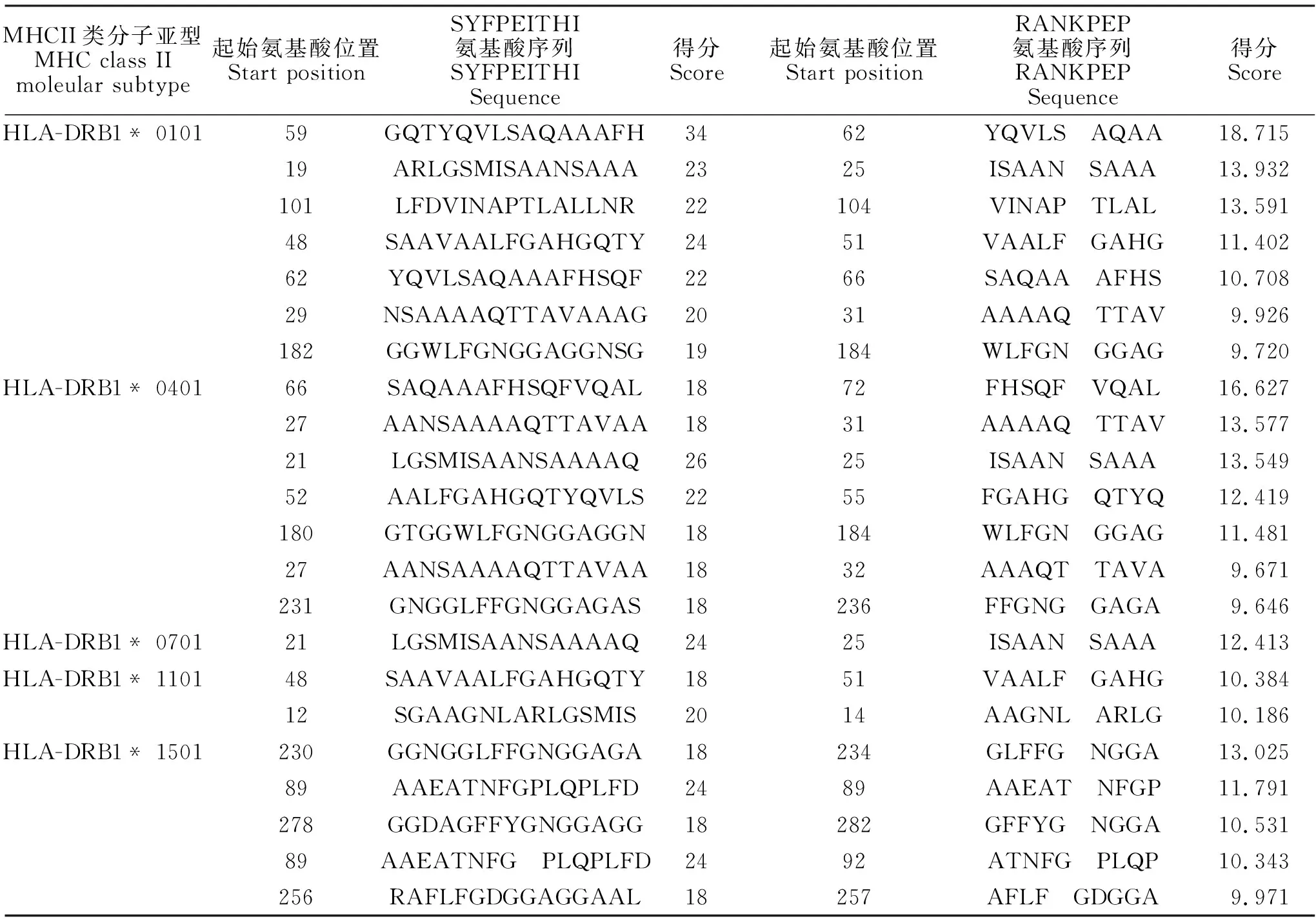
表3 PE_PGRS29蛋白Th细胞表位综合分析Table 3 Comprehensive analysis of Th cell epitopes of PE_PGRS29 protein
2.7 PE_PGRS29蛋白的细胞毒性T淋巴细胞抗原表位预测
限制性CTL表位HLA-A2、HLA-A3和HLA-B7的预测结果见表4。HLA表型进行预测的分值较高的潜在CTL表位数目及选择出每一种表型分值均较高的多肽序列,可成为CTL候选表位,其结果见表5。

表4 不同软件预测的PE_PGRS29蛋白CTL细胞表位信息Table 4 Cell epitope information of PE_PGRS29 protein predicted

表5 PE_PGRS29蛋白CTL表位综合分析Table 5 Comprehensive analysis of PE_PGRS29 protein CTL epitopes
2.8 PE_PGRS29蛋白的相互作用蛋白
经STRING数据库预测PE_PGRS29蛋白与ctpD(可能输出钴/镍的p型ATP酶;参与重金属稳态,属于阳离子转运ATP酶)、Rv1745c、fadE15(酰基辅酶a脱氢酶)、mmpS1、Rv3096(假设蛋白质)、trxB1(硫氧还原蛋白)、Rv1682、Rv3732和trxA等蛋白发生相互作用(图10)。
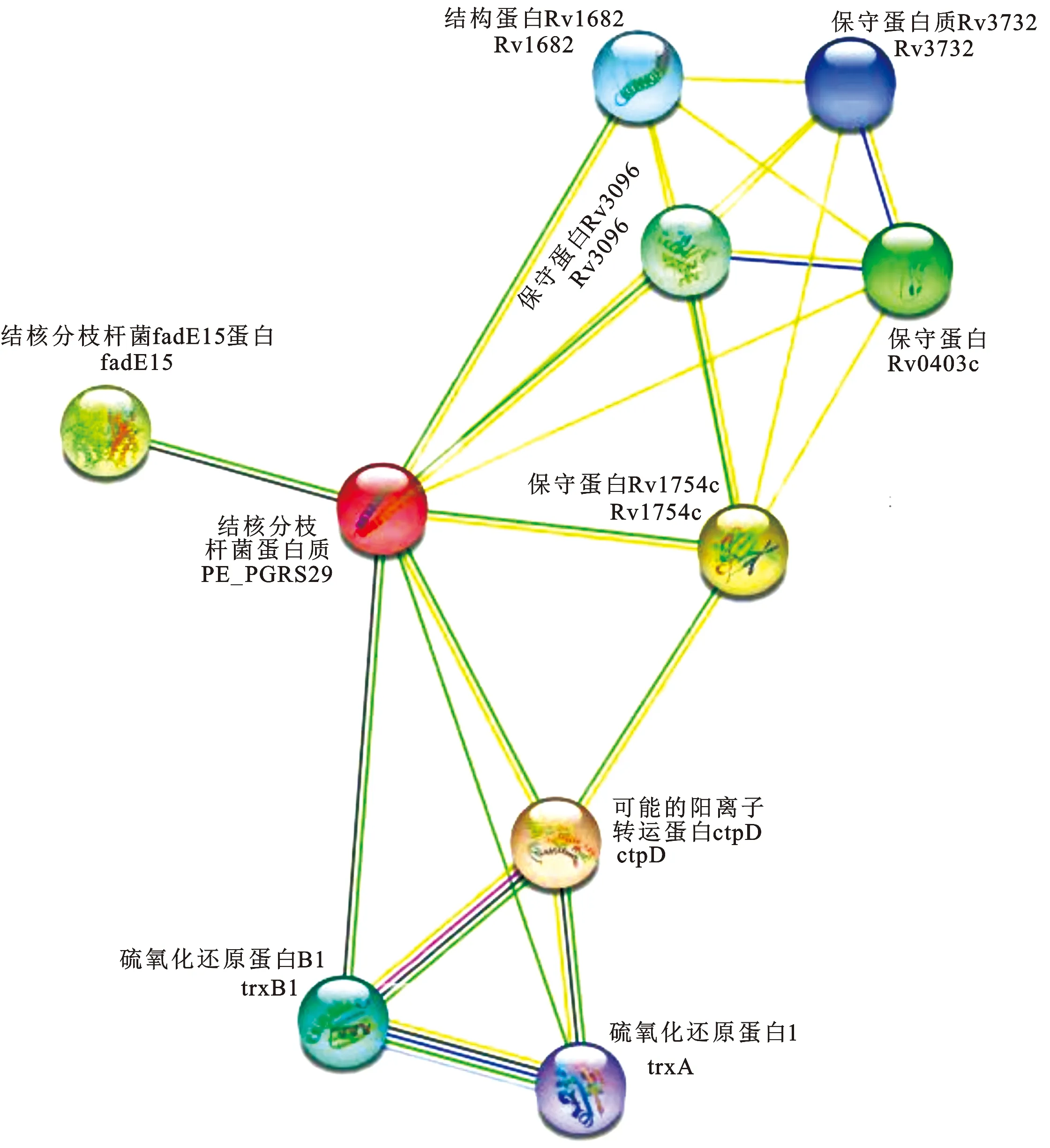
图10 PE_PGRS29蛋白的相互作用网络Fig.10 The interaction network of PE_PGRS29 protein
2.9 PE_PGRS29蛋白的同源性预测和进化树构建
经氨基酸序列比对,结核分枝杆菌(M.H37Ra)、结核分枝杆菌(M.H37Rv)、减毒牛型结核杆菌(M. bovis BCG)、结核分枝杆菌变种牛卡介苗(M.variant bovis BCG )、orygis 分枝杆菌(M. orygis)、卡内蒂分枝杆菌(M. canettii)、变种非洲K85结核分枝杆菌(M. tuberculosis variant africanum K85)、变种鳍足类结核分枝杆菌(M. tuberculosis variant pinnipedii)、decipiens分枝杆菌(M. decipiens)的序列覆盖区域分别是100%、100%、100%、100%、100%、99.7%、99.6%、99.5%、72.6%。PE_PGRS29蛋白与以上物种的氨基酸序列相似度较高(图11)。用MEGA-X软件构建系统进化树,PE_PGRS29蛋白在结核分支杆菌属同源性较高(图12)。
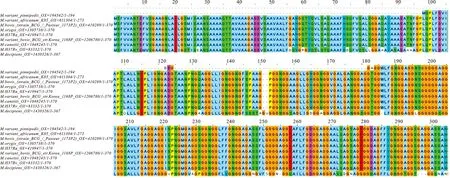
图11 PE_PGRS29多序列比对分析Fig.11 Sequence alignment of PE_PGRS29

图12 PE_PGRS29进化树Fig.12 Evolutionary tree of PE_PGRS29
3 讨 论
1998年,法国Pasteur和英国Sanger研究所对结核分枝杆菌进行了全基因组测序,因此一大类富含甘氨酸、N端有保守脯氨酸-谷氨酸序列(Proline-Glutamate,PE)的蛋白被发现,并被命名为PE蛋白家族,约占全部编码基因的6%[11]。随后,根据编码蛋白质C端含有串联重复序列的不同,PE蛋白家族进一步分为单独PE(PE_only)和PE_PGRS两个相近亚家族。PE_PGRS家族因C端编码基因富含鸟嘌呤-胞嘧啶基序(polymorphic guanine-cytosine rich sequences,PGRS)而得名,是Mtb独有的多基因亚家族,只存在于分枝杆菌属,且绝大多数仅存在于致病性结核分枝杆菌中,与免疫逃逸和抗原变异密切相关[12],近年来成为开发新药物、研制疫苗和诊断试剂特异性靶点的热点之一[13-14]。
PE_PGRS家族蛋白是由一组约104~1 901个氨基酸构成的酸性蛋白,参与构成Mtb的表面抗原成分,主要位于细胞壁和细胞膜上,与分枝杆菌的致病性与毒力息息相关[15]。PE_PGRS29作为该蛋白家族的一员,经预测分析,PE_PGRS29蛋白为亲水性蛋白,无信号肽,不存在跨膜螺旋,且该蛋白具有多个T、B细胞抗原表位,佐证了该蛋白位于Mtb胞膜上,参与机体对结核分枝杆菌免疫反应。
基因序列和ORF分析结果显示,PE-PGRS29基因编码的蛋白由370个氨基酸构成,包含18种氨基酸,其中以甘氨酸(34.6%)、丙氨酸(19.2%)、天冬氨酸(7.0%)、丝氨酸(5.7%)的组成比例较高。PGRS结构域的特征性标志是包含大量甘氨酸和丙氨酸重复序列,通常以Gly-Gly-Ala或Gly-Gly-X(X为任意氨基酸)形式存在[12]。大量甘氨酸的GGXGXD/NXUX(U指非极性或疏水性大残基)非肽序列(nonapeptide sequence)形成一个平行β滚筒或平行β螺旋结构,可与Ca2+连接,大量Ca2+与PE_PGRS蛋白结合,从而导致巨噬细胞中Ca2+浓度显著下降,吞噬溶酶体的程度受到抑制,则巨噬细胞内的结核分枝杆菌无法被溶酶体及时清除,造成持续的慢性感染[16-17]。因此,控制巨噬细胞中Ca2+浓度或抑制Ca2+与PE_PGRS蛋白结合,可能有助于结核分枝杆菌的免疫调控。
理化性质分析显示PE_PGRS29蛋白带负电荷的残基(Asp+Glu)总数为12,带正电荷的残基(Arg+Lys)总数为5,该蛋白的理论等电点pI为4.66,消光系数为9 970,280 nm处的吸光度为0.309。预测分析PE_PGRS29蛋白不稳定性指数为15.60,表明该蛋白为稳定蛋白。PE_PGRS29蛋白的二级结构分析显示无规则卷曲(Cc)169个,占45.68%,而酶活性部位和其他蛋白质特异的功能部位经常由这类有序的非重复性结构构成,BLAST软件分析证实PE_PGRS29蛋白含有1个结构域。Chai等[5]研究表明,PE_PGRS29蛋白含有真核生物样泛素化相关(UBA)结构域,UBA介导的泛素与PE_PGRS29的直接结合,招募自噬受体p62将分枝杆菌递送至LC3相关的自噬体,完成巨噬细胞中Mtb的异种清除。Bachhawat[18]研究发现,PE_PGRS蛋白中PE结构域普遍存在保守的DEVS(1个保守的四肽基序DEVS或DXXS,X为任意氨基酸),与蛋白caspase-3的识别位点DEVD/E基序高度相似,caspase-3蛋白可竞争性与磷酸化后的丝氨酸残基结合,抑制细胞凋亡,有助于逃避宿主细胞的免疫反应。预测分析PE_PGRS29蛋白含有10个丝氨酸磷酸化位点,表明PE_PGRS29蛋白可能参与这一抑制凋亡途径。本研究构建了PE_PGRS29蛋白的进化树以及多序列比对,发现PE_PGRS29蛋白与人类基因并无同源性,但在结核分枝杆菌菌属中同源性极高,保守性很强,含有多个T、B细胞抗原表位,提示PE_PGRS29蛋白可作为结核病治疗的潜在分子靶点以及对结核病新型候选疫苗有一定的参考价值。
结核分枝杆菌引起被感染的机体细胞自噬是降解细菌的重要过程,然而引发异种自噬的确切机制仍不清楚。本研究对PE_PGRS29蛋白进行了生物信息学分析,可为进一步研究PE_PGRS29蛋白的结构功能和理化性质提供参考,以期为结核病的预防和治疗提供新的分子靶点。

