基于改进ResUNet的乳腺癌细胞核图像分割
陈 立,魏钰欣,刘 斌,李 硕
(陕西科技大学 电子信息与人工智能学院,陕西 西安 710021)
0 引言
世界卫生组织国际癌症研究机构发布了2020年全球最新癌症数据,数据显示全球新发癌症病例达1 929万例,其中乳腺癌新发病例高达226万例,已经成为全球第一大癌[1]。在癌症的早期诊断中,细胞核的形态特征不仅有助于癌症评级,还有助于预测治疗效果[2]。因此,细胞核的精准分割成为医学图像分析和处理的基础,对病理诊断工作有十分重要的研究价值。
近年来,深度神经网络算法的崛起为细胞核分割带来了新进展,国内外很多研究者们已经在乳腺癌细胞分割领域做出了一系列研究。Su等[3]提出一种基于快速扫描深度卷积神经网络(Fast Scan Deep Convolution Neural Network, FSDNN)的组织病理学乳腺癌图像区域分割方法,应用于像素区域分割,在不牺牲性能的情况下去除原始卷积网络中的冗余计算。Xu等[4]提出深度卷积神经网络(Deep Convolution Neural Network, DCNN),以端到端的特征提取方式直接在原始像素强度值中学习,用于乳腺癌图像的上皮和间质区域的分割。Saha等[5]针对人表皮生长因子受体乳腺癌细胞图像提出了Her2Net分割网络,但多次特征融合增大了计算复杂度和参数量。Wang等[6]提出一种基于多层次特征和级联SVM分类的计算机辅助分级方法,用于自动区分低、中、高级别乳腺癌的组织病理图像。与传统算法相比,卷积神经网络算法效果显著优于人工提取特征的模型,在乳腺癌细胞的分割方面,上述分割网络已经取得较好的效果,但由于抗干扰能力不强,难以提取出更加抽象的图像特征,对于细胞核的分割效果不够精准。
近些年,U-Net模型[7]及以U-Net为基础进行改进的模型大范围地用于医学图像的分割,但还很少将其应用于乳腺癌细胞核图像分割中,故本文基于ResUNet[8]模型进行结构改进,使其能够更好地对乳腺癌细胞核进行分割。
1 ResUNet模型
ResUNet模型是在U-Net模型的基础上进行的改进,主要改进为在每层卷积处加入了残差连接,ResUNet模型的结构如图1所示。
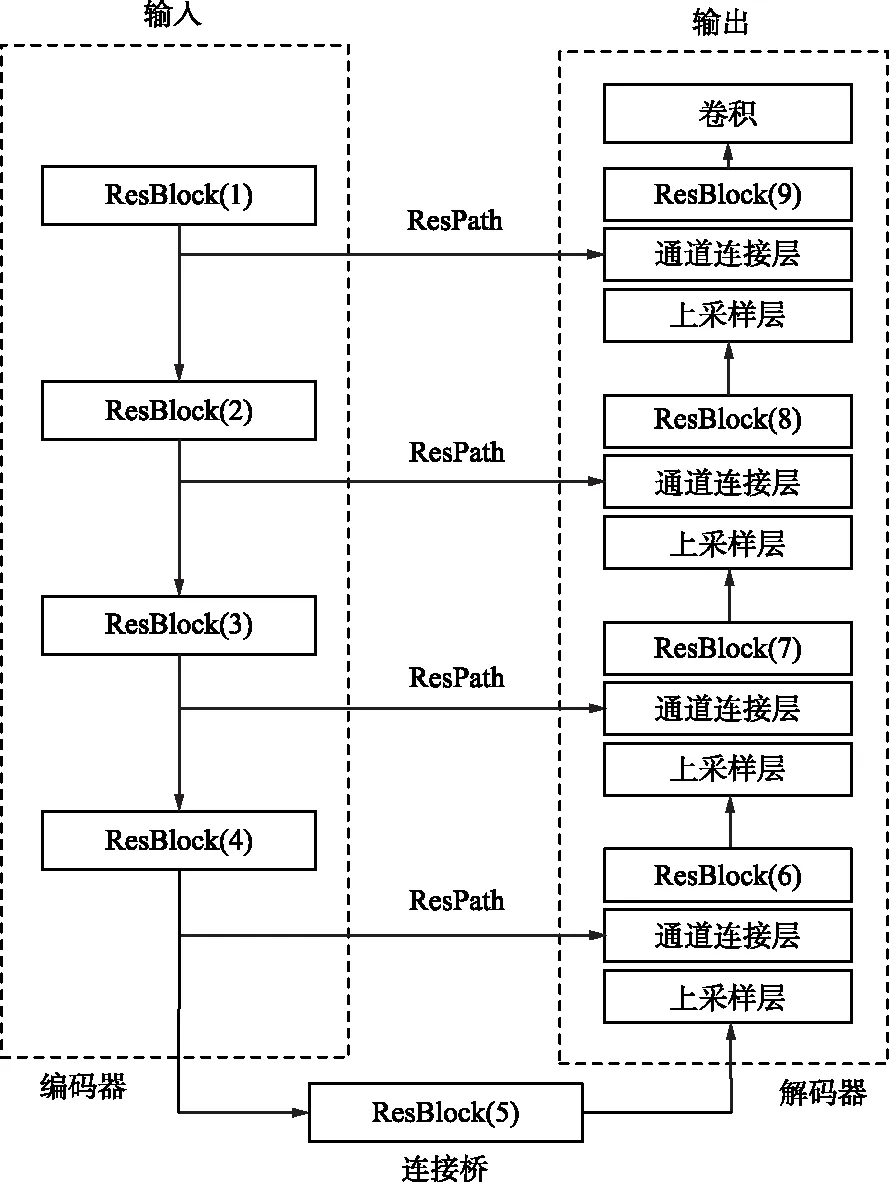
图1 ResUNet模型结构Fig.1 Structure of ResUNet model
ResUNet模型主要包括3部分:编码器、连接桥和解码器。编码器、连接桥和解码器均由如图2所示的ResBlock组成,用来提取图像特征。ResBlock在每层第2个卷积操作结束后添加了残差连接,残差连接需要和卷积层的初始输入具有相同分辨率,通过批归一化操作将每批输入的样本归一化为标准正态分布,缓解了模型训练中的梯度爆炸和消失现象,同时加速了收敛[9]。
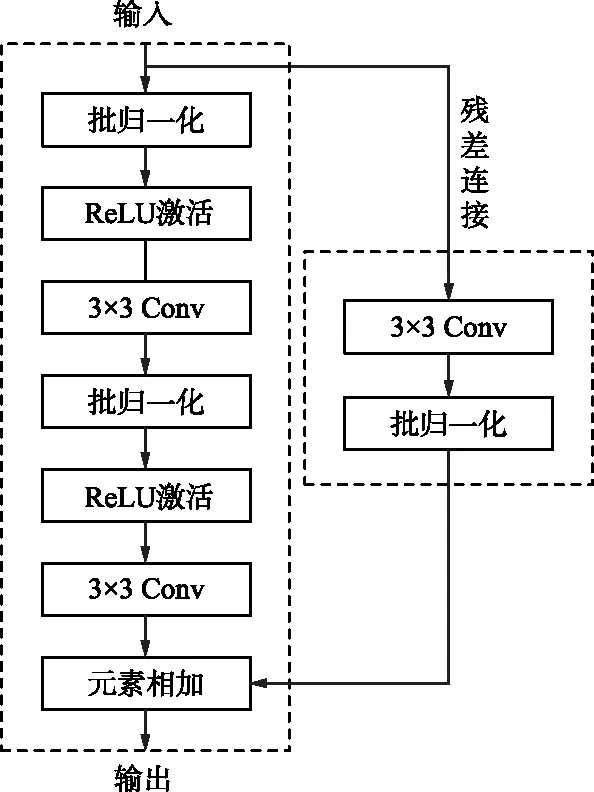
图2 ResBlock结构Fig.2 Structure of ResBlock
编码器部分由ResBlock连续经历4个下采样层组合而成,经过连接桥的一个ResBlock后进入解码阶段。解码器部分由采样层、通道连接层和ResBlock组合而成。通过上采样层将特征通道减半,尺寸扩大为原来的2倍,然后将对应下采样的特征图通过通道连接层拼接起来,再用1个ResBlock提取特征,此操作执行4次,最后得出分割结果。
2 本文方法
2.1 残差路径
ResUNet模型通过通道连接层将编码层的特征图与解码层的特征图进行拼接,通过通道连接层结合下采样和上采样过程中的深浅层次信息。浅层特征涵盖了更多的空间位置信息,这些丰富的浅层特征不可忽略,有利于细胞核的定位和分割,对分割结果影响很大。
编码器中的特征由于卷积层较浅,提取到的是浅层特征,而解码器中对应的特征由于卷积层更深,提取到是较深层次的特征,二者在语义上有较大差距,故本文提出了由残差块组成的残差路径(Residual Path,ResPath)。在模型的通道连接处加入ResPath,弥补编码器卷积层空间特征和解码器卷积层的语义特征的差距,使其在拼接前尽可能具有一致的深度,同时通过ResPath也可以更好地促进信息传播,保留图像特征信息。
本文提出的ResPath由残差块组成,使用1个3×3卷积和1个1×1卷积组成的固定残差块,如图3所示。与ResUNet中ResBlock不同的是,提出的残差块用1×1卷积代替了原3×3卷积作为残差连接,用1×1卷积做通道上的信息整合,ResPath的主要作用是进行图像间深、浅层特征信息的融合,为防止信息损失舍去了批归一化和ReLU激活函数[10]。

图3 残差块Fig.3 Residual block
本文在ResUNet模型每个同层上的编码器和解码器之间的通道连接层处添加1条ResPath,通道连接层的位置见图1。随着卷积层的增加,逐渐减少ResPath中残差块的个数。原ResUNet模型中共进行4次通道连接,实验选定从上至下分别使用包含4,3,2,1个残差块的ResPath。本文选取最上层通道连接处的ResPath结构进行展示,即从上至下第1条ResPath,包括4个残差块,如4图所示。
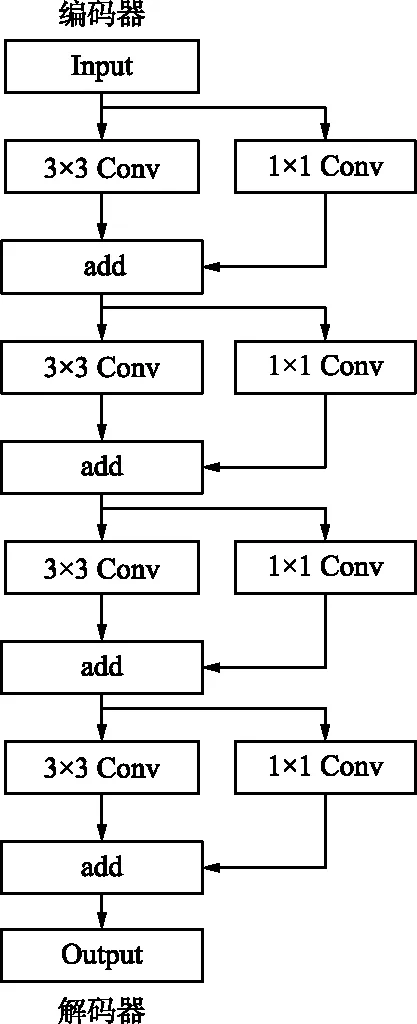
图4 残差路径Fig.4 Residual path
2.2 改进后ResUNet模型
改进后的ResUNet模型整体主要包括3部分:编码器、连接桥和解码器,共形成了9个ResBlock和一个普通卷积。
编码器除ResBlock(1)中最后一个卷积操作因不需要将特征图大小减半,设置卷积步长为1,其他ResBlock最后一个卷积操作的步长均为2,以此将特征图减小为原来的1/2。经过4个ResBlock后通过连接桥的ResBlock(5)后进入上采样阶段。上采样阶段首先经过Upsample将特征图大小扩大为原来的2倍后,将同层中编码器输出的特征经过ResPath后再与Upsample后的特征进行拼接,完成特征拼接后再进经过一个ResBlock,重复进行此操作4次后,最后通过一个1×1卷积和Softmax函数输出分割结果。
改进后ReUNet模型结构参数及每个步骤的输出大小如表1所示。

表1 改进后ResUNet模型各层参数设置Tab.1 Parameter setting of each layer of the improved ResUNet model
3 实验结果与分析
3.1 实验环境
使用TensorFlow架构来构建模型,实验平台配备Inter Core i7-6700HQ CPU,NVIDIA GeForce GTX 1080Ti 8 GB显存GPU,CUDA版本为10.0,编程语言为Python3.8。
3.2 数据集介绍
为验证改进后ResUNet模型的有效性,采用Naylor等[11]提供的乳腺癌数据对模型进行训练、验证和测试。该数据集选自11个病人的乳腺癌病理切片图像,再从每个病人的病理图中裁取了3~8幅图像并通过ITK-snap软件对每个细胞的细胞核核进行标注,共得到32幅图像。为验证改进后ResUNet模型是否具有普适性,用Kaggle平台提供的Data Science Bowl-2018公共医学细胞核数据集,该数据集包括670幅细胞核图像,含约29 000个细胞核。
3.3 数据预处理
为深度学习需要训练大量的数据来优化模型,而Naylor等提供的实验数据集的数量较少,故对数据集进行数据增强,提高模型的泛化能力。采用的数据增强操作包括水平翻转、平移缩放和随机裁剪等操作,共得到400张图片,按照7∶2∶1的比例随机将这400张图片分为训练集280张,验证集80张和测试集40张。Data Science Bowl-2018数据集不进行数据增强操作,将670张图片分为训练集480张,验证集120张和测试集70张。
考虑到特征并非总是连续值而是一些分类值[12],对标签进行One-Hot编码,将分类映射到欧式空间,使特征之间的距离计算更加合理[13]。
3.4 损失函数
NLLLoss(Negative Log-likelihood Loss)衡量模型语义分割的精度,该损失函数表达式如式(1)所示;Dice Loss衡量分割细胞核和对应标注图在形态特征方面的相似度,该函数表达式如式(2)所示:
(1)
(2)

本文按1∶1比例将NLLLoss和Dice Loss两个损失函数搭配使用,具有以下优点:
① Dice Loss从全局角度对分割结果进行衡量,NLLLoss从微观角度逐像素进行逼近,二者衡量方式的角度互补;
② 当出现图像前后景极不均衡情况时,NLLLoss无法解决这种极度不均衡情况,但Dice Loss不受前景大小的影响;
③ 当出现分割内容不均衡时,Dice Loss会趋向于学习大的块,而忽略小样本,NLLLoss依旧会对小样本进行学习;
④ NLLLoss对Dice Loss起到引导作用,如果在初始化时,只有Dice Loss则无法学习到正确的梯度下降方向,加上NLLLoss可以给网络一个学习的方向。
实验结果显示,使用NLLLoss和Dice Loss相结合的损失函数能够取得更好的分割效果。
3.5 优化函数
本文实验采用Adam(Adaptive Moment Estimation)优化函数,动态调整每个参数的学习率。与其他优化算法相比,Adam优化函数具有计算高效、占用内存较少等优点。
3.6 评价指标
为验证本文模型对乳腺癌细胞核分割的效果,采用平均交并比(Mean Intersection over Union,MIoU)、Dice系数、准确率(Accuracy,Acc)、精确率(Precision)、召回率(Recall)等评价指标对模型的分割性能进行评估。
MIoU表示真实值与预测值之间的交集同真实值与预测值之间并集的比值。Dice用于衡量分割结果和真实值之间的重合度。Acc表示预测结果中预测正确的比例。Precision表示正确分割的细胞核部分占预测结果为细胞核的部分的比例。Recall表示正确分割的细胞核部分占图像中真实的细胞核部分的比例。
(3)
(4)
(5)
(6)
(7)
式中,TP为被预测为正类的正样本;TN为被预测为负类的负样本;FP为被预测为正类的负样本;FN为被预测为负类的正样本;(k+1)为类别数。
3.7 实验过程结果及分析
实验使用除已提到的U-Net,ResUNet模型外,还选用了SegNet[14],Fcn8s[15],VggUNet[16],DenseUNet[17]与改进后的ResUNet模型(简称IResUNet模型)等7种分割模型分别在乳腺癌数据集和DSB数据集上进行实验,进行分割效果对比,验证模型的分割效果。
训练过程中利用Adam优化函数对网络进行优化,NLLLoss和Dice Loss函数共同衡量模型分割的精度,训练轮次数(epoch)设置为120,使用批训练,每批输入的样本数(Batch Size)设置为4,动量参数(Momentum)设置为0.9,初始学习率(Learning Rate)设置为0.000 1。用MIoU,Dice,Acc等评价指标对分割结果进行评判。
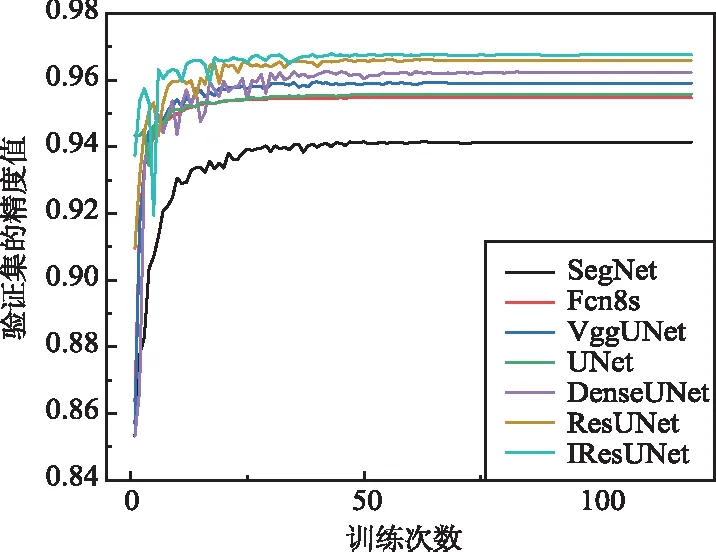
7种分割模型在2个数据集上训练过程中精度值和损失值变化情况分别如图5和图6所示。
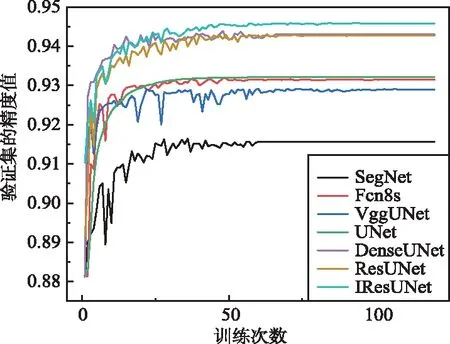
(a) 各模型的精度值变化

(b) 各模型的损失值变化图5 各模型在乳腺癌数据集训练过程中的精度和损失值变化Fig.5 Change of accuracy and loss values of each model during training on breast cancer datasets
(a) 各模型的精度值变化

(b) 各模型的损失值变化图6 各模型在DSB数据集训练过程中的精度和损失值变化Fig.6 Change of accuracy and loss values of each model during training on DSB datasets
7种分割模型在乳腺癌数据集和DSB数据集上的分割结果图在MIoU,Dice,Acc,Precision,Recall等评价指标上计算得出的结果如表2和表3所示。

表2 乳腺癌数据集上的分割结果Tab.2 Segmentation results on breast cancer dataset

表3 DSB数据集上的分割结果Tab.3 Segmentation results on DSB dataset
由表2可以看出,IResUNet模型在乳腺癌细胞核数据集上分割结果的MIoU,Dice,Acc等评价指标较ResUNet模型分别上分别提升了1.3%,1.2%,0.4%。为了更直观地展现各模型的分割结果,给出了各模型的分割结果,如图7所示。图7(a),(b)分别是乳腺癌细胞核的原图、对应标注图,图7(c)~(i)分别为SegNet,Fcn8s,VggUnet,U-Net,ResUNet,DenseUNet和IResUNet的分割结果。

(a) 乳腺癌细胞原图

(b) 乳腺癌细胞核标注图

(c) SegNet

(d) Fcn8s
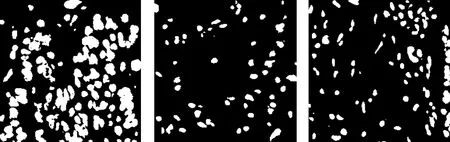
(e) VggUNet

(f) U-Net

(g) DenseUNet

(h) ResUNet

(i) IResUNet图7 分割结果对比 Fig.7 Comparison of segmentation results
由表3可以看出,IResUNet模型在DSB细胞核数据集实验结果的Miou,Dice,Acc等评价指标较ResUNet模型分别上分别提升了0.6%,1.6%,0.4%。由于分割结果较多,仅节选IResUNet模型在DSB数据集上的分割细节,如图8所示。图8(a),(b)分别表示DSB数据集上对应的细胞核的原图、标注图,图8(c)表示IResUNet模型的分割效果图。
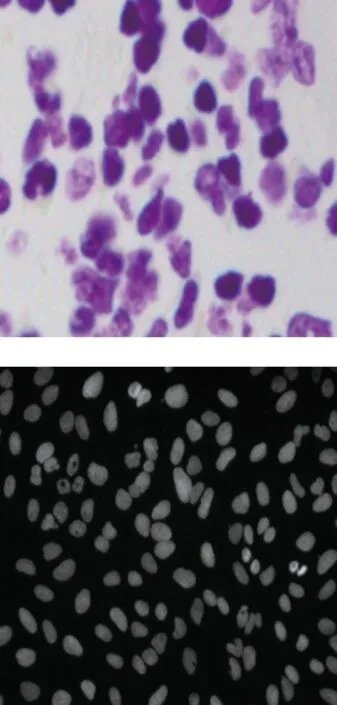
(a) 细胞核原图

(b) 标注图

(c) IResUNet图8 分割结果对比Fig.8 Comparison of segmentation results
训练过程中发现,IResUNet模型在epoch 80后验证集上的精度值和损失值趋于平稳达到瓶颈状态,此时进行参数微调,导入保存的模型,冻结前面部分训练权重[18]。冻结阶段模型的主干被冻结可以防止权值被破坏,特征提取网络不发生改变、占用的显存较小,仅对模型进行微调,可以加快训练效率,得到更有效的特征表示。经测试,该策略可小幅度提升模型的分割精度。训练过程中IResUNet模型在训练集和验证集上的精度值和损失值变化情况如图9和图10所示。

图9 冻结训练过程中IResUNet模型在乳腺癌数据集上精度值变化Fig.9 Change of accuracy values of IResUNet model on breast cancer dataset during freezing training
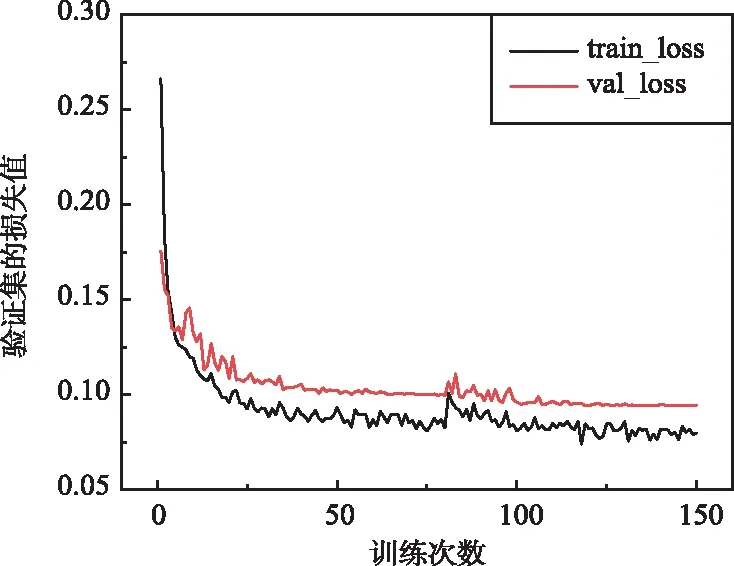
图10 冻结训练过程中IResUNet模型在乳腺癌数据集上损失值的变化Fig.10 Change of loss values of IResUNet model on breast cancer dataset during freezing training
冻结训练前后IResUNet模型在乳腺癌数据集上的分割结果与未进行冻结训练得到的分割结果如表4所示。由表4可以看出,使用冻结训练后IResUNet模型得到的预测图在MIoU,Dice,Acc等评价指标较未进行冻结训练前分别提升了0.4%,0.9%,0.1%,证明该策略是有效的。

表4 使用冻结策略前后分割结果对比Tab.4 Comparison of segmentation results before and after using freezing strategy
从图7和图8可以看出,SegNet,Fcn8s和VggUNet对于密集型细胞核图像的中心部分分割效果较差,对于一些颜色较浅的细胞核未能识别,存在欠分割现象较强。边缘细节部分的分割结果较为模糊,整体效果较差,但对于稀疏型细胞的中心部分预测效果相对较好。
U-Net对于细胞核图像的中心部分、边缘细节部分和较小的细胞核分割效果都比SegNet,Fcn8s和VggUNet好,对于一些独立的小细胞核存在过分割现象,对于密集型细胞核图像的边缘部分分割能力较差,整体效果更好。ResUNet和DenseUNet对于边缘细节部分和较小的细胞核分割效果比U-Net好,少数细胞核图像的中心部分分割效果不如U-Net。总体来说,ResUNet和DenseUNet的整体分割结果与标注图对比仍存在过分割和欠分割现象。
通过SegNet,Fcn8s,VggUnet,U-Net,DenseUNet,ResUNet的分割结果和IResUNet模型的分割结果对比分析得出,IResUNet模型分割结果最好。对于密集型细胞核图像的中心部分、边缘细节部分和较小的细胞核等分割细节的分割效果比其他模型的效果好,与给定标注图结果最接近。由以上结果得出,各模型在2个数据集上对于稀疏型细胞核的分割结果整体效果都较为可观,区别体现在对于密集型细胞核的分割。本文提出的IResUNet模型对于密集型细胞核的分割效果有明显提升。
4 结束语
乳腺组织切片细胞的医学图像常常存在细胞重叠、粘连频繁、细胞大小不一、细胞边界模糊等问题,成为细胞分割的难点,本文提出的IResUNet模型,提升了模型对背景中干扰元素的区分能力,改善了分割结果中常出现干扰点的情况,对于密集型细胞核的分割效果有明显提升,较ResUNet模型在MIoU,Dice,Acc等评价指标分别提升了1.8%,2.1%,0.6%。在DSB数据集上进行了验证,结果证明IResUNet模型具有较好的网络泛化能力和分割准确率,能够提高乳腺癌细胞核分割的精确度,可以更精准实现细胞核的分割,减少影像医师的工作负担,有助于乳腺疾病诊断及预后。
目前医学图像的数据集稀缺性导致可训练的数据量较少,不同医学中心的细胞核数据集中细胞核的形态、大小差异较大,因此如何在小数据集中防止过拟合问题的同时设计出分割精度更高的分割模型还有待研究。

