秦艽WRKY转录因子家族生物信息学分析
何懿菡,尹洋洋,胡 伟,李 铂,孙晓春,王 楠,黄文静,岳正刚
• 药材与资源•
秦艽WRKY转录因子家族生物信息学分析
何懿菡,尹洋洋,胡 伟,李 铂,孙晓春,王 楠,黄文静,岳正刚*
陕西中医药大学陕西省中药资源产业化省部共建协同创新中心秦药资源研究开发国家重点实验室(培育),陕西 咸阳 712046
利用生信技术分析秦艽WRKY家族,为进一步研究GmWRKY转录因子对秦艽次生代谢产物生物合成的分子调控机制提供信息。基于秦艽转录组注释结果挖掘WRKY家族成员,通过NCBI-tBlastx和SMART保守结构域预测进一步筛选得到41条WRKY序列并进行生信分析。基于getorf预测开放阅读框(open reading frame,ORF)区,对41条序列的ORF片段进行MEME保守基序分析和ClustalW比对,采用MEGAX构建邻接法(neighbor-joining,NJ)系统发育树。利用Omicshare GO对41个WRKY成员进行富集分析,利用String进行WRKY成员蛋白互作网络预测分析。通过转录组FPKM值对成员的基因表达情况进行热图分析,并对蛋白互作核心成员进行基因相对表达分析。41个秦艽WRKY成员中大部分成员的保守基序为“WRKYGQK”,仅GmWRKY27保守基序发生氨基酸突变,为“WRKYGKK”。依据WRKY特征结构域将41条序列的51个结构域分为3大类和8个亚类型,第I大类为氮端(N)和碳端(C),第II大类下设5个亚类型,以及第III大类;此外,系统进化树可将51个WRKY结构域分成4大组,Group I [I(N)]、GroupII(IIa+IIb)、GroupIII [I(C)+IIc]、GroupIV(IId+IIe和III),该结果与经典的WRKY家族分类特点基本一致。GO分析发现秦艽WRKY家族成员广泛参与代谢调控过程。当茉莉酸甲酯(jasmonic acid methyl ester,MeJA)诱导秦艽后,蛋白网络互作的核心成员GmWRKY1和GmWRKY17表达显著提高,推测这些WRKY成员与秦艽次生代谢产物的调控关系密切。为进一步开展秦艽WRKY转录因子功能研究奠定基础。
秦艽;WRKY转录因子;生物信息学;转录组分析;次生代谢产物
秦艽Pall.为“秦药”大宗道地中药材,是龙胆科(Gentianaceae)龙胆属L. 的一种多年生草本植物,以干燥根入药,具有祛风湿、清温热、止痹痛、退虚热之功效[1-2],其主要的次生代谢产物是以龙胆苦苷(gentiopicroside)为代表的裂环环烯醚萜苷类化合物[3],这类化合物是秦艽发挥临床疗效的物质基础,也是评价秦艽药材质量的重要指标[4]。
植物次生代谢产物也称“植保素”,是植物为了适应环境胁迫而产生的物质[5]。当植物受到外界胁迫后,体内会有一系列信号连锁反应,其中,转录调控是细胞内部调控网络中的重要一环,而转录因子能够通过协调转录酶与DNA模板的相互作用,来激活或抑制下游基因的表达[6]。WRKY是植物中重要的一类转录因子,可参与植物的生长发育[7],调控植物次生代谢产物的生物合成[8],以及响应各种生物和非生物胁迫[9]等生物进程。
WRKY转录因子调控药用植物次生代谢产物生物合成是当前研究的热点之一。研究发现,过表达可促进丹参毛状根中丹参酮含量的积累,其主要通过激活丹参酮合成途径酶关键基因来提高丹参酮的合成[10]。人参基因可响应病原菌的诱导,同时过表达基因可显著提高人参皂苷含量[11]。过表达基因可将短小蛇根草中喜树碱的含量提高3倍,干涉基因则显著降低喜树碱的含量[12]。过表达长春花基因可以显著提高代谢途径的关键酶基因色氨酸脱羧酶和转录抑制因子ZCT的表达,并抑制转录激活因子ORCA2、ORCA3和CrMYC2,从而使长春花转基因毛状根中的蛇根碱含量显著增加[13]。这些研究都表明WRKY转录因子在调控植物代谢产物积累中发挥着重要的作用。
本研究建立在秦艽转录组数据的基础上,对WRKY转录因子家族成员保守结构域、系统发育关系、基因表达水平、转录因子蛋白互作等方面进行了分析,为后期研究WRKY转录因子调控秦艽次生代谢产物的机制奠定基础。
1 材料
样品采自甘肃庆阳正宁县由陕西中医药大学李铂教授鉴定为秦艽Pall.的种子,于温室撒播种子给予16 h 光照,8 h 黑暗处理,覆膜出苗后培养一年生用于试验。多通道荧光定量PCR仪qTOWER 2.0(德国耶拿公司)。
2 方法
2.1 秦艽WRKY转录因子家族成员的鉴定
研究前期以50 μmol/L柠檬酸铵(ammonium citrate,AC)、200 μmol/L茉莉酸甲酯(jasmonic acid methyl ester,MeJA)为诱导子,水处理为对照分别处理秦艽幼苗6 h,送于深圳千年基因(Macrogen公司,中国)完成秦艽转录组测序和分析。本研究基于转录组基因注释结果,初步筛得74条潜在的秦艽基因,逐条进行NCBI-tBlastx序列相似度比对,参数为默认,以判定序列确为WRKY家族。并进一步利用SMART数据库(http://smart. embl.de/)比较序列家族特征,去掉WRKY结构域不完整的序列,最终筛选得到41条基因。
2.2 GmWRKY保守基序分析、蛋白多重比对以及系统进化树的构建
首先利用getorf(http://emboss.bioinformatics.nl/ cgi-bin/emboss/getorf)在线分析41条序列的开放阅读框(open reading frame,ORF)区,并选择含有WRKY基序的ORF长片段。随后,通过MEME在线软件(https://meme-suite.org/meme/)对秦艽WRKY蛋白序列进行保守基序鉴定,参数选择分析3个保守区。利用MEGAX[14]对秦艽WRKY家族序列的ORF区域进行ClustalW比对,选取保守区,采用邻接法(neighbor-joining,NJ)进行系统进化树的构建,Bootstrap检测值为1000,其他使用参数均为默认值。
2.3 GmWRKY转录因子家族成员(GO)注释及蛋白质网络互作
利用Omicshare GO富集分析工具建立WRKY家族转录组注释信息。利用String蛋白互作数据库(http://string-db.org/)对秦艽WRKY蛋白进行互作网络预测分析,选择拟南芥为参考物种。并利用Cytoscape 3.8.0[15]对互作结果进行视图优化。
2.4 GmWRKY转录因子家族成员的表达分析
基于转录组数据挖掘MeJA、AC和对照组的GmWRKY转录因子的FPKM值,用Omicshare热图分析工具对FPKM值进行聚类分析和可视化,分析类型选pearson。
对蛋白互作网络核心成员和进行基因相对表达分析,为内参[16],引物信息见表1。用200 μmol/L MeJA和对照水分别喷施处理秦艽幼苗6 h,收集对照和处理组的叶,重复3组,分别提取RNA,对1%凝胶电泳和Nanodrop检测合格的RNA用于后续的定量实验。使用德国耶拿荧光定量梯度PCR仪qTOWER 2.0,反应条件为95 ℃、30 s预变性,95 ℃、5 s,60 ℃、30 s,40个循环。利用2−ΔΔCt计算基因相对表达水平[17]。
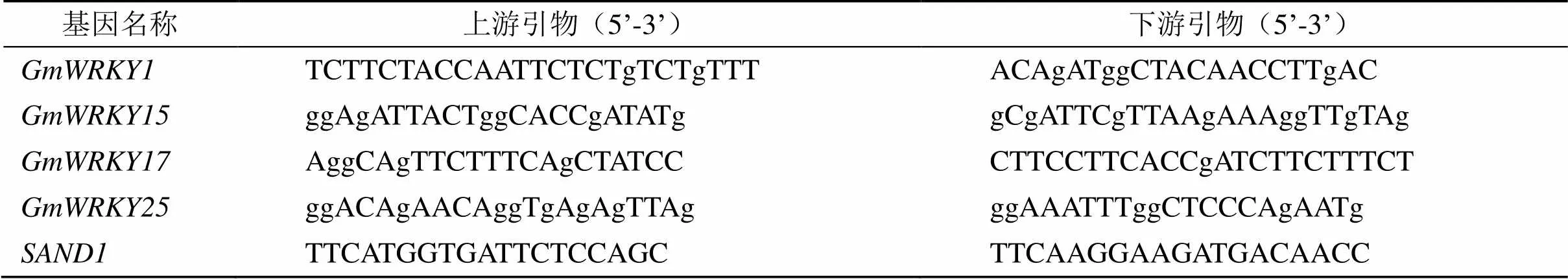
表1 实时荧光定量基因表达序列引物
3 结果
3.1 GmWRKY转录因子家族成员的鉴定
在秦艽转录组数据有74条基因被注释为秦艽基因,序列最短为209 bp,最长为2671 bp。通过对74条基因逐条进行NCBI-tBlastx比对,初步判定序列均为WRKY家族成员,但因部分序列信息不完整,结构域缺失,结合在线软件SMART分析结果,去除保守结构域不完整的序列,将明确具有WRKY保守结构域的41条序列列为秦艽WRKY家族成员,并依次命名为GmWRKY1~GmWRKY41,作为后续研究的对象。
3.2 GmWRKY家族成员蛋白质保守区分析
使用在线软件MEME分析41条秦艽WRKY家族蛋白的保守基序,经分析比对后3个保守区Motif1、Motif2、Motif3的基序长度分别为29、29、50个氨基酸。其中,Motif 1和Motif 3是基因的七肽保守序列,Motif 2是秦艽基因的特征基序(图1)。41条序列中,其中34条序列包含Motif1和Motif2,这34条序列中又有10条序列含Motif3。此外,GmWRKY7、24、29和31只含有1个保守区Motif1;GmWRKY16、26和30只含有1个保守区Motif2。
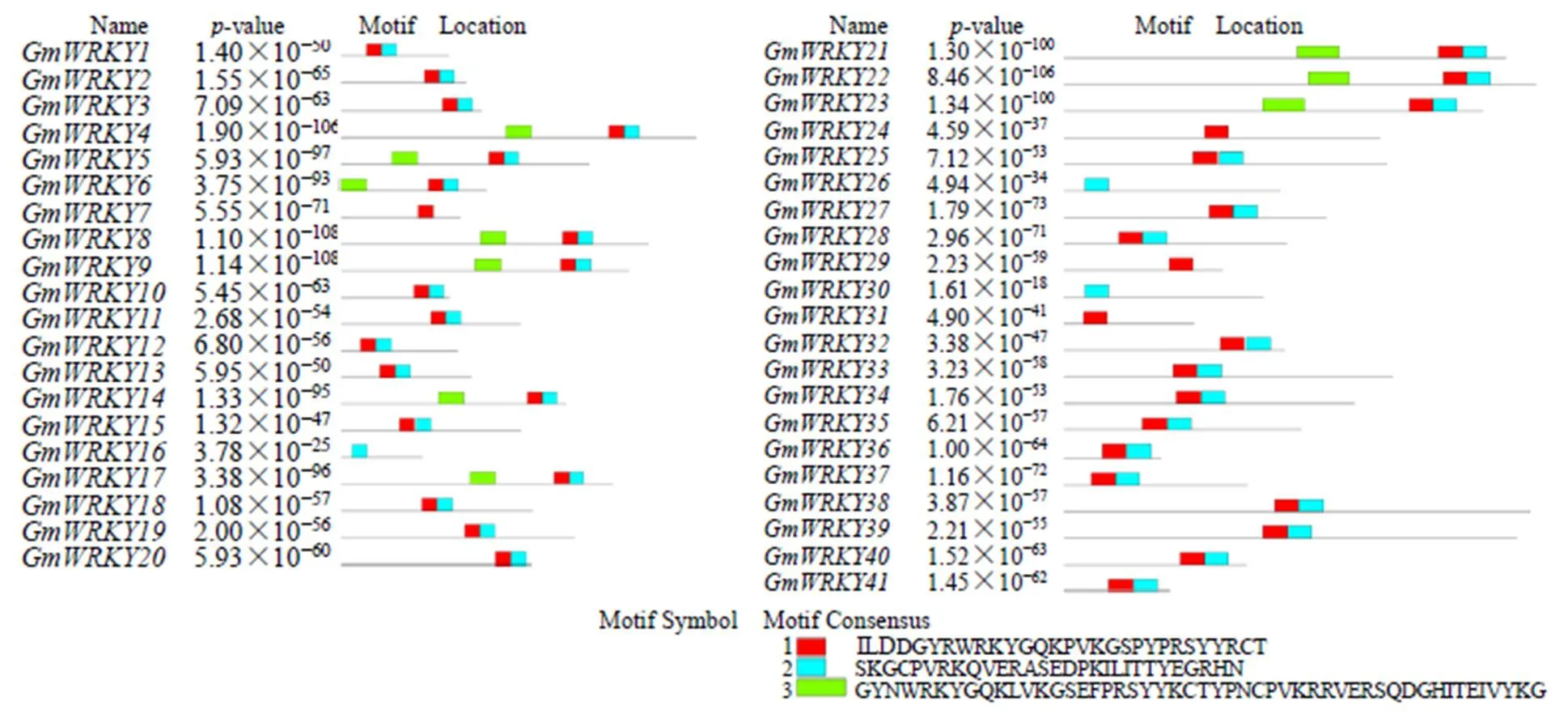
图1 秦艽41个WRKY家族成员蛋白保守基序分析
3.3 蛋白质序列比对及系统进化分析
多重序列比对分析结果发现41个GmWRKY蛋白均含有完整的结构域核心序列(图2)。大部分序列具有WRKY家族的保守基序“WRKYGQK”,仅GmWRKY27蛋白保守基序中的“Q”变成“K”。依据WRKY家族的结构特征,将41个WRKY蛋白分为3大类,其中10个蛋白为第I类,该类蛋白含有2个“WRKYGQK”序列,分别位于序列的N端和C端,以及C2H2型锌指结构。第II类有23个蛋白,特点是含有1个WRKY结构域,锌指结构为C2H2型,该大类可根据序列差异可进一步细化为5个小类,即IIa~IIe。第III类有6个WRKY转录因子,含有1个WRKY结构域,锌指结构为C2HC型。GmWRKY7和GmWRKY16与其他家族成员之间的保守氨基酸位点存在较大差异,分类不明确。
基于MEGAX比对秦艽41个WRKY家族成员的51个WRKY保守域,并构建系统进化树,其结果与保守结构域分类情况基本一致。系统进化树可将秦艽的3大类WRKY家族成员分为4大组(图3),Group1 [I (N)]、Group2 [IIc+I(C)]、Group3(IIa+IIb)、Group4(IId+IIe和III)。其中,Group2与Group1亲缘关系较近,聚为一大支。Group3与Group4亲缘关系近,被聚为另一大支。对于分类不明确的GmWRKY7和GmWRKY16分别聚类到了I(C)和I(N),与第I类亲缘关系较近。
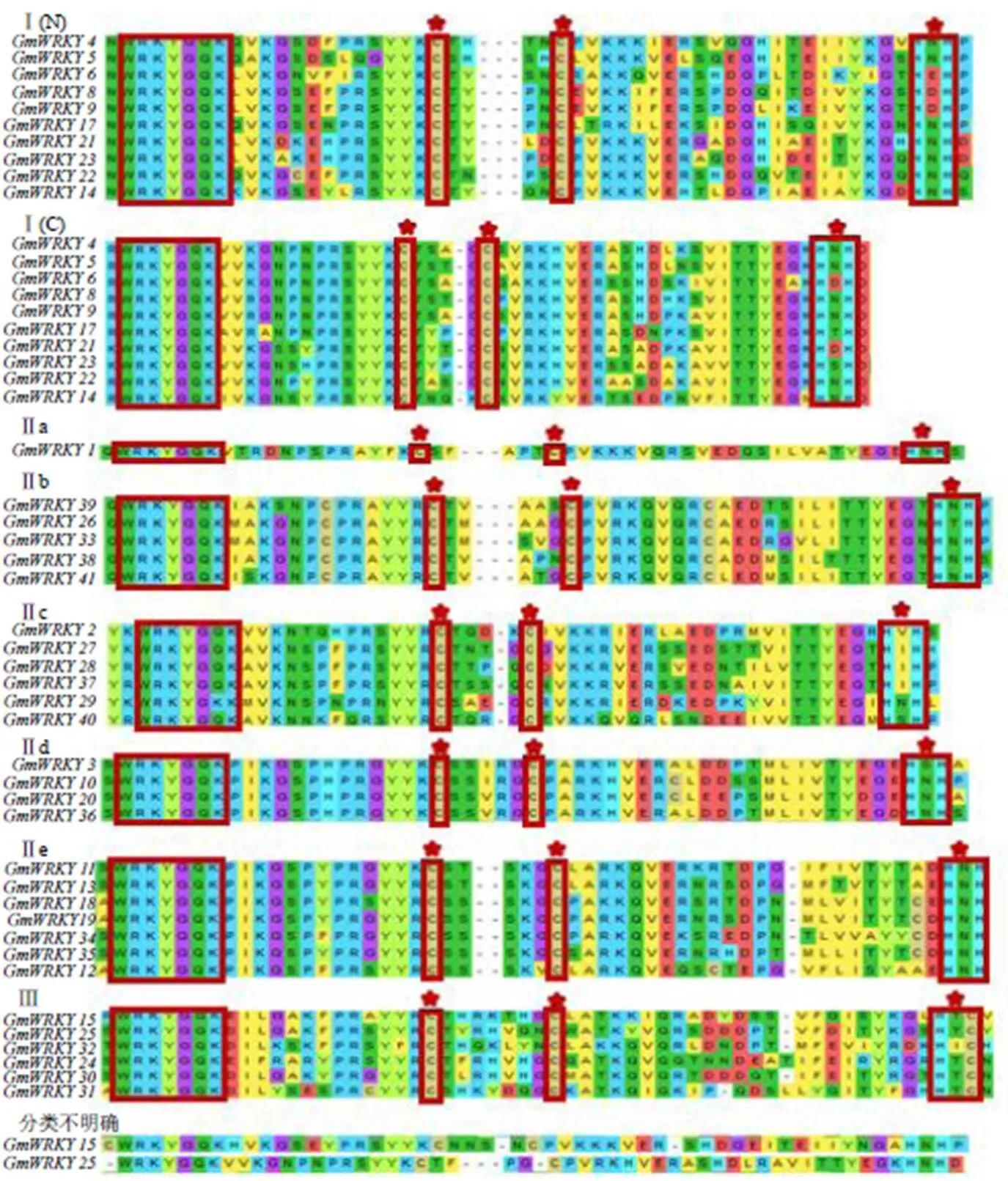
红色方框表示WRKY核心结构域,红色方框+ 表示锌指结构;I类的2个WRKY结构域,分别标识为“N”和“C”端
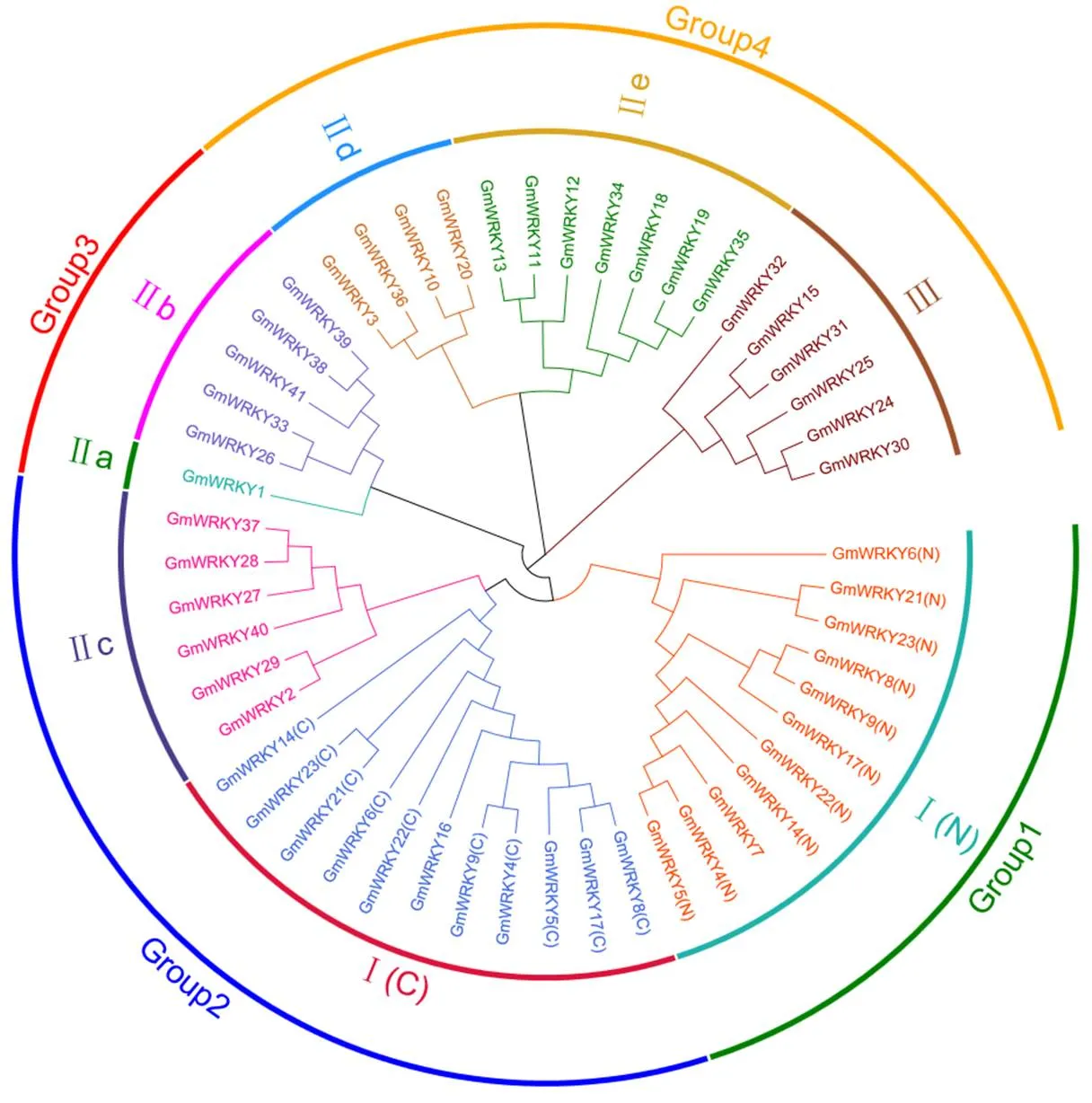
图3 WRKY家族的系统发育树
3.4 GmWRKY转录因子家族成员GO注释
基于转录组GO数据库信息,对秦艽WRKY转录因子进行功能注释。对于大部分秦艽基因在GO的二级分类的生物过程(biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)3个大类中得到注释(图4)。在生物过程中细胞过程(cellular process)和代谢过程(metabolic process)富集了40条序列信息。在细胞组分中有40条可富集于细胞(cell)、细胞部分(cell part)和细胞器(organelle)。分子功能中有40条序列可富集于绑定(binding)和核酸结合转录因子活性(Nucleic acid binding transcription factor activity)。
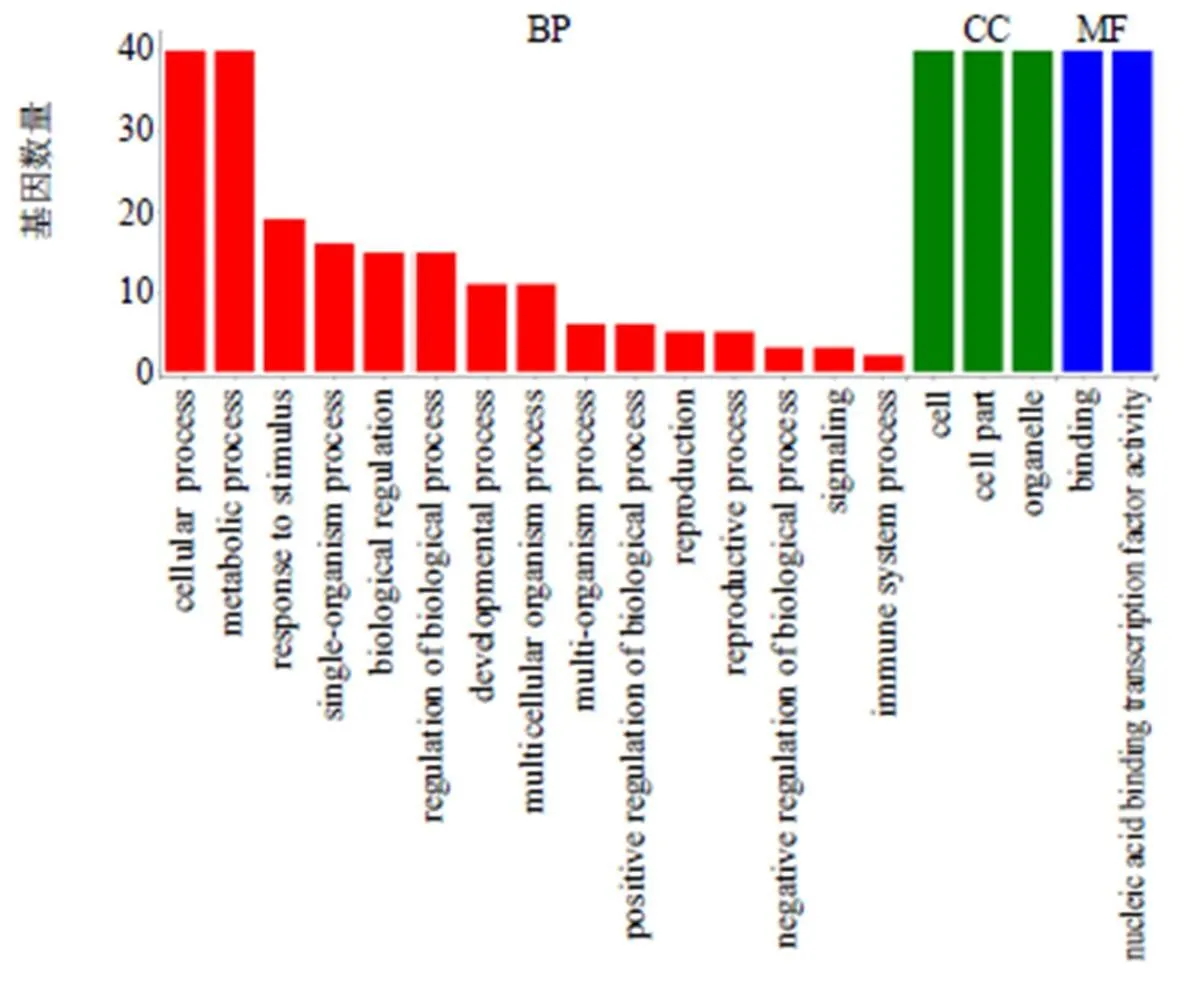
图4 秦艽WRKY家族成员的GO注释信息
3.5 GmWRKY蛋白互作网络预测
基于String数据库,构建了秦艽41条WRKY转录因子家族成员的蛋白互作网络,去除没有相互关系作用的成员,秦艽WRKY家族中的11个成员间有蛋白质互作的关系(图5)。可依据相互作用分为3块互作网络。其中,GmWRKY1、GmWRKY17、GmWRKY25和GmWRKY15为互作网络中的4个主要中心节点,相互作用关系最强。这4个主要中心节点与GmWRKY31、GmWRKY13和GmWRKY28之间也存在着相互作用关系。GmWRKY4和GmWRKY16,GmWRKY39、GmWRKY19和GmWRKY41为2块具有独立互作的蛋白网络。
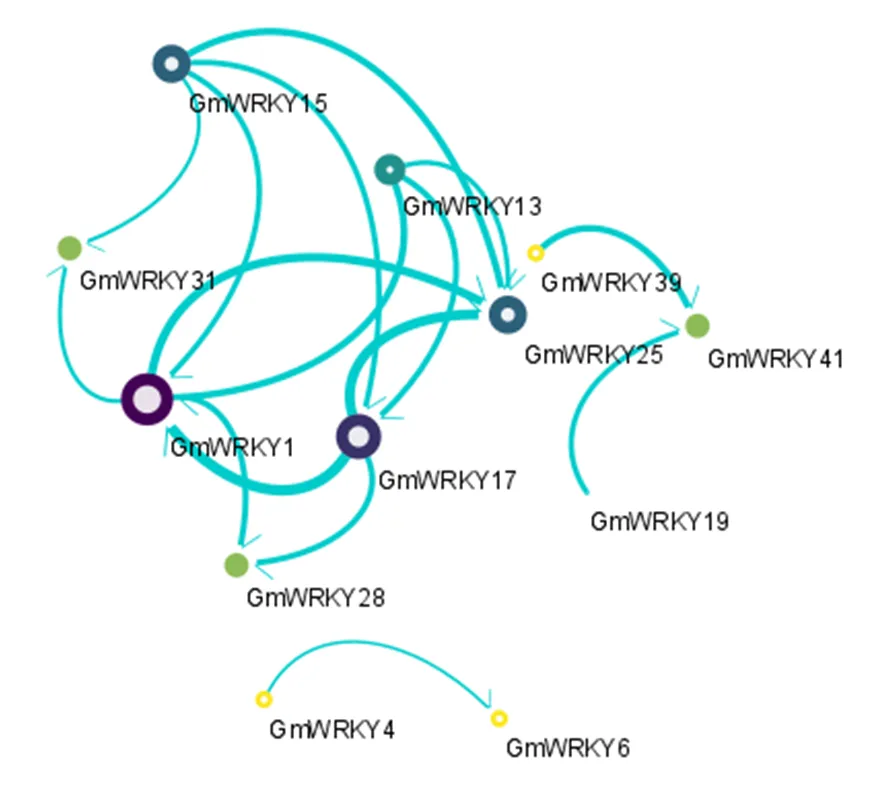
图5 秦艽WRKY家族成员的蛋白互作网络
3.6 GmWRKY转录因子家族成员表达模式分析
基于秦艽转录组数据的FPKM值,对WRKY基因在MeJA和AC诱导后的表达水平差异进行热图分析,和对照(CK)相比,41条GmWRKY家族成员受诱导调控显著,且不同成员对于MeJA和AC诱导响应强度不同(图6)。基因表达聚类结果显示,至,和对照相比,MeJA诱导后表达明显降低。至在MeJA和AC诱导后普遍降低。至在MeJA和AC诱导后普遍升高。至在AC诱导后表达显著升高。至在AC诱导后表达显著升高,而对MeJA诱导响应不强。至,和对照相比,MeJA诱导后表达明显升高而AC诱导后表达普遍降低。

图6 基于转录组FPKM值分析秦艽GmWRKY基因表达差异
以秦艽WRKY家族成员蛋白互作网络关系最强的4个节点GmWRKY1、GmWRKY17、GmWRKY25和GmWRKY15进行qRT-PCR基因表达分析(图7)。当秦艽受到MeJA诱导后,叶中表达量显著升高,其次是,和差异不显著。
4 讨论
WRKY转录因子家族因N端包含高度保守的WRKY结构域“WRKYGQK”而得名,该结构域能专一地与靶基因启动子区中的W盒(TTGACC/T)序列结合,激活或抑制基因的表达,进而调控植物的生长代谢,参与响应应激等各种生理活动[18]。秦艽41条GmWRKY家族成员WRKY结构域保守,大部分为“WRKYGQK”,仅GmWRKY27突变为“WRKYGKK”。在丹参、水稻、拟南芥等物种中也发现了类似的突变体,这种突变的产生也可能与物种长期进化有关[19]。有研究发现突变基序中谷氨酰胺(glutamine,G)氨基酸的突变会降低保守基序与DNA结合的能力[20]。对于WRKY结构域突变体的进化事件、以及突变体参与植物代谢调控的机制还需要进一步的研究。
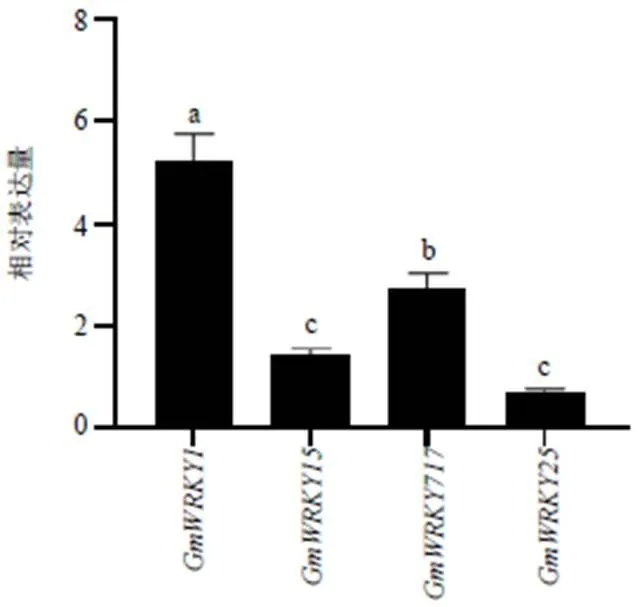
不同字母表示差异显著P<0.05
Eulgem等[21]依据WRKY家族成员的结构特点将其分为3大类型I(N端+C端)、II(a、b、c、d、e)和III。本研究对GmWRKY蛋白家族成员进行多重比对、保守基序分析结果发现秦艽WRKY家族成员也符合该分类特点,这些结果表明,不同物种的WRKY结构保守,功能可能也会比较相似。此外,系统进化树依据亲缘关系将GmWRKY保守域分为2大支,4大组。II类型的WRKY成员不单独分为一类,说明其并不是单系起源,且具有两个WRKY结构域基因的C端可能是具有一个WRKY域编码基因的祖先[22]。有研究发现许多来自不同系统发育群的基因参与植物响应同样的胁迫应激,说明WRKY家族可广泛参与植物的各种生理代谢调控网络,对WRKY家族的起源、进化关系的了解可以为其功能的研究提供有价值的信息[22]。
随着转录组测序技术的发展,越来越多药用植物的WRKY转录因子家族被挖掘和研究。WRKY家族在阳春砂和丹参中被挖掘,并预测了参与次生代谢产物合成相关的WRKY转录因子[23-24]。在红豆杉中挖掘了61条WRKY家族,并发现过表达WRKY成员能够提高目标药用成分的含量[25]。本研究基于秦艽转录组挖掘了41条WRKY转录因子,通过GO数据分析发现这些家族成员中有40条具有与核酸结合的转录因子活性,并广泛参与代谢调控过程。基于转录组数据的基因表达热图显示,WRKY成员对MeJA和AC的响应强度不同,推测不同成员在植物体内参与代谢调控的方式可能存在差异。此外,网络互作中的核心成员在受到MeJA诱导后基因表达均有所升高,其中和升高最为显著,推测这些WRKY成员与秦艽次生代谢产物的调控关系密切。对秦艽WRKY转录因子家族的分析将为进一步研究WRKY转录因子参与调控裂环环烯醚萜苷类代谢产物合成途径奠定基础。
利益冲突 所有作者均声明不存在利益冲突
[1] 中国药典 [S]. 四部. 2020: 45.
[2] 胡本祥, 彭亮, 杨冰月, 等. “秦药”的现代研究概况 [J]. 中草药, 2018, 49(21): 4949-4959.
[3] 陈千良, 石张燕, 涂光忠, 等. 陕西产秦艽的化学成分研究 [J]. 中国中药杂志, 2005, 30(19): 1519-1522.
[4] 马丽娜, 张铁军, 田成旺, 等. 大孔树脂分离纯化川西獐牙菜中环烯醚萜苷类和酮类成分的工艺研究 [J]. 中草药, 2010, 41(2): 227-231.
[5] 郭兰萍, 周良云, 康传志, 等. 药用植物适应环境胁迫的策略及道地药材“拟境栽培” [J]. 中国中药杂志, 2020, 45(9): 1969-1974.
[6] Lee T I, Young R A. Transcription of eukaryotic protein-coding genes [J]., 2000, 34: 77-137.
[7] Yang Y, Chi Y J, Wang Z,. Functional analysis of structurally related soybean GmWRKY58 and GmWRKY76 in plant growth and development [J]., 2016, 67(15): 4727-4742.
[8] Schluttenhofer C, Yuan L. Regulation of specialized metabolism by WRKY transcription factors [J]., 2015, 167(2): 295-306.
[9] Wani S H, Anand S, Singh B,. WRKY transcription factors and plant defense responses: Latest discoveries and future prospects [J]., 2021, 40(7): 1071-1085.
[10] Deng C P, Hao X L, Shi M,. Tanshinone production could be increased by the expression of SmWRKY2inhairy roots [J]., 2019, 284: 1-8.
[11] Yao L, Wang J, Sun J C,. A WRKY transcription factor, PgWRKY4X, positively regulates ginsenoside biosynthesis by activating squalene epoxidase transcription in[J]., 2020, 154: 112671.
[12] Hao X L, Xie C H, Ruan Q Y,. The transcription factor OpWRKY2positively regulates the biosynthesis of the anticancer drug camptothecin in[J]., 2021, 8(1): 7.
[13] Suttipanta N, Pattanaik S, Kulshrestha M,. The transcription factor CrWRKY1positively regulates the terpenoid indole alkaloid biosynthesis in[J]., 2011, 157(4): 2081-2093.
[14] Kumar S, Stecher G, Li M,. MEGA X: Molecular evolutionary genetics analysis across computing platforms [J]., 2018, 35(6): 1547-1549.
[15] Shannon P, Markiel A, Ozier O,. Cytoscape: a software environment for integrated models of biomolecular interaction networks [J]., 2003, 13(11): 2498-2504.
[16] He Y H, Yan H L, Hua W P,. Selection and validation of reference genes for quantitative real-time PCR in[J]., 2016, 7: 945.
[17] Erickson H S, Albert P S, Gillespie J W,. Assessment of normalization strategies for quantitative RT-PCR using microdissected tissue samples [J]., 2007, 87(9): 951-962.
[18] Bakshi M, Oelmüller R. WRKY transcription factors: Jack of many trades in plants [J]., 2014, 9(2): e27700.
[19] 田云, 卢向阳, 彭丽莎, 等. 植物WRKY转录因子结构特点及其生物学功能 [J]. 遗传, 2006, 28(12): 1607-1612.
[20] Duan M R, Nan J, Liang Y H,. DNA binding mechanism revealed by high resolution crystal structure ofWRKY1protein [J]., 2007, 35(4): 1145-1154.
[21] Eulgem T, Rushton P J, Robatzek S,. The WRKY superfamily of plant transcription factors [J]., 2000, 5(5): 199-206.
[22] Zhang Y J, Wang L J. The WRKY transcription factor superfamily: Its origin in eukaryotes and expansion in plants [J]., 2005, 5: 1.
[23] He X Y, Wang H, Yang J F,. RNA sequencing onLour. induced by MeJA identifies the genes of WRKY and terpene synthases involved in terpene biosynthesis [J]., 2018, 61(2): 91-102.
[24] Yu H Z, Guo W L, Yang D F,. Transcriptional profiles offamily genes and their putative roles in the biosynthesis of tanshinone and phenolic acids in[J]., 2018, 19(6): E1593.
[25] Zhang M, Chen Y, Nie L,. Transcriptome-wide identification and screening of WRKY factors involved in the regulation of taxol biosynthesis in Taxus chinensis [J]., 2018, 8(1): 5197.
Bioinformatics analysis of WRKY transcription factor family of
HE Yi-han, YIN Yang-yang, HU Wei, LI Bo, SUN Xiao-chun, WANG Nan, HUANG Wen-jing, YUE Zheng-gang
State Key Laboratory of Research & Development of Characteristic Qin Medicine Resources (Cultivation), Shaanxi Collaborative Innovation Center of Chinese Medicinal Resources Industrialization, Shaanxi university of Chinese, Xianyang 712046, China
To analyze theWRKY family of(GmWRKY) using biotransformation techniques in order to provide information for further research on the molecular regulation mechanism of GmWRKY transcription factor on the biosynthesis of the secondary metabolites of.Based on the annotation results of WRKY family members, 41 WRKY sequences were further screened by NCBI-tBLastx and SMART domains for bioinformatics analysis. Based on getorf prediction of open reading frame (ORF) region, MEME conservative motif analysis and ClustalW alignment were performed on ORF fragments of 41 sequences, and the neighbor-joining (NJ) phylogenetic tree was constructed by MEGAX. The enrichment analysis of 41 WRKY members was performed using Omicshare GO. And prediction analysis of WRKY member protein interaction network was performed by String. The expression of WRKY members was analyzed by heat map based on the FPKM value of the transcriptome, and the relative gene expression of the core members of GmWRKY protein interaction was analyzed.Most of the 41 GmWRKY members had a conserved motif of "WRKYGQK", and only THE GmWRKY27 conserved motif had an amino acid mutation of "WRKYGKK". According to the WRKY characteristic domain, 51 domains of 41 sequences were divided into three categories and eight subtypes. The first category consisted of nitrogen terminal (N) and carbon terminal (C), the second category consisted of 5 subtypes and the third category. Phylogenetic tree can divide 51 WRKY domains into four groups: GroupI [I(N)], GroupII (IIa+IIb), GroupIII [I(C)+IIc], GroupIV (IId+IIe and III). These results were consistent with the classic WRKY family classification. GO analysis revealed that members of the GmWRKY family were extensively involved in metabolic regulation. The expression ofand, the core members of protein network interaction, were significantly increased whenwas induced by MeJA, suggesting that these WRKY members were closely related to the regulation of secondary metabolites of.This study can lay a foundation for further research on the function of GmWRKY transcription factors..
Pall.; WRKY transcription factors; bioinformatics analysis; transcriptome analysis; secondary metabolites
R286.12
A
0253 - 2670(2022)23 - 7499 - 08
10.7501/j.issn.0253-2670.2022.23.021
2022-05-06
国家自然科学基金项目(81903753);陕西中医药大学科学基金项目(2017PY06)
何懿菡,讲师,研究方向为药用植物资源及次生代谢产物调控。Tel: (029)38183207 E-mail: annaaid@126.com
通信作者:岳正刚,副教授,研究方向为中药药效物质基础。Tel: (029)38183301 E-mail: liuxingjian1981@163.com
[责任编辑 时圣明]

