蝽科线粒体基因组tRNA和rRNA基因的比较及应用(半翅目:蝽总科)
廉 丹, 魏久锋, 丁晓飞, 刘迎香, 赵 清
(山西农业大学植物保护学院,山西 太谷 030801)
蝽科是半翅目异翅亚目中最常见的大科之一,世界性分布,全世界已知900多个属近5 000种[1].蝽科昆虫均为陆生,大多数为植食性种类,是重要的农业和园艺植物害虫.如二星蝽属的二星蝽(Eysarcorisguttiger)、拟二星蝽(E.annamita)和广二星蝽(E.ventralis)是水稻的重要害虫[2].部分种类如益蝽亚科的蠋蝽(Armachinensis)能够有效控制鳞翅目、鞘翅目、膜翅目和半翅目等的多种害虫,是重要的捕食性天敌昆虫[3].因此,该科昆虫具有十分重要的经济意义.
昆虫线粒体基因组是长度为15~20 kb的闭合环状双链DNA分子,编码37个基因,包括13个蛋白质编码基因(PCG),2个核糖体RNA基因(rRNA)和22个转运RNA基因(tRNA)[4-5].作为一种分子标记,线粒体基因组以其独特的优势,如体积小、拷贝数高、缺少重组子和内含子等特点,在物种鉴定、系统发育学以及谱系遗传学等研究领域得到广泛应用[6].研究表明[7],相比于以单基因进行的系统发育分析,线粒体基因组提供了相对丰富的遗传信息,可以更好地推断不同类群之间的系统发育关系和亲缘程度.早前研究中,蝽科的系统发育分析大多基于单个蛋白质编码基因,如卜云等[8]基于cox2基因序列数据,分别采用邻接法、最大简约法和贝叶斯推论法3种方法建立蝽科4亚科间的系统发育关系;代金霞等[9]基于cytb基因探讨了蝽科8个物种之间的系统发育关系,而对多个RNA基因包含的系统发育信息并未较好地挖掘和利用.
线粒体tRNA是具有携带并转运氨基酸功能的一类小分子核糖核酸,其核苷酸序列、基因含量和基因排列方式等,均可为系统发育树的构建提供信息[10-11].其序列较短,包含的系统发育信息较少,所以,在长期以来的系统发育研究中,大多只关注其在线粒体基因组中的位置变化和二级结构,而对于不同分类阶元水平上基于tRNA基因的系统发育研究十分欠缺[10].事实上,基于tRNA基因串联数据集构建系统发育树时,相比于单个tRNA基因可以得到较多的有效信息.在已有的研究中,Wang et al[11]通过对盲蝽科22个tRNA基因串联数据集的K-2-P遗传距离和Ka值的分析以及系统发育树的构建,表明tRNA基因在系统发育研究中具有较大的潜力.同时,近年来的研究表明[12-13],12S rRNA和16S rRNA序列远长于tRNA,含有较多的系统发育信息,可作为系统发育推断的良好分子标记,用于不同分类水平的系统发育研究.在蝽科系统发育研究中,较多的是基于13个蛋白质编码基因进行的,而基于tRNA和rRNA数据的研究较少,因此基于tRNA以及rRNA对蝽科线粒体基因组学进行比较分析是有必要的.
1 材料与方法
1.1 试验材料
本研究以半翅目蝽科昆虫为研究对象,选取NCBI网站已公布的蝽科4亚科26个物种的线粒体全基因组序列,对其tRNA和rRNA序列特征、遗传距离和二级结构进行比较分析,同时基于校正后的tRNA串联数据集和2个rRNA串联数据集,采用贝叶斯法对26种蝽科昆虫进行系统发育树的构建,探讨蝽科4亚科间的系统发育关系,并对tRNA和rRNA序列在蝽科系统发育关系构建中的有效性进行验证,补充tRNA和rRNA在蝽科线粒体基因组研究中的不足.

表1 本研究所选物种Table 1 List of species used in this study
以半翅目蝽科4亚科26个物种为研究对象,覆盖了中国蝽科的所有亚科,所使用的序列从NCBI(https://www.ncbi.nlm.nih.gov/)数据库下载(表1).
1.2 分析方法
将获得的26个物种的线粒体全基因序列,在Geneious Prime 2022.1[14]中提取22个tRNA和2个rRNA,并通过MITOS在线网站(http://mitos.bioinf.uni-leipzig.de/index.py/)推测其二级结构[15].通过Sequencematrix分别将22个tRNA和2个rRNA序列串联,利用MEGA11.0[16]计算tRNA和rRNA的碱基长度、碱基组成、AT含量,AT偏斜和GC偏斜(AT-skew=[A-T]/[A+T]和GC-skew=[G-C]/[G+C])[17],同时计算其基因保守位点、变异位点、简约信息位点和单一位点,基于Kimura-2-Parameter(K-2-P)计算亚科间和蝽亚科属间的遗传距离,并且用Muscle对每个同源基因进行比对,基于比对结果在二级结构中标注26个物种的保守位点.
选取蝽科26个物种的22个tRNA和2个rRNA序列串联数据集,以盾蝽科2个物种作为外群构建系统发育树.使用PartitionFinder v.2.1.1[18]计算每个数据集的替代模型后,在MrBayes v.3.2.6[19]中,使用贝叶斯推断方法以每个数据集的最优替代模型进行系统发育分析.其中,4个独立的马尔可夫链(1条冷链,3条热链)运行10 000 000代,当分裂频率的平均标准偏差值低于0.01时停止,然后“burnin”25%的样本.
2 结果与分析
2.1 tRNA序列特征及遗传距离
蝽科26个物种的22个线粒体tRNA长度为60~80 bp,将比对后的22个同源tRNA串联,检测到866个保守位点、738个变异位点、504个简约信息位点和225个单一位点,其中保守位点所占的比例为51.8%.串联后的22个tRNA长度为1 454~1 502 bp,AT含量为74.7%~78.6%,平均含量为77.0%,反映了碱基组成具有明显的AT偏斜.26个物种的tRNA均表现为GC正偏斜,除拟绿蝽(C.ubica)为AT负偏斜外,其余均为AT正偏斜(表2).
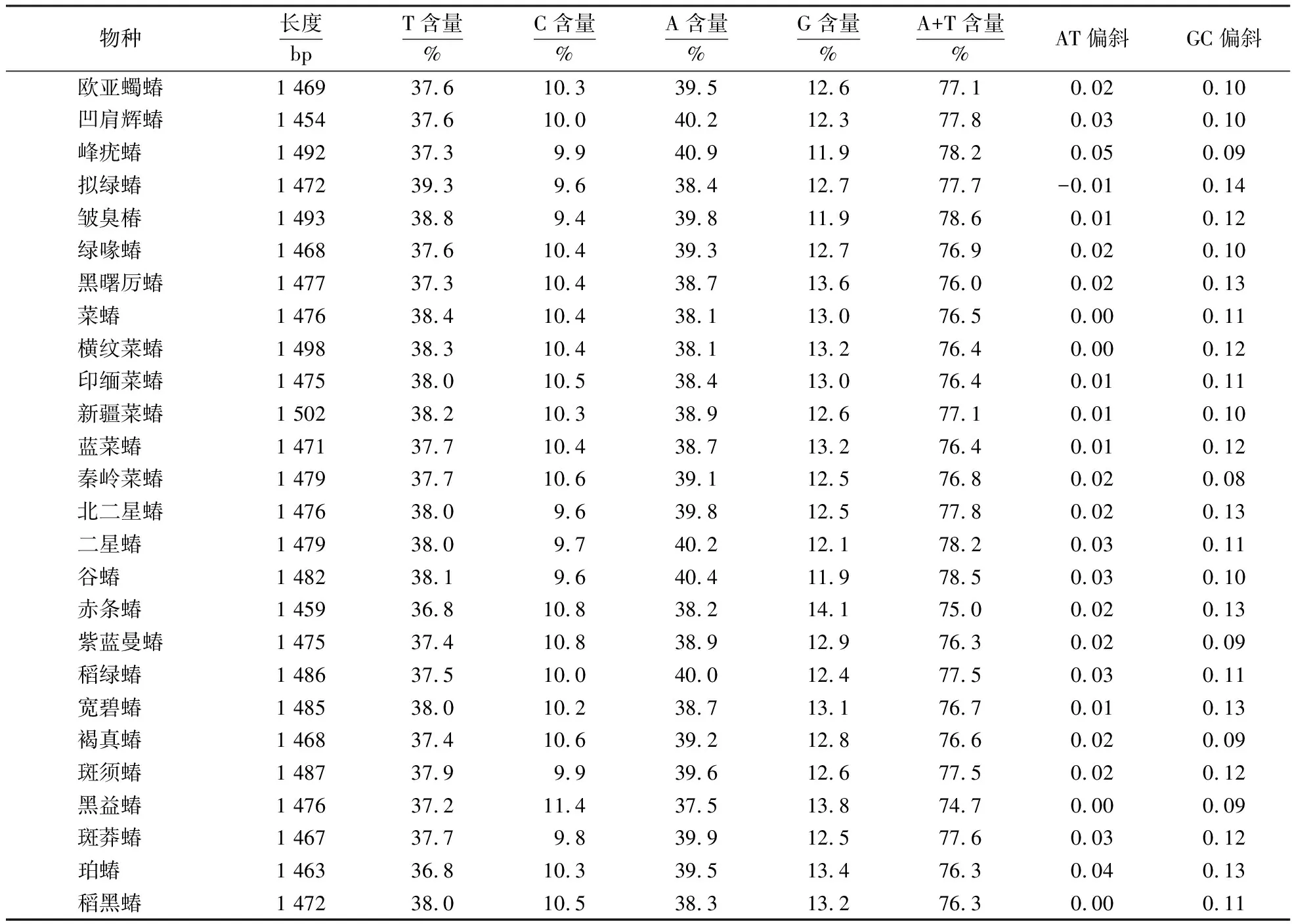
表2 蝽科22个tRNA核苷酸组成分析Table 2 Nucleotide composition analysis of 22 tRNAs in Pentatomidae
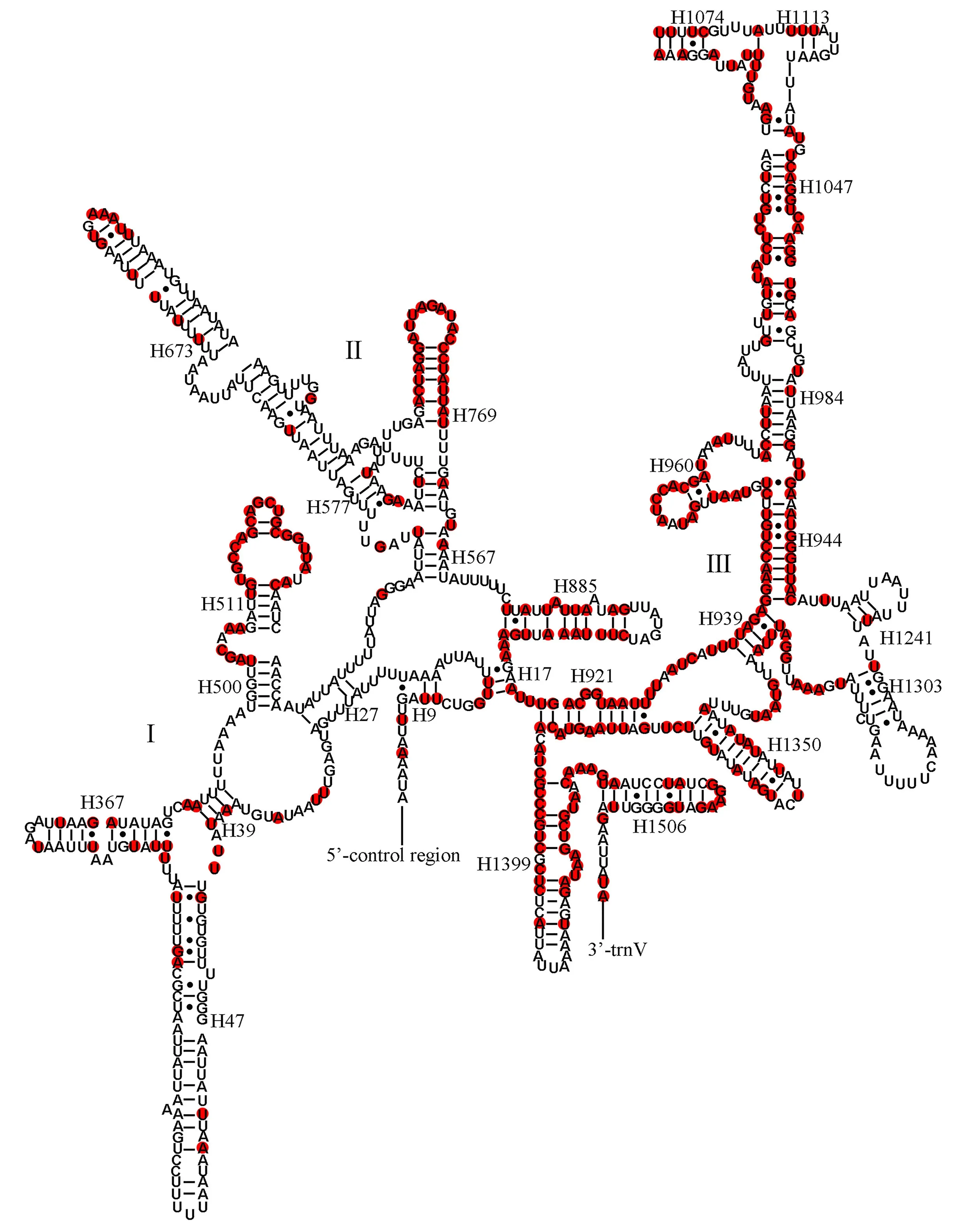
蝽科26个物种的基因排列方式较为保守,均和假定的祖先昆虫亚库巴果蝇(Drosophilayakuba)线粒体基因组的基因顺序一致,未发生重排[20].蝽科昆虫线粒体tRNA序列的保守性也反映在二级结构上,除tRNA-S1由于二氢尿嘧啶臂(dihydrouracil stem,DHU)茎缺失无法形成典型的茎环结构外,所有的tRNA都可折叠为典型的三叶草结构.22个tRNA二级结构均出现非经典的G-U配对,如欧亚蠋蝽(Armacustos)的8个tRNA(tRNA-C、tRNA-D、tRNA-G、tRNA-H、tRNA-I、tRNA-L1、tRNA-P、tRNA-V),除此之外还出现U-U和A-C配对,其中G-U配对对于维系tRNA二级结构的稳定性起着非常重要的作用(图1).
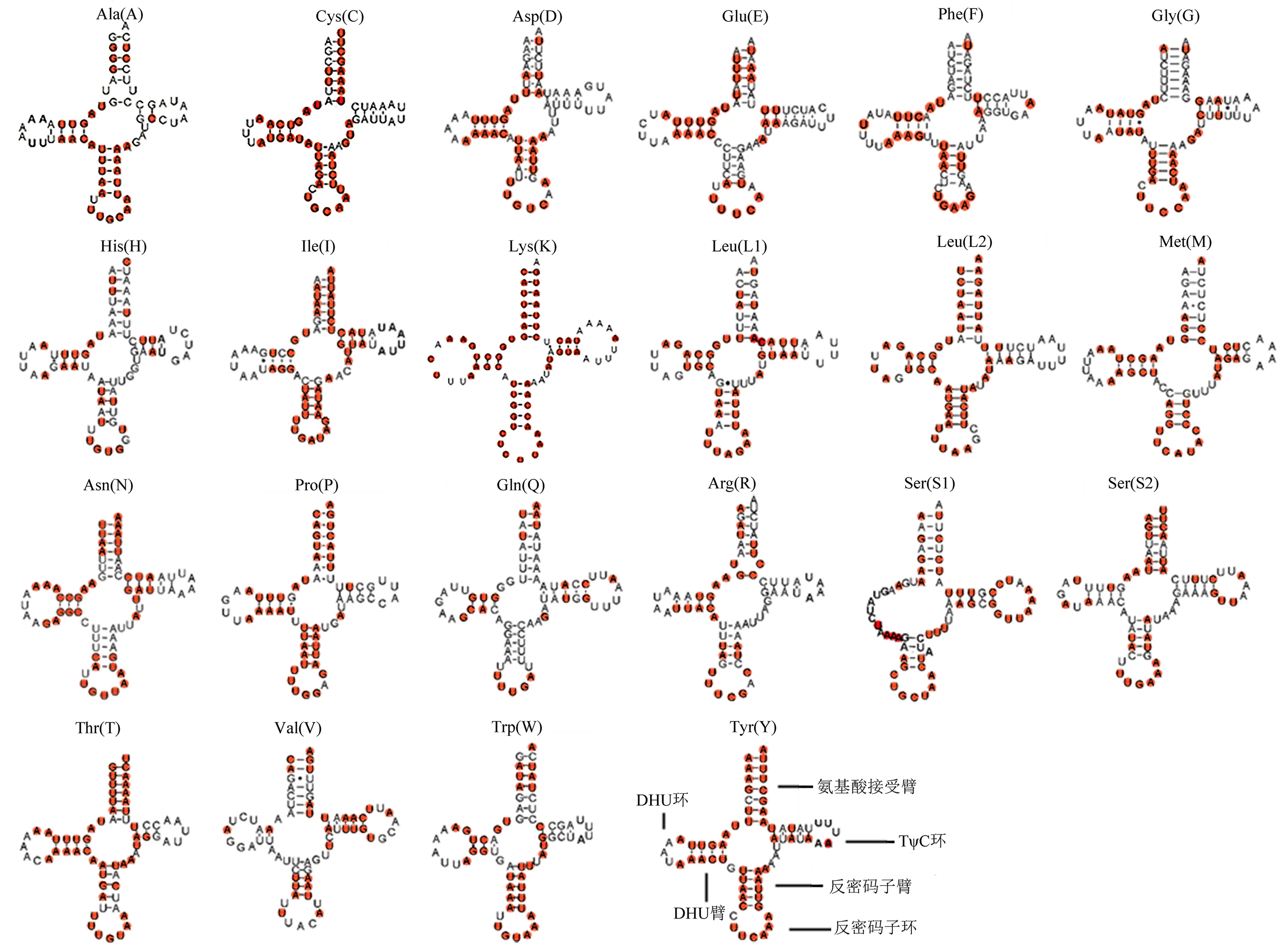
红色圆圈代表在蝽科26个物种中碱基100%保守.图1 蝽科22个tRNA的二级结构Fig.1 Secondary structure of the 22 tRNAs identified in the mitochondrial genome of Pentatomidae
蝽科26个物种的22个tRNA的一级序列和同源tRNA的二级结构均展现了不同的保守性.在22个tRNA中,tRNA-K保守性最高,而tRNA-Q的保守性最低.在同源tRNA的二级结构中,茎区相对于环区来说表现出较高的保守性,而环中除了反密码子环相对保守外,其余环的碱基变异程度都较高.tRNA茎中,DHU表现出极高的保守性,长度为2~4 bp,26个物种的22个tRNA中超过1个碱基不保守的只有tRNA-S2;反密码子臂表现出高的保守性,25个物种的tRNA-S1的反密码子臂的长度由典型的5 bp变为9 bp,而谷蝽(Gonopsisaffinis)变为4 bp,26个物种的tRNA-M反密码子臂长度为4 bp,剩余的26个物种的20个tRNA反密码子臂长度均为5 bp,全部碱基保守的有tRNA-A、tRNA-C、tRNA-K、tRNA-L2、tRNA-P、tRNA-W、tRNA-Y.氨基酸接受臂保守性较低,长度均为7 bp,全部碱基保守的有tRNA-C、tRNA-I、tRNA-K、tRNA-Y.假尿嘧啶臂(TψC臂)保守性极低,长度变异较大,没有全部碱基保守的tRNA.tRNA的4个环中只有反密码子环保守性高,长度均为7 bp,超过2个碱基不保守的只有tRNA-V.
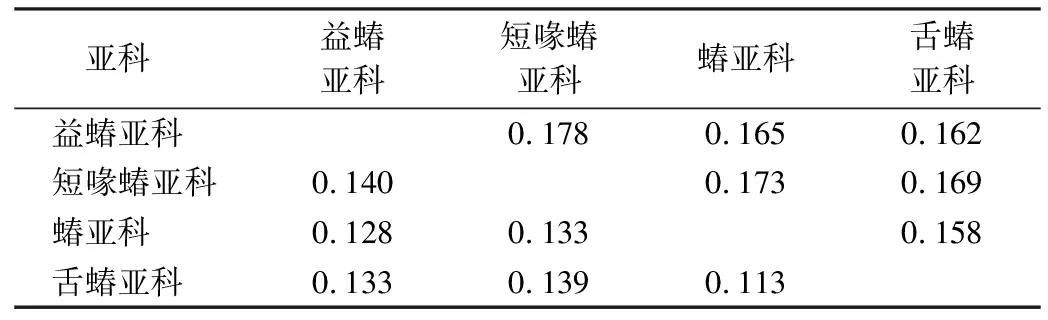
表3 蝽科4亚科间的22个tRNA(下三角)和2个rRNA基因(上三角)K-2-P遗传距离Table 3 K-2-P parameter distances of 22 tRNAs (lower triangle) and two rRNA genes (upper triangle) among four subfamilies of Pentatomidae
基于22个tRNA串联数据集计算蝽科4亚科之间和蝽亚科11个属之间的遗传距离,结果表明4个亚科之间的遗传距离为0.113~0.140(表3),舌蝽亚科和蝽亚科距离最小,益蝽亚科与短喙蝽亚科距离最大.蝽亚科11个属之间的遗传距离为0.082~0.160(表4),二星蝽属和辉蝽属遗传距离最小,绿蝽属和斑须蝽属遗传距离最大.
2.2 rRNA序列特征及遗传距离
蝽科26个物种的12S rRNA序列长度为795~829 bp,其中基因序列长度最长的是褐真蝽(Pentatomasemiannulata),最短的是紫蓝曼蝽(Menidaviolacea)和凹肩辉蝽(Carbulasinica).12S rRNA序列中,检测到370个保守位点、528个可变位点、375个简约信息位点和143个单一位点.16S rRNA序列长度为1 254~1 364 bp,其中基因序列长度最长的是斑须蝽(Dolycorisbaccarum),最短的是绿喙蝽(Dinorhynchusdybowskyi).16S rRNA序列中,检测到610个保守位点、803个可变位点、572个简约信息位点和202个单一位点.2个rRNA的串联长度为2 050~2 174 bp,AT含量为75.7%~80.0%,平均含量为78.3%,说明碱基组成具有明显的AT偏斜,26个物种碱基A和T含量均高于G和C,所有物种T含量均高于A,G含量均高于C,表现出AT负偏斜和GC正偏斜(表5).

表4 蝽亚科11个属间的22个tRNA(下三角)和2个rRNA基因(上三角)K-2-P遗传距离Table 4 K-2-P parameter distances of 22 tRNAs (lower triangle) and two rRNA genes (upper triangle) among 11 genera of Pentatominae
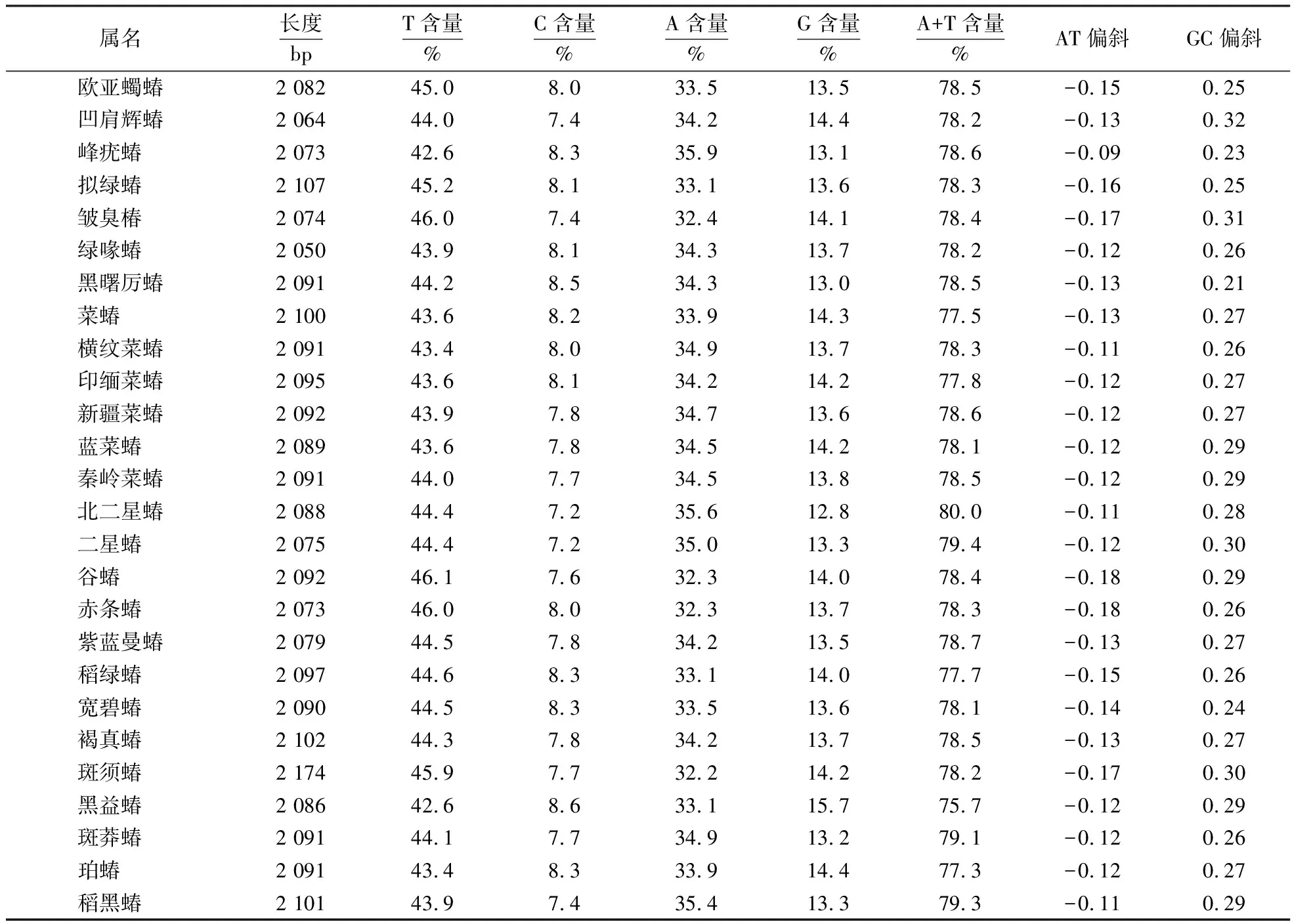
表5 蝽科2个rRNA核苷酸组成分析Table 5 Nucleotide composition analysis of two rRNAs in Pentatomidae
选取欧亚蠋蝽绘制其12S rRNA和16S rRNA的二级结构,并对其保守位点进行了标注.12S rRNA二级结构包括3个结构域和26个茎环结构(图2),蝽科H511、H769、H921、H1399序列和二级结构较为保守,相比结构域Ⅰ和Ⅱ,结构域Ⅲ更保守.16S rRNA二级结构包含6个结构域(节肢动物普遍缺少结构域Ⅲ)和44个茎环结构(图3),蝽科H563、H1775、H1906、H1925、H2507、H2547的序列和二级结构较为保守,16S rRNA的结构域Ⅳ和Ⅴ比结构域Ⅰ、Ⅱ、Ⅵ更保守.rRNA的同源性更多的体现在二级结构保守性上,而不是具有某段保守的序列.
红色圆圈代表在蝽科26个物种中碱基100%保守.图2 蝽科12S rRNA基因二级结构图Fig.2 Secondary structure of 12S rRNA gene of Pentatomidae

红色圆圈代表在蝽科26个物种中碱基100%保守.图3 蝽科16S rRNA基因二级结构图Fig.3 Secondary structure of 16S rRNA gene of Pentatomidae
分别对蝽科4亚科和蝽亚科11个属的2个rRNA串联数据集计算遗传距离,结果表明,4个亚科之间的遗传距离为0.158~0.178(表4),舌蝽亚科和蝽亚科距离最小,益蝽亚科和短喙蝽亚科距离最大.蝽亚科内11个属之间的遗传距离为0.120~0.209(表5),二星蝽属和辉蝽属遗传距离最小,斑须蝽属和辉蝽属遗传距离最大.
2.3 系统发育
以22个tRNA(图4)串联数据集基于贝叶斯推断法构建的系统发育树拓扑结构与2个rRNA(图5)的基本一致.结果均表明:益蝽亚科各个属聚合在一起形成严格的单系群,但内部属之间的姐妹群关系未得到解析.短喙蝽亚科是严格的单系群,亚科中的谷蝽和皱臭蝽(Dalsirascabrata)形成姐妹群后,又与蝽亚科中莽蝽属聚合在一起;蝽亚科中的各个属内种间的个体聚合在一起,二星蝽属和辉蝽属形成姐妹群,拟绿蝽属与绿蝽属聚合在一起后又与碧蝽属以高置信度形成姐妹群,曼蝽属、真蝽属、斑须蝽属和珀蝽属由于选取物种的数目太少而单独成一支;舌蝽亚科中的赤条蝽(Graphosomarubrolineatum)和稻黑蝽(Scotinopharalurida)并未聚合在一起,而是和蝽亚科中的物种聚集在一起.值得注意的是,在rRNA数据集构建的系统发育树中,益蝽亚科和蝽亚科中的曼蝽属形成姐妹群.
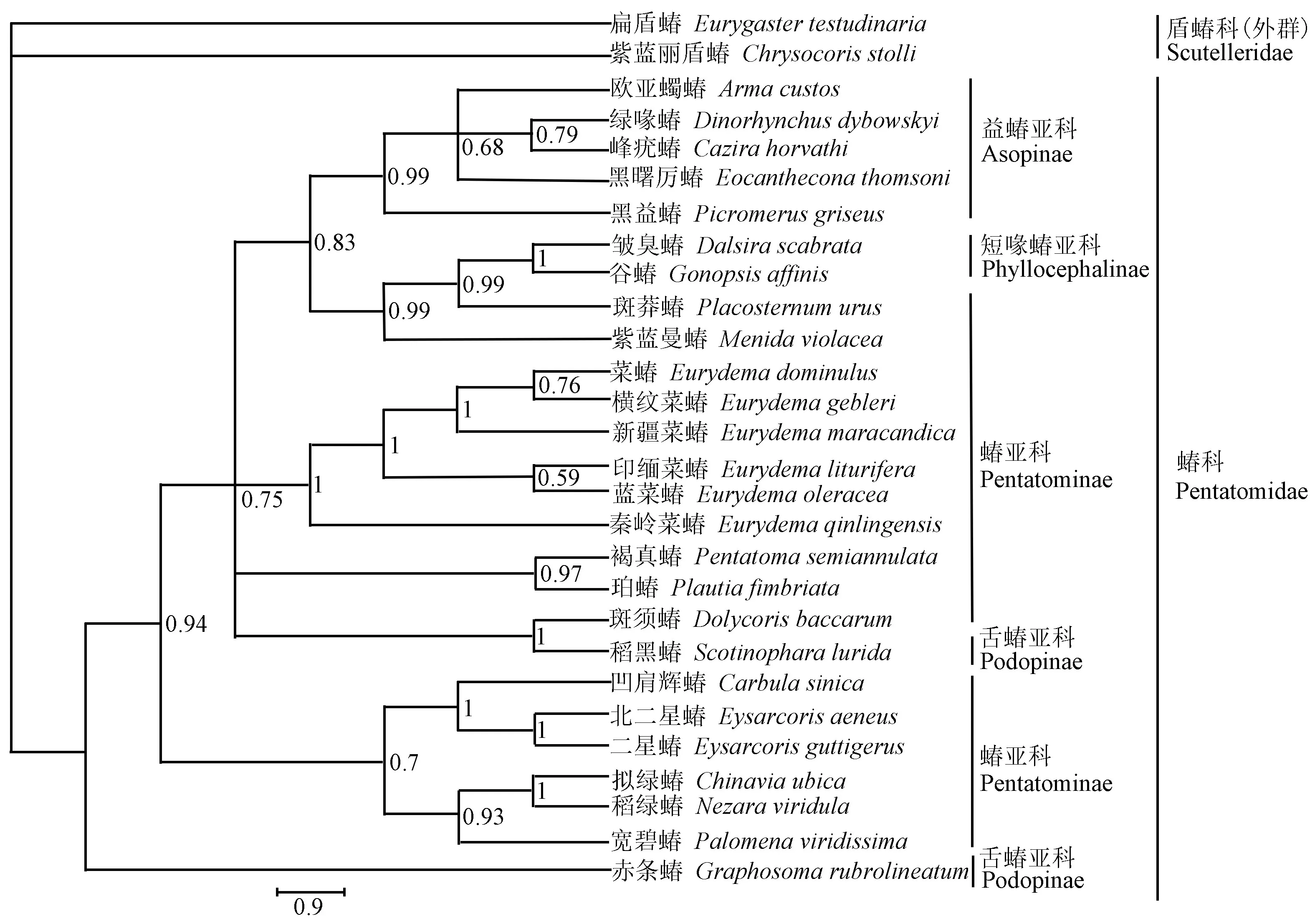
图4 基于22个tRNA 基因构建的26个蝽科物种的BI系统发育树Fig.4 Phylogenetic tree of 26 species inferred from 22 tRNA genes constructed using BI analysis
3 讨论与结论
在蝽科昆虫系统发育研究中,目前应用最为广泛的是PCG基因的核苷酸序列及氨基酸序列,而rRNA和tRNA大多是与PCG基因组合用于系统发育关系的分析.近年来,12S rRNA和16S rRNA被作为分子标记用于系统发育关系的推断,tRNA也逐渐被重视且用于系统发育树的构建[10-13].
线粒体基因的核苷酸组成和取代偏差会影响系统发育组学分析的最终结果,所以,碱基组成的偏向性对研究线粒体基因组复制转录的机理和系统发育关系具有重要意义[13].碱基组成的偏好性一般是由于4种碱基不对称的突变和选择压力,主要来自基因的复制和转录.有研究表明,CG含量的变异导致了基因间不同的进化模式,GC之间的相对含量与复制起点的方向相关,而AT之间的相对含量因复制起点方向、基因方向以及密码子位置的变化而改变[21-22].本研究对蝽科26个物种的tRNA的碱基组成分析表明,tRNA在碱基组成上具有明显的AT偏斜.26个物种tRNA均表现为GC正偏斜,除了拟绿蝽表现为AT负偏斜外,其余均为AT正偏斜.对rRNA的碱基组成分析发现rRNA碱基组成同样具有明显的AT偏斜,平均含量为78.3%,略高于tRNA的平均含量,且26个物种rRNA均表现为AT负偏斜和GC正偏斜,这与大多数昆虫线粒体基因组呈现的AT正偏斜和GC负偏斜不一致[23].

图5 基于2个rRNA 基因构建的26个蝽科物种的BI系统发育树Fig.5 Phylogenetic tree of 26 species inferred from two tRNA genes constructed using BI analysis
tRNA和rRNA的遗传距离结果均表明,蝽科4亚科间舌蝽亚科和蝽亚科亲缘关系最近,益蝽亚科和短喙蝽亚科亲缘关系最远,这与徐志强[24]基于28个新征对蝽总科进行支序分析的结果一致,同时,蝽亚科11个属间二星蝽属和辉蝽属亲缘关系最近,这与形态上两属为近缘属的研究结果一致,此结果也支持2个属归于二星蝽族的观点[25].之前的研究表明,蝽次目73个物种cox1基因的属间遗传距离为0.118~0.244[26],本研究基于rRNA对蝽亚科属间遗传距离的计算结果(0.120~0.209)与之接近,而基于tRNA的结果偏低(0.082~0.160),这表明在选择进化中tRNA的变异较慢,研究结果证明,tRNA和rRNA均可以作为解析蝽科亚科间和属间亲缘关系的分子标记.
本研究通过对tRNA和rRNA序列以及二级结构比较分析得出,不同物种的线粒体同源基因、不同线粒体RNA基因及同一线粒体RNA基因不同区域内的核苷酸变异均存在显著多态性.(1)不同物种的线粒体同源tRNA和rRNA不仅在核苷酸组成上存在差异,在二级结构上茎区和环区的长度也存在差异,如横纹菜蝽的tRNA-V的DHU臂缺失,而欧亚蠋蝽的为2 bp.(2)相同物种中,tRNA序列比rRNA序列更加保守,tRNA序列的保守率为51.8%,rRNA序列的保守率为40%.在22个tRNA中,tRNA-K的保守性最高,保守位点占核苷酸总数的75%,而tRNA-Q的保守性最低,保守位点占核苷酸总数的35%.在2个rRNA中,16S rRNA序列比12S rRNA序列保守,16S rRNA序列的保守率为40.9%,12S rRNA序列的保守率为39.3%,表明tRNA基因适用于高阶元的分类研究,而rRNA基因可以用于解析近缘属的系统发育关系.(3)同一tRNA中,茎区由于在进化选择中需要维持其二级结构而比环区保守性更高,且环区中的反密码子环由于要行使翻译功能较为保守.同一12S rRNA结构域Ⅲ比结构域Ⅰ和Ⅱ更保守,16S rRNA的结构域Ⅳ和Ⅴ比结构域Ⅰ、Ⅱ、Ⅵ更保守.同源RNA基因不同的核苷酸组成和相同或近似的二级结构,如16S rRNA的H1196在26个物种中没有相似的序列,但却有着相似的二级结构,这与陈国忠等[27]提出的RNA结构构成上的生物学意义要大于它的序列组成的结论一致.
基于tRNA和rRNA串联数据集构建的BI系统发育树结果基本一致,均表明短喙蝽亚科、益蝽亚科为单系群,这与Xu et al[28]的研究结果一致,证明了tRNA和rRNA在蝽科系统发育研究中的有效性.蝽亚科的单系性在许多研究中都受到了广泛的质疑[29-30],本研究的2个数据集的系统发育结果也表明,蝽亚科不是单系群.基于2个数据集的系统发育树结果均表明,舌蝽亚科不是单系群,这与基于核基因和线粒体基因的系统发育结果一致[29].其中条蝽属的分类地位极其模糊:Li et al[25]通过分子数据证明条蝽属隶属于蝽亚科;而Rider et al[31]根据形态特征把条蝽属划归为舌蝽亚科;本研究基于tRNA构建的系统发育树中,条蝽属是蝽科最早分化出来的单独一支,而基于rRNA构建的系统发育树中,条蝽属混合在蝽亚科中,这与Wang et al[32]的研究结果一致,表明舌蝽亚科可能不是有效的分类单元,后续仍需要对更多舌蝽亚科的物种序列进行分析.基于2个数据集构建的系统发育树虽然都无法准确解析蝽科4亚科间的姐妹群关系,但是,系统发育结果和部分基于13个PCGs的系统发育树结果一致,这可能是因为蝽亚科内部的系统发育关系较为混乱,也与数据库有限的物种有关[29].
总之,本研究通过对蝽科26个物种的tRNA和rRNA的比较分析发现,这两类基因在物种的亲缘关系及系统进化树分析上具有应用价值,在对数据进行分析时不应该只考虑蛋白编码基因,应充分利用各种数据为昆虫的系统学研究提供更多的有效证据.

