光谱法结合凝胶电泳和荧光显微镜研究萘与DNA的相互作用及其应用
马 纪, 黄国霞, 姚运乾, 李军生, 阎柳娟
(1.广西糖资源绿色加工重点实验室/生物与化学工程学院,广西科技大学,广西柳州 545006; 2.广西柳州螺蛳粉工程技术研究中心,广西柳州 545006)
工业化进程加剧了自然环境的污染。多环芳烃类(Polycyclic Aromatic Hydrocarbons,PAHs)化合物是含有两个及以上苯环的碳氢化合物,如萘(NAP)、蒽、菲和芴等。PAHs主要来源于森林和草原火灾、农业生产、工业“三废”排放、发电、汽车尾气、生活垃圾焚烧、香烟烟雾等[1]。NAP是自然界中含量最丰富的多环芳烃,也是工业中最重要的芳烃,在炼焦和石油蒸馏过程中大量产生[2]。NAP具有疏水性、易燃、高挥发性和在水介质中的化学稳定性[3],主要存在于驱虫剂、香烟、除臭剂、树脂、木材防腐剂、润滑剂和燃料中[4]。由于NAP的毒性强和环境持久性,是美国列为重点污染物的16种多环芳烃之一[5]。NAP被世界卫生组织的国际癌症研究机构列为第二类致癌物[6]。研究结果表明,NAP具有强致癌性和诱变性[7],增加人类喉癌和结直肠癌的风险[8],可诱发人类白内障[9],破坏人体红细胞引起溶血性贫血[10]。因此,采取有效的措施控制NAP污染已迫在眉睫。目前,去除NAP常用方法主要有氧化法[11]、电氧化法[12]、吸附法[13]和生物降解[14]等。但部分方法对NAP去除材料成本高,去除剂难以再生和重复利用,去除效率低,且造成的二次污染严重。因此,开发具有快速、高效、经济、易于分离回收和可重复利用的去除剂具有重要的意义。
实验表明PAHs可以插入双螺旋DNA的层状结构中,导致DNA构象改变,从而影响基因的复制和表达,显示出细胞毒性或基因毒性[15,16]。因此,根据PAHs嵌入DNA的特性,利用磁珠固定DNA选择性捕获PAHs,从而间接清除PAHs。本文采用紫外光谱、熔融法、荧光光谱、共振散射光谱、圆二色谱、琼脂糖凝胶电泳和荧光显微镜等技术,研究了NAP与DNA的相互作用机制。通过结合过程的热力学参数,探讨了NAP与DNA结合的主要作用力。以磁珠固定DNA为吸附剂去除溶液中的NAP,并测定其去除率。
1 实验部分
1.1 仪器与试剂
Cary60紫外-可见分光光度计(安捷伦科技有限公司,美国);RF-5301PC荧光分光光度计(岛津公司,日本);CS30116圆二色谱仪(Applied photophysics公司,英国);DYY-6D电泳仪、WD-9413A凝胶成像记录仪(北京六一生物科技有限公司);DM4B荧光显微镜(徕卡公司,德国)。
萘(NAP,≥98%,Sigma-Aldrich公司,美国),用无水乙醇配制成浓度为1.0×10-3mol/L储备溶液;磁珠(上海富康生物科技有限公司);鲱鱼精DNA(优级纯,北京Solarbio生物科技有限公司);Tris-HCl缓冲溶液(pH=7.4)。
1.2 实验方法
1.2.1 紫外-可见吸收光谱固定DNA浓度,在一系列5 mL EP管中依次加入0、80、160、240、320、400、480、560、640和720 μL 1.0×10-5mol/L的NAP溶液,再在每个EP管中分别加入1 mL浓度为1.0×10-4mol/L的DNA溶液;固定NAP浓度,在一系列5 mL EP管中依次加入0、5、10、15、20、25、30和35 μL DNA溶液(1.0×10-6mol/L),再向每个EP管中加入4.0×10-6mol/L NAP溶液500 μL。用Tris-HCl缓冲溶液定容至5 mL,25 ℃水浴反应30 min后,扫描紫外光谱。
1.2.2 DNA热变性实验移取1 mL 1.0×10-5mol/LNAP溶液,1 mL 1.0×10-4mol/L的DNA溶液于10 mL比色管中,用Tris-HCl缓冲溶液定容至10 mL。从25 ℃升温至100 ℃,温度梯度为5 ℃,每个梯度反应5 min,测量260 nm波长处的吸光度值。
1.2.3 荧光光谱在26 ℃条件下,于5 mL比色管中加入0.4 mL 1.0×10-6mol/L NAP溶液,用Tris-HCl缓冲溶液定容至5 mL。准确移取3 mL该溶液于比色皿中,采用滴定法,逐次往比色皿中滴加1.0×10-7mol/L DNA溶液10 μL。以260 nm为激发波长,记录荧光发射光谱。改变温度条件,以相同方法分别测量36 ℃和46 ℃时的荧光光谱。
1.2.4 KI猝灭实验在5 mL比色管中,加入0.15 mL 1.0×10-3mol/L NAP溶液,0.30 mL 1.0×10-3mol/L DNA溶液,用Tris-HCl缓冲溶液定容至5 mL。准确移取3 mL该溶液于比色皿中,采用滴定法,逐次往比色皿中滴加1.5×10-2mol/L KI溶液10 μL。激发波长为260 nm,记录荧光发射光谱。以F0/F对KI浓度作图,分析NAP与DNA的相互作用模式。
1.2.5 粘度实验在含有5.0×10-5mol/L DNA的Tris-HCl缓冲溶液中,加入不同浓度的NAP溶液,定容摇匀,将其加入乌氏粘度计中,在恒温水浴槽中25±0.1 ℃反应30 min,使用数字秒表记录不同浓度NAP溶液流过毛细管所用的时间,每个样品测量3次,得到平均流动时间,以(η/η0)1/3对R(R=[NAP]/[DNA])作图,得出NAP对DNA粘度的影响。
1.2.6 圆二色谱(CD)在7支2.5 mL EP管中,依次加入0、10、20、40、50、60和80 μL 1.0×10-4mol/L NAP溶液,再分别向EP管中加入5.0×10-4mol/L DNA溶液50 μL,用Tris-HCl缓冲溶液定容至2.5 mL,扫描圆二色谱。
1.2.7 共振光散射光谱(RLS)在11支5 mL EP管中,依次加入0、10、20、30、40、50、60、70、80、90和100 μL浓度为1.0×10-6mol/L DNA溶液,再分别向EP管中加入1.0×10-6mol/L NAP溶液200 μL,用Tris-HCl缓冲溶液定容至2.5 mL,扫描共振散射光谱。
1.2.8 琼脂糖凝胶电泳将0.16 g干琼脂糖溶解于20 mL二次蒸馏水中,加热至沸腾后,立即倒入模具中冷却至固体,得到0.8%琼脂糖凝胶。分别将10 μL 5.0×10-5mol/L溴酚蓝、6.0×10-5mol/L DNA、5.0×10-5mol/L NAP、5.0×10-5mol/L EB、NAP-DNA和EB-DNA加入样品槽中。电泳条件为:电压100 V,电流100 mA。当溴酚蓝条带达到琼脂糖凝胶约1/3时,停止电泳。利用凝胶成像记录系统拍摄图像。
1.2.9 NAP去除实验和荧光显微镜在1.5 mL塑料离心管中加入10 μL磁珠,再加入10 μL 5.0×10-5mol/L DNA和20 μL结合缓冲溶液(100 mmol/L Na3PO4,200 mmol/L NaCl,6.0 mmol/L EDTA-2Na),混匀。40 ℃反应1 h,磁选机离心,弃去上清液。将10 μL NAP加入上述溶液中,放入培养箱摇床300 r/min 反应20 min。取上清液,用荧光光谱测定NAP溶液的含量。移取1.0 μL的磁珠溶液,滴在载玻片上,置于荧光显微镜观察磁珠表面的变化。
2 结果与讨论
2.1 紫外吸收光谱分析
通过紫外吸收光谱的变化可以判断药物小分子与DNA的作用模式。由图1A可知,只有DNA存在时,最大吸收波长在260 nm处。随着NAP浓度的增加,最大吸收波长由260 nm向长波方向移动到270 nm处,红移了10 nm,并且吸光度值增大,即产生增色效应。保持NAP浓度不变,与不同浓度的DNA相互作用后的紫外吸收光谱,如图1B所示。只有NAP存在时,最大吸收波长在276 nm处。随着DNA浓度的增加,最大吸收峰由276 nm向短波方向移动至265 nm处,Δλ=11 nm,出现蓝移现象,并且吸光度值减小,出现减色效应。当谱带呈现红移或蓝移、吸光度减小或增大,表明小分子与DNA是通过嵌入模式结合[17]。这是由于DNA的结构发生了改变,即DNA的碱基π轨道与NAP的π轨道相互耦合引起的[18]。因此,初步判断DNA-NAP的作用模式为嵌入结合。
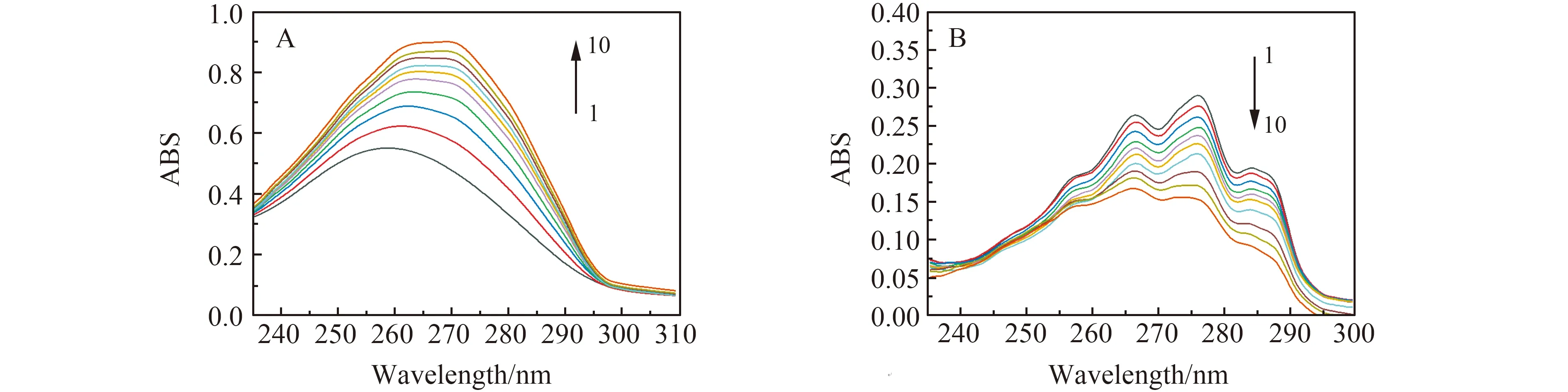
图1 不同浓度NAP与DNA作用的紫外吸收光谱图(A);不同浓度DNA与NAP作用的紫外吸收光谱图(B)Fig.1 UV absorption spectra of DNA interaction with different concentrations of NAP(A);UV absorption spectra of NAP interaction with different concentrations of DNA(B)(A) cDNA=1.0×10-4 mol/L,1 - 10:cNAP(×10-5 mol/L)= 0,0.4,0.8,1.2,1.6,2.0,2.4,2.8,3.2,3.6;(B) cNAP =4×10-6 mol/L,1 - 10:cDNA (×10-6 mol/L)= 0,0.2,0.4,0.6,0.8,1.0,1.2,1.4,1.6,1.8.
2.2 DNA热变性实验
双链DNA暴露在高温环境中,其结构会被破坏形成单链DNA,破坏DNA结构变化的温度也被称为热变性温度(Tm)。DNA和DNA-NAP混合溶液的Tm曲线如图2所示。通过Tm曲线的过渡中点确定DNA的Tm[19]。

图2 DNA分别在有和无NAP下的热变性曲线Fig.2 Melting curves of DNA in the absence and presence of NAP with various concentrationscDNA=1.0×10-4 mol/L,cNAP=1.0×10-5 mol/L.
嵌插入DNA碱基对的小分子,与DNA形成稳定的复合物,阻碍DNA双链结构向单链结构转变,要破坏DNA的结构需要更高的温度,这导致DNA的Tm升高[20]。Tm越高,DNA的结构越紧密,而双螺旋的稳定性也越强,反过来Tm越低,DNA双螺旋结构稳定性就越弱[21]。从图2可以看出,DNA的Tm为58.72 ℃,NAP-DNA混合溶液的Tm升高至80.21 ℃,加入NAP后DNA的Tm增加了21.49 ℃,说明NAP与DNA相互作用后,DNA的内部结构发生了改变,相比与NAP结合之前DNA结构变得更加稳定。因此,推断NAP与DNA是通过嵌入结合,这与紫外光谱法分析的结果一致。
2.3 荧光光谱分析
2.3.1 荧光猝灭机理通过测定小分子自身的荧光强度变化来研究小分子的结构变化,当结合后有复合物产生时,在测定中可以观察到猝灭现象。猝灭现象分为静态、动态猝灭以及非辐射能量转移,根据Stern-Volmer方程[22]表示:
(1)
其中,F0、F分别为NAP与DNA作用前后的荧光强度,τ0表示只有NAP时的荧光寿命为1.0×10-8s,[Q]为猝灭剂的浓度。
如图3所示,NAP的荧光峰位于325 nm附近,随着DNA溶液的不断加入,NAP的荧光发射峰呈有规律的趋势下降,且有1~2 nm的轻微红移,这表明NAP溶液分子与DNA之间发生了相互作用。以F0/F对[Q]拟合可得到Stern-Volmer猝灭曲线(图3D),根据直线斜率可得出NAP与DNA作用的猝灭常数KSV、Kq(表1)。KSV值随着温度的升高而降低,为静态猝灭,反之为动态猝灭[23]。从图3D和表1可以看出,随着温度的升高,KSV降低,当温度升高到46 ℃时,KSV降低的幅度减小,说明不能单独根据KSV值判断荧光猝灭的类型。由表1的结果可知,不同温度下反应的猝灭速率常数(Kq)均大于生物最大猝灭常数2.0×1010L/(mol·s-1),表明DNA对NAP的猝灭方式为静态猝灭,DNA与NAP作用后形成了NAP-DNA复合物。因此,DNA与NAP的猝灭为静态猝灭和动态猝灭相结合过程,以静态猝灭为主,动态猝灭为辅。
2.3.2 结合常数静态猝灭过程可用结合常数表示结合亲和力。根据Lineweaver-Burk双倒数曲线方程表示[24]:
(2)
根据公式(2),以log[(F0-F)/F]对log[Q]作双倒数线性图,根据直线的截距和斜率可求得NAP与DNA的结合常数Kb和结合位点数n,如表1所示。在26 ℃时,NAP与DNA的结合常数最大,达到2.9×106L/mol,结合位点数为1.30;在36 ℃时,NAP与DNA的结合常数最小,为9.3×105L/mol。一般小分子与DNA的结合常数在1.0×105~1.0×1011L/mol范围内属于嵌入结合,结合常数大于1.0×1011L/mol为沟结合。因此,NAP与DNA相互作用方式为嵌入结合。

表1 在不同温度下NAP与DNA相互作用的结合常数、结合位点及双分子猝灭速率常数Table 1 Binding constant,number of binding site and bimolecular quenching rate constant of interaction between NAP and DNA at different concentrations
2.3.3 热力学参数结合反应的焓变(ΔH)、熵变(ΔS)和自由能变(ΔG)等热力学参数是决定结合作用力的主要依据。根据Van’t-Hoff方程和Gibbs-Helmholtz方程[25],从logKa与1/T图中获得的截距和斜率计算得到ΔH和ΔS的值分别是-91.20 kJ/mol和-128.46 J/(mol·K)。进一步计算得到ΔG的值为-52.92 kJ/mol。ΔH和ΔS的计算值均为负值,说明在NAP嵌入DNA过程中,氢键和范德华力是主要的结合力。ΔG<0表明NAP-DNA复合物的嵌入结合过程是自发的。
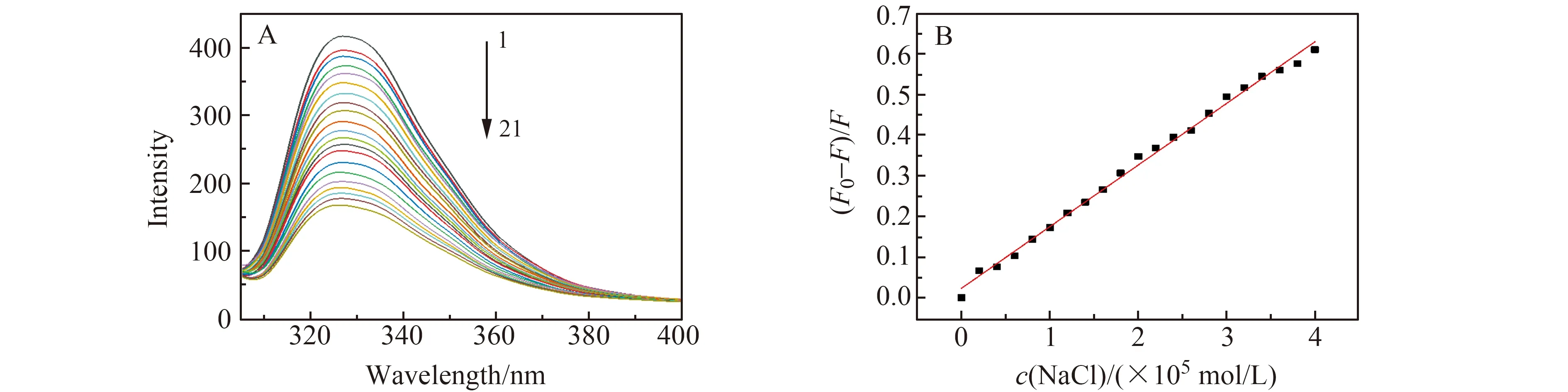
图4 NaCl对NAP荧光光谱的影响(A);不同浓度NaCl的猝灭率(B)Fig.4 Effects of NaCl on fluorescence spectra of NAP (A);Quenching rates under NaCl with different concentrations(B)cDNA=2.0×10-7 mol/L,cNAP=2.0×10-6 mol/L,1 - 12:cNaCl(×10-5)=0,0.2,0.4,0.6,0.8,1.0,1.2,1.4,1.6,1.8,2.0,2.2,2.4,2.6,2.8,3.0,3.2,3.4,3.6,3.8,4.0 mol/L.
2.4 KI猝灭实验
为了解NAP与DNA的结合方式,进行了KI猝灭的实验。I-是一种众所周知的猝灭剂,由于DNA的磷酸基团带负电荷对I-有排斥作用,因此,如果有机小分子插入到双链DNA中,小分子就会受到有效保护而使其荧光强度猝灭程度减小。而在凹槽结合和静电态结合的情况下,小分子暴露于溶剂中,更容易被I-攻击而导致其荧光强度显著猝灭。因此,在DNA的存在下,I-对嵌插结合的有机小分子的荧光猝灭程度比没有DNA要小[26]。图5为I-对NAP及NAP-DNA的荧光猝灭情况。随着I-浓度的增大,NAP荧光强度不断减小。在NAP-DNA体系中,I-对NAP的猝灭程度明显降低,说明NAP通过嵌插模式与DNA结合。
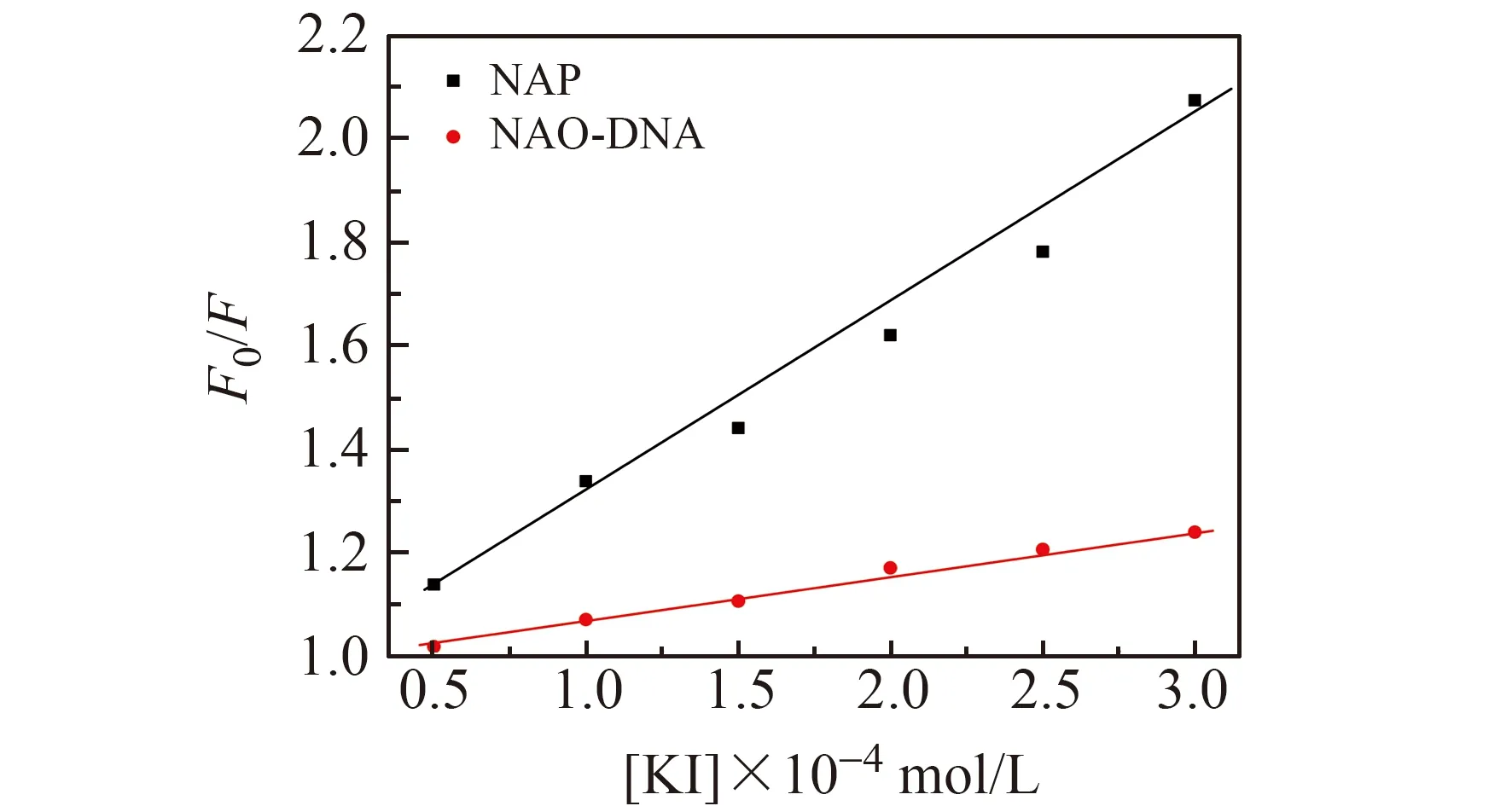
图5 DNA存在和不存在时KI对NAP的荧光猝灭图Fig.5 Fluorescence quenching plots of NAP by KI in the absence and presence of DNAcDNA=6.0×10-5 mol/L,cNAP=3.0×10-5 mol/L.
2.5 粘度实验
粘度测量是研究溶液状态下小分子与双链DNA结合模式的有效方法[27]。当小分子嵌插双链DNA的碱基中时,使DNA的螺旋长度增加,从而导致DNA的相对粘度增大;而当小分子以沟槽或静电作用与DNA结合时,DNA的相对粘度基本不发生明显变化。因此可以通过DNA粘度的变化来判断小分子与DNA结合的模式。如图6所示,DNA的相对粘度随着NAP的浓度的增加而增大,表明NAP与DNA的作用方式为嵌插结合,与上述KI猝灭实验得到的结果一致。

图6 不同浓度NAP对DNA溶液相对粘度的影响Fig.6 Effect of increasing concentrations of NAP on relative viscosity of DNA at room temperaturecDNA=5.0×10-5 mol/L,cNAP=0 - 10.0×10-5 mol/L,([NAP]/[DNA])(×10-5 mol/L)=0,0.2,0.4,0.6,0.8,1.0,1.2,1.4,1.6,1.8,2.0.
2.6 圆二色谱(CD)分析
圆二色谱法是一种有效且高敏感度的技术,是研究大分子与小分子结构成的主要手段之一,可以根据正负峰的变化判断药物分子与DNA的作用模式。当小分子与DNA发生作用后,会使DNA平行G4构象和DNA反平行G4构象中的正峰和负峰发生变化,其中平行G4构象275 nm附近处的正峰与碱基对堆积有关,而245 nm附近处的负峰表征为右旋B-DNA。从图7可知,当溶液中只有DNA存在时,在波长275 nm处存在正峰,在波长245 nm处存在负峰,这说明DNA是右旋B-DNA,此时出现的275 nm附近的正峰与碱基堆积存在一定的关系。DNA与NAP反应后,275 nm处的正峰向上移动,245 nm处的负峰则向下移动,这可能是DNA碱基的电子跃迁偶极矩偶合的变化引起的[28]。此外,还可以从药物与DNA作用后诱导的CD信号强度变化来判断其作用模式。一般诱导的CD信号小于10 L/(mol·cm)的属于嵌入结合[29]。因此,证实DNA与NAP通过嵌入方式发生作用。

图7 不同浓度的萘与DNA作用的CD光谱Fig.7 CD spectra of DNA interacting with NAP with different concentration1:cNAP=2.0×10-5 mol/L;2 - 8:cNAP(×10-6)=0,0.4,0.8,1.6,2.0,2.4,3.2 mol/L;cDNA=1.0×10-5 mol/L.
2.7 共振光散射光谱(RLS)分析
共振光散射光谱法是通过测定不同浓度的DNA与平面分子的共振散射信号,当散射信号随着DNA浓度上升而不变或者下降时,表示DNA和平面分子的相互作用达到饱和,计算其饱和值(BSV)[30],通过结合饱和值判断分子与DNA结合能力强弱,由此可以判断分子对DNA毒性的强弱。从图8可知,没有加入DNA的情况下,浓度为1.0×10-6mol/L的NAP在0~700 nm范围内有两个共振散射峰,分别在297 nm和540 nm处,在297 nm处RLS信号最强。随着DNA的浓度不断增大,其共振散射光谱强度逐渐增强,最大共振散射峰位置也发生了红移。当加入的DNA浓度达到2.5×10-6mol/L时,其共振散射光谱强度不再增强,说明NAP与DNA结合达到饱和状态。此时其最强RLS信号在545 nm附近。计算出NAP-DNA结合饱和值为2.5,说明NAP与DNA相互作用的结合力比较强。

图8 不同浓度DNA与NAP作用的共振光散射(RLS)光谱图Fig.8 Resonance scattering spectra of NAP interacting with different concentrations of DNAcNAP=1×10-61×10-6 mol/L;1 - 6:cDNA(×10-6)=0,0.5,1.0,1.5,2.0,2.5×10-6 mol/L.
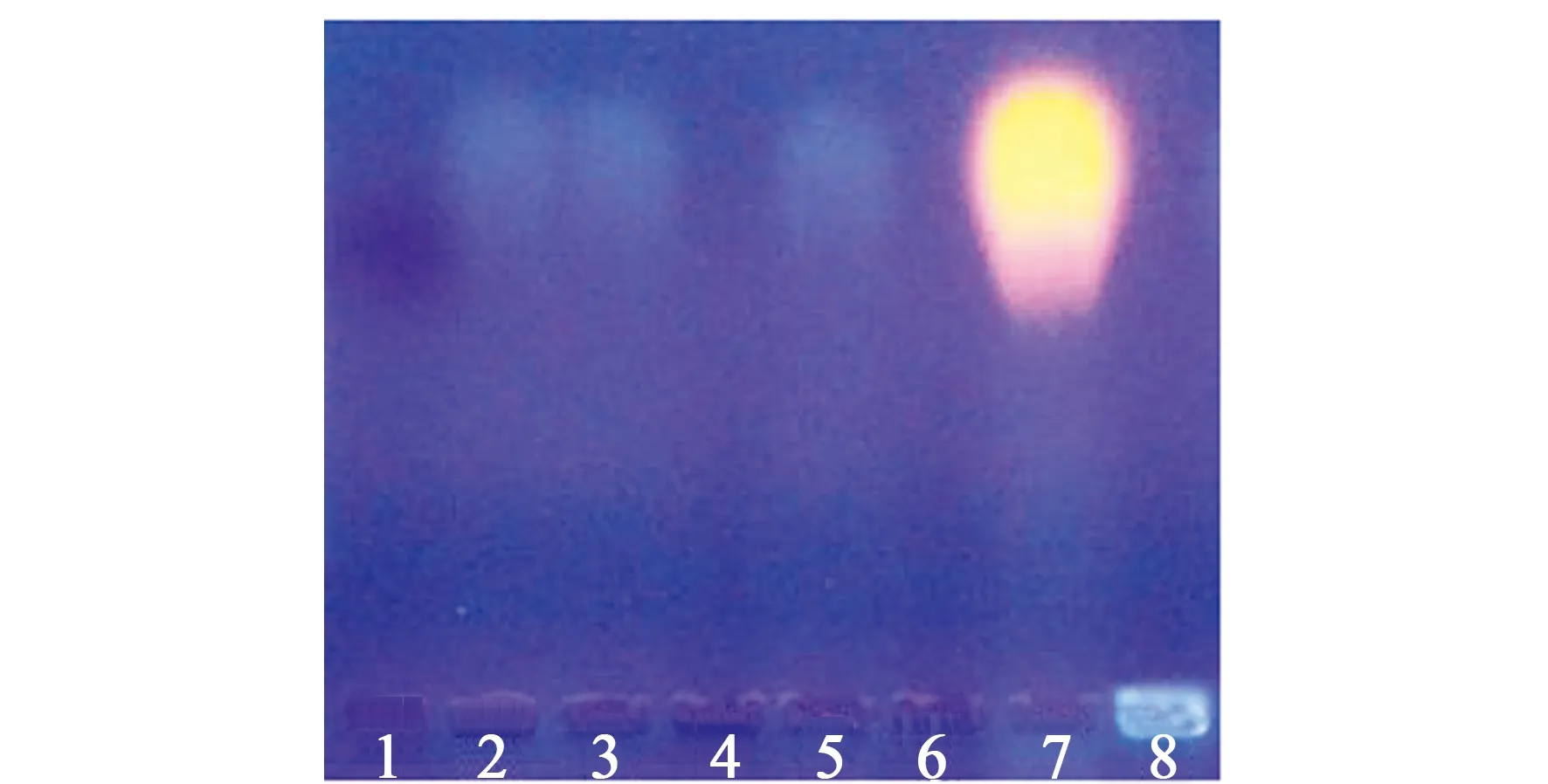
图9 琼脂糖凝胶电泳图Fig.9 Results of agarose gel electrophoresisLane 1:Bromophenol blue,lane 2、3、5:NAP-DNA,4:DNA,6:EB,7:EB-DNA,8:NAP.
2.8 琼脂糖凝胶电泳分析
由于DNA分子中的磷酸基团带负电荷,在电场中DNA分子会向正极方向移动。溴化乙锭(EB)是一种典型的DNA嵌入剂,很容易嵌入到DNA的碱基之间,在电泳过程中,EB-DNA复合物中的EB则会随着DNA向正极移动,在紫外光下呈现橙色的荧光。同样,NAP-DNA体系电泳结束后,在紫外光下泳道2、3、5中呈现蓝色的荧光,而单独NAP在电场中不会发生移动,说明DNA分子与NAP分子一起迁移,因此可以推测NAP分子嵌入到DNA碱基对之间,如果NAP分子在沟区或双螺旋外侧,则会快速向正极或者其它方向迁移。
2.9 NAP去除实验和荧光显微镜分析
磁珠是一种有效的核酸吸附剂,具有操作简单、分离快、纯化容易等优点。本实验中使用的磁珠是Fe3O4外层包裹SiO2的超顺磁纳米材料。如图10所示,在荧光显微镜下EB、DNA和磁珠单独存在时均不显示荧光。磁珠与EB混合,在荧光显微镜下未观察到荧光(图10A),而磁珠与DNA和EB反应后显示许多明亮的荧光点(图10B)。EB作为一种典型的DNA插层物,不能单独发射荧光,但与DNA相互作用后可以产生强荧光。因此,可以推断EB成功插入游离DNA的碱基对中,并可用磁珠将其分离。与EB不同,NAP自身有荧光,但NAP与磁珠反应洗脱后磁珠表面未出现荧光(图10C),说明磁珠不能吸附NAP。在NAP与DNA-磁珠反应洗脱后,磁珠表面呈现大量的荧光点(图10D),说明NAP嵌入DNA的碱基间。以上结果再次直观证实了NAP与DNA以嵌入模式结合,并通过磁珠吸附DNA将NAP间接吸附到磁珠上。

图10 EB与磁珠结合的荧光显微镜图(A);EB与DNA和磁珠结合的荧光显微镜图(B);NAP和磁珠结合洗脱后的荧光显微镜图(C);NAP与DNA和磁珠结合洗脱后的荧光显微镜图(D)Fig.10 Fluorescent image of magnetic beads-EB(A);magnetic beads-DNA-EB after washing(B);magnetic beads-NAP after washing(C) and magnetic beads-DNA-NAP after washing(D)
根据NAP的浓度和荧光强度拟合的线性方程(Ex=265 nm,Em=325 nm),通过荧光光谱检测上清液中NAP的残留量。利用NAP吸附量Q和去除率(%)来测定NAP与磁珠的结合量[31]。本文通过测定NAP的残留量得到NAP的去除效率,DNA对NAP的吸附量为3.2×10-4mol/g(41.01 mg/g),去除率为99.35%。结果表明,磁珠固定化DNA对NAP有较好的吸附能力。基于多环芳烃-DNA嵌入技术,DNA在多环芳烃的去除中具有良好的应用前景。
3 结论
采用紫外光谱、荧光光谱、圆二色谱、共振散射光谱、粘度实验、琼脂糖凝胶电泳和荧光显微镜对NAP与DNA的相互作用进行了系统的研究。紫外光谱显示,NAP与DNA作用后谱带呈现红移增色、蓝移减色效应,说明NAP通过嵌入与DNA结合并形成了稳定的复合物。热变性实验结果显示,NAP的加入导致DNA热熔温度增加了21.49 ℃,说明NAP与DNA嵌插结合使DNA双螺旋结构的稳定性增加。荧光猝灭结果表明,NAP与DNA相互作用中主要以静态猝灭机制为主,动态猝灭为辅。热力学数据表明,NAP与DNA相互作用属于自发过程,二者嵌插结合的主要作用力以氢键和范德华力为主。离子强度实验、KI猝灭实验和粘度实验结果排除了静电作用和沟槽结合模式。圆二色光谱结果进一步证实NAP与DNA嵌插结合方式。通过共振散射光谱实验计算得到NAP与DNA的结合饱和值为2.5,说明NAP与DNA相互作用具有较强的结合力。琼脂糖凝胶电泳和荧光显微镜实验直观地验证了NAP和DNA嵌插结合方式。以上实验结果揭示了DNA能选择性捕获NAP。基于此,利用磁珠固定DNA选择性吸附NAP,饱和吸附量为41.01mg/g,其去除率达到99.35%。该研究结果为探索NAP与DNA作用机理提供了理论依据,也为建立基于PAHs-DNA相互作用的新型PAHs去除方法提供了新途径。因此,这项工作的结果有望提供新颖的应用信息,例如从鲑鱼精中提取廉价DNA,设计磁珠固定DNA去除PAHs的过滤装置,并将其应用于饮用水中去除PAHs。

