云南玉溪绿孔雀遗传多样性分析:与圈养种群的比较
刘思霞 张馨元 晏鸣霄 杜焓瑜 杨金城 李广龙 官文渊 杨守庄 徐艳春,7*
(1.东北林业大学野生动物与自然保护地学院,哈尔滨,150040;2.中国科学院西双版纳热带植物园,勐腊,666303;3.浙江大学生命科学学院,杭州,310058;4.国家林业和草原局林产工业规划设计院,北京,100010;5.衢州越鸟农业发展有限公司,衢州,324000;6.秦皇岛野生动物园,秦皇岛,066100;7.国家林业和草原局野生动物保护与利用工程技术研究中心,哈尔滨,150040)
绿孔雀(Pavomuticus)是一种大型鸡形目(Galliformes)鸟类,曾在中国南方[1-2]和东南亚[3]广泛分布,并在世界各地成为民族文化的重要载体。绿孔雀被分为3个亚种,即原产于马来西亚和爪哇的指名亚种(P.m.muticus),分布于印度东北部至缅甸西北部的印度亚种(P.m.spicifer)和分布于印支地区的云南亚种(P.m.imperator)[4]。在过去的半个多世纪里,由于狩猎[5-6]、栖息地退化和丧失[5,7]以及人类干扰[8-9],所有分布区的绿孔雀种群数量都显著下降,2009年绿孔雀被IUCN濒危物种红色名录列为濒危(EN)级(https://www.iucnredlist.org/species/22679440/131749282)。
历史上,绿孔雀在中国的分布很广,包括湖南、湖北、四川、广西、广东、云南和西藏等地理范围[10],而今仅在云南省西部、中部和南部的22个县有所分布[5]。1993年,实地调查记录的种群总数量在800~1 100只[11],此后下降到大约500只[12],最新调查为235~280只[13]。我国境内的绿孔雀栖息地,除了占种群数量60%以上的双柏和新平外,近60%的分布区域都在过去的30年里相继消失,而双柏和新平2个地区也面临着栖息地破碎化的风险[5,14]。本研究借助云南省玉溪市新平和峨山交界处的羽毛样本,分析该种群的遗传多样性,为深入了解绿孔雀种群状况,制定保护计划提供基本信息。
1 材料与方法
1.1 样品采集
2018—2019年,在云南省玉溪市新平与峨山交界处的野外调查中,从自然栖息地共收集到29份羽毛样品(图1)。每份样品单独包装在一个纸质信封里,标明收集地点和时间。羽毛样本编码为YBD01~YBD29,4 ℃保存,命名为YBD种群。另外,选择9只纯种绿孔雀(编码GP01~GP09)和6只纯种蓝孔雀(编码BP01~BP06)作为参考,以鉴定羽毛所属物种。为了研究YBD种群与中国现有圈养种群之间的遗传关系,从不同的养殖机构共采集了33份绿孔雀样品,其中16只是从中国云南瑞丽捕获的野生绿孔雀的后代,其余个体的地理来源不明。所有样品均为羽毛样品,编码为CAP01~CAP33。
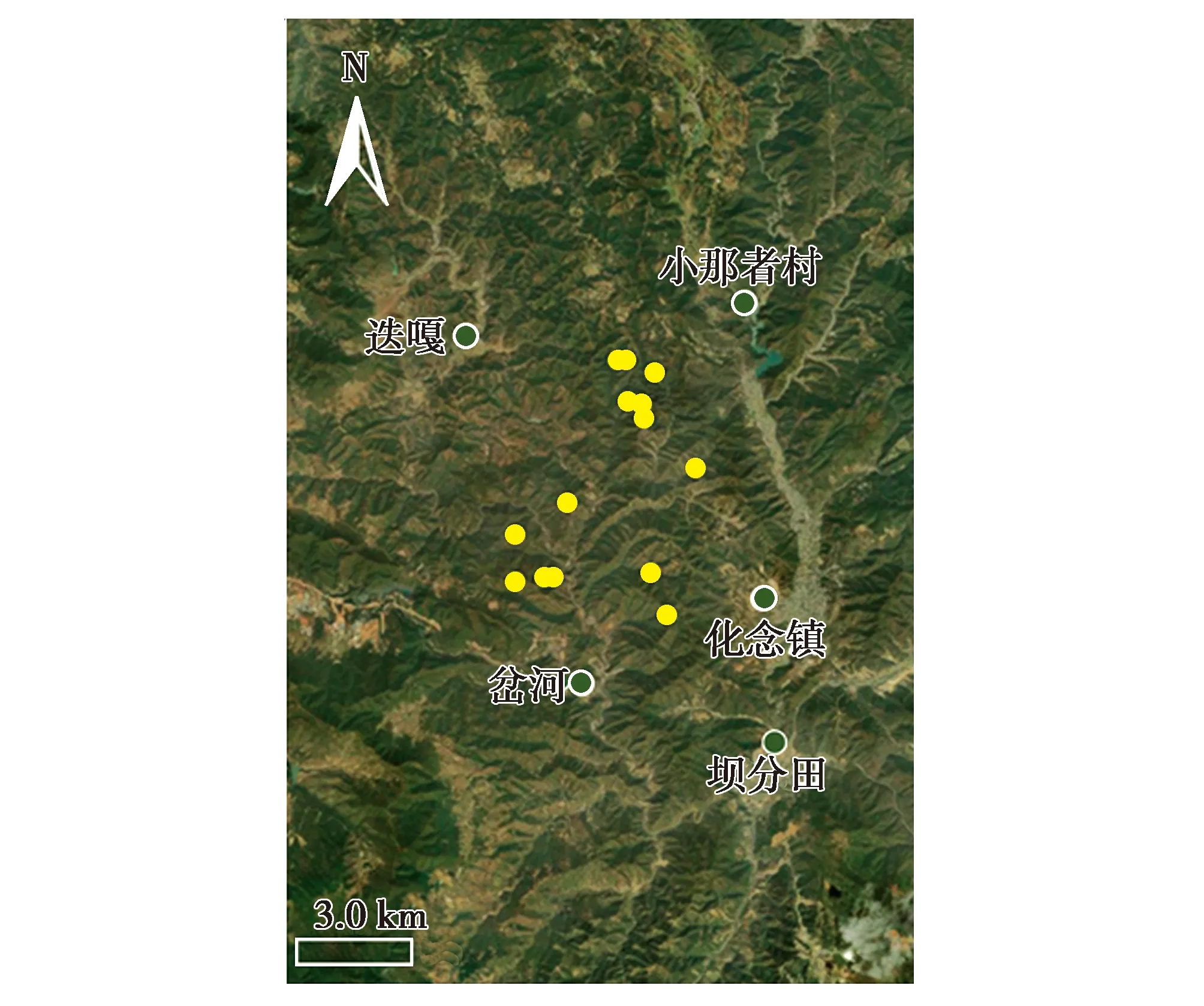
图1 云南省玉溪市新平与峨山交界处孔雀羽毛的收集地点Fig.1 Geographic location of the collected feather samples in the juncture of Xinping and Eshan of Yuxi City,Yunnan Province,China
1.2 DNA提取
从每根羽毛上切下约5 mg的羽枝,放入离心管中。向试管中加入约1 000 μL 2% SDS溶液,把羽毛没入液面以下,在65 ℃的水浴中孵育30 min。将羽毛样品转移到另一个含有75%乙醇的烧杯中漂洗30 min,再转移到含有20 mL ddH2O的50 mL烧杯中,振荡洗涤约30 min。清洗干净的羽毛样品放在滤纸上自然干燥,移入1.5 mL离心管中,剪成小块备用。DNA的提取按照Volo等[15]的方法,消化时间为2 d,异丙醇沉淀冷冻12 h,冷冻离心16 000 r/min 30 min。使用Nanodrop 2000 C(Thermo Scientific,USA)测定DNA的浓度,并稀释至~15 ng/μL用于PCR 扩增。
1.3 Cyt b基因片段的扩增和测序
Cytb基因 PCR 扩增引物参考Verma等[16]使用的鸟类常用扩增引物mcb398(5′-TACCATGAGGACAAATATCATTCTG-3′)和mcb869(5′-CCTCCTAGTTTGTTAGGGATTGATCG-3′)。采用50 μL的PCR反应体系:DNA(~45 ng)3 μL,上下游引物各1 μL(10 pmol/μL),2×EasyTaq®PCR SuperMix(全式金生物技术有限公司)25 μL和ddH2O 20 μL。反应程序:94 ℃预变性5 min;94 ℃变性30 s,52 ℃退火45 s,72 ℃延伸30 s,循环35次;72 ℃终延伸7 min,4 ℃保存。扩增产物在1.5%的琼脂糖凝胶上80 V电泳40 min,用AxyPrepTMDNA Gel Extraction Kit试剂盒纯化回收,采用Sanger法双向测序,用DNASTAR软件包上的SeqMan Pro对序列进行拼接[17]。
1.4 微卫星的分型
参考Du等[18]的方法,选择PC3、PC19、PC25、PC36、PC41、PC46、PC67、PC125、PC142、PC148、PC151、PC201、PC236、PC281[19]、MCW0069和MCW0098[20]16个微卫星位点对野生和圈养种群的样本分型,上游引物的5′端用FAM(蓝色)、JOE(绿色)和TAMRA(黑色)3种荧光素标记。PCR反应体系均为15 μL:DNA(~45 ng)3 μL,上、下游引物(10 pmol/μL)各1 μL,2×EasyTaq®PCR SuperMix(全式金生物技术有限公司)5 μL和ddH2O 5 μL。反应程序:94 ℃预变性5 min;94 ℃变性30 s +Ta℃退火45 s + 72 ℃延伸30 s,循环35次;72 ℃终延伸10 min,4 ℃保存。PCR产物在3100x Genetic Analyzer(Applied Biosystems,USA)上电泳分离,用GeneMarker®收集片段信号[21]。为避免基因分型错误,每个个体进行3次基因分型。
1.5 羽毛样品的母系物种鉴定
使用MEGA 5.05中的Clustal W程序[22],比较野外羽毛样品和已知绿孔雀、蓝孔雀的线粒体Cytb基因序列,确认它们的同源性,用EditSeq[17]将所有序列修剪到421 bp。根据Dinh等[23]的建议,用MEGA 5.05[22]和Kimura 2-parameter(K2P)模型计算每个待测羽毛样本与已知纯种绿孔雀和蓝孔雀样本之间的遗传距离,同时计算已知绿孔雀种内两两个体间的遗传距离(intra-d)与绿孔雀、蓝孔雀种间两两个体间的遗传距离(inter-d),这些遗传距离用于确定每个样本的母体物种起源。使用IBM SPSS Statistics 23(IBM Corp.New York,USA)检验每个羽毛样品与2组已知孔雀个体的遗传距离落入intra-d还是inter-d的范围(95%置信区间),从而确定其所属物种;用MEGA 5.05基于K2P距离构建系统发育树,用iTOL v4.4.2(https://itol.embl.de/)进行可视化。
1.6 个体识别
羽毛样本在野外捡拾,可能存在同一个体多次重复,因此在正式的种群分析之前,需进行个体识别以确定真实样本量(鸟类数量)。首先,用Micro-Checker v2.23[24]检测各个微卫星分型结果的无效等位基因的概率,据此矫正等位基因和基因型频率,用于进一步种群遗传分析。用Gimlet Software v1.3.1[25]计算多位点的累积个体识别概率(cumulative probability of identity,P(ID)),即2个无关个体或同胞之间在多个微卫星位点上基因型完全一致的可能性[26]。为了消除种群结构的影响,计算修正基因型相似概率(P(ID)biased);考虑到样本中存在血缘关系的可能,还计算了同胞或亲子之间基因型相似概率(P(ID)sibs),作为个体识别的最低可信度。最后,用Cervus 3.0确认被鉴定为同一个体的多个样品是否在所有位点的基因型完全相同,或者是符合最低接受标准:仅在1个位点相差1个等位基因[27]。
1.7 遗传多样性及种群遗传结构分析
使用Popgene 1.32计算观察等位基因数(Na)、有效等位基因数(Ne)、观察杂合度(Ho)和期望杂合度(He);用Cervus 3.0计算YBD种群和CAP种群的每个微卫星位点的多态信息含量(PIC);用Popgene 1.32计算2个种群的种群近交系数(fixation indices of subpopulation,FIS)、每个位点的遗传分化指数(FST)、基因流(Nm,Nm=0.25(1-FST)/FST)、Nei氏遗传距离和遗传相似度,并进行哈迪-温伯格平衡(H-W)检验。为了推断YBD和CAP种群之间的遗传分化程度,用Arlequin 3.5[28]计算2个群体之间的遗传分化指数FST,使用STRUCTURE 2.3.1分析种群的遗传结构[29]。最佳K值(理论种群数量)和种群遗传结构图形结果使用在线软件(http://clumpak.tau.ac.il/)生成。

附录1 云南玉溪绿孔雀种群的SSR分型结果
2 结果与分析
2.1 羽毛样品的母系物种鉴定
作为参考种群的已知绿孔雀(GP)和蓝孔雀(BP)的种间遗传距离(inter-d)为3.43%~3.68%,95%的置信区间为[3.41%~3.61%];参考绿孔雀种群种内遗传距离(intra-d)为0~0.72%,95%的置信区间为[0.26%~0.46%]。羽毛样品和已知绿孔雀之间的K2P距离为0.120~0.602,均落入intra-d的95%的置信区间,而与蓝孔雀的K2P距离在3.531%~3.788%,落入inter-d的95%的置信区间,且在intra-d的95%的置信区间之外(表1)。系统发育树显示29个羽毛样品都与绿孔雀聚为一支(图2),Bootstrap支持度为0.493~1.000。结果表明,所有的羽毛样品的母系均为绿孔雀。
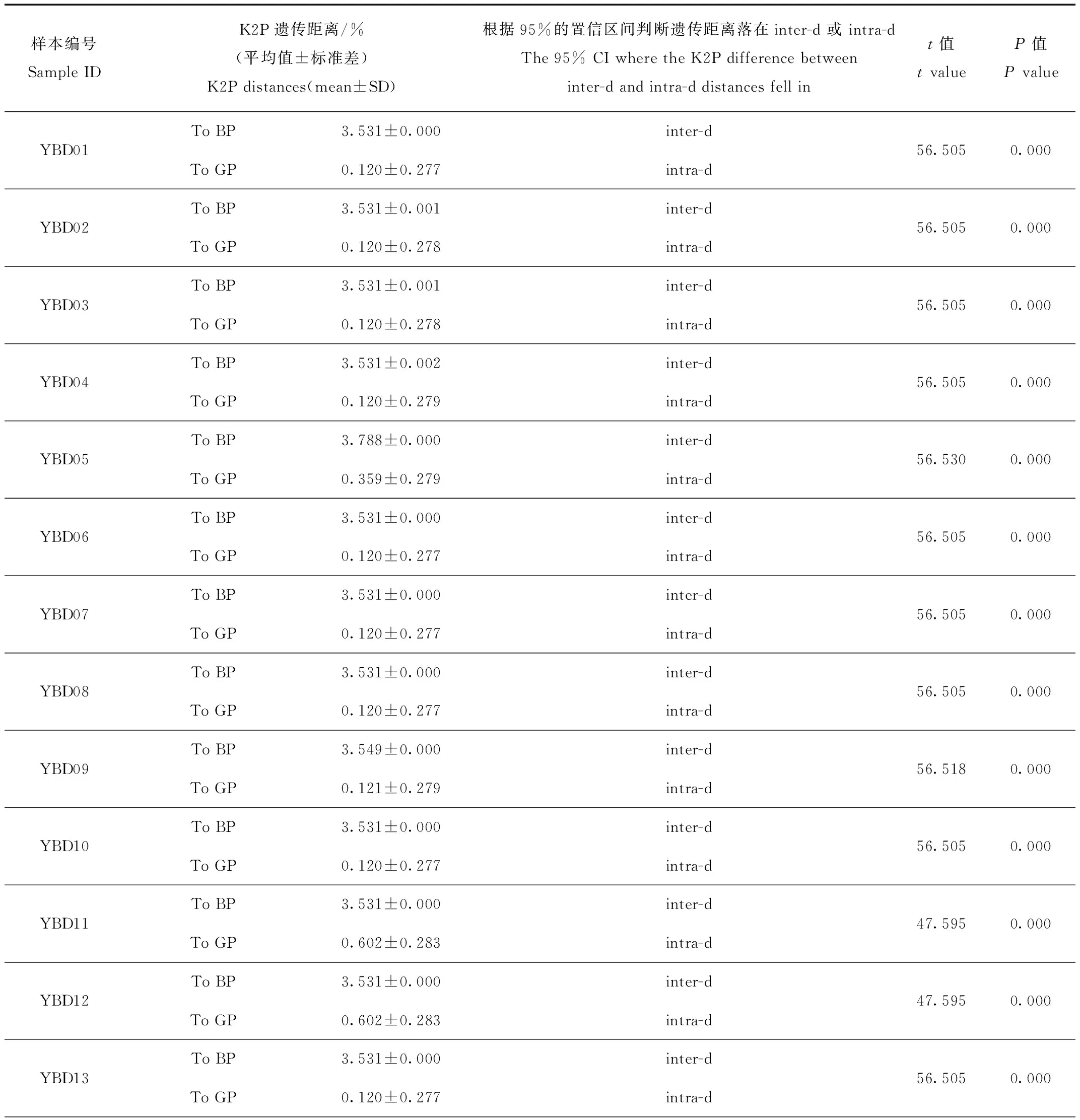
表1 以421 bp的Cyt b基因序列估算的羽毛样品YBD01~29与纯种绿孔雀(GP)和蓝孔雀(BP)之间的K2P遗传距离
续表1

图2 基于Cyt b基因421 bp序列的K2P遗传距离所做的NJ系统发育树显示羽毛样品(橙色)、纯种绿孔雀(绿色)和纯种蓝孔雀(蓝色)的关系Fig.2 The NJ tree of feather samples (orange) and reference samples of the green (green) and blue peafowl (blue) based on K2P genetic distance inferred from 421 bp sequence of Cyt b gene
2.2 羽毛样本的个体识别
Micro-Checker在PC36、PC67、PC125、MCW0069和MCW0098这5个位点检测到无效等位基因。为了保证个体识别结果的可靠性,后续分析中把这5个位点舍去,保留未检测到无效等位基因的其余11个位点,即PC46、PC148、PC151、PC142、PC19、PC236、PC41、PC25、PC281、PC201和PC3。图3显示,累积P(ID)值随着位点数的增加而下降。分子生态学研究中常以P(ID)值在0.001 0~0.000 1为同一性认定标准,即累积P(ID)达到0.001 0或0.000 1以下时,如果这2个样品具有不同的基因型,则可判定为来自不同的个体[30]。本研究数据显示,当微卫星位点数达到8个时,P(ID)sibs的联合P(ID)达到0.000 963,而对于相同的8个位点,P(ID)biased的联合P(ID)达到0.000 000 036 49。当位点更多时,累积P(ID)会进一步下降,增加基因型错配的机会,影响个体识别结果的准确性[30](图3)。同时考虑到绿孔雀在野外的稀有性,数值小于0.001 0的联合P(ID)足以可靠地识别个体。
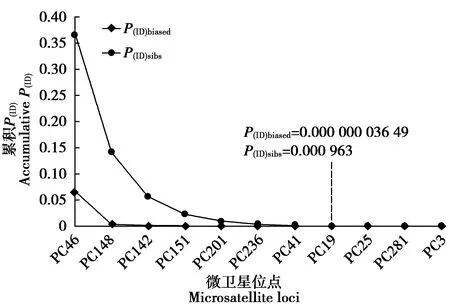
图3 微卫星位点累积P(ID)的变化趋势Fig.3 The declining trend of cumulative probability of identity (P(ID)) with addition of microsatellite loci
用8个微卫星位点通过Cervus软件进行个体识别,结果显示羽毛样本中YBD10和YBD25、YBD11和YBD27、YBD23和YBD24在8个位点上基因型相同,或者仅在1个位点上只有1个等位基因不同,其余样品显示1个以上等位基因的错配(附录1)。最后,确定野外绿孔雀样本为26只。
2.3 YBD和CAP种群的遗传多样性
利用Popgene检测了YBD种群和CAP种群在11个微卫星位点的种群遗传学参数。YBD种群的Na为2~8个,平均为(4.727±2.328)个;CAP种群的Na为2~6个,平均为(4.091±1.300)个;YBD种群和CAP种群之间等位基因频率差异为0~0.846。2个种群之间Na在PC46上差异最大,YBD种群有8个等位基因,而CAP种群只有4个。总的来说,Na在YBD种群中比在CAP种群中更多(表2)。
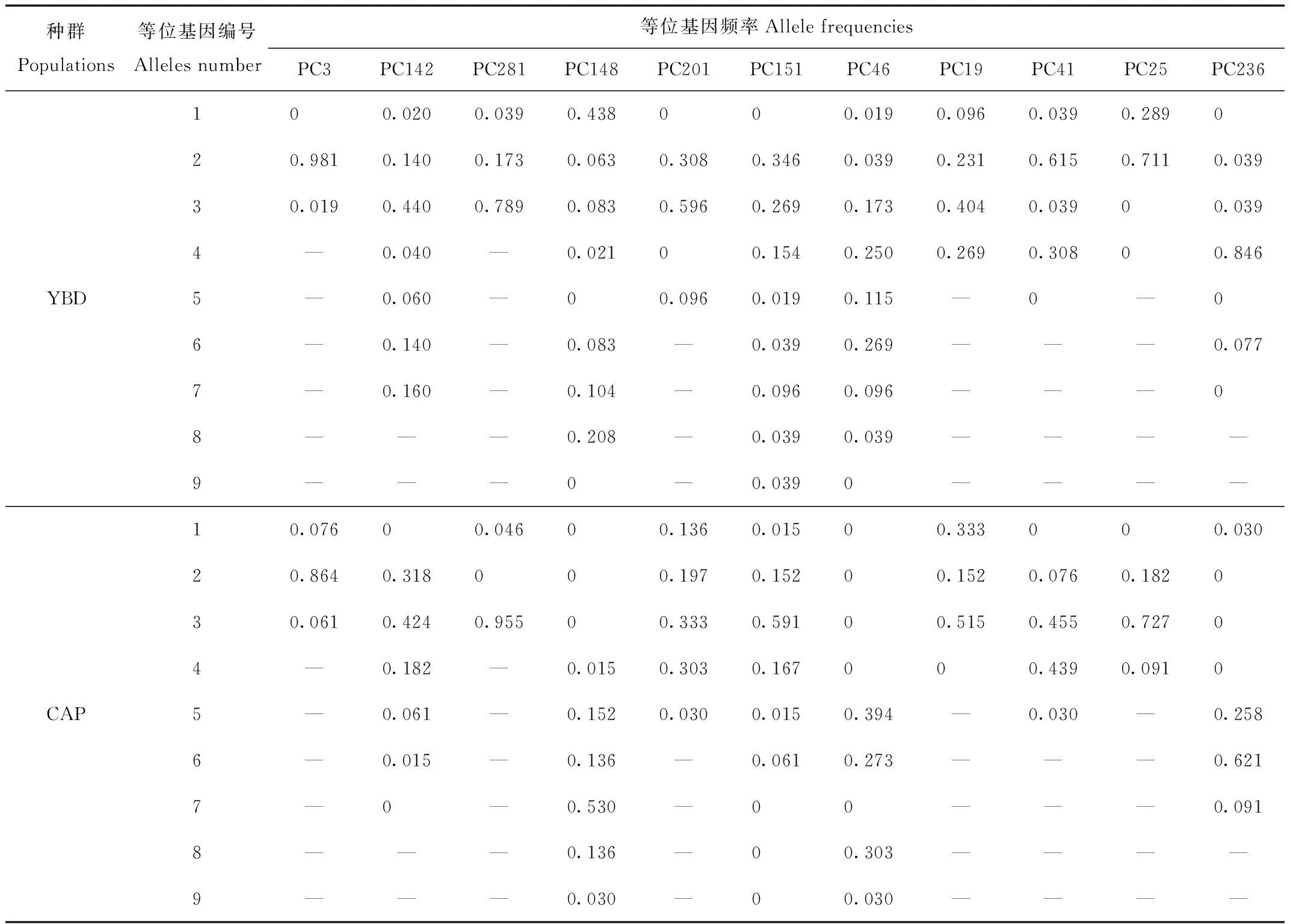
表2 11个微卫星位点在YBD和CAP种群中的等位基因和等位基因频率
表3显示,YBD种群中Ne的数量为1.039~5.240,平均为(2.768±1.294),而CAP种群Ne的数量为1.095~3.828,平均为(2.434±0.818)。在2个种群中,所有位点的Na都显著大于Ne,表明2个种群都面临遗传多样性丧失的风险。YBD种群的平均Ho和平均He分别为(0.258±0.164)和(0.546±0.251);CAP种群平均Ho和平均He分别为(0.479±0.228)和(0.531±0.201)。哈迪-温伯格平衡(H-W)检验表明,YBD种群中有2个位点(PC3和PC25)处于平衡状态(P>0.05),其余9个位点显著偏离平衡状态(P<0.05);相比之下,CAP种群中有7个位点处于平衡状态(P>0.05),其余4个位点(PC201、PC236、PC148和PC151)表现出显著偏离(P<0.05)。这11个位点在YBD和CAP种群中的平均PIC分别为(0.493±0.242)和(0.489±0.177)。YBD种群的FIS在0.720 (PC236)~-0.405 (PC25),平均值为(0.456±0.357),2个位点的FIS<0,9个位点的FIS>0,表明存在近亲繁殖的趋势;CAP种群的FIS在0.441 (PC151)~-0.199 (PC25),平均值为(0.102±0.217),5个位点的FIS<0,6个位点的FIS>0,表明近亲繁殖趋势不明显(表4)。


续表3

表4 YBD和CAP绿孔雀种群的11个微卫星位点的F检验结果
2.4 种群遗传分化与遗传结构分析
11个位点遗传分化指数FST的平均值为0.138。根据Wright的分类标准[26],3个位点(PC41、PC25和PC236)遗传分化程度较大(FST>0.150),6个位点(PC281、PC46、PC151、PC201、PC19和PC148)处于中度分化状态(0.050 在进行种群遗传结构分析时,预设K值(理论种群数)为2~4,每个K值重复运算10次,K=3时的DeltaK最大,表明本研究中所有参试个体可划分为3个种群(图4):CAP种群可划分为2个亚群(红色和绿色),而YBD种群为单一亚群(蓝色),2个CAP亚群的遗传成分占比很小。这说明CAP种群可能由来自不同地理区域的祖先发展而来,而2个亚群之间的基因交流十分有限。同时,CAP种群的祖先并非来自YBD种群。 图4 YBD和CAP种群的STRUCTURE分析(K=3)Fig.4 STRUCTURE analysis of YBD and CAP populations (K=3) 云南省各地蓝孔雀的养殖比较多,有研究怀疑养殖的蓝孔雀发生逃逸,并在野外建立了种群[13,31],因此,首先需要鉴定羽毛是否来自绿孔雀。物种鉴定采用母系遗传的线粒体DNA,如果母系来源于蓝孔雀,排除在后续研究;母系属于绿孔雀的样品,仍需采用核DNA鉴定其核基因组是否来自绿孔雀。Cytb基因片段鉴定母系物种是野生动物法医学鉴定的标准遗传标记[18]。鉴定时以转换(transition,Ts)为碱基替换模式计算K2P遗传距离,以种间和种内的K2P距离范围为参照[23],考察羽毛样品与已知纯种绿孔雀和蓝孔雀之间的距离,落在种内还是种间的范围内,95%的可信度确定其属于绿孔雀还是蓝孔雀;同时也用K2P遗传距离构建NJ系统发育树,通过Bootstrap再次考察其属于哪个物种,本研究2种方法都一致支持所有羽毛样本的母系来源均为绿孔雀(表1,图2)。 核基因的鉴定中,高质量DNA正常分型的16个位点[18],有5个检测到无效等位基因,说明从角化的羽翈获得的DNA质量较低,对核基因的分型质量有明显的影响。采用11个分析正常的位点计算累积P(ID),随着位点的增加,累积P(ID)下降的速度(图3),选定8个位点即可满足个体识别可靠性的评估,又能减少分型误差带来的影响。最终以极高的可信度从29份样品中鉴定了26只个体。 YBD种群的Na为2~8个,平均为4.727个,说明表观上含有的遗传变异较多,但是Ne平均值只有2.768,大多数位点(9/11)都显著偏离H-W,这意味着强烈的遗传漂变和非随机交配将拉高遗传多样性丢失的风险。相比而言,CAP种群的Na为2~6个,平均为4.091个,Ne平均为2.434,均明显地低于YBD种群,11个位点中有7个处于H-W状态,表明遗传多样性丢失的风险小于YBD种群。 2个种群的杂合度都不高,CAP种群的Ho平均为0.479,而YBD种群的Ho只有0.258,说明遗传多样性很低;2个种群的He都高于各自的Ho,表明杂合度低于预期主要源自近交。种群近交系数(FIS)进一步印证这一点,YBD种群的近交程度(平均0.456)明显高于CAP种群(平均0.102)。绿孔雀野外种群以小规模家族群为主[11,32],YBD种群很可能因为生境中的隔离因素导致族群间的基因交流困难,繁殖以局部近交为主。STRUCTURE分析显示,YBD种群内部没有发生显著的遗传分化(图4)。种群历史上的基因交流是比较频繁而有效的,表现出来的近交并非是生境中景观异质性形成长期的隔离,而是近期才出现的。 STRUCTURE分析表明(图4),CAP种群包含2个遗传谱系,一个是以秦皇岛野生动物园饲养的绿孔雀为代表,最初建群者来自瑞丽地区;另一个谱系的建群者来源地没有可靠的记录。瑞丽地区、玉溪地区和CAP种群中的第2个谱系的绿孔雀之间有着极其明显的遗传分化,预示着整个云南种群的内部可能遗传结构非常复杂,甚至有着深度的分化。 本研究显示我国云南绿孔雀的地理分化可能非常显著,需要开展系统的谱系地理学研究,厘清各地种群之间的遗传关系,确认进化显著单元。在开展迁地保护时,避免通过再引入打破进化显著单元的独特性。具体到玉溪地区的种群,内部没有显著的遗传分化,遗传多样性水平较低,近期的近交可能导致遗传多样性的进一步丢失。管理上以加强族群之间的基因交流为目标,通过生态廊道的修建,或在不同族群之间进行个体转移,增加遗传交流的机会,扭转遗传多样性随近交的深化而不断丢失的趋势。 致谢:感谢秦皇岛野生动物园和中国科学院西双版纳热带植物园提供研究样品。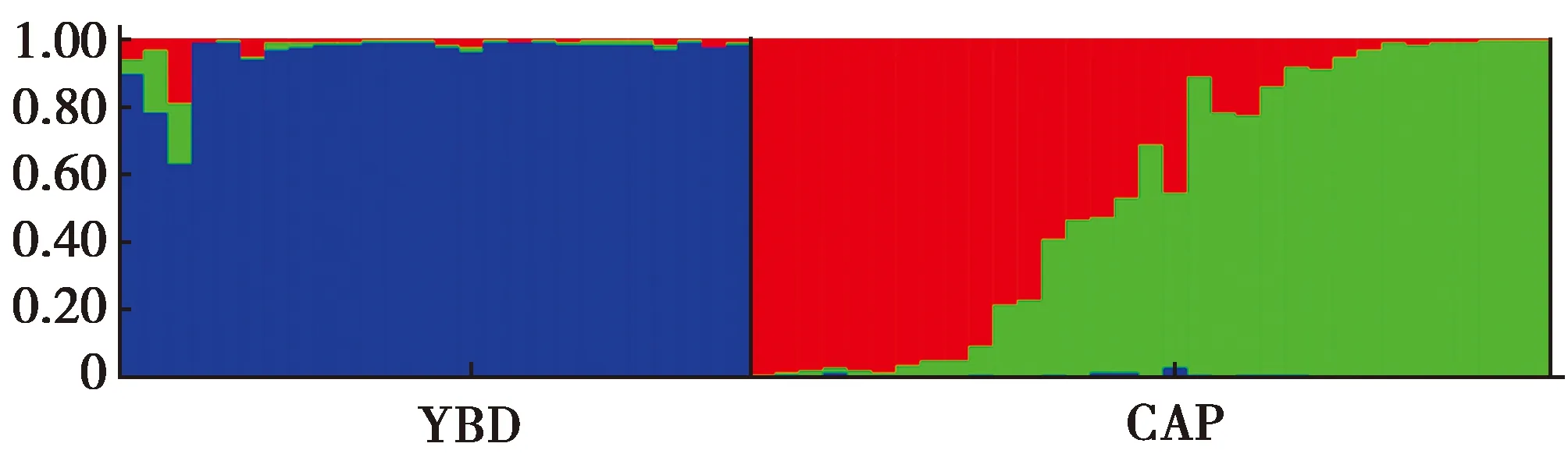
3 讨论
3.1 羽毛样品的物种鉴定和个体识别
3.2 YBD种群的遗传多样性
3.3 YBD种群与CAP种群的遗传关系
4 结论与建议

