南海北部表层海水微生物多样性及部分真菌活性物质研究
李 涵,樊成奇,陈 莎,周 进,周俊芳,陆亚男,马丽艳,田晓清,3
(1.上海海洋大学食品学院,上海 201306;2.中国水产科学研究院东海水产研究所,上海 200090;3.农业农村部远洋与极地渔业创新重点实验室,上海 200090;4.农业农村部东海渔业资源开发利用重点实验室,上海 200090;5.农业农村部低洼盐碱地水产养殖重点实验室,上海 200090)
地球表面约70%以上的面积都是海洋,海洋由于其特殊的环境(高压、高盐、低温等)拥有着非常丰富的微生物资源[1]。为了在海洋独特的环境中生存,海洋微生物产生了不同的适应机制,包括产生一些特定的生物分子,以及一些陆生环境中未曾发现的具有生物活性的化合物等[2]。种类丰富、代谢独特的海洋微生物是海洋天然产物的重要生产力,也是发现新药的一个重要来源[3],这一方面弥补了因新的陆生生物活性次级代谢产物越来越少的不足[4],另一方面也为发现新的活性次级代谢产物提供了新的途径。
海洋来源的真菌,其次级代谢产物因其独特的结构和活性的多样性,在近些年受到了研究者们的广泛关注。在来源于南海海域可培养真菌中已经分离到多种具有生物活性的次级代谢产物,例如核苷类[5]、萜类[6-7]、吲哚生物碱类[8]、聚酮类化合物[9]、生物碱[9-10]等,分离出来的次级代谢产物具有很好的抗菌活性[9]、抗肿瘤活性[9]、抗氧化活性[9]、抗细胞毒性[10]、抗烟草花叶病毒[11]等。由此可见,从海洋来源的真菌中发掘新的活性物质具有很好的发展前景。
南海是中国面积最大的海域,地处热带-亚热带地区,最大深处大于5 000 m,周围有多种生态系统分区如海岛、珊瑚、红树林等[12],蕴含着非常丰富的微生物资源,是研究海洋微生物多样性的理想区域之一。但近年来对南海微生物的多样性研究主要集中在沉积物中[13-15],仅有少数文献对海水中微生物的多样性进行报道[16-20]。本文通过对南海表层海水微生物的多样性及真菌粗提物的组蛋白去乙酰化酶(histone deacetylase,HDAC)抑制活性、抗肿瘤和抑菌活性进行研究,旨在筛选和发现可以作为海洋药物来源的天然产物,为南海微生物资源开发提供参考。
1 材料与方法
1.1 实验材料
海水样品的采集和保存:2019年10月于南海北部海域选取8个站位(15号、17号、19号、21号、39号、41号、43号和45号站位)采集样品,具体站位信息如表1。样品采集后冷藏运输至实验室4℃冰箱中保存。

表1 南海北部海域采样站位信息Tab.1 Information of sampling stations in the northern South China Sea
1.2 实验方法
1.2.1 菌株的分离和纯化
海水样品5 L,用0.22μm的无菌滤膜过滤,滤膜剪碎后放入无菌离心管中,加入10 mL无菌水充分振荡混匀后静置于4℃冰箱过夜。取菌悬液1 mL至9 mL无菌水中,常温混匀,并梯度稀释3次(10-1、10-2、10-3),取300μL混合液至细菌、真菌平板中,置于28℃恒温培养箱中培养3~28 d。所有分离培养基均按照文献[21]方法配制。真菌分离培养基:YPD合成培养基、PDA合成培养基和孟加拉红培养基;细菌分离培养基:2216E合成培养基;放线菌分离培养基:高氏1号培养基、R2A培养基、Qligo培养基、2216E合成培养基。肉眼观察菌落的特征(形态、大小、颜色等),初步筛选后挑选出不同形态的菌落至相对应的培养基中,三线法纯化至单菌落,斜面保存至4℃冰箱中。真菌分离过程中在培养基中两两加入抗生素:青霉素、链霉素、氯霉素各30 mg·mL-1。细菌分离过程中在培养基中两两加入抗生素:放线菌酮50 mg·mL-1,萘啶酮酸25 mg·mL-1,重铬酸钾50 mg·mL-1。
1.2.2 菌株鉴定
将微生物单菌落分别接种到5 mL相应液体培养基中,置于振荡培养箱中28℃,160 r·min-1培养2~5 d。细菌DNA提取,根据细菌基因组DNA提取试剂盒(Takara miniBest Bacteria DNA Extraction Kit Ver3.0,生工生物工程(上海)股份有限公司)说明书步骤提取目标基因组DNA。真菌DNA委托上海生工生物工程有限公司进行提取,根据真菌基因组DNA提取试剂盒(E.Z.N.ATMMag-Bind Soil DNA Kit(M5635-02),OMEGA)说明书步骤提取目标基因组DNA。
使用细菌通用引物:27F:(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R:(5′-GGTTACCTTGTTACGACTT-3′);真菌通用引物:ITS1:(5′-TCCGTAGGTGAACCTGCGG-3′)和ITS4:(5′-TCCTCCGCTTATTGATATGC-3′)。
细菌PCR反应体系为50μL:2X Taq PCR Master Mix 25μL,DMSO 0.5μL,上下游引物各1 μL,模板DNA 2μL,ddH2O 20.5μL。PCR反应程序:预变性95℃5 min;变性95℃30 s、退火56℃30 s、延伸72℃1 min,35个循环;延伸72℃10 min。真菌PCR反应系为25μL:10X PCR Buffer 2.5μL,MgCl(25 mmol·L-1)2μL,dNTP(2 mmol·L-1)0.5μL,上下引物各1μL,Taq DNA Polymerase(5 U·μL-1)0.5μL,DNA模板1μL,ddH2O 16.5μL。PCR反应程序:预变性94℃5 min;变性94℃30 s、退火60℃30 s、延伸72℃1 min,35个循环;延伸72℃8 min。扩增后的产物进行0.8%琼脂糖凝胶电泳检测后送至上海生工生物工程有限公司进行测序。将所测得细菌序列上传至EZbiocloud网站进行比对,真菌序列提交至NCBI网站并进行Blast比对,下载同源性较高的序列,用Clustal W对所得序列进行校正比对分析,利用Mega 7.0软件构建N-J系统发育树。
1.2.3 真菌培养与粗提物制备
结合真菌序列比对结果和真菌所处站位的不同,从中挑选出16株真菌(表2)在真菌平板上活化。挑取一定量活化好的菌丝体接种于500 mL YPD液体培养基中,28℃、120 r·min-1条件下培养18 d。培养结束后加入1.5 L无水乙醇浸泡,静置过夜,超声2次,每次30 min,待菌丝体沉降后,收集滤液,减压浓缩,浓缩液用DMSO溶解,制成10 mg·mL-1的粗提物备用。

表2 参与活性筛选的16株真菌的具体分类信息汇总Tab.2 Summary of specific taxonomic information of 16 strains of fungi in activity screening
1.2.4 活性筛选
1.2.4.1 HDAC抑制活性筛选
按照HDAC抑制剂药物筛选试剂盒(BioVision,货号K340-100)说明书操作。采用黑色96孔板,总反应体系为100μL。SAHA作为阳性对照1,以DMSO溶解,配置成100μmol·L-1的SAHA溶液;TSA(试剂盒自带的抑制剂)作为阳性对照2。Reaction Mix体系为50μL:10μL 10X HDAC Assay Buffer,2 μL Hela Nuclear Extract,5μL HDAC Substrate,33μL超纯水。待测粗提物反应体系为50μL的Reaction Mix,49 μL的超纯水,1μL的待测粗提物。阳性对照1的反应体系为50μL的Reaction Mix,49μL的超纯水,1μL的SAHA溶液。阳性对照2的反应体系为50μL的Reaction Mix,47μL的超纯水,2 μL的DMSO溶液,1μL的TSA溶液。阴性对照的反应体系为50μL的Reaction Mix,49μL的超纯水,1μL的DMSO。每个样本设置3个平行重复,计算HDAC的抑制活性。
1.2.4.2 抗肿瘤活性筛选
按照MTT[22]法测定16株真菌粗提物对HeLa细胞(购自中科院细胞库,上海)的活性。真菌粗提物稀释到60μg·mL-1,紫杉醇作为阳性对照其浓度为2μg·mL-1,实验重复3次。
1.2.4.3 抑菌活性筛选
按照K-B纸片法[23]测定16株真菌粗提物的抑菌活性。具体步骤为:将10 mg·mL-1的真菌粗提物稀释至0.2 mg·mL-1,将待测的两株指示菌嗜水气单胞菌(Aeromonas hydrophila)、维氏气单胞菌(Aeromonas vickers)(来源于中国水产科学研究院东海水产研究所)接种到液体培养基中,28℃振荡培养12 h进行菌株活化,调整其菌液浓度为104个·mL-1。将两株指示菌液分别吸取100μL均匀涂布在LB固体平板上。等待菌液吸收后,在平板上放置灭菌的滤纸片,加入稀释后的真菌粗提物5μL,28℃培养12 h,空白平板作为阴性对照,以抑菌圈的直径来表示粗提物抑菌活性的强弱,实验重复3次。
2 结果与分析
2.1 可培养细菌的分离结果
采用稀释涂布的方法对2019年采集的8个站位的海水样品中微生物进行菌株分离培养。经16S rRNA基因序列比对分析,合并相似度在98%以上的菌株为同一种[24],共分离得到162株细菌。对162株细菌进行16SrRNA基因序列比对结果显示:分属于4个门,5个纲,15个目,21个科,35个属的61个种,系统进化树构建如图1。从门水平上来看,变形菌门(Proteobacteria)和放线菌门(Actinomycetes)为优势类群,变形菌门一共分 离 出105株,其 中α-变 形 菌 纲(α-Proteobacteria)84株,占51.8%,γ-变形菌纲(γ-Proteobacteria)21株,占12.9%。放线菌门50株,占30.8%,拟杆菌门(Bacteroidetes)4株,占2.5%,厚壁菌门(Firmicutes)3株,占1.9%。
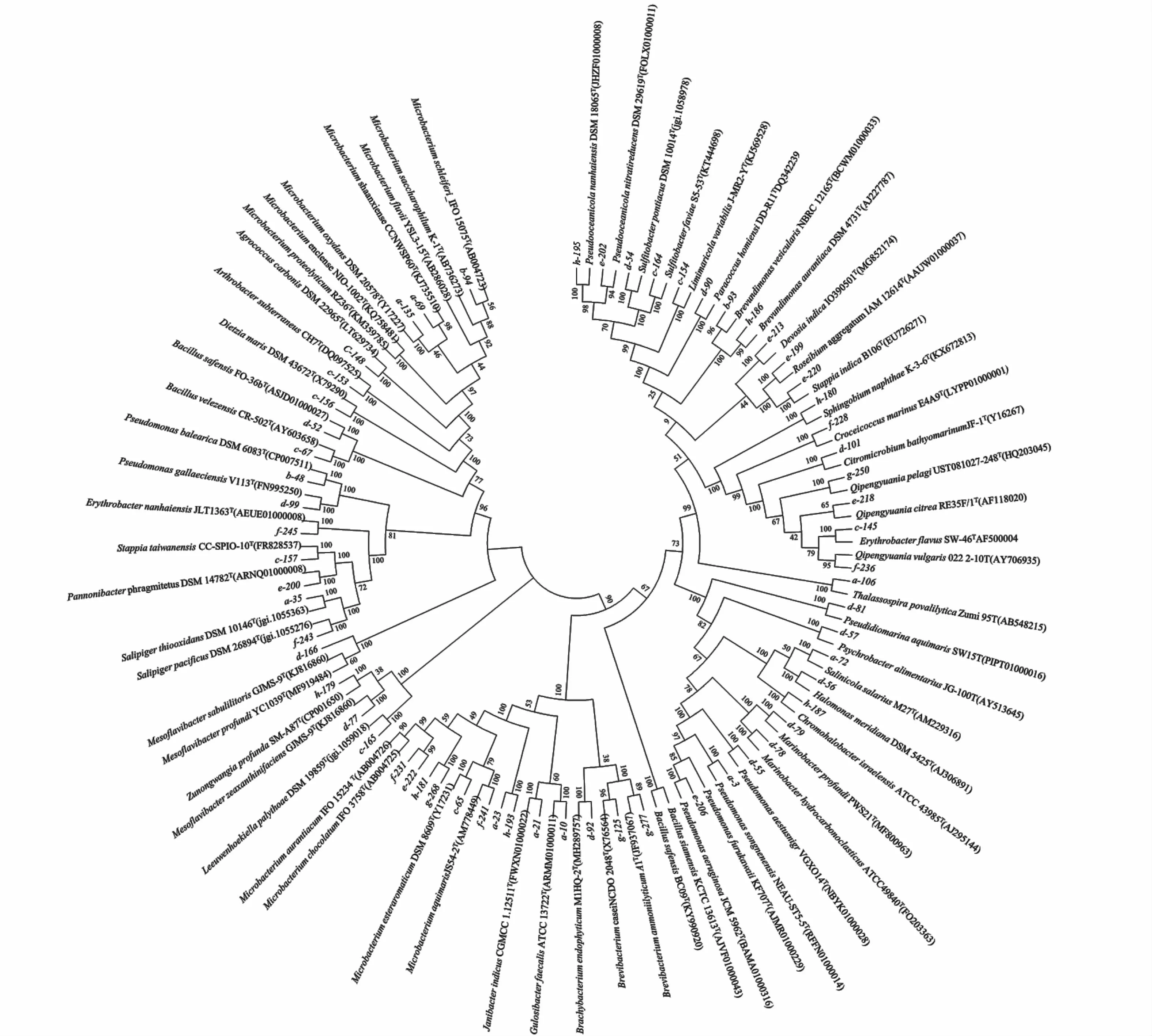
图1 基于NJ法对部分细菌代表菌株16S r RNA序列构建系统进化树Fig.1 Neighbour-joining(NJ)tree based on bacterial 16S r DNA sequences of some representative bacterial strains
从属水平上来看(图2),微杆菌属(Microbacterium)和赤杆菌属(Erythrobacter)为优势属,分别分离得到40株和23株,袁其鹏属(Qipengyuania)、假单胞菌属(Pseudomonas)和斯塔普氏菌属(Stappia)分别分离得到14株、10株和9株。Salipiger属 分 离 得 到6株,Brachybacterium、Citromicrobium、Marinobacter、Sulfitobacter属各分离得到5株,Pseudooceanicola属 分 离 得 到4株,Bacillus、Brevundimonas、Croceicoccus、Roseibium属 各 分 离 得 到3株,Limimaricola、Mesoflavibacter、Psychrobacter、Paracoccus、Sphingobium属各分离得到2株,Agrococcus、Arthrobacte、Chromohalobacter、Devosia、Dietzia、Gulosibacter、Halomonas、Janibacter、Leeuwenhoekiella、Pannonibacter、Pseudidiomarina、Salinicola、Thalassospira和Zunongwangia属都只分离得到1株。

图2 分离得到的细菌属分类及数量占比情况Fig.2 Classification of isolated bacteria strains by genus and number proportion
从菌株数量上来看(图3),Erythrobacter pelagi分离得到的菌株数量最多,共12株;E.flavus和Microbacterium aurantiacum、M.schleiferi次之,分别分离得到10、9、9株。

图3 分离得到细菌种分类及数量占比情况Fig.3 Classification of specific species of isolated bacteria strains and number proportion
2.2 不同站位可培养细菌多样性
在不同站位间分离出来的可培养细菌多样性有明显差异。从门水平上来看(图4-A),8个站位都分离到变形菌门和放线菌门的细菌,且在每个站位中变形菌门菌株数均高于放线菌门菌株数,只有3个站位分离到厚壁菌门和拟杆菌门。19号站位和21号站位分离得到细菌菌落组成相同,变形菌门、放线菌门、拟杆菌门和厚壁菌门菌株在这两个站位中都有分离到。17号、39号和41号站位的细菌菌落为放线菌门和变形菌门。15号和45号站位虽然都分离到3个门的细菌,但是它们的菌落组成不一样,两个站位均分离到来自放线菌门和变形菌门中的细菌,15号站位未能分离到拟杆菌门细菌但分离到厚壁菌门的芽孢杆菌属,45号站位未能分离到厚壁菌门但有来自拟杆菌门的细菌。
从属水平上来看(图4-B),15、19和21号站位分离到的属种类最丰富,分别为15、13、14种,17号站位中分离出来的细菌数最少且属的种类也最少,只有4个属。Erythrobacter、Microbacterium、Qipengyuania这3个属分布较广,从7个 站 位 中 分 离 出 来。Stappia和Citromicrobium属从5个站位中分离出来、Pseudomonas和Brevundimonas属从4个站位中分离出来,Roseibium、Pseudooceanicola、Marinobacter和Bacillus属从3个站位中分离出来,Salipiger、Paracoccus、Croceicoccus和Brevibacterium属从2个站位中分离出来。另有20个属的细菌都只在特定的站位中被分离出来,分属于21、19、15、45和39号站位,占总属多样性的57%,其中21号站位分离出特定属的数量是最多的(6个属),19号站位中有5个属,15号站位中有4个属,45号站位中有3个属,39号站位中有2个属。
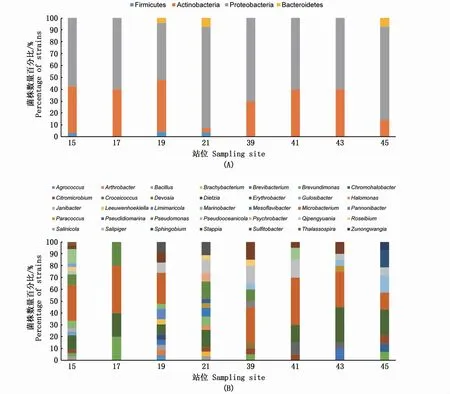
图4 不同站分离得到细菌门(A)和属(B)水平多样性Fig.4 Phylum(A)and genus(B)level diversity of bacteria isolated from different sites
2.3 可培养真菌的多样性
从8个站位的海水样品中分离出真菌,共43株。经过ITS基因序列比对后发现这43株真菌分属于2个门,5个纲,7个目,7个科,7个属的14个种,系统进化树构建如图5。
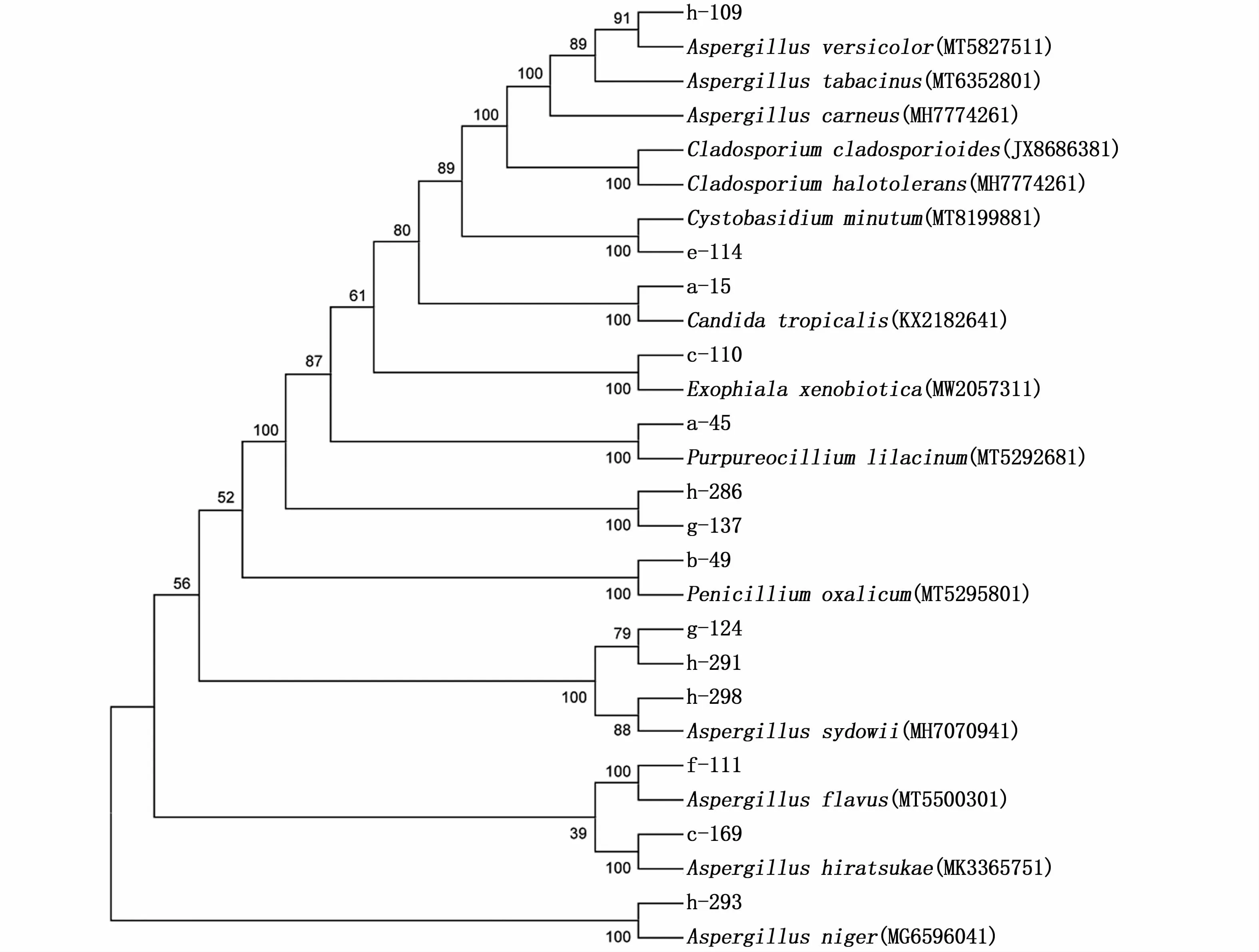
图5 基于NJ法对ITSr DNA序列的部分真菌所属菌株构建系统进化树Fig.5 Neighbour-joining(NJ)tree based on fungi ITS r DNA sequences of some representative fungi strains
从门水平上来看,这43株真菌分属于子囊菌门(Ascomycota)和担子菌门(Basidiomycota),子囊菌门中所分离出来的菌株数量是最多的,有40株,其余3株是担子菌门。从属水平上来看,Aspergillus为优势属,共分离得到21株,占48.8%;其次是Penicillium,分离得到9株,占20.9%; Cladosporium、 Cystobasidium、Purpureocillium、Exophiala和Candida分别分离得到5、3、2、2和1株,各占11.6%、7%、4.7%、4.7%和2.3%。从菌株数量上来看,Penicillium oxalicum分离得到的菌株数量是最多的,共分离得到9株,其次是Aspergillus versicolor,分离得到7株。A.sydowii和A.carneus则分别分离得到4株。Cystobasidium minutum和Aspergillus flavus分别分离得到3株。Purpureocillium lilacinum、Cladosporium halotolerans、 Cladosporium sphaerospermum和Exophiala xenobiotica分别分离得 到2株,Aspergillus hiratsukae、A.niger、A.tabacinus、Candida tropicalis和Cladosporium cladosporioides都分别只分离得到1株。从分离到的真菌序列与NCBI数据库中已有的序列相比,其相似性都大于98%。
2.4 部分真菌的活性分析
HDAC抑制活性筛选结果显示(图6),这16株真菌粗提物对HDAC均有一定的抑制作用,但与阳性对照组相比抑制率较低,抑制率在44%~54%之间,阳性对照组SAHA和TSA对HDAC的抑制率达92.3%和94.7%。Aspergillus sydowii b-96、Aspergillus versicolor h-291、Aspergillus versicolor e-118、Cladosporium halotolerans b-61、Cystobasidium minutum e-114、Penicillium oxalicum f-112、Cystobasidium minutum e-129、Penicillium oxalicum b-49的真菌粗提物对组蛋白去乙酰化酶的抑制率在50%~54%之间,其余真菌粗提物对HDAC的抑制率在44%~49%之间,A.sydowii b-96的真菌粗提物的抑制率最高,为54%,A.versicolor h-291的抑制率次之,为53%,A.flavus f-111的抑制率最低,为44%。
抗癌活性结果显示(图6),这16株真菌粗提物在浓度为60μg·mL-1时对Hela细胞的抑制率较低,其中编号为P.oxalicum h-287和A.versicolor h-291的真菌粗提物对肿瘤细胞的抑制率为37%和35%,A.sydowii b-96和E.spinifera c-110这两株真菌粗提物对Hela细胞的抑制率最低,仅为2%。
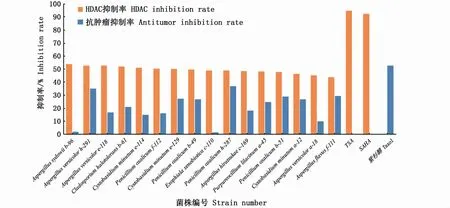
图6 16株真菌HDAC、抗肿瘤活性筛选结果Fig.6 Screening results of HDAC and antitumor activity of 16 strains of fungi
抑菌活性测试发现,这16株真菌粗提物对嗜水气单胞菌和维氏气单胞菌没有抑菌效果。
3 讨论
本研究从南海局部地区采集了8个站位的表层海水样品,经过基因序列比对分析后共分离得到162株细菌和43株真菌。162株细菌是由变形菌门、放线菌门、拟杆菌门和厚壁菌门组成的,其中α-变形菌纲和放线菌纲为优势类群,与苏悦[16]从南海北部海域分离到的细菌的优势类群相同。本实验中分离到的厚壁菌门数量最少,且都是芽孢杆菌属,与徐重[17]、卢靖雯等[18]从南海表层海水中分离的结果一致,而苏悦[16]未能从南海北部海域表层海水中分离到厚壁菌门菌株。有研究显示,可培养的芽孢杆菌属在南海沉积物中分离到的菌株数量多于表层海水[25-27],推测是芽孢杆菌属在海水中的分布少于沉积物中,原因可能是海水中的营养物质相较于沉积物中偏少,导致芽孢杆菌未能在海水中形成芽孢帮助它抵御复杂的海洋环境,不足以支撑其在海水中存活下来;亦或是实验室中使用的培养基选择性不强,不适合芽孢杆菌的生长,对于海水中分离芽孢杆菌属较少的原因还需要进行更深入的探讨。
基于本研究分离结果,不同站位分离得到的可培养细菌多样性、数量存在差异。从采样的空间水平分布来看,15~21号这4个站位的可培养细菌多样性多于39~45号站位,造成站位间分离到的可培养细菌多样性差异的原因,可能是受到各个站位水文因素的影响,如洋流、营养组成[28]。
从8个站位中分离得到43株真菌,经过与数据库比对后其ITS序列与真菌相似性均大于98%。大多数的海洋真菌都属于子囊菌门,从中分离到的担子菌门代表性却不足[29],本实验中真菌分离结果与其相同。其中曲霉属(Aspergillus)和青霉属(Penicillium)为优势属,分别占48.8%和20.9%。曲霉属和青霉属在自然界中分布较广,从冯丽等[30]、曾奇等[15]和曲佳等[31]从南海沉积物中分离出的真菌多样性结果来看,曲霉属和青霉属在沉积物中也为优势属。
抗肿瘤活性实验结果表明,真菌P.oxalicum h-287和A.versicolor h-291粗提物对Hela细胞活性的抑制率最高,分别为37%和35%。AWAD等[32]对A.versicolor Faesay4进行深层发酵后获得L-谷氨酰胺酶,对其进行抗肿瘤活性研究,发现其对人肝癌细胞(HepG-2)、结肠癌细胞(HCT-116)、乳腺癌细胞(MCF-7)、肺癌细胞(A-549)和宫颈癌细胞(Hela)细胞具有较强的抗癌活性,IC50分别为39.61、12.8、6.18、11.48、7.25μg·mL-1。SHI等[33]从海洋真菌P.oxalicum中分离出一种新型二氢噻吩缩合色酮(POA),POA是具有较强的抗肿瘤活性,被认为是一种具有生物活性的抗癌药物。本实验中,从两株不同属真菌中获得的粗提物对Hela肿瘤细胞有一定的抑制作用,与既往文献研究结果相似,后续可以对这两株真菌扩大发酵,以期从中分离出具有抗肿瘤活性的次级代谢产物。
WANG等[34]从中国西沙群岛采集到的海绵上分离到一株Aspergillus sydowii,对其进行发酵培养后从乙酸乙酯提取部位分离得到一个已知化合物aspergillusene A,这种化合物对嗜水气单胞菌有显著的抗菌活性。LI等[35]从深海沉积物真菌A.versicolor SD-330中分离鉴定了1个新的芳香族双烯型倍半萜和4个已知的类似物,其中3个化合物对嗜水气单胞菌、大肠杆菌、迟缓爱德华菌和哈维弧菌等人畜共患致病菌具有选择性抑制活性,最低抑菌浓度(MIC)为1.0~8.0 μg·mL-1。通 过 已 有 文 献 表 明[34,35],部 分Aspergillus真菌的次级代谢产物对水生致病菌如嗜水气单胞菌有抑菌作用,而本实验中16株真菌的粗提物对嗜水气单胞菌均没有抑菌作用,推测是因为本实验中只测试了单一粗提物浓度,其中可能的活性次级代谢产物浓度低于其最低抑菌浓度,后续研究将继续开展对这16株真菌分离鉴定的次级代谢产物的抑菌作用的筛选,有望发现活性化合物。
综合以上分析,编号为P.oxalicum h-287和A.versicolor h-291两株真菌的粗提物在活性筛选实验中表现优异,HDAC抑制率为49%和53%,抗肿瘤抑制率为37%和35%,可以作为潜在的目标活性菌株对其次级代谢产物和活性进行深入研究。
4 小结
本文对南海局部地区表层海水中分离到的微生物进行初步鉴定,共分离得到162株细菌和43株真菌。样品中162株细菌属于由变形菌门、放线菌门、拟杆菌门和厚壁菌门组成的35个属,43株真菌属于由子囊菌门和担子菌门组成的14个种,该地区的微生物多样性较高。通过对分离得到的部分真菌进行HDAC活性、抗肿瘤活性和抑菌活性筛选后发现,16株真菌粗提物对HDAC都一定的抑制作用,对人宫颈癌细胞的抑制率在2%~37%范围内,对两株细菌指示菌没有抑菌效果。本研究结果可为研究南海菌株资源及其代谢产物提供资料,也可为研究人员从南海中发现有生物活性的天然药物提供参考。
致谢:感谢浙江工业大学占扎君教授团队完成了体外抗肿瘤活性筛选实验。

