中国荷斯坦牛PIK3CB基因多态性及其与繁殖和产奶性状的关联分析
徐昊祺,许静漪,胡丽蓉,张 帆,罗汉鹏,张海亮,师 睿,李 想,刘 林,刘巧香,郭 刚,王雅春
(1.中国农业大学动物科技学院,农业农村部动物遗传育种与繁殖(家畜)重点实验室,畜禽育种国家工程实验室,北京 100193;2.北京奶牛中心,北京 100192;3.北京生物种业创新联合体,北京 101206;4.北京首农畜牧发展有限公司,北京 100029)
长期以来,育种者对奶牛的产奶性能进行了高强度的选育,其遗传进展取得了一定程度的改善[1]。然而,由于产奶性状与繁殖性状之间存在的不利遗传相关,产奶性状的高强度选择造成了繁殖、健康、长寿等功能性状的衰退,严重影响了奶牛养殖的效益[2]。由于繁殖性状的遗传力较低,常规选育进展速度较慢;针对繁殖性状进行相关研究,筛选候选基因和分子标记,采用分子标记进行辅助选择对繁殖性状的遗传改良具有重要意义。师睿等[3]前期对中国荷斯坦牛繁殖性状进行了全基因组关联分析,获得了包含磷脂酰肌醇-4,5-二磷酸3-激酶催化亚单位β(PIK3CB)在内的多个候选基因。随后,采用蛋白互作网络分析发现PIK3CB基因与多个候选基因存在互作关系,表明PIK3CB基因可能是影响中国荷斯坦牛繁殖性状的重要基因(未发表)。PIK3CB基因可以编码磷脂酰肌醇3-激酶(PI3K)/蛋白激酶B(Akt)信号转导通路中磷脂酰肌醇3-激酶的p110催化亚单位PI3Kp110β[4],PI3K/Akt通路广泛存在细胞中,对细胞周期、凋亡、糖代谢及其他重要生理过程产生调节作用[5]。
研究表明,PI3K/Akt通路可调节奶牛等哺乳动物颗粒细胞的凋亡进程,参与卵泡发育和排卵,以调控卵巢活动周期[6-7]。畜禽生殖器官的细胞凋亡与繁殖性能存在关系,在雌性动物卵巢中,卵母细胞、颗粒细胞和黄体细胞的凋亡会使卵泡闭锁,从而影响卵巢发育[8-9]。排卵后残留的颗粒细胞和膜细胞形成黄体,黄体的正常退化是启动下一个发情周期和卵泡发育的重要条件,黄体的溶解也同样与细胞凋亡有密切联系[10]。输卵管上皮细胞和子宫内膜细胞的周期性生长、分化、退化和再生与生殖周期有密切联系;在不同繁殖力的母羊生殖器官中,细胞凋亡情况和细胞凋亡基因Bad的表达存在显著差异,Bad基因编码PI3K/Akt通路下游Bcl-2家族中的促进细胞凋亡蛋白,受上游PIK3CB基因表达的调控[11]。马鸿程等[12]发现,在牦牛不同大小级别卵泡壁颗粒细胞及卵母细胞中均有PIK3CB基因表达,其参与了PI3K/Akt介导的卵泡发育调控。上述研究表明PIK3CB基因可能在动物繁殖生理中扮演着重要角色。此外,PI3K/Akt通路通过调控葡萄糖摄取和脂质合成,参与奶牛乳腺上皮细胞的功能分化和乳汁合成的代谢途径[13]。葡萄糖是泌乳奶牛合成乳糖的前体物质,与乳脂、乳蛋白的合成密切相关,对泌乳奶牛具有重要的营养生理功能[14]。同时,乳糖可以维持牛奶的渗透压,其合成速率是产奶量的主要控制因素[15]。在敲除PIK3CB基因的小鼠肝脏中,p110β的缺失会影响胰岛素敏感性和葡萄糖稳态[16]。PI3K/Akt通路上游PIK3CB基因参与表达的PI3K蛋白受胰岛素受体激活后催化4,5-二磷酸脂酰肌醇(PIP2)生成第二信使PIP3,可以调控糖原合成、抑制糖异生,降低血糖[17],从而影响奶牛的产奶性能。
综上,PIK3CB基因可能与中国荷斯坦牛繁殖和产奶性状有关,但在荷斯坦牛中鲜见关于PIK3CB基因多态性与繁殖和产奶性状的关联分析。本研究拟通过筛查PIK3CB基因的单核苷酸多态位点(SNP),并与荷斯坦牛的5个繁殖性状(初产日龄、初配日龄、产犊至首次配种间隔、青年牛首末次配种间隔、经产牛首末次配种间隔)和6个产奶性状(产奶量、乳蛋白量、乳蛋白率、乳脂量、乳脂率、体细胞评分)进行关联分析,以探究该基因与荷斯坦牛繁殖和产奶性状的关系,获得中国荷斯坦牛繁殖和产奶相关的遗传标记,为中国奶牛的标记辅助选择和基因组选择提供有用信息。
1 材料与方法
1.1 DNA混池构建
选取70头无亲缘关系或亲缘关系较远的中国荷斯坦牛公牛构建DNA池,冻精样品由北京奶牛中心提供。用高盐法提取冻精样品基因组DNA,使用NanoDrop 2000检测浓度后将所有DNA稀释到100 ng/μL,于-40 ℃保存备用。
1.2 样品采集和DNA提取
挑选北京地区8个牛场(金银岛、半截河、南三、渠头、长三、长四、金星和中以)1 160头健康的泌乳荷斯坦牛,采集血液样品,每头牛采集尾根静脉血约10 mL,使用血液基因组DNA提取试剂盒(DP318,天根生化科技(北京)有限公司)从抗凝血样提取基因组DNA,利用NanoDrop 2000测定样品DNA浓度和纯度,并通过2.0%琼脂糖凝胶电泳鉴定DNA片段完整性,―40 ℃保存备用。
1.3 基因多态性筛查
根据GenBank中牛PIK3CB基因的序列(登录号:NC_037328.1),采用Primer-BLAST设计引物,引物信息见表1。引物均由北京擎科生物科技有限公司合成。PCR反应体系25 μL:DNA(100 ng/μL)1 μL,1×CataAmpTaqPlus PCR Mix(北京开拓赛思生物技术有限公司)22 μL,上、下游引物(10 μmol/μL)各1 μL。PCR反应程序:98 ℃预变性2 min;98 ℃变性10 s,退火(温度见表1)30 s,72 ℃延伸10 s,共35个循环;72 ℃延伸1 min。PCR产物经2.0%琼脂糖凝胶检测后,送至北京擎科生物科技有限公司测序,利用R语言对测序峰图进行可视化,识别双峰,并比对参考序列筛选SNP位点。

表1 引物信息

续表
1.4 基因分型
针对筛选出的SNP,根据其峰值比例、距离、所处区域及突变类型等确定了7个SNPs用于在1 160头泌乳荷斯坦牛中进行基因分型。采用竞争性等位基因特异性PCR(KASP)技术进行SNP分型,其检测引物和通用引物见表2,KASP分型由中玉金标记(北京)生物技术股份有限公司完成。

表2 多态位点竞争性等位基因特异性PCR(KASP)分型引物信息
1.5 育种值估计
本研究收集了北京地区荷斯坦牛群(与基因分型个体属于相同群体)的初产日龄(AFC)、初配日龄(AFS)、产犊至首次配种间隔(ICF)、青年牛首末次配种间隔(IFL_H)、经产牛首末次配种间隔(IFL_C)5个繁殖性状以及产奶量(MY)、乳蛋白量(PY)、乳蛋白率(PP)、乳脂量(FY)、乳脂率(FP)、体细胞评分(SCS)6个产奶性状的估计育种值(EBV)作为表型,用于关联分析。基于牛群的生产性能测定记录和牧场管理记录,进行上述性状的常规遗传评估工作,进而获得上述性状的估计育种值,繁殖和产奶性状的遗传评估模型参考Liu等[18]和任小丽等[19]的研究。
1.6 数据处理与统计分析
对7个SNPs进行分型后,统计具有多态位点的基因型频率、基因频率、多态信息含量(PIC)、群体杂合度(He)和有效等位基因数(Ne),并检验各SNP基因型分布是否符合哈代-温伯格平衡定律。使用Haploview 4.2软件对PIK3CB基因SNP进行连锁不平衡分析,并构建单倍型块。
采用SAS 9.2软件的GLM过程对5个繁殖性状(AFC、AFS、ICF、IFL_H和IFL_C)和6个产奶性状(MY、PY、PP、FY、FP和SCS)与PIK3CB基因的7个SNPs进行基于单位点和单倍型组合的关联分析,关联分析模型为:
Y=μ+G+e
式中,Y,泌乳牛各繁殖或产奶性状的EBV;μ,群体均值;G,基因型或单倍型固定效应;e,随机残差。采用Bonferronit检验进行组间比较。结果以最小二乘均值±标准误表示。P<0.05表示差异显著;P<0.01表示差异极显著。
2 结 果
2.1 PIK3CB基因的SNP
根据双峰筛查与序列比对,确定了17个位于1号染色体上的候选SNPs位点(表3),均已被数据库收录,其中15个SNPs位于内含子区域,2个SNPs位于外显子区域。最终确定7个SNPs用于在泌乳牛群体中分型,其测序峰图如图3所示。由图3可知,g.130430832 A>-位点可见由缺失导致的片段错位,其余SNP位点可见明显套峰。
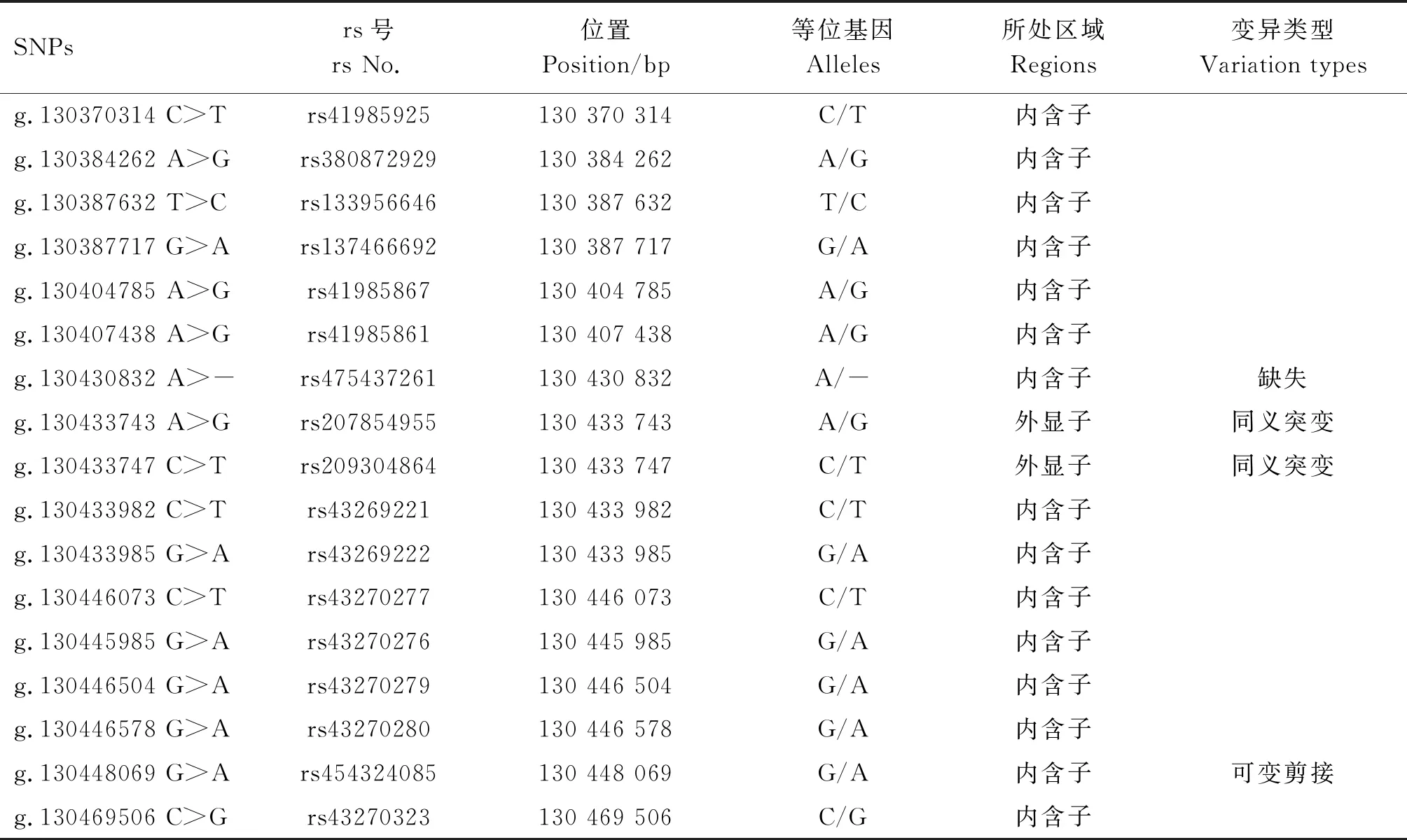
表3 PIK3CB基因候选SNPs信息

箭头所示为SNPs位点
2.2 SNP的群体遗传学分析
通过KASP分型技术,本研究在1 160头泌乳荷斯坦牛中获得了7个SNPs的基因型,部分结果如图3所示。由图3可以看到3种基因型聚类明显,说明SNP分型结果可靠。

左上和右下区域点分别代表纯合基因型;中间区域点代表杂合基因型;圆圈中点代表无明确分型的信号;左下区域点为空白对照
由表4可知,PIK3CB基因7个SNPs的MAF的范围为0.12~0.44。位点g.130430832 A>-、g.130433743 A>G和g.130448069 G>A的PIC分别为0.21、0.23和0.22,为低度多态;其余SNP的PIC位于0.41~0.49之间,为中度多态。g.130448069 G>A位点处于哈代-温伯格不平衡状态(P<0.05),其余SNPs均处于哈代-温伯格平衡状态(P>0.05)。7个SNPs的遗传杂合度(He)较低,说明其在测定群体中变异较小。g.130430832 A>-、g.130433743 A>G和g.130448069 G>A位点的有效等位基因数(Ne)较小,等位基因在测验群体中分布较不均匀,其余SNPs均接近于2,说明等位基因在测验群体中分布均匀。
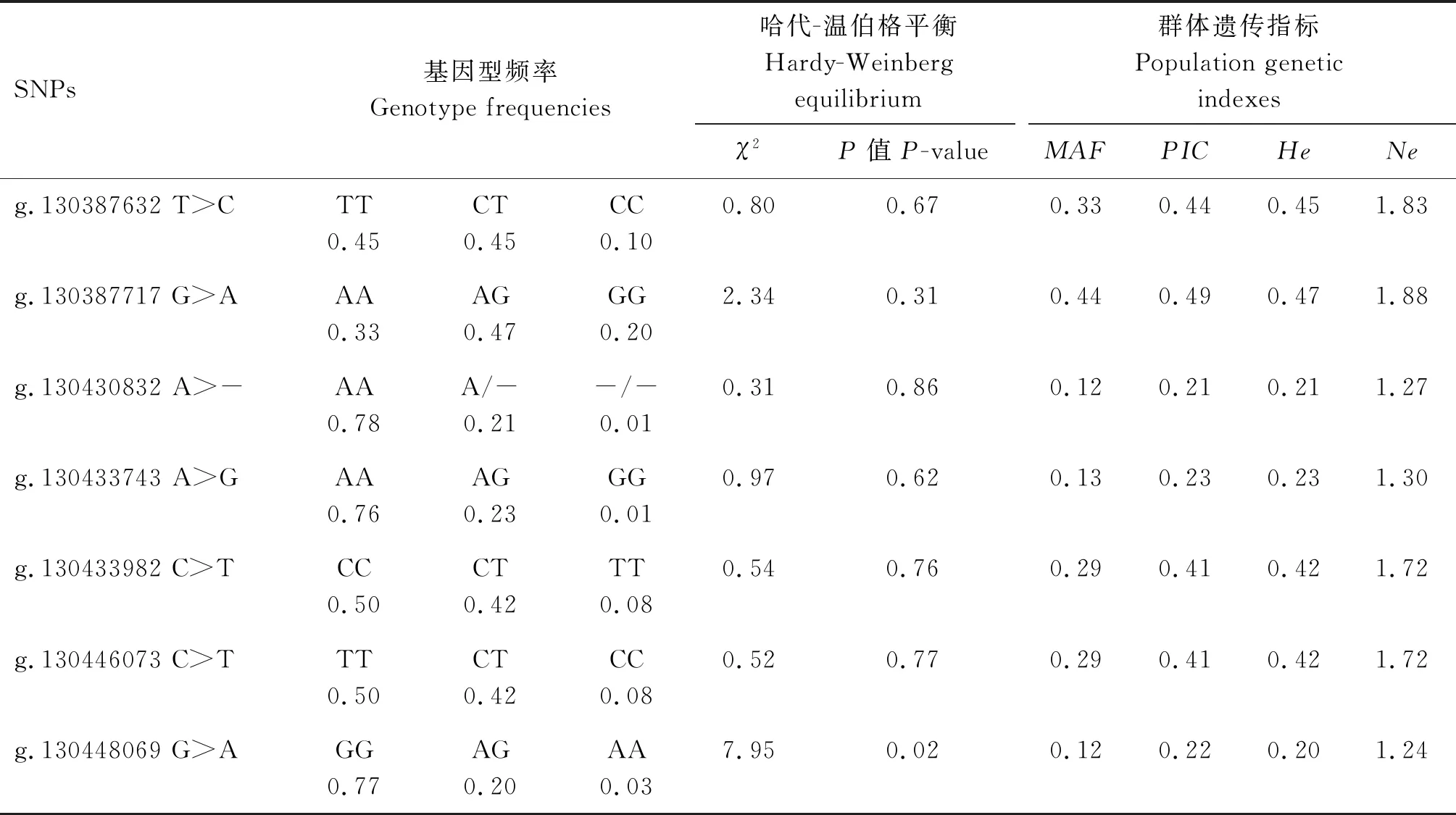
表4 PIK3CB基因7个SNPs的遗传多样性
2.3 PIK3CB基因SNPs与繁殖和产奶性状的关联分析
2.3.1PIK3CB基因SNP与繁殖性状关联分析 由表5可知,7个SNPs位点与AFC均无关联(P>0.05),g.130387632 T>C位点3种基因型彼此间IFL_H差异极显著(P<0.01),表现为CC
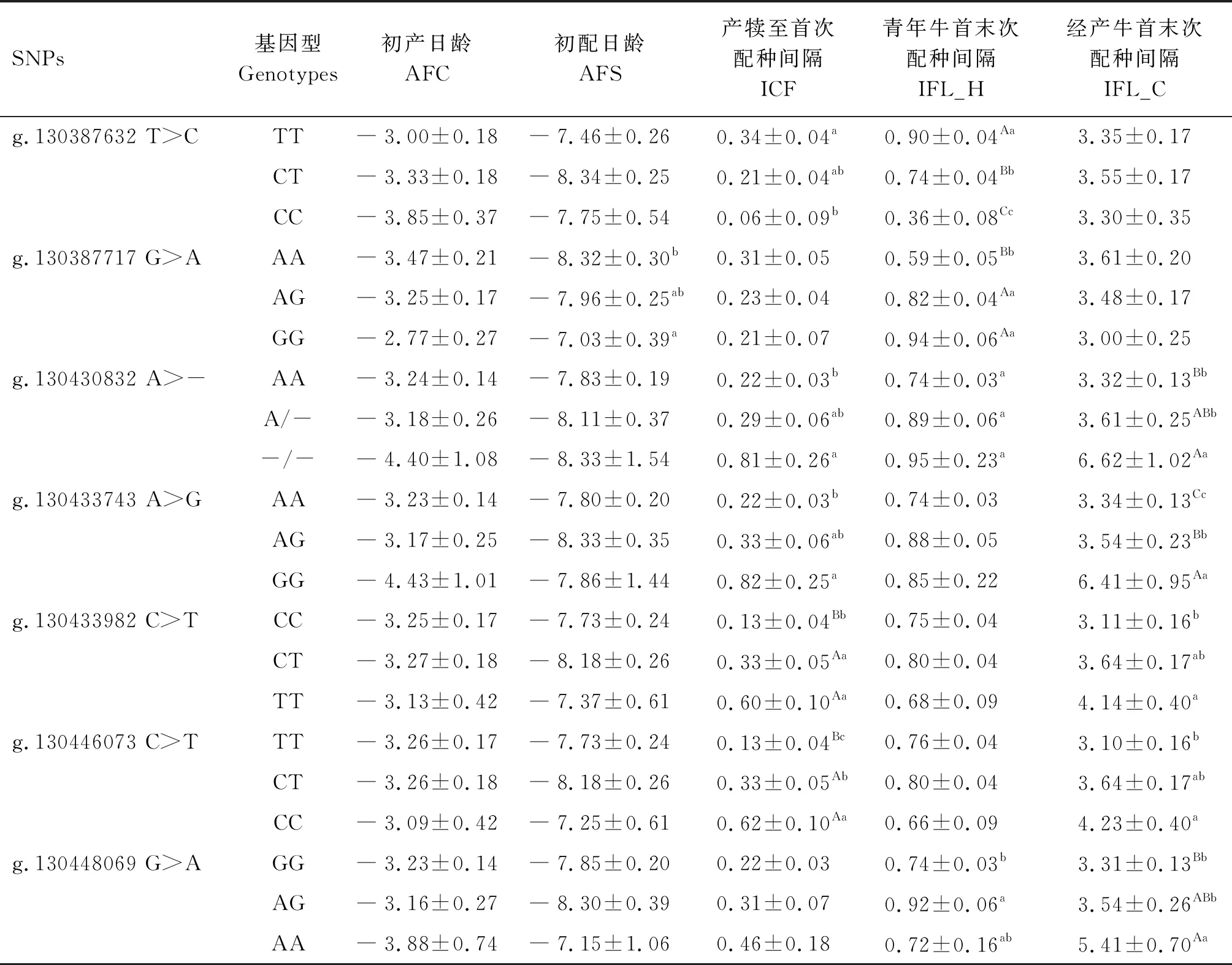
表5 PIK3CB基因与繁殖性状关联分析结果
2.3.2PIK3CB基因SNP与产奶性状关联分析 由表6可知,g.130387632 T>C位点TT基因型个体的PP极显著高于CC基因型(P<0.01),CT基因型个体显著高于CC基因型(P<0.05);g.130387717 G>A位点AA基因型个体MY极显著高于另外2种基因型(P<0.01),PY显著高于另外2种基因型(P<0.05),PP极显著低于另外2种基因型(P<0.01),FY极显著高于AG基因型(P<0.01);g.130430832 A>-位点AA基因型个体SCS极显著高于A/-基因型,显著高于-/-基因型(P<0.05),其MY、PY和FY均显著低于A/-基因型(P<0.05);g.130433743 A>G位点AA基因型个体SCS极显著高于另外2种基因型(P<0.01),MY、PY和FY显著低于AG基因型(P<0.05);g.130433982 C>T位点CC基因型个体MY、PY和FY极显著低于CT基因型(P<0.01),MY和PY显著低于TT型(P<0.05),PP、FP和SCS显著高于TT基因型(P<0.05);g.130446073 C>T位点TT基因型个体MY、PY和FY极显著低于CT基因型(P<0.01),MY和PY显著低于CC基因型(P<0.05),PP、FP和SCS显著高于CC基因型(P<0.05);g.130448069 G>A位点GG基因型个体FY极显著低于AG基因型(P<0.01),SCS极显著高于AG基因型(P<0.01),显著高于AA基因型(P<0.05),MY和PY显著低于AG基因型(P<0.05)。
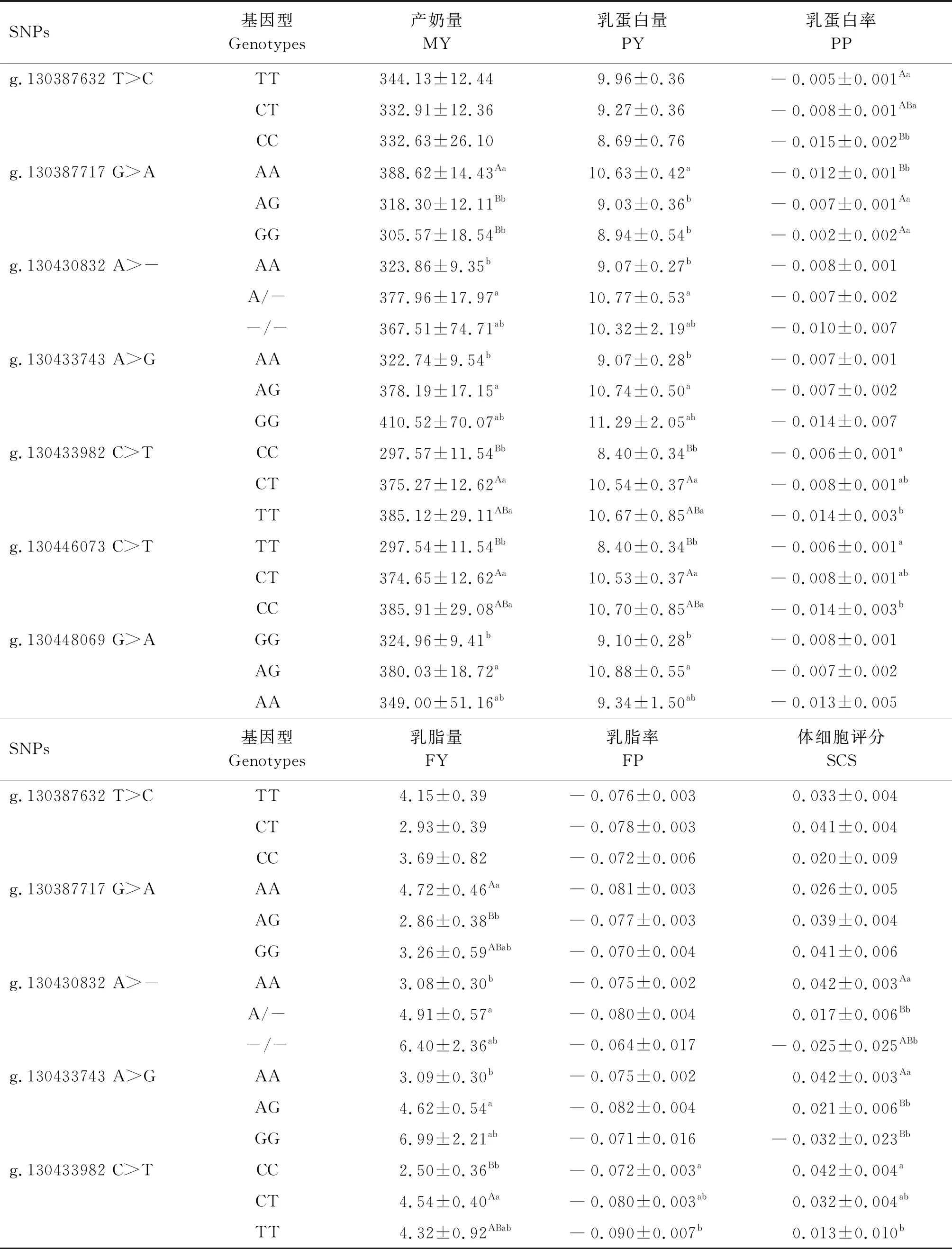
表6 PIK3CB基因SNPs与产奶性状关联分析结果

续表
2.4 PIK3CB基因单倍型组合与繁殖和产奶性状关联分析
2.4.1 单倍型构建 由图3可知,PIK3CB基因中7个SNPs的连锁不平衡分析显示,g.130387717 G>A、g.130430832 A>-、g.130433743 A>G、g.130433982 C>T、g.130446073 C>T和g.130448069 G>A共6个SNPs紧密连锁,可形成一个单倍型块。该单倍型块共有4种单倍型,各单倍型的基因型频率均>10%;其中,H1为优势单倍型,频率为42.9%。为了准确估计各单倍型组合的总体平均值,本研究以个体百分比>5%为标准,筛选出了7种单倍型组合(即H1H1、H1H2、H1H3、H1H4、H2H2、H2H3和H2H4)与繁殖和产奶性状进行关联分析。

方块颜色表示连锁程度,颜色越深,连锁程度越高;方块中数值为位点间相关性值的百分数,无数值代表两位点完全连锁
2.4.2PIK3CB基因单倍型组合与繁殖性状的关联分析 由表7可知,H2H2组合个体IFL_H极显著低于H1H1、H1H2、H1H3、H1H4和H2H4组合(P<0.01);H2H3组合个体IFL_H极显著低于H1H1组合(P<0.01),显著低于H1H3和H1H4组合(P<0.05);H2H4组合个体AFS显著低于H1H1组合(P<0.05);各单倍型组合间ICF无显著差异(P>0.05)。

表7 PIK3CB基因单倍型组合与繁殖性状关联分析结果
2.4.3PIK3CB基因单倍型组合与产奶性状的关联分析 由表8可知,H2H3组合个体MY极显著高于H1H1和H1H2组合(P<0.01),PY和FY极显著高于H1H2组合(P<0.01),PP极显著低于H1H1组合(P<0.01);H1H4组合个体PY和FY极显著高于H1H2组合(P<0.01),MY显著高于H1H2组合(P<0.05),SCS显著低于H1H2组合(P<0.05);H2H2组合个体PP极显著低于H1H1组合(P<0.01);H2H4组合个体FY显著高于H1H2组合(P<0.05)。
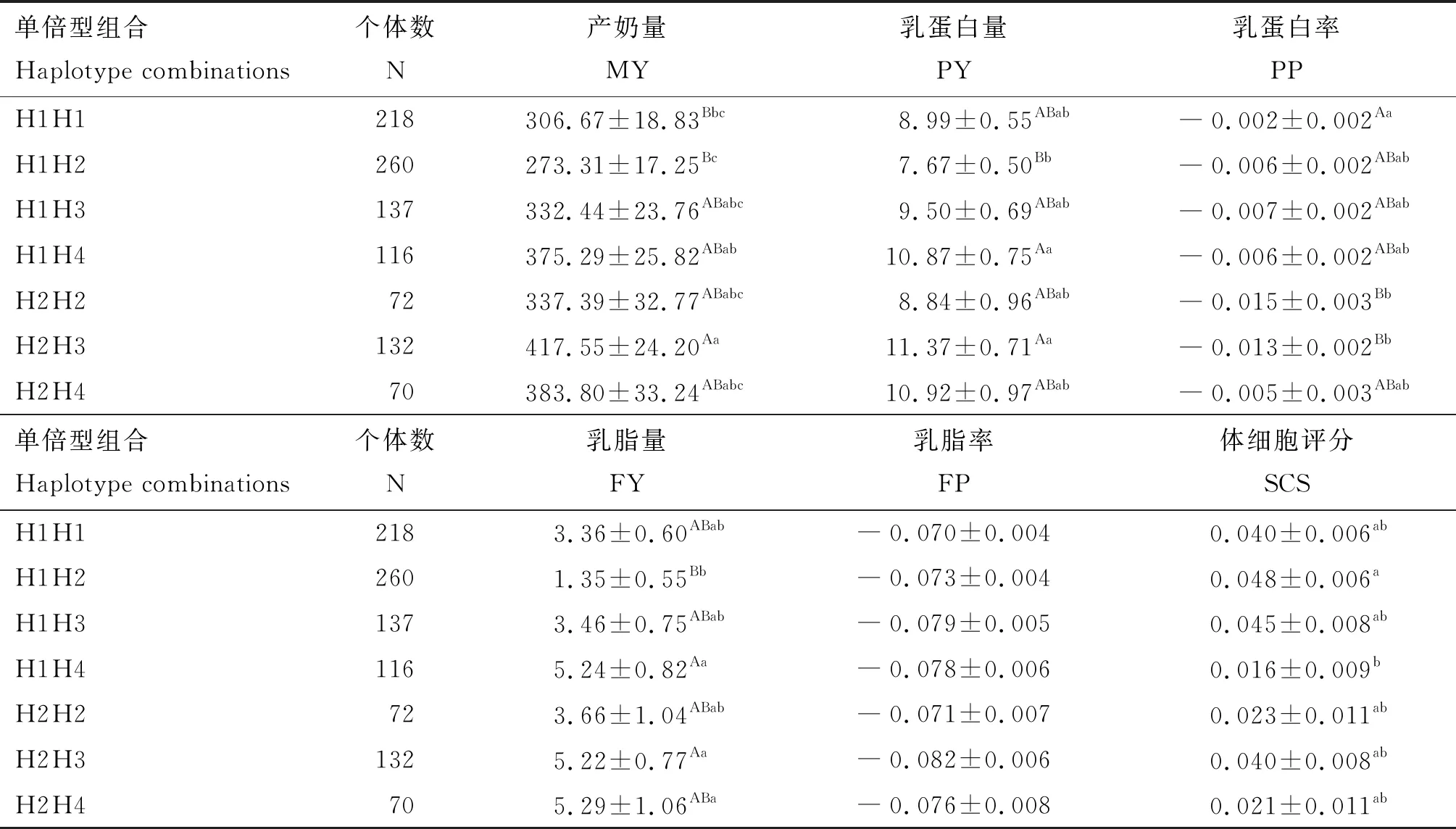
表8 PIK3CB基因单倍型组合与产奶性状关联分析结果
3 讨 论
通过混池测序法,本研究鉴定到PIK3CB基因在中国荷斯坦牛中共有17个SNPs,其中15个SNPs位于内含子,2个SNPs位于外显子,表明该基因内含子区存在丰富的遗传变异。为有效进行KASP分型,筛选多态性较高的位点;同时考虑到对表型的影响,优先保留位于外显子或有特殊变异,即缺失和可变剪接的位点;此外,考虑到连锁不平衡现象,相邻200 bp内只保留1个SNP,最后筛选得到7个SNPs进行基因分型,并与繁殖和产奶性状进行关联分析,发现7个SNPs与目标性状具有显著或极显著的相关性。其中,g.130433743 A>G位点与ICF、IFL_C、MY、PY、FY和SCS具有显著或极显著的相关性,且AA基因型个体ICF、IFL_C、MY、PY和FY最低,而SCS最高。本研究所关注的5个繁殖性状的EBV越小代表繁殖性能越好,配种时间越短,这将减少奶牛养殖场饲养管理成本[20]。对于产奶性状,除SCS外,其余6个性状的EBV越高,代表个体产奶性能较好。因此,g.130433743 A>G位点的AA基因型个体具有较好的繁殖性能,而产奶性能较差。该位点为外显子区域的同义突变,同义突变可以影响共翻译折叠的时间,从而改变基因的功能[21];或通过改变mRNA局部茎环结构,影响蛋白质的翻译过程,使得不同基因型的表型存在差异[22]。因此,该位点可能通过改变mRNA的二级结构影响PIK3CB基因的表达,进而影响表型。g.130448069 G>A位点处于可变剪接区域,与IFL_H、IFL_C、MY、PY、FY和SCS性状均呈显著或极显著相关,且GG基因型个体的IFL_C、MY、PY和FY性状最低,SCS性状最高,表明该基因型个体繁殖性能较好,而产奶性能较差。可变剪接能使单个基因产生多个mRNA异构体和蛋白质亚型[23-24],可能产生异常转录本和蛋白质[24]。推测g.130448069 G>A位点的不同基因型可能产生不同的转录本,从而对繁殖和产奶性状产生影响。针对该位点对PIK3CB基因进行转录组分析,有望进一步揭示导致表型差异的分子机制。
除g.130387717 G>A位点外,其他位点不同基因型间繁殖和产奶性状的变化趋势表现出颉颃效应。但在g.130387717 G>A位点上,AA基因型个体AFS和IFL_H性状最低,MY、PY和FY性状最高,即AA基因型个体的繁殖和产奶性状均表现良好,可作为中国荷斯坦牛繁殖和产奶性状的候选位点重点关注,该位点位于内含子区域。通常认为内含子不参与蛋白质的翻译过程而难以对表型产生影响,但内含子区域的变异也可以影响基因的表达,如PIK3CB基因内含子rs361072位点G等位基因能增加GATA结合位点从而促进转录[25],该位点与血清胰岛素样生长因子1水平和寿命有关[26]。在文惠等[27]对富亮氨酸重复序列G蛋白偶联受体5(LGR5)基因的研究中,同样有3个内含子SNPs与中国荷斯坦牛副乳头的发生存在关系。本研究中,PIK3CB基因内含子SNP位点对繁殖和产奶性状的影响可能与内含子区突变的调控修饰有关,可以通过分子生物学试验进一步验证。
动物基因对表型的影响可能受到多个突变位点的共同作用[28]。研究发现,一个单倍型上的多个突变会对性状产生更大的协同影响[29]。PIK3CB基因的g.130387717 G>A、g.130430832 A>-、g.130433743 A>G、g.130433982 C>T、g.130446073 C>T和g.130448069 G>A共6个SNPs紧密连锁形成一个单倍型块。以产奶性状为例,形成单倍型块的6个SNPs位点均与MY、PY和FY性状呈显著或极显著相关,且单倍型块与MY、PY和FY性状均呈极显著相关。其中,H1H2组合个体的3个性状最低,而H1H2正是g.130430832 A>-、g.130433743 A>G、g.130433982 C>T、g.130446073 C>T和g.130448069 G>A位点的最低基因型组合,且不包含g.130387717 G>A位点的最高基因型AA;H1H4、H2H3和H2H4组合个体均排在3个性状的前3位,H1H4组合不包含各位点最低的基因型,H2H3和H2H4组合在包含g.130387717 G>A位点的最高基因型AA的同时,伴有多个位点的杂合基因型,使得组合EBV较高;此外,H2H3组合个体AFS和IFL_H性状较低,H2H4组合个体AFS性状最低,因此H2H3和H2H4组合的繁殖和产奶性状均表现良好,与关于g.130387717 G>A位点AA基因型的结果一致。本研究的单位点关联分析和单倍型组合关联分析可以相互佐证,H2H3和H2H4组合作为繁殖和产奶性状的优势单倍型组合,可为奶牛的平衡育种提供理论依据。
4 结 论
本研究在PIK3CB基因上共检测到17个SNPs,其中7个SNPs与繁殖和产奶性状具有显著或极显著关联,g.130433743 A>G位点的AA基因型和g.130448069 G>A位点的GG基因型个体具有较好的繁殖性能,而其产奶性能相对较差;g.130387717 G>A位点的AA基因型个体繁殖和产奶性状均表现良好,上述3个位点可作为奶牛选育中繁殖和产奶性状的潜在分子标记。PIK3CB基因单倍型组合中,H2H3和H2H4组合个体的繁殖和产奶性状均表现较好,为优势单倍型组合。本研究揭示了PIK3CB基因在中国荷斯坦牛群体中的多态性,并重点筛查了该基因中与繁殖和产奶性状相关的多态位点,可为使用分子标记辅助选择改善中国荷斯坦牛的繁殖和产奶性状提供参考。

