藜麦WRKY基因的进化与多胁迫条件下的转录应答
侯丽媛,贾举庆,姜晓东,王育川,赵菁,陈禺怀,黄胜雄,吴慎杰*,董艳辉*
(1.山西农业大学生命科学学院,山西 太原 030031;2.山西农业大学农学院,山西 晋中 030801;3.华中师范大学数学与统计学院,湖北 武汉 430079;4.合肥工业大学食品与生物工程学院,安徽 合肥 230009)
藜麦(Chenopodium quinoa),苋科藜亚科藜属,为异源四倍体真双子叶植物。全基因组测序表明藜麦起源于拥有A基因组的苍白茎藜(Chenopodium pallidicaule)和B基因组的瑞典藜(Chenopodium suecicum),由这两个祖先二倍体杂交而来[1]。藜麦在安第斯山脉区域的玻利维亚、秘鲁、厄瓜多尔等国家有5000~7000年的种植历史[1],富含膳食纤维、矿物质、各种维生素和人体必需的氨基酸,特别是赖氨酸和蛋氨酸[2-4],被称为“母亲粮食”,更被联合国粮食及农业组织(Food and Agriculture Organization of the United Nations,FAO)评选为21世纪与食品安全相关的最有前景的作物之一,抗寒抗旱,有着比其他粮食作物更强地适应恶劣气候和贫瘠土壤的能力[5]。
WRKY是植物转录因子中成员数目庞大的家族之一,特征在于其DNA结合域:WRKY结构域[6-8]。WRKY结构域包括一个非常保守的七肽WRKYGQK和位于其C端的C2H2或C2HC类型的锌指结构。基于WRKY结构域数目和锌指结构类型,WRKY家族成员可以被划分为3组[6-9]:Ⅰ组成员含有2个WRKY结构域,Ⅱ和Ⅲ组成员分别只有1个WRKY结构域。Ⅰ组与Ⅱ组成员都具有C2H2类型的锌指结构;而Ⅲ组成员的锌指结构为C2HC。基于WRKY结构域中氨基酸保守位点的不同,Ⅱ组成员进一步划分为5个亚组:Ⅱ-a、Ⅱ-b、Ⅱ-c、Ⅱ-d和Ⅱ-e[9]。
WRKY转录因子广泛参与了植物对各种生物和非生物胁迫的应答反应,是一种重要的抗逆调节基因[10-12]。鉴于其重要性,目前该基因在抗逆方面的功能研究在粮食油料作物和蔬菜水果类园艺植物中被广泛开展。基于干旱转录组数据,从玉米(Zea mays)中鉴定得到一个受干旱诱导表达的WRKY基因,ZmWRKY106[13]。ZmWRKY106的表达水平受干旱、高温和外源脱落酸(abscisic acid,ABA)的诱导。异源过表达该基因的拟南芥(Arabidopsis thaliana)植株表现出对干旱和高温胁迫的耐受性增强[13]。大豆(Glycine max)的GmWRKY12在正常条件下,其表达维持在低水平;干旱和盐胁迫条件下,显著地被诱导高表达。干旱和盐胁迫下,GmWRKY12转基因大豆植株表现出耐受性增强;同时转基因大豆中脯氨酸(proline,Pro)含量大幅增加[14]。过表达番茄(Lycopersicon esculentum)SlWRKY8的转基因番茄植株显示出对丁香假单胞杆菌番茄治病变种(Pseudomonas syringaepv.tomato,Pst)Pst.DC3000的抗性显著增强。抗病基因SlPR1a1和SlPR7在转基因植株中的表达水平显著上调。同时,转基因番茄植株表现出对干旱和盐的耐受性[15]。小麦(Triticum aestivum)的TaWRKY46在聚乙二醇(polythylene glycol,PEG)处理下上调表达[16]。过表达TaWRKY46的拟南芥植株在含有甘露醇的1/2 MS培养基上具有更快的萌发速度和更长的根长,表现出更强的耐渗透胁迫能力[16]。
藜麦基因组测序研究完成时间较晚[1],WRKY基因的功能研究相对很少。高温胁迫条件下,部分藜麦WRKY基因表现出显著差异的表达水平[17]。部分藜麦WRKY基因被推测可能是cqu-miR156b的靶基因,通过小分子RNA的调控,参与了植物的发育和抗逆调控[18]。本研究利用生物信息学方法,在全基因组水平对藜麦WRKY基因家族进行系统地研究和分析,包括家族成员分类、WRKY结构域的氨基酸保守位点比较、系统进化树构建、不同胁迫条件下WRKY基因表达模式解析,以及WRKY家族成员数目扩增的动力分析。本研究结果可为藜麦进一步的遗传改良提供基础资料。
1 材料与方法
1.1 藜麦和祖先二倍体WRKY基因的挖掘
从藜麦基因组数据库(www.cbrc.kaust.edu.sa/chenopodiumdb)下载藜麦、藜麦祖先二倍体苍白茎藜和瑞典藜的基因组和全部编码蛋白序列。利用HMMER 3.0软件[19],基于从Pfam数据库[20](pfam.xfam.org)下载的WRKY结构域文件(编号:PF03106),对藜麦基因组编码的蛋白序列进行WRKY蛋白序列的比对。比对得到的WRKY蛋白序列中的WRKY结构域,在InterPro数据库[21](www.ebi.ac.uk/interpro)中进行WRKY结构域的确认,确认后的序列即为准确的藜麦WRKY转录因子。
1.2 藜麦WRKY蛋白序列的保守域组成鉴定
参考已发表物种的WRKY转录因子的研究数据和InterPro数据库[21]WRKY结构域的确认结果,对WRKY结构域,结构域大小和边界进行确定。同时,利用InterPro和SMART(smart.embl-heidelberg.de)数据库,对WRKY结构域以外的其他蛋白保守域进行鉴定。
1.3 藜麦WRKY基因的系统进化树构建
利用MAFFT程序[22]对藜麦WRKY成员的WRKY结构域的蛋白序列进行多序列比对,默认参数。基于比对结果,通过IQ-TREE 1.6.12程序[23]构建系统进化树,最适合建树模型由内置的ModelFinder[24]检测设定,自展值设置为1000。
1.4 藜麦WRKY基因的扩增动力分析
从葡萄(Vitis vinifera)基因组数据库(www.genoscope.cns.fr/externe/GenomeBrowser/Vitis/)下载葡萄的基因组和全部编码蛋白序列。利用本地BLASTP程序构建了藜麦基因组编码所有蛋白的本地数据库,分别与葡萄(V.vinifera)、祖先二倍体苍白茎藜(C.pallidicaule)和瑞典藜(C.suecicum)进行全基因组范围的蛋白序列比对,E值≤1E-05。利用JCVI程序[25],提取物种间的同源基因组模块。将位于同源基因组模块的WRKY基因利用Circos软件[26]进行可视化,并分析藜麦WRKY基因的扩增动力。
1.5 藜麦WRKY基因在不同组织和胁迫条件下的表达谱
从NCBI的Bioproject下载藜麦转录组数据:1)叶、茎和花簇的转录组(编号:PRJNA394651);2)干旱、高温、盐和低磷胁迫条件下藜麦幼苗的转录组(编号:PRJNA306026);3)花生(Arachis hypogaea)褪绿扇形斑病毒(groundnut chlorotic fan-spot virus,GCFSV)侵染藜麦幼苗的转录组(编号:PRJNA349075)。
1.6 数据统计与分析
采用R包Pheatmap,对藜麦WRKY基因在不同组织叶、茎、花簇中的表达情况,以及干旱、高温、盐、低磷胁迫和GCFSV侵染下转录组数据表达变化进行分析,采用R包Pheatmap软件,以Euclidean距离衡量标准和Median聚类方法作图。
2 结果与分析
2.1 藜麦WRKY转录因子家族成员
通过系统的生物信息学分析,藜麦WRKY家族成员数目最终确定为90个(表1)。依据WRKY基因在藜麦染色体上的相对位置,依次对藜麦WRKY家族成员进行编号。详细的藜麦WRKY家族成员信息见表1,包括数据库编号、染色体定位、蛋白序列长度、WRKY结构域数目和成员分组。藜麦WRKY家族成员蛋白序列的平均长度为367 aa,其中蛋白序列最长和最短的成员分别为CqWRKY38(1178 aa)和CqWRKY07(84 aa)。
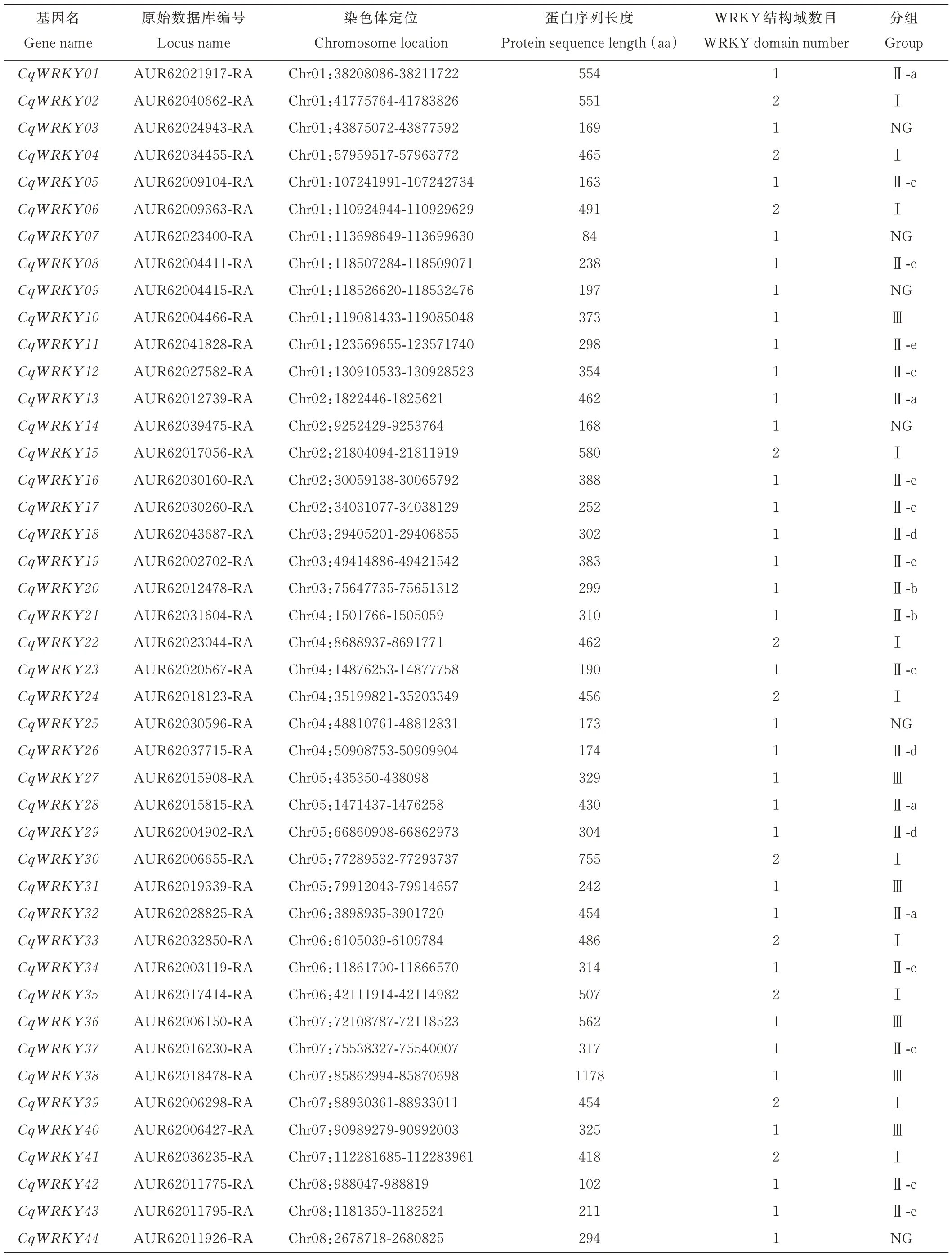
表1 藜麦WRKY家族成员Table 1 The WRKY family members in C.quinoa
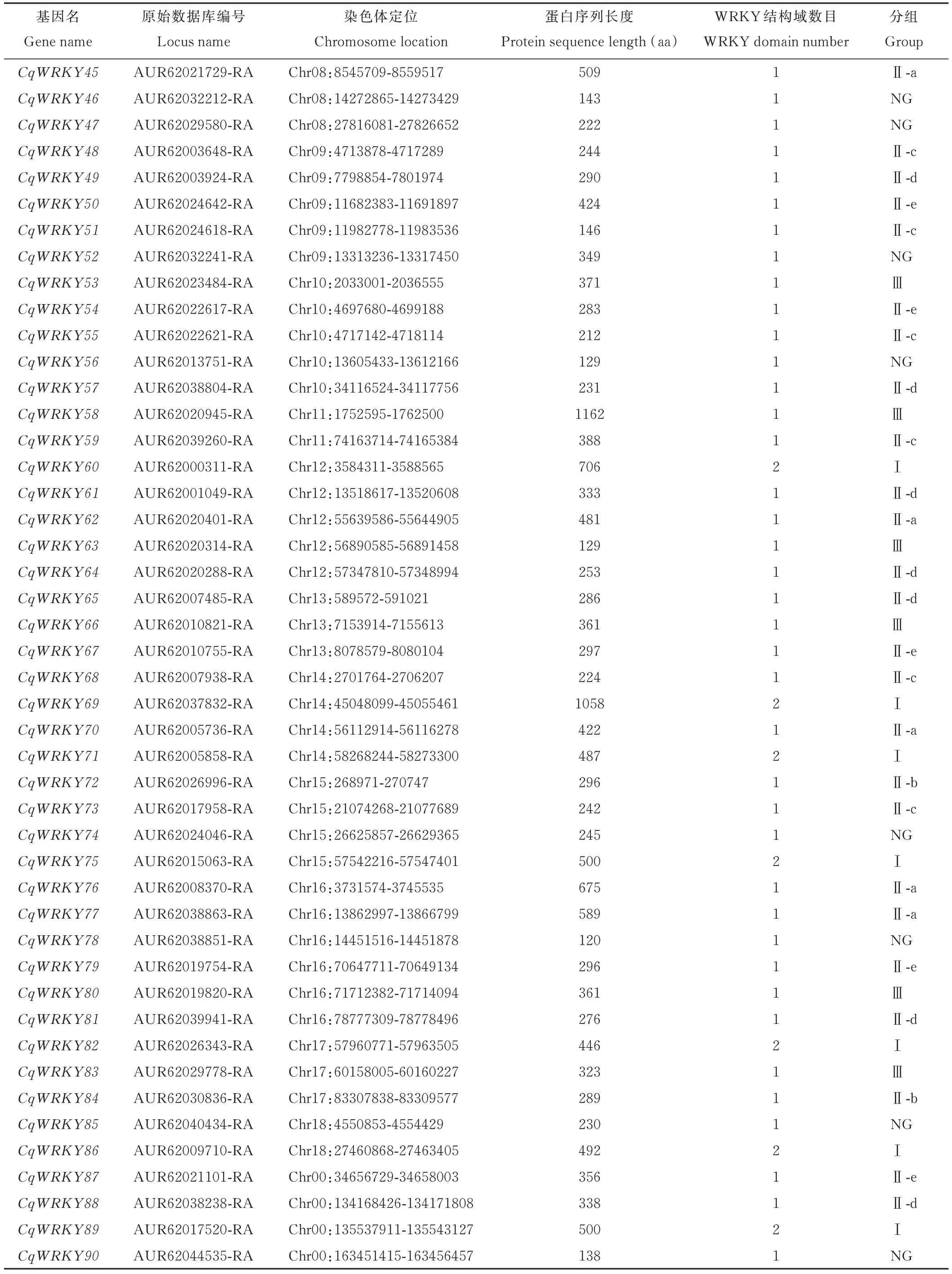
续表Continued Table
藜麦基因组每条染色体上都存在WRKY基因,定位信息显示基因主要分布在染色体的两端。藜麦WRKY基因染色体定位呈现出染色体特异的分布;其中1号染色体上分布多达12个WRKY基因;而3、17、11、18号染色体上分别只有3、3、2、2个WRKY基因(图1)。
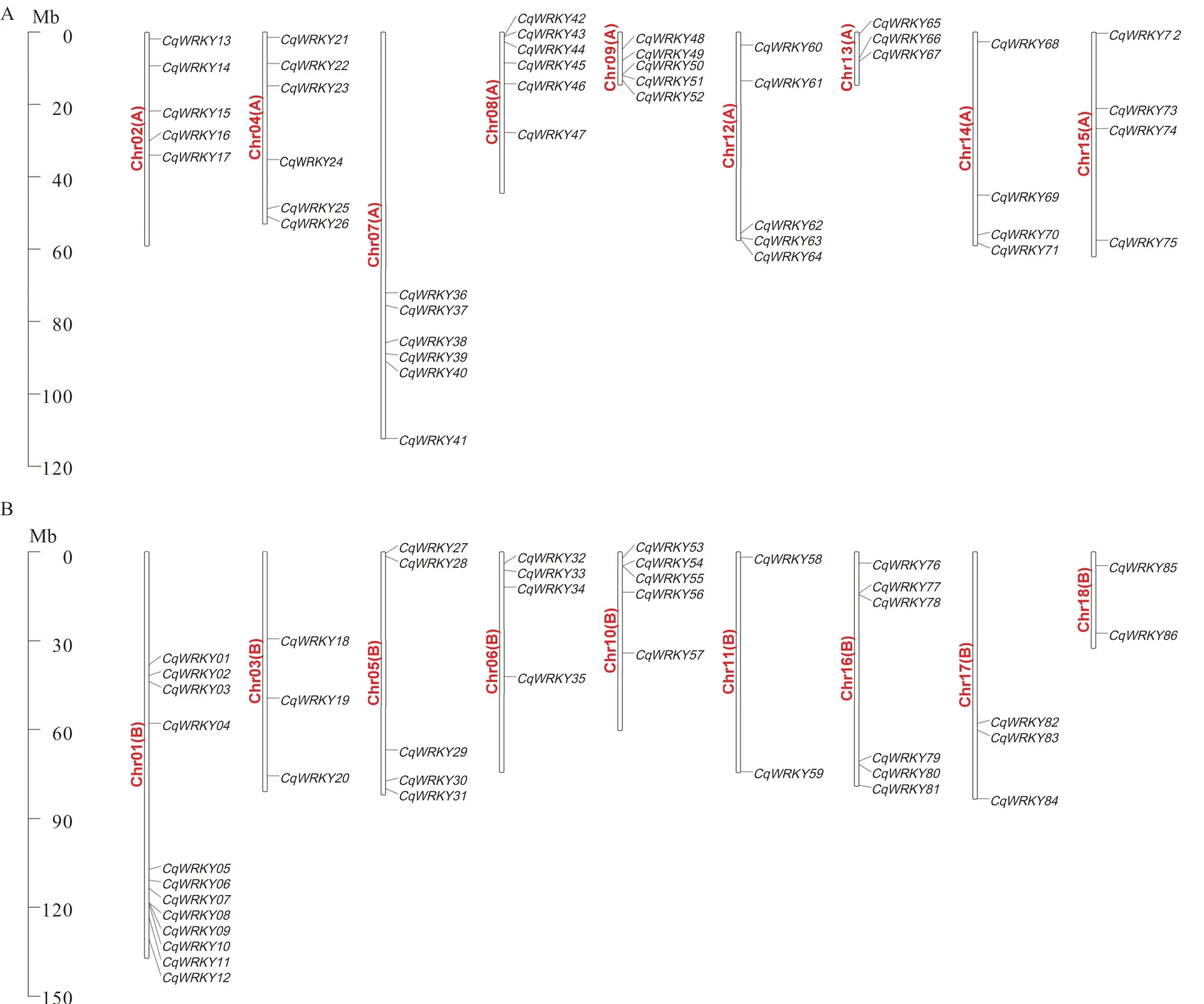
图1 藜麦WRKY基因的染色体定位Fig.1 The chromosomal location of C.quinoa WRKY genes
2.2 藜麦WRKY家族成员的分组
基于WRKY保守域的数目和锌指结构类型,将90个藜麦WRKY转录因子划分为3个大类群:其中18个含有2个WRKY结构域,且锌指结构类型为C2H2的成员被划分为Ⅰ组;12个含有1个WRKY结构域,且锌指结构类型为C2HC的成员划分为Ⅲ组;Ⅱ组含有46个WRKY转录因子,含有1个WRKY结构域,且锌指结构为C2H2类型。此外,还有14个WRKY转录因子因WRKYGQK短肽的缺失,以及锌指结构变异较大而未划分到任何分组(表1)。
进一步提取了Ⅱ组46个成员的WRKY结构域的蛋白序列。基于多序列比对结果中氨基酸保守位点的差异(图2),Ⅱ组成员进一步被划分为5个亚组:Ⅱ-a(9个),Ⅱ-b(4个),Ⅱ-c(13个),Ⅱ-d(10个)和Ⅱ-e(10个)(表1和图2)。分别在藜麦WRKY转录因 子Ⅱ-c组CqWRKY05和CqWRKY23,Ⅱ-e组CqWRKY67和CqWRKY79的蛋白序列中,发现了变异的WRKYGQK短肽(图2)。WRKYGQK的变异短肽为WRKYGKK(CqWRKY05、CqWRKY23)和WRKYGEK(CqWRKY67、CqWRKY79)。CqWRKY19蛋白序列中发生了WRKYGQK短肽的缺失。此外,CqWRKY45(Ⅱ-a)、CqWRKY12(Ⅱ-c)、CqWRKY42(Ⅱ-c)、CqWRKY51(Ⅱ-c)、CqWRKY65(Ⅱ-d)其锌指结构存在组氨酸位点的缺失(图2)。

图2 藜麦Ⅱ组WRKY成员的WRKY结构域蛋白序列比对Fig.2 The alignment of WRKY domain peptide sequences in groupⅡC.quinoa WRKYs
2.3 藜麦WRKY基因的系统进化树
本研究提取了能够分组的76个藜麦WRKY转录因子中的84个完整的WRKY结构域蛋白序列进行比对分析,继而构建了藜麦WRKY基因的系统进化树(图3)。Ⅰ组成员同一蛋白序列中的两个WRKY结构域,依据其在序列中的相对位置,分别命名为N端和C端结构域(图3)。
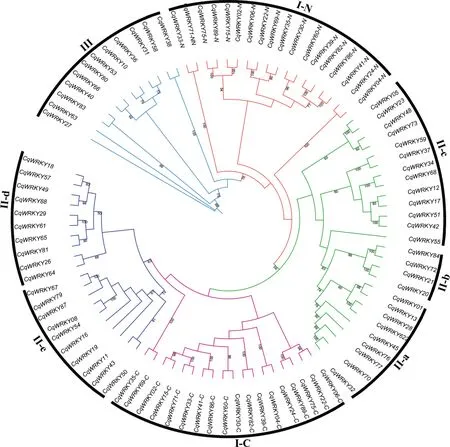
图3 藜麦WRKY基因的系统进化树Fig.3 The phylogentic tree of WRKY genes in C.quinoa
进化树中聚类的大分支非常明显,来自不同组别的藜麦WRKY基因都聚成了组别特异的分支(图3),包括分支Ⅰ-N(Ⅰ组N端的WRKY结构域)、分支Ⅰ-C(Ⅰ组C端的WRKY结构域)、分支Ⅲ(Ⅲ组成员),这3个分支都属于单系群。Ⅱ组的成员主要呈现出两个分支聚类,一个分支包括了Ⅱ-a、Ⅱ-b和Ⅱ-c组的成员,另外的则由Ⅱ-d和Ⅱ-e构成。整个进化树中,来自不同组别的分支进一步聚成5个主要的分支,包括group 1(Ⅱ-d和Ⅱ-e)、group 2(Ⅰ-C)、group 3(Ⅱ-a、Ⅱ-b和Ⅱ-c)、group 4(Ⅰ-N)和group 5(Ⅲ)。进化树中,藜麦WRKY成员的聚类结果与之前基于结构域特征的分组完全一样,进一步支持了本研究的准确性。
2.4 藜麦WRKY转录因子蛋白序列的保守域组成
基于InterPro数据库,对藜麦WRKY的蛋白序列的保守域组成进行分析。在17条WRKY蛋白序列中,发现了除WRKY结构域以外的其他保守域的存在(图4)。这17条WRKY蛋白序列主要分布在Ⅱ-d(10条)。另外7条序列分别分布在Ⅰ组(1条)、Ⅱ-a(2条)、Ⅱ-c(2条)和Ⅲ组(2条)。

图4 藜麦WRKY蛋白序列中保守域的分布Fig.4 The distribution of conserved domains in C.quinoa WRKY protein sequences
保守域分析结果表明:分组间各自存在着特异的保守域组成,且同一分组中的WRKY成员拥有一致的保守域组成(图4)。相似的氨基酸保守域组成暗示着可能存在相近的基因功能。Ⅲ组成员的保守域为WRKY、NBARC和LRR_8;Ⅱ-a成员的保守域为WRKY和UQ_con;Ⅰ组的CqWRKY69的保守域为WRKY、DUF4371和Dimer_Tnp_hAT。此外,Ⅱ-c和Ⅱ-d在进化关系上属于相近的组,两个组成员的保守域组成均为WRKY和Plant_zn_clust。
2.5 藜麦WRKY基因的扩增动力
藜麦为异源四倍体植物,来源于分别含有A基因组的苍白茎藜和B基因组的瑞典藜两个祖先二倍体的杂交[1]。如果两个亲本的所有遗传物质都被继承,且祖先物种基因组中WRKY基因在后续进化中没有丢失,那么祖先二倍体A基因组的苍白茎藜和藜麦、B基因组的瑞典藜和藜麦之间的WRKY同源基因对基本上应均为1对应2的同源关系:即苍白茎藜和瑞典藜的1个WRKY基因分别在藜麦中能找到2个同源的WRKY基因(在本研究的后续表示中,将不同对应的同源关系用Vs符号表示)。
基于藜麦WRKY基因的鉴定方法,在祖先二倍体苍白茎藜和瑞典藜的基因组序列中分别鉴定得到46和45个WRKY基因。运用本地BLASTP程序,对全基因组编码蛋白序列进行两两比对,并用JCVI程序分别进行了藜麦基因组与两个祖先二倍体苍白茎藜和瑞典藜基因组之间同源基因组模块和模块上面同源WRKY基因的鉴定。结果显示:苍白茎藜和藜麦之间,瑞典藜和藜麦之间存在1 Vs 2同源关系的WRKY基因对分别有52和32对(图5)。此外,两个祖先二倍体苍白茎藜、瑞典藜和藜麦的同源WRKY基因之间还存在着1 Vs 1同源关系的WRKY基因分别有10和13对(图5),2 Vs 1同源关系的分别有5和7对(图5)。这些复杂的WRKY同源基因对进一步说明藜麦进化过程中存在着WRKY基因的丢失与扩增。
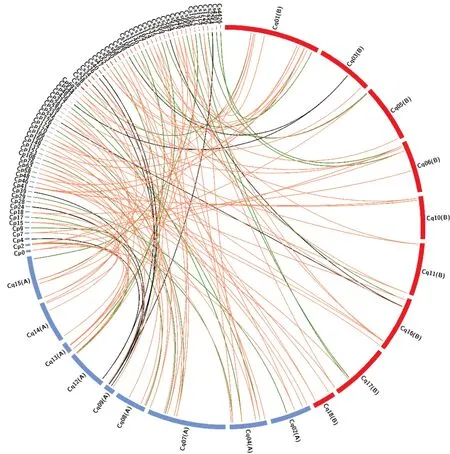
图5 苍白茎藜、瑞典藜和藜麦之间的WRKY基因的保留与丢失Fig.5 Reservation and loss of the WRKY genes between C.pallidicaule,C.suecicum and C.quinoa
本研究将苍白茎藜和藜麦、瑞典藜和藜麦之间1 Vs 2同源关系的WRKY基因对进行展示(图6)。藜麦中严格意义上分别遗传自苍白茎藜和瑞典藜的1 Vs 2同源关系的WRKY基因对有20对,这些来自祖先种的WRKY基因在藜麦杂交形成后得到了保留(图6A)。此外,祖先二倍体和藜麦之间存在一些特殊的1 Vs 2同源关系的WRKY基因对,一共有44对(图6B)。这些祖先二倍体的WRKY基因在藜麦中对应的2个同源WRKY基因只落在了A或B亚基因组。表明这些藜麦WRKY基因所在的基因组区域发生了染色体的重组。
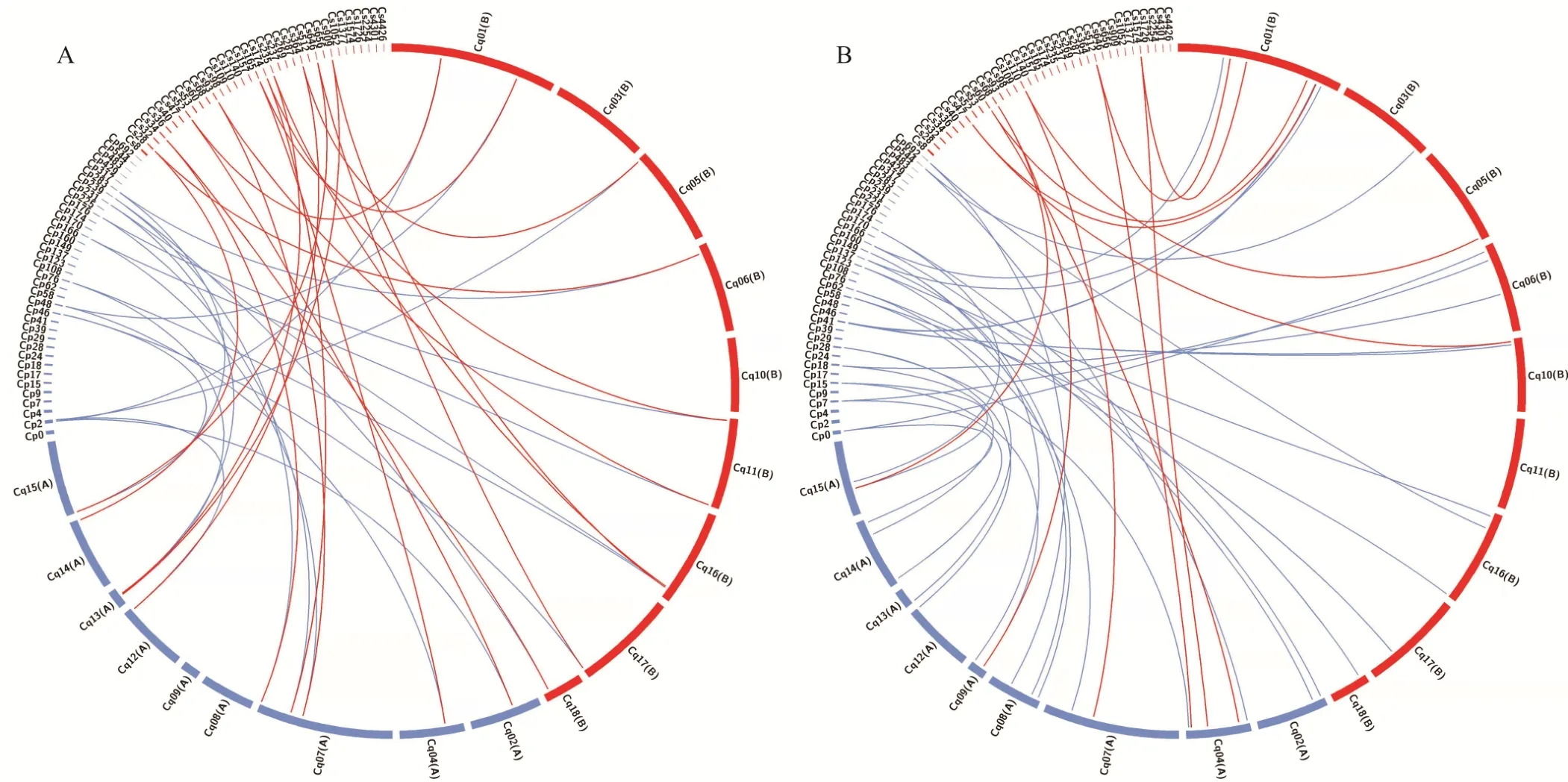
图6 苍白茎藜、瑞典藜和藜麦之间的WRKY基因的保留与丢失Fig.6 Reservation and loss of the WRKY genes between C.pallidicaule,C.suecicum and C.quinoa
2.6 藜麦WRKY基因在多个胁迫条件下的表达谱
基于干旱、高温、盐和低磷胁迫,以及GCFSV侵染下藜麦幼苗的RNA-seq数据,解析了藜麦WRKY基因的表达谱(图7)。在干旱、高温、盐和低磷等不同胁迫条件下,25个WRKY基因明显地被诱导或抑制表达(图中呈现的是在至少一个胁迫条件下的基因表达值≥5)(图7A)。CqWRKY76和CqWRKY83在对照组和胁迫条件下,都显现比较高的表达水平(最低基因表达值22.53)。盐和低磷胁迫下,CqWRKY76表达水平大约只有对照组的50%;干旱条件下,CqWRKY83表达水平下降到对照组的50%,低磷胁迫下上升了1.6倍以上(74.01/46.20)(图7A)。此外,同样在对照组和胁迫条件下呈现出较高表达水平的CqWRKY26和CqWRKY40,分别在干旱和盐胁迫、干旱和高温胁迫条件下的表达水平与对照组相比下调近50%。CqWRKY72和CqWRKY84呈现出胁迫特异性的诱导表达,在高温条件下,表达上调了2~3倍(图7A)。此外,CqWRKY05、CqWRKY07、CqWRKY20、CqWRKY21、CqWRKY23和CqWRKY48在干旱和高温条件的表达几乎被完全抑制,呈现出极低的表达水平;CqWRKY21在低磷胁迫下表达却被上调了2倍以上(图7A)。

图7 藜麦WRKY基因在干旱、高温、低磷和盐胁迫和GCFSV侵染下的基因表达谱Fig.7 The CqWRKY genes’expression profiles under the stresses of drought,heat,low P and salt and GCFSV infection
在GCFSV侵染藜麦幼苗条件下,42个WRKY基因表现出明显地被诱导或抑制表达(基因表达值≥10)(图7B)。在干旱、高温、低磷和盐胁迫中显著上调或下调表达的基因中,CqWRKY05、CqWRKY20、CqWRKY21、CqWRKY23同样在GCFSV侵染下呈现出强诱导上调表达趋势(图7B)。相较对照,CqWRKY20、CqWRKY21表达分别被上调了10和20倍。推测这2个WRKY基因可能属于藜麦中的重要调控基因,广泛参与了藜麦生物与非生物胁迫(干旱、高温和低磷)下的调控应答。此外,CqWRKY36、CqWRKY42、CqWRKY47和CqWRKY78在GCFSV侵染下,也呈现出强诱导上调表达趋势(图7B)。
3 讨论
藜麦在进化过程中发生过一次祖先二倍体种间杂交所带来的全基因组倍增事件[1],藜麦基因组保留了大量祖先种基因组的同源序列。两个祖先二倍体苍白茎藜和瑞典藜的基因组序列在藜麦基因组中分别存在着14.6%和46.5%的同源DNA序列[1]。此外,作为很早分化出来的植物,葡萄基因组中只存在着一次大多数真双子叶植物所共有的全基因组倍增,γ倍增事件[27]。然而,葡萄基因组倍增后,其发生了大规模的片段丢失和重组。虽然都只经历了一次全基因组倍增,但与葡萄相比,藜麦的基因组进化更为保守。
为了比较WRKY基因在上述两物种间的进化差异,在藜麦和葡萄基因组间进行了同源基因组模块的鉴定,对定位其中的WRKY同源基因对进行了统计。藜麦和葡萄基因组中分别有68和41个WRKY基因可以定位在两物种之间的同源基因组模块中。将上述同源模块中的WRKY同源基因对进行统计,发现葡萄和藜麦WRKY基因之间存在着1 Vs 1、1 Vs 2、2 Vs 1和多Vs多同源关系的WRKY基因对分别有5、44、2和30对(图8)。以上数据表明:基因组倍增以后,藜麦基因组保留了相当数目的WRKY同源基因。相反,在葡萄基因组中,大量倍增基因被丢失。

图8 以葡萄为参照的藜麦WRKY基因的倍增Fig.8 Conservation of the WRKY genes in C.quinoa based on the comparisons to those in V.vinifera
干旱、高温、盐、低磷胁迫及GCFSV侵染藜麦幼苗的RNA-seq数据揭示了受到这些胁迫诱导而显著上、下调表达的WRKY基因(图7)。此外,对藜麦WRKY基因在叶片、花和茎的表达也进行了分析。44个WRKY基因在上述组织中存在一定的表达水平(至少一个组织中表达值≥10)(图9)。13个WRKY基因,包括CqWRKY26、CqWRKY39、CqWRKY40、CqWRKY41、CqWRKY64、CqWRKY66、CqWRKY72、CqWRKY76、CqWRKY80、CqWRKY82、CqWRKY83、CqWRKY84和CqWRKY86在这3个组织中恒定地高水平表达(表达值>40)(图9)。这些组成型高表达成员中的6个WRKY基因,包括CqWRKY26、CqWRKY40、CqWRKY72、CqWRKY76、CqWRKY83和CqWRKY84,在干旱、高温、盐、低磷胁迫或GCFSV侵染下显著诱导或抑制表达(图7)。CqWRKY72在拟南芥中的同源基因AtWRKY40被证明是ABA信号途径[28](调控种子萌发、根长和幼苗的发育)和包括白粉病侵染[29]、高盐[30]、渗透胁迫[30]、强光胁迫[31]等逆境应答的转录抑制因子基因。

图9 藜麦WRKY基因在花簇、叶和茎中的表达谱Fig.9 The expression profiles of CqWRKY genes in inflorescence,leaf and stem
此外,部分WRKY基因呈现出组织特异性的表达。CqWRKY07、CqWRKY48、CqWRKY56、CqWRKY68、CqWRKY87在花和叶片中表达水平极低,在茎中高表达(图9);CqWRKY01和CqWRKY13在叶片中相对高表达(图9);CqWRKY43和CqWRKY59在花中相对高表达(图9)。同时,在茎中高表达的CqWRKY07和CqWRKY48的表达水平受到干旱和高温的抑制(图7A),以及GCFSV侵染诱导其显著上调(图7B)。CqWRKY48在拟南芥中的同源基因AtWRKY13的突变体促进了开花[32],却影响了茎秆的正常发育[33],其突变体中大量木质素合成相关基因的表达被抑制。此外,AtWRKY13可以通过正调控PDR8[34]和D-CYSTEINE DESULFHYDRASE[35]编码基因的表达,提高拟南芥对镉的耐受性。上述藜麦WRKY基因在藜麦的逆境应答和生长发育的过程中,推测属于关键的调节基因。
4 结论
基于系统的生物信息学方法,在藜麦基因组中共鉴定得到90个WRKY基因;划分为3组:Ⅰ组(18个)、Ⅱ组(46个)和Ⅲ组(12个),其中Ⅱ组成员进一步被划分为5个亚组:Ⅱ-a(9个),Ⅱ-b(4个),Ⅱ-c(13个),Ⅱ-d(10个)和Ⅱ-e(10个)。藜麦WRKY基因进化树中家族成员的聚类结果和分组完全一致,进一步支持了成员分组的可靠性。藜麦和祖先二倍体苍白茎藜、瑞典藜的同源基因组模块分析表明,藜麦WRKY基因数目增加主要源于全基因组的倍增。干旱、高温、盐、低磷胁迫和GCFSV侵染下,大量WRKY基因的表达水平被显著诱导或抑制,这些WRKY基因很可能参与了藜麦逆境应答的调控。
——紫 苏

