基于实体识别和信息融合的知识图谱研究
——以新冠肺炎疫情为例
刘华玲,孙 毅
(上海对外经贸大学 统计与信息学院,上海 201620)
0 引 言
2020年初,新冠肺炎疫情的突然爆发对中国乃至全球范围内的经济、贸易、环境、医疗等领域产生了巨大的冲击与影响。中国政府及时发现疫情、快速做出响应、制定相关政策计划来应对这一突发性公共医疗事件,最终在较短时间里迅速地取得了对疫情的控制。但全球范围内的情况依然非常严峻。截至2021年7月,全球范围内累计确诊数已超过1.9亿。虽然相较于疫情初期已经略有好转,并且研制出了针对性疫苗,但是随着病毒的变异与部分地区的不利防疫措施,疫情依然存在很高的风险性。
PubMed包含了全世界范围内大量学者、科研机构、医生的研究成果,对医学领域的发展起了至关重要的作用,也是对抗新冠疫情的前沿阵地。网站中伴随着大量无序信息的庞大数据急需寻找有效方法,从中快速发现有用知识。而医疗领域的相关知识具有专业性与特殊性,存在同一个对象拥有多种不同的命名方式或描述方式的情况,这阻碍了医学领域的知识传播与发展。
该文以新冠肺炎疫情为例,使用深度学习与知识图谱技术提取并整合各类相关医学信息,构建新冠肺炎疫情知识图谱,向用户提供一种方便快速地获取知识的方法。帮助研究人员、医疗机构、投资者寻找合适的合作对象或了解最新的研究成果与前沿方向。
1 相关研究
1.1 命名实体识别研究
命名实体识别是自然语言处理中的一项基础任务,用于从非结构化文本数据中提取特定专有名词。传统的规则法[1]需要依赖已有词典和既定表达式,但准确率受限且可扩展性较差。使用统计学习模型如最大熵模型[2]、马尔可夫模型[3]、支持向量机[4]等虽然准确率有所提升,但需要人工提取特征,在专业性较强的医学文本识别领域普遍表现不佳。
近年来,有学者开始尝试从深度学习的角度使用深度神经网络和CRF模型进行实体识别。Suman等[5]使用混合深度神经网络、双向LSTM和CNN对多模态Twitter数据中的实体进行识别。吴俊等[6]使用BERT预训练模型及中文预训练字嵌入向量,融合BiLSTM与CRF对《深度学习500问》书中的专业术语进行抽取。这一类方法解决了人工构造特征的不稳定性,大量研究证实了深度学习在公开的文本数据集中能够取得较好的效果。但是,不同深度学习模型在各个数据集中的效果差异性较大,同时对于非日常使用的文本,这一类方法的效果有待进一步的研究与验证。
在医学领域,实体识别是智慧医疗、智能问答等任务实现的前提。罗凌等[7]以笔画序列为输入对ELMo模型进行改进,学习中文电子病历文本中上下文相关的笔画向量,构建多任务学习神经网络,提升了模型性能。Luo等[8]在BiLSTM-CRF模型中引入Attention机制对全局信息进行学习,实现文档级化学命名实体识别。扈应等[9]提出了结合CRF的边界组合识别方法,使用多输入卷积神经网络进行实体筛选并分类,有效识别出生物医学文本中嵌套和不连续实体。目前医学文本的研究主要集中于从诊断或病例中提取相关实体进行辅助医疗,而针对医学科研文本的相关研究较少,该文使用在公开数据上表现优秀的BERT-BiLSTM-CRF模型对PubMed中获取的医学文献进行命名实体识别,取得了较好的识别效果。
1.2 同名作者消歧方法研究
作者名字是文献最重要也是最常用的标识符,在知识图谱构建、文献检索中都起到了重要的区分作用。作者名字通常不是唯一的,所有语言中作者同名现象都不可避免。此外,全球不同国家的作者将名字转为英文时使用的翻译标准不同,更加剧了这一歧义性。同名作者消歧是自然语言处理领域实体消歧的衍生问题,主要可以分为融合外部知识库方法和比较特征相似性方法。
随着Google Scholar、ORCID这类大规模学者信息数据库在全球范围的普及,通过融合这些公开数据中的信息,研究者可以较为简便地消除作者名字歧义。Kang[10]从Google Scholar中自动收集共同引用信息,使用聚类方法进行同名作者消歧。白海燕等[11]从ORCID构建机制的角度进行分析,基于记录的权威度和信任值建模实现消歧。这类方法基于作者本人在数据库中的信息进行匹配,准确率相对较高,但是如果信息不完整则无法完成消歧。
另一类方法通过对同名作者的特征进行提取,比较作者特征相似性来完成聚类任务,判断两个同名实体是否代表同一作者。Emami[12]从网页中提取待消歧实体的个人属性和社会关系,将其映射至无向加权图中,使用基于图的聚类算法对节点进行分组以消除歧义。Niu等[13]基于Skip-gram框架提出了三种编码模型对词语语义进行学习,在实体消歧的任务上取得了较好的结果。阮光册等[14]将外部特征与语义特征相结合,使用BERT+XGBoost对英文作者进行消歧。王若琳等[15]不再局限于文本特征,提出论文嵌入网络PaperEmbNet对作者姓名构建异质信息网络,使用循环神经网络算法AR4CPM与层次凝聚聚类对同名作者进行消歧。这一类方法有效克服了数据不完整情况下的消歧问题,但在准确率与适应性上有待进一步提升。该文将两类消歧方法进行结合,使用多源数据对作者信息进行补充,并使用机构与领域信息的相似性对作者进行判别。
1.3 知识图谱构建方法研究
2012年,Google提出知识图谱的概念,随后广泛应用在智能问答、知识推理、个性化推荐等领域,为知识的发现提供了可借鉴的手段。在医学领域中,知识图谱相关研究多集中于智能诊疗、文献检索、生物关系构建等方向。研究的应用场景不同,使用的方法与数据结构也都不尽相同。
Ping等[16]针对心血管疾病患者构建个体知识图谱,将生物学知识与患者的病史及健康状况进行集成,为临床医生与研究者提供信息查询与交换服务。廖开际等[17]使用BiGRU-Attention模型抽取实体间的关系,将非结构化文本数据转为结构化数据,用Neo4j图数据库构建医疗社区问答知识图谱。Xu等[18]使用Bio-BERT模型提取生物实体,整合多源数据构建知识图谱PKG,基于生物实体间的关联对作者和实体的关系进行描述。Odmaa等[19]参考权威的国际医学标准术语集、多源异构临床路径指南、医学百科等构建知识图谱CMeKG,包含了100余万个医学概念关系实例。目前对于特定医疗实体相关的图谱研究相对较少,该文以新冠作为研究对象,提取各类实体进行关系关联,融合作者信息与文章出版信息,使用图结构对信息进行整合与呈现,构建完整简洁的新冠疫情图谱。
2 新冠肺炎疫情知识图谱构建
2.1 BERT-BiLSTM-CRF模型
命名实体识别模型整体结构如图1所示,分为三个部分。首先使用BERT模型获取原始语料的语义表示,再通过BiLSTM模型进一步编码得到每个词的词向量,最后经过softmax分类器和CRF层进行识别与标注,提取出语料中的命名实体,为后续知识图谱的构建奠定基础。

图1 BERT-BiLSTM-CRF模型结构
BERT模型全称Bidirectional Encoder Representation from Transformers,是Google于2019年提出的基于多层双向Transformer结构的预训练词向量模型,能够同时捕获前后文的语义特征。其中最核心的Transformer模块[20]是基于self-attention机制的一种文本序列架构,由编码器和解码器构成,比传统RNN模型在训练速度上有了很大提升,兼容了并行计算来进一步提升其运算速度。
由于医学领域的名词具有专业性,直接使用基于日常用语语料库预训练得到的BERT模型识别效果不佳,需要使用医学领域语料对模型进行微调。输入序列的长度选取常用的参数设定为512,在每个句子的开头添加[CLS]标志,在序列结尾添加[SEP]标志,对于不满足序列长度的句子用[PAD]标志填充,用[X]对分词后的单词后缀进行标注。将医疗语料数据依上述方法处理后,加入模型中进行训练,得到调整后的模型。
BERT在训练中使用了masked language model (MLM)和next sentence prediction(NSP)。MLM在模型每一次训练时随机遮盖一定比例的单词,再通过上下文的联系来预测被遮盖掉的词。NSP判断两个句子是否是前后文,输出一个判断结果并保存在输出序列的[CLS]标志位中。
使用BERT模型训练得到每个序列的特征向量表示之后,将向量输入BiLSTM模型中编码。基本的LSTM的结构可以形式化表示为:
(1)
(2)
(3)
(4)
(5)

BiLSTM在单个LSTM的基础上采用正序和倒序计算得到两组不同的隐藏层表示,将其拼接得到隐藏层最终表示。这一改进能更好地捕捉双向的语义依赖关系与语义共现信息,从而提升模型的性能。BiLSTM的输出经过softmax分类后,对应得到输入序列中每个词的标签概率分布。
为了解决BiLSTM不考虑BIO标注前后文关系的问题,在最后引入条件随机场(CRF)来获得全局最优的序列标记。定义矩阵P为BiLSTM的输出矩阵,矩阵元素Pij代表第i个单词属于第j个标签的概率。整体预测序列y={y1,y2,…,yn}的概率如公式(6)所示,矩阵A是转移矩阵;Aij表示由标签i转移到标签j的概率。
(6)
(7)
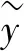
2.2 模型准确性验证
使用LitCOVID数据集[21-22]对模型进行训练并对识别效果进行检验,LitCOVID精选了PubMed中与新冠肺炎疫情相关的文献,并已经对文献题目与摘要中的实体进行了标注,医学实体标签分为六类:Gene,Disease,Chemical,Mutation,Species,CellLine。但是由于每天都有新的文章被发表刊登,大部分最新的文献并没有及时地标注实体信息,且数据集中有部分文献的标注信息存在缺失。因此,筛选出数据集中命名实体标注完整的文章用于模型的训练,取其中70%作为训练集、30%作为测试集来检验模型的识别效果。
为了更好地对模型效果进行对比,在同一数据集上使用BiLSTM-CRF模型进行重复实验,并与使用医学语料调整后的BioBERT模型进行对照,结果如表1所示。可以看出,该文使用的模型在各个实体类别中的F1分数都较高,总体平均F1分数达到了92.86%。
表1 不同模型验证集F1分数比较 %
在实体识别模型训练完成后,将其应用于在PubMed上以关键词“COVID-19”和“novel coronavirus pneumonia”进行检索后获取的所有论文。截至写稿日共计获得149 058篇文献。对每篇文章的题目和摘要使用BERT-BiLSTM-CRF模型进行医学实体提取,共得到34 802个实体,其中包含20 051个Disease类,6 488个Chemical类,5 019个Gene类,2 488个Species类,489个Mutation类,176个CellLine类。
2.3 同名作者消歧
首先对从PubMed获取的新冠相关文献作者进行分析,发现72.84%的作者名出现过两次及以上,存在严重的姓名歧义问题,需要判断不同文献中重复出现的姓名是否属于同一个人。对于同一作者拥有多个英文名的问题也需要进行处理,如Zhong Nanshan与Nanshan Zhong代表同一作者,但按照字符串匹配会被视为两个不同作者。对作者名字进行消歧是构建知识图谱前的必要工作,具体流程如图2所示。
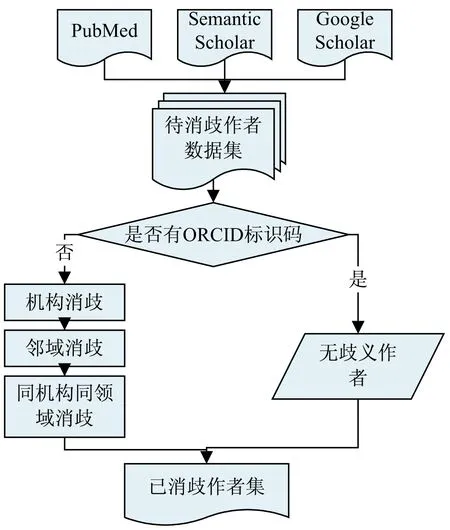
图2 同名作者消歧流程
为了解决作者信息不完整问题,特别是论文合著者大量存在的信息缺失。使用Semantic Scholar和Google Scholar对作者及其文章进行二次搜索以补充信息并提升后续消歧准确率。Semantic Scholar在信息筛选中使用了AI技术,覆盖了各个领域的学者、期刊、会议等信息,并且可以方便地访问作者的主页获取相关信息。Google Scholar是全球最大的搜索引擎之一,搜索到的内容从质量和数量上都具有保证。
获取每个作者的机构、邮箱、电话信息作为后续歧义消除的判别条件。为了解决缩写或名称变化,参照中科院全球科研项目数据库中的科研机构名称对数据进行清洗,如果机构名称相似度超过设定的阈值则认为是同一机构,对于不同搜索引擎存在少量信息冲突的情况进行了人工修正,最终将不同来源的数据整合成具有统一规范的数据集。
随着ORCID的普及,逐渐有学者开始在论文中附上自身唯一标识码,将拥有ORCID的名字认定为不存在歧义,并进行编号。之后参考林克柔[23]的方法使用主成分分析对不同机构的作者进行消歧,在此基础上结合昌宁等[24]学者的方法对姓名存在的学术圈进行划分,解决同机构不同领域的同名不同人问题,并合并不同机构中领域相同的同名同人。最后,使用邮箱和电话信息对同机构同领域的学者进行消歧。最终为每一个名字所代表的作者分配一个唯一编号增量加入到作者集合中,构成最终消歧完成的作者集。
由于该文使用的作者信息未经过人工标注,常用的机器学习评价指标无法计算,为确保所构建图谱的准确性在最后引入了人工验证,对重复率较高、特征相似度较低的名字进行检查。总计得到397 163个消歧后的作者名字。
2.4 知识图谱构建
新冠肺炎知识图谱整体构造流程如图3所示。将2.2节提取到的实体信息以及2.3节的消歧作者信息进行结合,依据实体-实体、作者-作者和实体-作者三类关系进行关联,构建出知识图谱的基础框架。
图3 知识图谱构建流程
但仅有基础信息的知识图谱显然无法完整全面地获知新冠疫情相关信息,对于促进知识传播的作用有限。因此为了增加知识的丰富程度,把从Semantic Scholar和Google Scholar中获取的作者隶属关系、教育背景、邮箱信息融入作者节点以减小其粒度。对于医学实体,从PubMed中获取相关文献的出版信息、期刊名称、发表时间,将信息融入实体节点来描述其研究状况。在后续有新的文献需要加入图谱时,可以通过增加新的节点或修改节点属性的方式增量完成添加,无需重复构建图谱。同时,对于文中暂未涉及到的其他信息来源,如SCI、Embase数据库等,也可以通过这一方法,先与图谱中的文献进行匹配,如果已经存在于图谱中,则无需再次添加,如果图谱中尚未收录,则可以方便地通过模型提取实体信息后增量添加到图谱中。最终,完整地构建了一个新冠肺炎疫情知识图谱。
3 知识图谱可视化及使用方法
本节的知识图谱是基于PubMed中与新冠疫情相关的所有文献构建的,图谱中包含巨大的信息量,各节点之间连接众多且互相交错,使用普通的二维图像无法对整个图谱进行清晰的可视化呈现。为此,在使用图谱时提供了便捷的搜索接口,允许使用者仅提取感兴趣的相关领域内容,把信息量缩减到二维图像能够承载的程度。图4从知识图谱中提取了发文量最高的部分作者和被研究次数最多的医学实体进行可视化,一类节点代表医学实体,一类节点代表作者节点。其中作者节点的大小由作者累计发表文献的数量决定,医学实体节点的大小由被提及的次数决定。总体来看,发文量排名前五的国家分别为:中国,美国,意大利,英国,印度。

图4 知识图谱部分节点可视化
除了聚焦某个研究领域的整体研究状况之外,还可以进行更为精细的检索。一方面可以定位到某位领域内的知名学者,了解他的研究方向与主要成果,另一方面也能聚焦某一个特定的医学实体,聆听其他人对于这个问题的意见或寻找未来潜在的合作伙伴和投资对象。
例如钟南山院士对中国疫情的控制、治疗、研究做出了巨大的贡献,可以快速地从知识图谱中提取他的研究成果、共同合作者等相关信息并形成子图。子图的两类节点分表表示钟南山院士所有已发表研究中包含的医学实体,与钟南山院士的主要合作研究者。可以分析出钟南山院士与关伟杰、李益民的关联最大,他们之间的合作最为紧密,同时排名靠前的实体包含了“patients”、“critically ill”和“oxygen”,这与钟南山院士战斗在抗疫第一线,更关心具体的病例症状有关。
从知识图谱中提取药物相关信息后发现,研究最热门的药物排名前列的分别是羟氯喹(Hydroxychloroquine)、瑞德西韦(Remdesivir)、氯喹(Chloroquine)、托珠单抗(Tocilizumab)。以Chemical类的瑞德西韦(Remdesivir)为例,作为医学领域内的专业学者,或许会关心它究竟是否已经被证实有效,或者是否存不良反应,该方法也可以提取出瑞德西韦的相关子图,包含与瑞德西韦相关的其他医学实体与已发表论文中多次出现瑞德西韦的作者作为节点。由于版面大小限制,该文无法展示具体可视化结果。
从相关作者的隶属信息发现,中国科学院上海药物研究所是国内研究瑞德西韦较多的机构,美国国立卫生研究院转化科学促进中心在所有国外机构中,对于瑞德西韦的研究做出了最大贡献。从图谱中可以查询得到与瑞德西韦关联较大的实体包括“chloroquine”、“hydroxychloroquine”、“lopinavir/ritonavir”和“favipiravir”。大部分是常用的新冠疫情治疗药物,在临床中会根据不同情况进行组合使用,研究者热衷于对比不同药物之间的治疗效果,来确定药物的相对有效性。
新冠肺炎的患者在染病期间通常会伴有各类并发症,如何处理这些病症也是研究的重要方向。对知识图谱中疾病类(Disease)信息进行提取,发现研究最多的疾病主要包括:肺炎(Pneumonia)、癌症(Cancer)、炎症(Inflammation)、焦虑(Anxiety)等。图5对实体节点中发文的时间序列进行计数可以进一步看出,在其他并发症的研究呈现出平稳或下降的趋势时,“焦虑”的相关研究却呈现不断上升的趋势,说明对患者在感染期间或者治愈之后可能产生的心理问题正逐步成为研究热点。
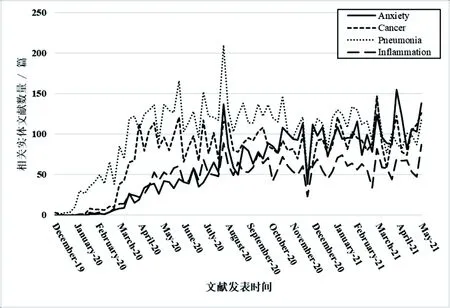
图5 相关疾病时间趋势
4 结束语
正如开篇中所说,新冠肺炎疫情是对全球每一个国家每一个家庭的严峻考验,大家应该齐心协力,共同度过这一难关。以PubMed中所有疫情相关文献作为基础,使用BERT-BiLSTM-CRF模型解决了关键的命名实体识别问题,并对同名的研究者进行消歧处理,以最终处理后的作者名字、医学实体之间的互相联系为依据建立起一个全面、完备的知识图谱。将世界各地的顶尖科学家、医药学家、医生等专家对于疫情的研究成果总结、归纳到一起后进行可视化处理。不仅可以快速查询新冠疫情相关的研究现状、前沿热点、研究进程,也让研究人员与投资者快速有效地寻找特定课题的意见领袖成为可能。这种及时精准的信息共享及顶尖学者之间的精诚合作无疑对早日战胜疫情,进而减少失业、重启经济、恢复教育起到关键的推动作用。
构建的知识图谱在纵向和横向上都具有非常优秀的可扩展性。纵向来看,当有新的文献发表时,可以迅速提取其中的医学实体,向知识图谱中增量添加新信息,无需进行复杂且耗时的重建工作。横向来看,该图谱构建方法可以无障碍地应用到任何同类型的领域(比如癌症、心脏病等)。未来甚至可以不再局限于医疗领域,在诸如全球变暖、环境污染、信息安全等热点问题上也可以进行很好的扩展。

