胃癌组织神经元再生相关蛋白的表达及临床意义研究
徐园 贾黎华 虞伟明
胃癌是常见的实体恶性肿瘤之一,已成为全球第五大最常见恶性肿瘤和第三大恶性肿瘤相关死亡原因[1-3]。尽管近年来临床在胃癌诊断和治疗方面取得了较大进展,但其仍然是一种死亡率极高和预后不良的疾病[4]。根治性手术切除是胃癌的重要治疗方法。然而,由于胃癌缺乏早期特异性症状,多数患者被诊断时已是晚期,失去了手术治疗的最佳时机。另外,在接受根治性手术切除的胃癌患者中,肿瘤复发和转移也常发生。为了改善胃癌患者的预后,在过去的几十年里,世界范围内已经尝试了多种治疗方法,包括化疗、放疗、靶向治疗等,但效果并不理想[5-6]。探寻胃癌新的生物标志物或治疗靶点意义重大。神经元再生相关蛋白(neuronal regeneration associated protein, NREP),又称C5orf13或P311,是一种高度保守的8 kDa胞内蛋白,含有68个氨基酸[7-8]。研究表明,NREP在胚胎、小鼠大脑中高表达,可参与细胞增殖、迁移和分化等多种生物学功能[9-10]。且NREP在恶性肿瘤进展中发挥重要作用,包括胶质母细胞瘤[11]、前列腺癌[12]、食管癌[13]等。然而NREP在胃癌中的研究报道不多。基于此,本研究旨在探讨NREP在胃癌中的表达情况,并分析其临床意义。通过挖掘不同公共在线生物信息数据库的数据,探讨NREP在胃癌组织中的表达情况,并应用基因芯片和免疫组化方法验证其表达情况,分析NREP表达的临床意义,现报道如下。
1 材料和方法
1.1 研究标本 选取2021年3月至8月在宁波市李惠利医院接受D2根治术的胃癌患者20例,术前均未接受放化疗。其中男12例,女8例;年龄18~85岁,平均年龄56岁。留取患者胃癌组织和对应的癌旁正常组织作为研究标本。本研究经医院医学伦理委员会批准(批件号:KY2021PJ122),患者家属知情同意并签署知情同意书。
1.2 在线生物信息数据分析胃癌组织NREP mRNA的表达 使用3个不同在线生物信息数据库分析NREP mRNA在胃癌中的表达情况。(1)使用Oncomine数据库(http://www.oncomine.org),其为公共在线癌症微阵列数据库和数据挖掘平台。本研究阈值如下:P=0.01;差异倍数=2.0;数据类型:mRNA;范围:前10%的基因。(2)使用基因表达谱交互分析(Gene Expression Profiling Interactive Analysis,GEPIA)数据库,其为中国学者设计的一个Web服务器,基于癌症基因组图谱计划(The Cancer Genome Atlas Program,TCGA)和基因型组织表达数据库(Genotype-Tissue Expression,GTEx)中的9 736个肿瘤样本和8 587个正常样本。(3)使用Ualcan数据库(http://ualcan.path.uab.edu.),其为一个综合的计算工具,使用3级RNA测序和从TCGA数据库中的31种癌症类型中提取的临床病理参数,用于分析癌症转录组数据。基于UCSC Xena数据库分析胃癌患者预后不良的危险因素。Kaplan-Meier plotter数据库分析胃癌患者NREP mRNA表达与预后的关系。
1.3 基因芯片分析胃癌组织差异表达基因 使用Trizol试剂提取20例患者的胃癌组织与癌旁正常组织的总RNA,操作参照试剂说明书进行。使用Affymetrix Human Genome U133 Plus 2.0基因芯片(美国昂飞公司)确定基因表达谱。杂交后,对芯片进行清洗和染色。然后,使用Gene Chip Scanner 3000基因芯片扫描仪对微阵列进行扫描。采用MAS 5.0算法和Gene Spring 11.0软件(美国安捷伦技术公司)进行数据读取和分析。差异表达基因经微阵列显著性分析筛选。上调和下调基因的阈值设置采用≥2或≤0.5倍的变化。
1.4 人类蛋白质图谱(Human Protein Altas,HPA)分析胃癌组织NREP蛋白的表达 HPA(https://www.Proteinatlas.org/)是一个免费的公共生物信息数据库,包括蛋白质的差异表达分析和对大约20种恶性肿瘤的免疫染色,可用于分析靶基因的蛋白表达。本研究通过HPA数据库分析胃癌组织NREP蛋白的表达水平,并分析NREP在胃癌组织和正常组织中的免疫组化结果。
1.5 免疫组化检测胃癌组织和癌旁正常组织NREP蛋白的表达 从石蜡包埋的胃癌组织、癌旁正常组织标本中分别切取5 μm薄切片,脱蜡并提取抗原。组织切片用兔多克隆NREP抗体(英国Abcam公司,1∶200稀释)在4℃过夜,然后用生物素化的二抗孵育,并用二氨基联苯胺(DAB)染色(丹麦Dako公司)。染色以细胞质或细胞膜呈棕黄色为阳性,由2名病理科医生根据染色深度及染色细胞百分比进行评定,采用双盲及统一标准。不染色为0分,染色淡为1分,染色中度为2分,染色深为3分。染色细胞百分比≤5%为0分,6%~30%为1分,31%~60%为2分,≥60%为3分。将每张切片的染色深度与染色细胞百分比得分相乘,其乘积为最后得分。0~1分为(-),2~3分为(+),4~6分为(++),>6分为(+++)。将(-)及(+)设定为NREP蛋白低表达,(++)及(+++)设定为NREP蛋白高表达。
1.6 胃癌患者预后不良的危险因素分析 从UCSC Xena数据库[14]下载患者生物信息,其为一个癌症基因组数据库,可为公共数据提供可视化分析。临床参数包括患者性别、年龄、T期、N期、M期、病理分期和组织学分级,采用单因素和多因素分析胃癌患者预后不良的危险因素,以评估NREP蛋白表达对胃癌患者预后的预测价值。
1.7 Kaplan-Meier plotter数据库分析胃癌患者NREP mRNA表达与预后的关系 Kaplan Meier-plotter是一种基于GEO、TCGA和Cancer Biomedical Informatics Grid(caBIG)的网络服务器,用于评估各种基因对生存信息的影响。本研究通过Kaplan-Meier plotter数据库分析胃癌患者NREP mRNA表达与预后的关系。
1.8 NREP功能的富集分析 采用基因本体(Gene Ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析方法研究NREP可能影响胃癌的生物学功能和信号通路,通过Pearson相关分析寻找与NREP表达相关的基因(|r|≥0.4,P<0.05)。采用R软件包对共表达基因进行功能分析。
1.9 统计学处理 采用GraphPad Prism 5统计软件。非正态分布的计量资料组间比较采用Mann-WhitneyU检验。计数资料组间比较采用Fisher确切概率法。危险因素分析采用Cox比例风险模型。采用Kaplan-Meier法绘制生存曲线,两组生存曲线的比较采用logrank检验。P<0.05为差异有统计学意义。
2 结果
2.1 胃癌组织与癌旁正常组织NREP mRNA表达水平比较 来自Oncomine数据库的(Chen数据集)分析均显示,胃癌组织NREP mRNA表达水平高于癌旁正常组织(P<0.05),见图1a。GEPIA数据库分析显示,胃癌组织NREP mRNA表达水平明显高于癌旁正常组织(P<0.05),见图1b。Ualcan数据库分析显示,胃癌组织NREP mRNA表达水平高于癌旁正常组织(P<0.05),见图1c。

图1 胃癌组织与癌旁正常组织NREP mRNA表达水平比较(a:Oncomine数据库;b:GEPIA数据库;c:Ualcan数据库)
2.2 基因芯片检测胃癌组织NREP mRNA表达结果基因芯片微阵列表达谱分析发现NREP mRNA在胃癌组织中高表达,胃癌组织中NREP mRNA的表达水平比癌旁正常组织高至少2.8倍,差异有统计学意义(P<0.05),见表1。
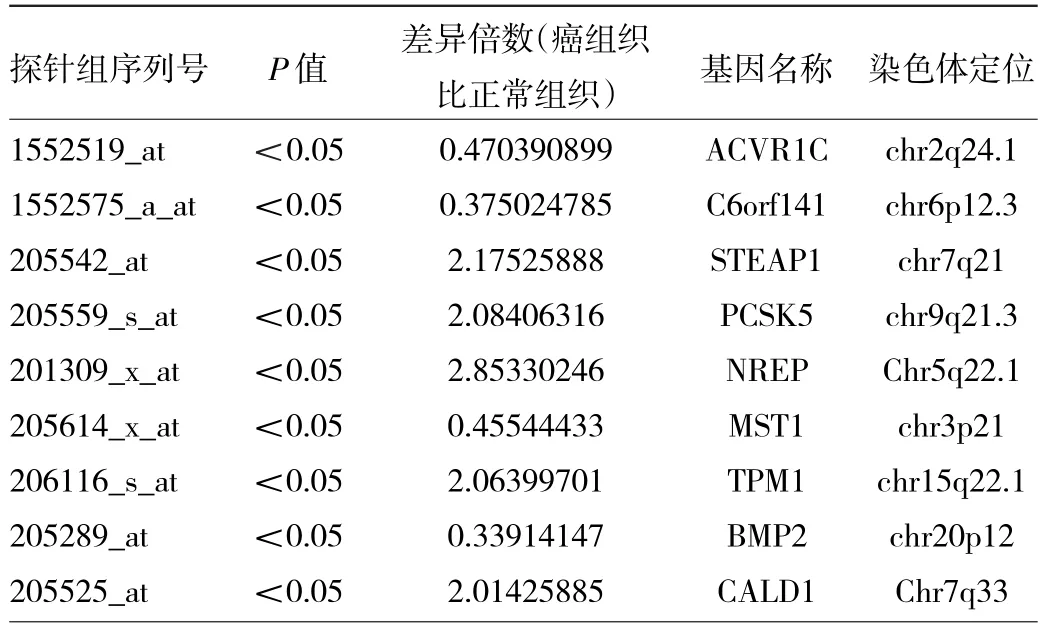
表1 基因芯片检测胃癌组织部分呈现异常高表达的基因
2.3 胃癌组织和癌旁正常组织NREP蛋白表达情况比较 通过HPA数据库分析NREP在胃癌中的蛋白表达水平,显示NREP在胃癌组织中高表达,呈中、高胞质胞膜染色(图2a,见插页)。20例胃癌患者的免疫组化检测结果显示,NREP在胃癌组织中细胞质或胞膜呈棕色强染色(图2b,见插页),即高表达,正常组织中呈低染色或不染色(图2c,见插页),即低表达;胃癌组织NREP蛋白高表达13例(65%),低表达7例(35%);癌旁正常组织中NREP高表达4例(20%),低表达16例(80%),胃癌组织NREP蛋白高表达比例高于癌旁正常组织(P<0.05)。

图2 胃癌组织和癌旁正常组织NREP蛋白表达情况比较(a:基于HPA数据库的NREP在胃癌组织中的表达情况,免疫组化染色,×100;b:NREP在胃癌组织细胞质或细胞膜中呈阳性染色,DAB染色,×200;c:NREP在癌旁正常组织中呈阴性染色,DAB染色,×400)
2.4 胃癌患者预后不良的危险因素分析 单因素分析显示,年龄>65岁、病理分期Ⅲ/Ⅳ期、T分期高、N分期高、M分期高及NREP高表达是胃癌患者预后不良的危险因素(均P<0.05)。多因素分析显示,年龄>65岁、M分期高、NREP高表达是胃癌患者预后不良的独立危险因素(均P<0.05),见表2。

表2 胃癌患者预后不良的危险因素分析
2.5 胃癌患者NREP mRNA表达与预后的关系分析Kaplan-Meier plotter数据库分析显示,NREP mRNA低表达胃癌患者总生存时间、无进展生存时间均优于高表达胃癌患者(均P<0.05),见图3(插页)。

图3 胃癌患者NREP mRNA高表达与低表达患者生存曲线比较(a:总生存曲线比较;b:无进展生存曲线比较)
2.6 NREP基因功能富集分析结果 GO富集分析显示,NREP基因编码蛋白质主要定位于含胶原的细胞外基质、内质网腔、细胞外基质成分胶原三聚体、基底膜。这些蛋白主要参与细胞外结构组建、细胞外基质组建、成骨、细胞底物黏附和结缔组织发育。KEGG通路分析显示NREP主要富集于细胞外基质受体的相互作用、黏着斑、PI3K-Akt信号通路、人乳头瘤病毒感染和细胞骨架作用调控等方面,见图4。

图4 NREP基因功能富集分析结果(a:在生物过程、细胞成分和分子功能等方面的GO富集分析;b:KEGG通路分析)
3 讨论
由于缺乏早期诊断方法和肿瘤的强侵袭性,胃癌已成为全世界最致命的恶性肿瘤之一,其发病率仍居高不下。寻求新型的功能性生物标志物来帮助早期诊断和预测预后显得尤为重要。在生物信息学工具和数据库的帮助下,本研究探索了新的早期诊断生物标志物,并试图研究其参与胃癌发生和发展的潜在分子机制。
NREP最初研究报道其主要存在于神经元和肌肉中[15]。现有关NREP的研究在逐渐增多,已有多项研究表明,NREP可参与促进神经和肺组织再生[16-17]、肾纤维化[18]、皮肤创伤血管生成[19]、肌成纤维细胞分化和迁移。此外,NREP在肿瘤中的作用和发挥的功能也引起了诸多学者的关注。
Mariani等[11]研究发现NREP在胶质细胞瘤中高表达,具有促进胶质母细胞瘤发展的作用。McDonough等[20]则进一步研究了NREP促进胶质瘤发生、发展的可能机制,认为NREP高表达可能通过激活Rac1,调节细胞迁移,进而影响细胞外周肌动蛋白,引起细胞骨架重构,在调节胶质瘤的运动和侵袭方面发挥了重要作用。唐春兰等[21]发现NREP在肺小细胞癌中呈现过表达,且NREP与ITGB4BP存在相互作用,NREP可能通过NREP-ITGB4BP信号通路调节小细胞肺癌的迁移,从而参与肺癌的侵袭和转移。
在本研究中,GEPIA、Oncomine和Ualcan等在线数据库均显示NREP mRNA在胃癌中呈高表达。此外,我们利用基因芯片、免疫组化等方法也验证了NREP在胃癌中的表达水平。KM-Plotter和GEPIA数据库显示NREP高表达与胃癌的总生存时间和无进展生存时间不良密切相关。基于TCGA的原始数据,多因素Cox回归分析显示NREP是胃癌患者预后的独立预测因子之一。据此,笔者推测NREP或可作为评价胃癌预后的生物标志物。
在肿瘤发生、发展过程中,包括胃癌在内,肿瘤细胞必须与外基质紧密结合,并与其他细胞沟通,形成适合增殖并最终转移的间质微环境[22-23]。GO富集分析表明,NREP编码的蛋白质主要定位于含胶原的细胞外基质、内质网腔、基底膜等处。这些蛋白在细胞外结构组织形成、细胞外基质组建、细胞底物黏附和结缔组织的发育中发挥了重要作用,它们还参与胶原结合、整合素结合、糖胺聚糖结合和形成具有抗拉强度的细胞外基质结构成分,在肿瘤细胞增殖过程中提供了良好的间质微环境,并为肿瘤细胞最终发生侵袭,转移提供条件。
KEGG通路分析显示NREP主要富集于细胞外基质受体相互作用、局灶黏附、PI3K-Akt信号通路、人乳头瘤病毒感染和细胞骨架作用调控等。近年来研究发现局灶黏附与细胞外基质受体相互作用是胃癌发生发展的重要途径[24]。局灶黏附是一种细胞底物黏附结构,在整合素介导的信号转导通路中发挥功能作用[25]。局灶性黏附与细胞外基质受体相互作用与整合素有关,这是一种异质二聚体细胞表面受体,参与细胞与细胞之间的特异性相互作用和细胞与细胞外基质的黏附。由此笔者推测,NREP可能为胃癌形成并最终转移提供了良好的间质微环境,并可能通过调控肿瘤黏附与ECM受体的相互作用,参与胃癌的发生、发展。
综上所述,NREP在胃癌组织中高表达,且其高表达与患者预后不良密切相关。NREP或参与了胃癌的发生、发展,可作为胃癌诊断和治疗的新靶点。

